基于医疗文本数据聚类的帕金森病早期诊断预测
2020-10-18张晓博李天瑞彭莉兰
张晓博,杨 燕*,李天瑞,陆 凡,彭莉兰
(1.西南交通大学信息科学与技术学院,成都 611756;2.西南交通大学人工智能研究院,成都 611756;3.综合交通大数据应用技术国家工程实验室(西南交通大学),成都 611756)
(*通信作者电子邮箱yyang@swjtu.edu.cn)
0 引言
目前,帕金森病已成为除老年痴呆症以外最常见的神经退行性和致残性疾病,通常发生在老年人中,临床表现主要包括静止性震颤、运动迟缓、肌强直和姿势性步态障碍[1-5]。帕金森病不仅影响患者的生活质量,而且会给家庭和社会带来沉重的负担。
在我国,年龄65 岁以上人群帕金森病的患病率约占1.7%[6],年龄超过80 岁的患病率约占2.65%[7]。我国患病率与世界发达国家相近,目前全世界有大约1 000 万帕金森病人,而我国的患者超过200万人[8]。
帕金森病最主要的病理改变是中脑黑质多巴胺能神经元的变性死亡,由此而引起纹状体黑质多巴胺能神经元含量显著性减少而致病。导致这一病理改变的确切病因现阶段仍不清楚,遗传因素、环境因素、年龄老化、氧化应激等均可能参与帕金森病多巴胺能神经元的变性死亡过程[9]。目前尚无有效的预防措施阻止疾病的发生和进展。当患者出现临床症状时黑质多巴胺能神经元死亡至少在50%以上,纹状体黑质多巴胺能神经元含量减少在80%以上。因此,早期借助人工智能技术预测并发现帕金森病临床患者,有利于采取有效的措施阻止多巴胺能神经元的变性死亡,以阻止疾病的发生与进展。本文基于一个国际上研究帕金森病进展指标的临床研究平台即PPMI(Parkinson’s Progression Markers Initiative)公开提供的临床医疗检查文本信息数据集[10],结合主成分分析(Principal Component Analysis,PCA)[11],5 种传统的经典聚类K均值(K-Means)[12]、K中心点(K-Medoids)[13]、高斯混合模型(Gaussian Mixture Model,GMM)[14]、亲和力传播(Affinity Propagation,AP)[15]、谱聚类(Spectral Clustering,SC)[16],以及基于聚类的相似性划分算法(Cluster-based Similarity Partitioning Algorithm,CSPA)、元聚类算法(Meta-CLustering Algorithm,MCLA)、超图分割算法(Hypergraph Partitioning Algorithm,HGPA)共3 种聚类集成方法[17],来分析并预测数据集中的多巴胺异常帕金森病患者、健康体和无多巴胺缺失患 者(Scans Without Evidence of Dopamine Deficiency,SWEDD)。该应用方法能够辅助早预防、早发现与早治疗,具有重要的临床研究意义与实际应用价值。
本文的主要贡献包括4个方面:
1)通过PPMI平台提供的公开医疗文本信息数据集,采用聚类等机器学习技术预测并辅助诊断帕金森病。
2)主成分分析方法被应用到医疗文本信息数据集中来降维不同维度的维度空间,不仅解决了数据维度的复杂问题,同时也为聚类提供了多层次可比较的多维度数据集。
3)降维后的不同维度数据集被5 个传统的经典聚类模型和3 种不同的聚类集成方法聚类后,得出特征维度值取30 时GMM聚类效果最佳的结论。
4)应用不同维度数据集的实验结果表明特征维度值小于40 时,高斯混合模型GMM 的聚类效果最佳;而当特征维度值大于40 时,谱聚类(SC)表现突出;3 种聚类集成方法中MCLA的聚类性能最好。
1 相关工作
帕金森病的预测和辅助诊断不论是基于单模态的医疗数据还是多模态的数据集,都被不少学者和研究人员尝试进行研究,应用于不同的数据集上的帕金森病辅助诊断也都有着重要的临床研究意义。接下来,本文概述已有的帕金森病分类、预测工作以及在不同医疗数据集上的应用。
近年来,核磁共振成像(Magnetic Resonance Imaging,MRI)、功能磁共振成像(Functional MRI,FMRI)、经颅超声检查(Transcranial Sonography,TCS)、单光子发射计算机断层成像(Single-Photon Emission Computed Tomography,SPECT)、正电子发射断层成像(Position Emission Tomography,PET)和定量磁化图(Quantitative Susceptibility Mapping,QSM)等单模态医疗数据被用来对帕金森病的辅助诊断进行研究,并取得一些成果。文献[18]开发了一种新颖的级联多列算法框架,通过对单模态神经影像学数据的分析来进行帕金森病辅助诊断。文献[19]中提出了用于检测帕金森病中形态学生物标记的基于多层次感兴趣区域特征提取的机器学习方法,对帕金森病的形态计量生物标志物具有很好的识别能力。文献[20]使用支持向量机技术和基于单光发射计算机断层扫描脑图像的体素特征方法制定了一种用于帕金森病辅助诊断的全自动计算解决方案。文献[21]设计支持向量机方法结合胸带重采样技术进行非分层的多类分类,并依据帕金森病患者的氟脱氧葡萄糖正电子发射断层扫描数据,区分帕金森病和系统萎缩症。文献[22]采用机器学习的方法分析FMRI 数据,根据认知状态来区分帕金森病患者。
另外,不少针对医疗图像数据特征提取并选择的技术也被用来研究帕金森病的辅助诊断。文献[23]定量比较了基于TCS数据的计算机辅助诊断和3种大小的感兴趣区域性能,对原始数据提取特征和降维特征的实验结果表明,覆盖整个中脑区域的感兴趣区域实现了总体最佳的帕金森病诊断性能。文献[24]中提出了通过核磁共振和扩散张量成像数据进行帕金森病诊断的联合回归和分类框架,并设计了统一的多任务特征选择模型,以探索特征、样本和临床医学病理知识之间的多种关系。文献[25]通过MRI 数据辅助诊断帕金森病,实现了一种联合特征样本选择方法,用于选择样本和特征的最佳子集。文献[26]研究了一种用于帕金森病辅助诊断的迭代典型相关分析特征选择方法,特点是以更全面的方式使用MRI数据,并将不同类型的特征融合到一个公共空间中进行分析和选择。文献[27]使用定量磁化图从黑质中提取放射学特征,并采用传统机器学习算法对帕金森病患者与正常人进行分类。
此外,还有深度学习方法也被用来对帕金森病的辅助诊断进行研究。文献[28]中提出了一种深度神经映射大幅度分布机器学习算法,该算法通过深度神经网络技术在大幅度分布中执行核映射而非隐式核函数进行帕金森病辅助诊断,可以克服核选择的困难,并进一步提高分类性能。文献[29]通过卷积神经网络来自动识别帕金森病患者,该深度学习采用的数据是通过由一系列可以提取信息的传感器组成的智能笔,从个人实验测试期间的手写动态中提取信号并学习特征。文献[30]研究了一种用深度学习技术辅助诊断帕金森病严重程度的方法,并在帕金森病人的远程监控语音数据集上进行训练和测试。文献[31]开发了基于深度学习的多巴胺转运蛋白成像解释系统,用来完善帕金森病的影像学诊断。该系统由帕金森病患者和正常人的影像数据训练而成,能够显示出高分类精度,也可对帕金森病不确定的患者进行影像学诊断,并在进一步的临床研究中提供客观的患者组分类。文献[32]中提出了一种深层神经网络分类器,其中包含堆叠的自动编码器和Softmax 分类器,并在两个有帕金森病患者语音障碍相关语音数据库上进行了模拟实验,验证了深度神经网络分类器识别帕金森病患者的有效性。
随着对帕金森病发展的不断研究和临床医学数据的日益积累,研究者们也开始尝试使用多模态的医学数据对帕金森病进行智能诊断。文献[33]中提出了一种深度学习方法,考虑到来自语音、手写和步态的多模态数据信息,对开始或停止运动的困难进行建模,并使用这些转换来训练卷积神经网络模型,实现对帕金森病患者和健康受试者进行分类。文献[34]实现了一种基于多模态神经影像数据的新型特征选择方法,可用于帕金森病检测和临床诊断预测。文献[35]通过25名帕金森病患者和25 位健康对照受试者的核磁共振全脑T1加权、弥散张量成像数据和神经心理学评估数据(含语言记忆测试和视觉空间记忆测试),发现了无痴呆的帕金森病患者的声明性记忆障碍可以通过弥散张量成像分析检测到的海马结构的微结构改变率来预测。文献[36]研究了一种统计方法,用于分析多种模态的神经影像数据,以确定可将帕金森病患者与健康受试者区分开的特征,该方法基于弹性网,执行正则化和变量选择,同时引入以简约性和可再现性为中心的附加条件,通过交叉验证进行评估显示出极高的准确性,成功分离出与帕金森病相关的大脑区域。文献[37]展示了一种基于通用规范相关分析的多视图表示学习的方法,用于学习从笔迹和步态等多模态数据中提取特征的表示形式,可以用作基于语音特征的补充,有效解决了帕金森病患者与健康对照的分类等问题。文献[38]使用具有多种录音类型的帕金森病相关语音数据集,并采用Softmax、神经网络、对数回归和决策树4 种技术对实验数据集进行分类,得出神经网络方法识别帕金森病准确率最高的结论。文献[39]设计了一种多类型的机器学习模型框架,用于捕捉并补充帕金森病患者的语音样本类型,并使用均值投票和多数投票的评估标准进行了评估,表明了元音样本具备帕金森病特征的补充信息。文献[40]研究了如何通过个体持续的发声和语音信号检测帕金森氏病,依据持续性发声和依赖文本的语音方式对帕金森病进行筛查的信号数据,使用随机森林技术作为机器学习算法,用于单个特征集和决策级融合,最后将基于随机森林的邻近矩阵非线性投影到2D空间中,丰富了医疗决策支持。
本文提出的基于医疗文本信息数据的帕金森病早期诊断预测研究,对单模态的医疗文本信息数据进行聚类分析。不仅有效利用了医疗检查过程中产生的各项人体指标信息数据,也在很大程度上挖掘了数据的特征信息。该应用方法可以根据医疗文本数据所具有的文字信息来判断被检查人是否患有帕金森病,也可以预测患病原因是否与其体内黑质多巴胺能神经元含量多少有关。
2 本文方法
本章主要介绍PCA、相关聚类算法和聚类集成等技术,并重点阐述了PCA 降维不同维度空间后结合聚类、聚类集成等方法处理数据的具体算法过程。
2.1 主成分分析PCA
针对医学文本数据特征维度数量多且复杂的情况,需对其数据维度进行降维处理。数据降维方法主要有两种:无监督降维和有监督降维。对于无监督的方法,数据的标签不能被标记,这意味着只能通过学习样本之间的相似特征来对数据样本进行分类或聚类;而对于有监督的方法,类标签学习被认为可以获得更稳健分类或聚类结果。考虑到临床实际和研究目标,本文选择无监督降维处理。无监督降维技术有很多,如PCA、独立成分分析和非负矩阵分解等。PCA 主要采用数学降维的方法,以综合变量来代替原来众多的变量,使得综合变量能尽可能地代表原来变量的信息量,而且彼此之间互不相关。这种把很多个变量转化为少数几个互相无关的综合变量的统计分析方法叫作主成分分析或主分量分析。对文本数据信息特征的降维处理,最佳的选择是主成分分析即PCA 方法,因为PCA 降维能够在保留数据集中大部分特征的同时降低数据的维数[11]。
2.2 聚类方法
1)K-Means 算法是聚类问题的基本方法之一。这是一种基于簇元素的重心表示簇的方法。K-Means 算法将用户输入系统的数据簇分为n个数据簇和K个用户再次输入的数据簇[12]。
2)K-Medoids算法只需计算一次距离矩阵,就可以在每次迭代中找到新的中心点,并使得中心和集群其他部分之间的距离之和最小化[13]。
3)GMM 主要用来估计样本的概率密度分布,估计模型是几个高斯模型的加权和,每个高斯模型代表一个簇。从样本数据在高斯模型上的投影中分别得到每个类的概率,并选择概率最大的类作为决策结果[14]。GMM被定义如下:

其中:参数K是模型个数;πk是高斯权重;p(x|k)是高斯模型排序到k的概率密度。
4)AP方法将数据点对点之间的相似度作为输入度量,在数据点之间交换实值消息,直到一组高质量的示例和相应的集群逐渐出现[15]。置信度被定义如下:

其中:以点i和点k之间的相似度r(i,k)作为聚类中心的输入,减去点i和其他所有候选聚类中心的最大相似度。
归属度a(i,k)被定义如下:

其中归属度a(i,k)设置为自吸引度r(k,k)与从其他点接收的候选聚类中心k点的正吸引度之和。
5)谱聚类(SC)是从图论中演化出来的算法,后来在聚类中得到了广泛的应用。它的主要思想是把所有的数据看作空间中的点,这些点之间可以用边连接起来。距离较远的两个点之间的边权重值较低,而距离较近的两个点之间的边权重值较高,通过对所有数据点组成的图进行切图,让切图后不同的子图间边权重和尽可能地低,而子图内的边权重和尽可能地高,从而达到聚类的目的。由于本身使用了降维,因此相较于传统聚类算法,该方法降低了处理高维数据聚类的复杂度[16]。
2.3 聚类集成方法
1)CSPA将每个数据点表示成一个顶点,两个点被分在同一个图中的次数占聚类集体中成员个数的比例为相应两顶点间边的权重,这样根据一个聚类集体生成一个图后,再利用图形划分算法来得到最终聚类结果,其时间复杂性是二次的[17]。
2)MCLA 则是将每个簇当成顶点,簇之间拥有的相同数据点数占所有数据的比例作为这两个顶点间边的权重,然后在此基础上再利用图形划分算法将簇划分成不同的组,最后每个点根据它在不同组中出现的次数来选择它所在的组从而构成最终的聚类集成结果,其时间复杂性是一次的[17]。
3)HGPA 把聚类集体中的每个簇表示成一条超边,它连接所有在此簇中的数据点,每条超边权重一样,然后利用超图划分算法得到最终聚类结果,其时间复杂性是一次的[17]。
2.4 聚类评估指标
1)聚类精确率(ACCuracy,ACC):ACC是聚类结果的近似值,可以用来评价聚类的准确性。ACC定义如下:

其中Nk是正确分类到每个类的数据项数。ACC越大,聚类性能越好[41]。
2)标准互信息(Normalized Mutual Information,NMI):互信息(Mutual Information,MI)是用来衡量两个数据分布的吻合程度,并计算正确率。MI的定义如下:
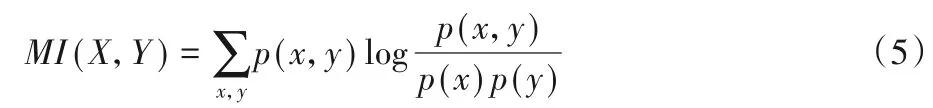
NMI是MI的标准化,用熵作为分母将互信息调整到[0,1]内,可用于聚类评价,定义[41]如下:
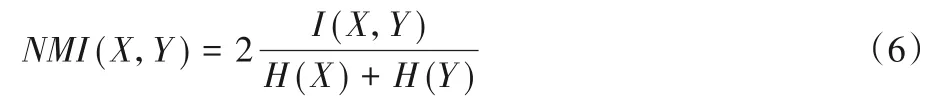
3)F1 值是精确率和召回率的调和平均值,可以准确地评价聚类算法的性能。F1值的定义如下:

其中:P、R分别表示聚类算法的精确率和召回率[41]。
4)调整兰德系数(Adjusted Rand Index,ARI):兰德指数(Rand Index,RI)需要给出实际的类别信息C,假设K是聚类结果,a表示C和K中同一类别元素的对数,b表示C和K中不同类别元素的对数,RI指数公式如下:


ARI的取值范围是[-1,1],值越大,聚类结果越符合实际情况。广义上讲,ARI是衡量两个数据分布的匹配程度[41]。
2.5 数据处理算法过程
本文医疗文本数据被处理的整个算法过程如下:

3 实验与结果分析
3.1 实验数据
本文所用的实验数据来源于PPMI 平台提供的公开文本数据集,共1 783 条数据记录,135 个特征。由于49 个特征存在数据缺失,最终选取了86 个有效特征,其中有代表性的10个特征说明如表1[10]所示。另外,针对表1 中特征gds 与p-tau之间的关系,样本数据分布如图1 所示,同样,gds 与rem 特征联系反映的样本分布如图2所示。

表1 特征变量说明Tab.1 Description of feature variables
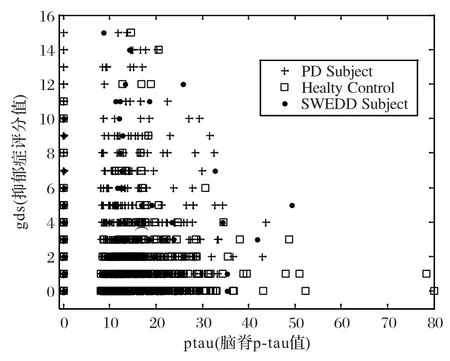
图1 基于脑脊液p-tau值与抑郁症评分值的样本分布Fig.1 Sample distribution based on ptau and gds

图2 基于脑脊液p-tau值与睡眠行为障碍评分值的样本分布Fig.2 Sample distribution based on ptau and rem
3.2 实验设置
所有实验均在一台工作站(Intel Core i7-3337U CPU@1.80 GHz,内存8 GB)上操作运行。首先,本文使用ActivePython-2.7.13.2716 软件和Python 代码来处理原始数据集,得到1 783 个数据样本,并选择了86 个有效特征;然后,应用PCA 方法将86 个特征分别降维到80、70、60、50、40、30、20 和10 共计8 个不同维度的维度空间;其次,选择K-Means、K-Medoids、GMM、AP 和SC 共5 种不同聚类方法对8 个维度空间数据进行聚类,并采用CSPA、MCLA 和HGPA 共3 种聚类集成方法对前面5 种聚类算法进行聚类集成,同时采用ACC、NMI、F1 和ARI共4 个指标在Matlab R2014a 软件平台上评价聚类性能;最后,本研究比较了5 个聚类方法和3 个聚类集成的实验结果。整体实验设计流程如图3所示。

图3 实验设计流程Fig.3 Flowchart of experimental design.
3.3 实验结果
本节描述了不同维度的聚类实验结果。
K-Means、K-Medoids、GMM、AP、SC 共5 种不同聚类方法及CSPA、MCLA 和HGPA 共3 种聚类集成在8 个不同维度即80、70、60、50、40、30、20和10的维度空间上的聚类结果如表2所示。从表2 可看出,5 个聚类和3 个聚类集成方法在不同维度上的ACC、NMI、F1 和ARI最佳性能值已被重点标注。不同维度上的评价指标最高值大小不同,不同维度范围下对应评价性能最好的聚类算法也有所差异。当特征维度大于40 时,SC 的ACC和F1 值效果最好;当特征维度取70 时,ACC值达到0.614 1;当特征维度选择小于40 时,GMM 的4 项评价指标都表现优异;而当特征维度取30时,GMM 的4项指标性能最佳,其中ACC值达到0.891 2;在3 个聚类集成方法中,不论维度取多少,MCLA 的两项指标ACC和F1 值均表现最好,当特征维度取80时,ACC值达到0.596 2。
8 个维度空间上GMM 与SC 聚类算法识别样本数据准确度效果,比较结果如图4 所示;3 个聚类集成方法在每个维度上的最高值表现如图5所示。
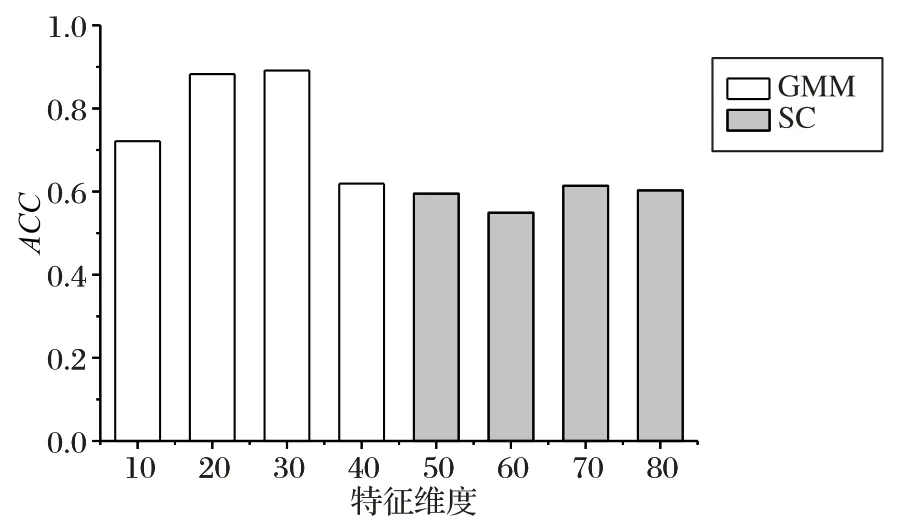
图4 GMM和SC在8个特征维度上的聚类性能比较Fig.4 Clustering performance comparison between GMM and SC on 8 feature dimensions
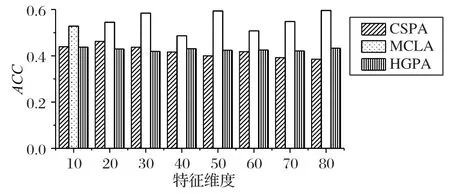
图5 聚类集成方法在8个特征维度上的聚类性能比较Fig.5 Clustering performance comparison of clustering ensemble methods on 8 feature dimensions
从图4 中分析得知,GMM 在维度取30 时,聚类准确度在所有维度上的5 个聚类算法中取值最大,明显高于SC 在维度为70 时的准确度;从图5 可看出,MCLA 聚类集成方法在3 个聚类集成中每个维度上的准确度值都是最大的。
4 结语
本文进行了基于医疗文本数据聚类的帕金森病早期诊断预测研究。首先对PPMI 平台提供的公开医疗文本数据集进行预处理后,选择有效的86 个数据特征;为降低数据复杂度,结合PCA 方法分别对原始数据进行80、70、60、50、40、30、20和10 不同维度的降维;最后引用K-Means、K-Medoids、GMM、AP 和SC 聚类方法对8 个维度空间数据进行聚类,并使用了CSPA、MCLA 和HGPA 聚类集成方法。在ACC、NMI、F1和ARI聚类评估指标上的实验结果显示,得出医疗文本数据特征维度降维到30 时,GMM 聚类效果最佳的结论,准确度达到89.1%,能够有效识别多巴胺异常帕金森病患者、健康体和无多巴胺缺失帕金森病患者。

表2 不同聚类方法在不同维度的实验结果Tab.2 Experimental results of different clustering algorithms on different feature dimensions
