钱塘江三角鲂线粒体基因组测序及其结构特征分析
2020-10-14冯晓宇马恒甲
刘 凯,冯晓宇,马恒甲,谢 楠
(杭州市农业科学研究院 水产研究所,浙江 杭州 310024)
线粒体DNA(mitochondrial DNA,mtDNA)广泛存在于真核生物细胞内,独立于核基因组之外,具有结构简单、多拷贝、能够独立复制、编码效率高、进化速率快,且一般无组织特异性等特点,被广泛应用于种群遗传、系统发育演化、适应性进化等领域中[1]。通过对鱼类线粒体基因组的比对和分析,获得了大量的鱼类线粒体基因组数据,为鱼类线粒体的起源与演化,以及种群遗传、适应性进化等研究提供了新的方向与依据。
三角鲂(MegalobramaterminalisRichardson),隶属于鲤形目(Cypriniformes),鲤科(Cyprinidae),鲌亚科(Culterinae),鲂属(Megalobrama),自然分布于长江、黄河和黑龙江等水系[2]。由于过度捕捞及生态环境变化,三角鲂野生资源量严重下降。钱塘江流域拥有一定的三角鲂种质资源且建有全国唯一的国家级三角鲂原种场[3],对三角鲂种质资源保护、增殖放流等工作较为重视。目前对于三角鲂繁育、养殖技术的研究较多,而对它的线粒体基因组、系统进化研究较少。基因测序技术的深入发展,使其能够广泛应用于三角鲂的种质研究。近年,有学者通过线粒体基因组序列特征探讨了珠江流域[4]和黑龙江流域[5]的三角鲂和团头鲂(MegalobramaamblycephalaYih)、广东鲂(MegalobramahoffmanniRichardson)以及厚颌鲂(Megalobramapellegrini Tchang)等4种鱼类的种间关系,而对于钱塘江流域的三角鲂则研究较少。本研究测定了钱塘江三角鲂的线粒体全基因组序列,分析了其主要结构特征,通过与珠江流域和黑龙江流域三角鲂线粒体基因组的BLAST比较及遗传距离分析,表明不同流域间三角鲂存在遗传学差异;通过比对包括钱塘江三角鲂在内的15种鲌亚科鱼类的线粒体基因组,构建了鲌亚科鱼类的系统进化树,探讨了不同流域间三角鲂的亲缘关系,以及三角鲂与团头鲂等其他鲂属鱼类的亲缘关系。该研究结果为钱塘江三角鲂的种质鉴定、地理种群划分和亲缘关系鉴定等方面提供了理论依据。
1 材料与方法
1.1 实验材料及DNA提取
以国家级钱塘江三角鲂原种场保存的钱塘江三角鲂为材料。采集鳍条组织样品,用无水乙醇固定,置于-20 ℃的冰箱中存储备用。使用柱式动物组织基因组DNA提取试剂盒(购自天根生化科技有限公司),按照试剂盒使用说明书步骤提取总DNA,分别提取3个样本的总DNA进行后续实验。
1.2 基因测序
基于GenBank中团头鲂(EU434747)和采集自珠江流域[4](JX242528)以及黑龙江流域[5](MH289765)的三角鲂线粒体基因组全序列,用Primer premier 5.0软件设计扩增了钱塘江三角鲂线粒体基因组全序列的19对引物(表1)。PCR扩增产物用琼脂糖凝胶电泳检测,纯化后用大肠埃希菌DH5α感受态细胞进行TA克隆,经蓝白斑筛选,挑选阳性克隆送往上海派森诺生物科技有限公司,使用ABI 3730XL型号测序仪进行测序。

表1 钱塘江三角鲂线粒体基因组序列扩增使用的引物序列Table 1 Sequences of primers used in amplification of complete mitochondrial genome of Megalobrama terminalis from Qiantang River
1.3 序列拼接与分析
使用UGENE软件[6]的CAP3组件进行序列拼接,获得了钱塘江三角鲂线粒体基因组全序列。通过BLAST[7]同源序列检索并使用UGENE软件[6],分析了钱塘江三角鲂线粒体基因组的蛋白编码基因、rRNA基因、tRNA基因和非编码区,以及统计了序列长度、碱基组成、GC含量等信息。利用RNAcentral网站[8]的Auto Traveler软件(https://rnacentral.orghelpsecondary-structure)绘制rRNA二级结构。使用tRNA scan-SE软件[9](http://lowelab.ucsc.edu/tRNAscan-SE/)预测tRNA的位置及二级结构,使用VARNA软件[10]绘制二级结构图。利用Mfold软件[11](http://unafold.rna.albany.edu/?q=mfold/DNA-Folding-Form)预测茎环结构。使用RepeatMasker软件[12](http://www.repeatmasker.org/cgi-bin/WEBRepeatMasker)搜索并标记重复序列。使用MitoAnnotator[13]软件(http://mitofish.aori.u-tokyo.ac.jp/annotation/input.html)进行线粒体基因组注释并结合相关物种信息加以校正。使用CGview软件[14](http://cgview.ca/)绘制线粒体基因组图谱及Blast比较圆形图。利用MEGA X软件[15]计算相对同义密码子使用度(relative synonymous codon usage,RSCU),以对密码子使用偏好进行评估。如果密码子使用无偏好性,则RSCU值为1;如果该密码子比其他密码子使用频率高,则RSCU值大于1;反之RSCU值小于1[16]。利用MEGA X软件[15]基于线粒体基因组全序列计算相对遗传距离(pairwise distances)。
1.4 系统进化树构建

2 结果与分析
2.1 线粒体基因组结构与组成
钱塘江三角鲂线粒体基因组序列经注释后提交GenBank(登录号:MN725725)。在其线粒体基因组中存在2个散在的重复序列,分别位于D-loop区的15 705~15 758 bp和16 498~16 517 bp位置;含有22个tRNA基因、2个rRNA基因和13个蛋白编码基因(图1)。钱塘江三角鲂线粒体基因组序列全长为16 621 bp,其碱基组成为A(31.23%)、G(16.17%)、C(27.87%)和T(24.73%);A+T含量(55.97%)高于G+C含量(44.03%),表明钱塘江三角鲂线粒体基因组具有AT偏好性。

图1 钱塘江三角鲂线粒体基因组图谱Fig.1 Map of complete mitochondrial genome of Megalobrama terminalis from Qiantang River
不同物种其线粒体基因的起止位置、长度和所在链有所不同,表2展示了钱塘江三角鲂所有rRNA基因、tRNA基因、蛋白编码基因和非编码区的长度、起止位置和编码链,以及蛋白编码基因的起始密码子与终止密码子等信息。

表2 钱塘江三角鲂线粒体基因组结构特点Table 2 Characteristics of complete mitochondrial genome of Megalobrama terminalis from Qiantang River
2.2 rRNA与tRNA编码基因结构特征
钱塘江三角鲂线粒体包含两个rRNA,分别为12S rRNA和16S rRNA,两者均位于H链上。12S rRNA在线粒体序列上的位置为70~1 030 bp,16S rRNA在线粒体序列上的位置为1 103~2 794 bp,两个rRNA之间夹着1个tRNA-Val基因。以CRW[18]网站提供的鲤鱼(CyprinuscarpioLinnaeus)线粒体12S rRNA(b.16.m.C.carpio)文件为模板,利用Auto Traveler软件绘制了钱塘江三角鲂线粒体12S rRNA二级结构(图2),此二级结构中第Ⅰ、第Ⅱ结构域称为可变区,而第Ⅲ、第Ⅳ结构域则为保守区。与鲤鱼相比,钱塘江三角鲂线粒体12S rRNA在第Ⅰ、第Ⅱ结构域多处出现碱基的替换、插入与重排现象,而第Ⅲ、第Ⅳ结构域则相对较少。以人(HomosapiensLinnaeus)线粒体16S rRNA(mHS_LSU_3D)文件为模板,利用Auto Traveler软件绘制了钱塘江三角鲂线粒体16S rRNA二级结构,此二级结构由6个结构域组成(图3),其中第Ⅰ-Ⅲ、第Ⅵ结构域称为可变区,而第Ⅳ、第Ⅴ结构域则为保守区。对比12S rRNA和16S rRNA的二级结构图可以发现,与16S rRNA基因相比,12S rRNA基因会显得更加保守。此外,从二级结构图中也可发现,rRNA茎区的碱基序列比环区的碱基序列要相对保守,较少出现碱基的替换、插入与重排现象。
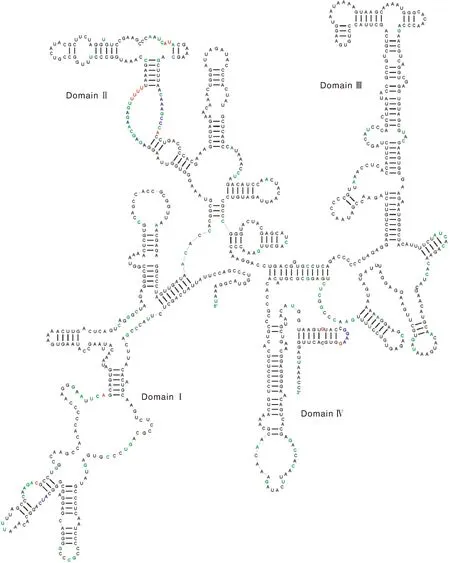
以CRW[18]网站提供的鲤鱼(Cyprinus carpio Linnaeus)线粒体12S rRNA(b.16.m.C.carpio)文件为模板,利用Auto Traveler软件绘制。黑色表示碱基与模板相同,绿色表示碱基与模板不同,红色表示插入的碱基,蓝色表示碱基的重排。Using the Cyprinus carpio Linnaeus mitochondrial 12S rRNA(b.16.m.C.carpio) file provided on the CRW[18] website as a template, it was drawn using Auto Traveler software. Black indicated that the base was the same as the template, green indicated that the base was different from the template, red indicated the inserted base, and blue indicated the reinsertion of the base.图2 钱塘江三角鲂线粒体12S rRNA二级结构预测图Fig.2 Predicted secondary structure of mitochondrial 12S rRNA of Megalobrama terminalis from Qiantang River
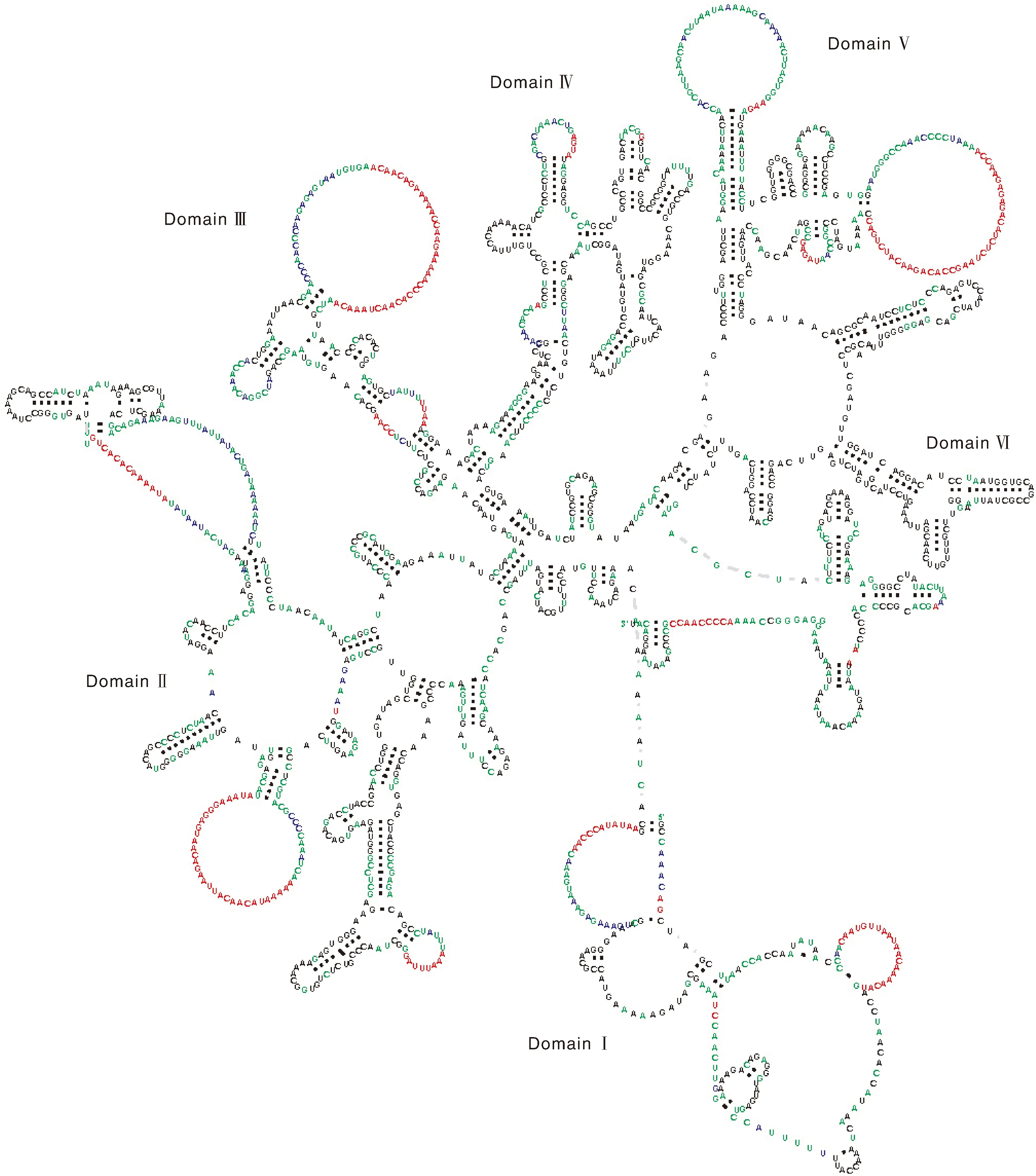
以人(Homo sapiens Linnaeus)线粒体16S rRNA(mHS_LSU_3D)文件为模板,利用Auto Traveler软件绘制。黑色表示碱基与模板相同,绿色表示碱基与模板不同,红色表示插入的碱基,蓝色表示碱基的重排。A Homo sapiens mitochondrial 16S rRNA(mHS_LSU_3D) file was used as a template and it was drawn using Auto Traveler software. Black indicated that the base was the same as the template, green indicated that the base was different from the template, red indicated the inserted base, and blue indicated the reinsertion of the base.图3 钱塘江三角鲂线粒体16S rRNA二级结构预测图Fig.3 Predicted secondary structure of mitochondrial 16S rRNA of Megalobrama terminalis from Qiantang River
与大多数鱼类线粒体类似[1],钱塘江三角鲂线粒体也包含22个tRNA基因,其序列长度从69 bp到76 bp不等,tRNA基因序列的总长度达到了1 566 bp(表2)。22个tRNA基因中,有2个tRNA-Ser(UCN,AGY)和2个tRNA-Leu(UUR,CUN);tRNA-Ser的反密码子分别为UGA和GCU,tRNA-Leu的反密码子分别为UAA和UAG。8个tRNA(tRNA-Gln、tRNA-Ala、tRNA-Asn、tRNA-Cys、tRNA-Tyr、tRNA-Ser(UCN)、tRNA-Glu和tRNA-Pro)位于L链上,其余14个tRNA则位于H链上。tRNA二级结构预测结果显示,钱塘江三角鲂线粒体中tRNA-Phe、tRNA-Val、tRNA-Leu(UUR)(图4)等21个tRNA均可形成典型的三叶草型二级结构,而tRNA-Ser(AGY)由于缺失二氢尿嘧啶臂(D臂)而不能形成典型的三叶草结构(图5)。tRNA scan-SE软件预测tRNA-Ser(AGY)的二级结构时,61号碱基G未与43号碱基C配对,却与44号碱基C形成配对,从而造成60号碱基未形成配对(图5-A)。比对mitotRNAdb数据库[20]上斑马鱼(DaniorerioHamilton)tRNA-Ser(AGY)的二级结构,tRNA-Ser(AGY)61号碱基G应该与43号碱基C形成了配对,而48号碱基G与56号碱基C则未形成配对(图5-B),否则tRNA-Ser(AGY)T臂的茎上将形成6个碱基对,而鱼类tRNA-Ser(AGY)T臂的茎上只有5个碱基对[21]。此外,在tRNA的二级结构中摆动配对现象比较常见[22],如tRNA-Leu(UUR)的7号碱基G与69号碱基U形成配对。在人线粒体tRNA-Leu(UUR)D臂上存在一段13碱基构成的功能DNA序列Motif(5′-TGGCAGAGCCCGG-3′),是线粒体转录终止因子(mTERF)的结合位点[23],参与调节2个rRNA和H-链下游基因的转录水平[24],在钱塘江三角鲂线粒体基因tRNA-Leu(UUR)D臂上也存在一段类似的Motif(5′-TGGCAGAGCATGG-3′),其主要碱基组成和排列顺序与人的Motif类似,仅仅是在第10、11位置的碱基与人有差异,表明该Motif很可能具有类似功能。

图4 钱塘江三角鲂线粒体tRNA-Leu(UUR)二级结构预测图Fig.4 Predicted secondary structure of mitochondrial tRNA-Leu(UUR) of Megalobrama terminalis from Qiantang River

A,tRNA scan-SE软件预测的tRNA-Ser(AGY)二级结构;B,重新绘制的tRNA-Ser(AGY)二级结构。A, The secondary structure of tRNA-Ser(AGY) predicted by tRNA scan-SE; B, The secondary structure of tRNA-Ser(AGY) redrawn.图5 钱塘江三角鲂线粒体tRNA-Ser(AGY)二级结构预测图Fig.5 Predicted secondary structure of mitochondrial tRNA-Ser(AGY) of Megalobrama terminalis from Qiantang River
2.3 非编码区
线粒体中的非编码区,在调节线粒体DNA复制和转录中起到非常重要的作用[1]。与大多数鱼类线粒体类似,钱塘江三角鲂线粒体的非编码区也包含2段,其中一段被称为控制区(control region),又叫作D-loop区(displacement-loop region),另一段则被称为L-链复制起始区,又叫作OL区(origin of L-strand replication region)[1]。此外,钱塘江三角鲂线粒体的非编码区还含有12个基因间隔序列,其长度从1 bp到13 bp不等,其中最长的序列间隔位于tRNA-Asp基因和COX2基因之间,其长度达到了13 bp。由5个tRNA基因串联组成的区域被称为WANCY区[25],位于此区域的tRNA-Asn基因和tRNA-Cys基因之间有一段启动L-链复制的序列,此序列即是OL区,该序列与tRNA-Asn基因和tRNA-Cys基因的部分序列可组成一个茎环结构,茎有12 bp,环有11 nt(图6)。在这个茎环结构的5′端(tRNA-Cys基因这一侧),有一段保守的功能DNA序列Motif(5′-GCCGG-3′),其对L-链的复制至关重要[26],钱塘江三角鲂线粒体中也具有这个结构(图6方框所示),与斑马鱼线粒体OL区的茎环结构也非常类似[21,27]。
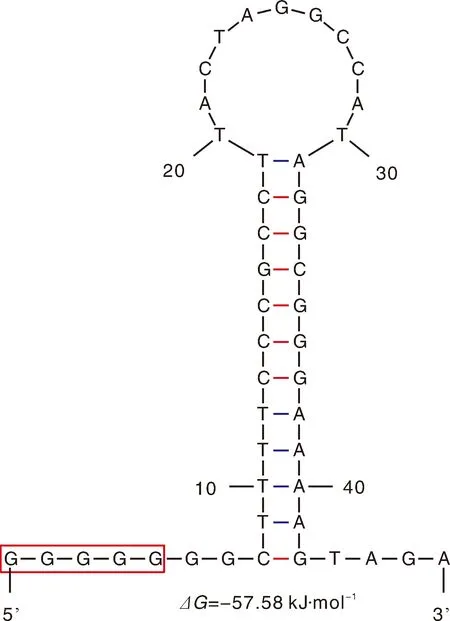
图6 钱塘江三角鲂线粒体L-链复制起始区茎环结构预测图Fig.6 Predicted secondary structure of origin of L-strand replication region of Megalobrama terminalis from Qiantang River
钱塘江三角鲂线粒体的D-loop区长度为937 bp,G+C含量为35.75%,低于A+T含量(64.25%),具有明显的AT偏好性,这个现象与其他脊椎动物以及鱼类线粒体控制区的碱基含量相似[1,28]。鱼类线粒体基因组变异大多发生在D-loop区,该区域位于tRNA-Pro基因和tRNA-Phe基因之间[1],一般可分为3个区段:分别为终止结合序列区(terminal associated sequences,TAS),中央保守区(central domain,CD)和保守序列区(conserved sequence blocks,CSB)[1,28]。根据已报道的鲂属鱼类D-loop区的结构特征[29],对钱塘江三角鲂线粒体D-loop区的相关区段进行了识别(图7)。钱塘江三角鲂线粒体D-loop区中TAS区的长度为76 bp,并在TAS区的前端发现了串联重复序列(图8-A),其可形成稳定的茎环结构(图8-B),此外还发现该重复序列包含一段5碱基构成的功能DNA序列Motif(5′-TACAT-3′),其被认为是D-loop区DNA转录终止的候选位点[30]。钱塘江三角鲂线粒体D-loop区中CD区的关键序列包括CSB-F、CSB-E、CSB-D这3个区段,CSB-F是区分TAS区和CD区的标志,CSB-E关键序列中含有GTGGG-box[31]的类似序列GAGGG,CSB-D与其他鱼类的相应序列也比较类似[21,29,31]。CSB区一般含有3个保守序列CSB-1、CSB-2和CSB-3。CSB-1一般在脊椎动物中普遍存在[28,32],可作为CSB区和CD区的分界标志,钱塘江三角鲂线粒体CSB-1的序列为ATTATTAAAAGACATA。CSB-2和CSB-3序列要比CSB-1序列保守,因此较容易识别,该CSB-2和CSB-3序列分别为CAAACCCCCCTACCCCC和TGTCAAACCCCGAAACCAA。此外,在D-loop区的近3′端也发现一个重复序列,这在康氏似鲹(ScomberoidescommersonnianusLacepède)[33]和黄条鰤(SeriolaaureovittataValenciennes)[34]等其他鱼类中也有存在。
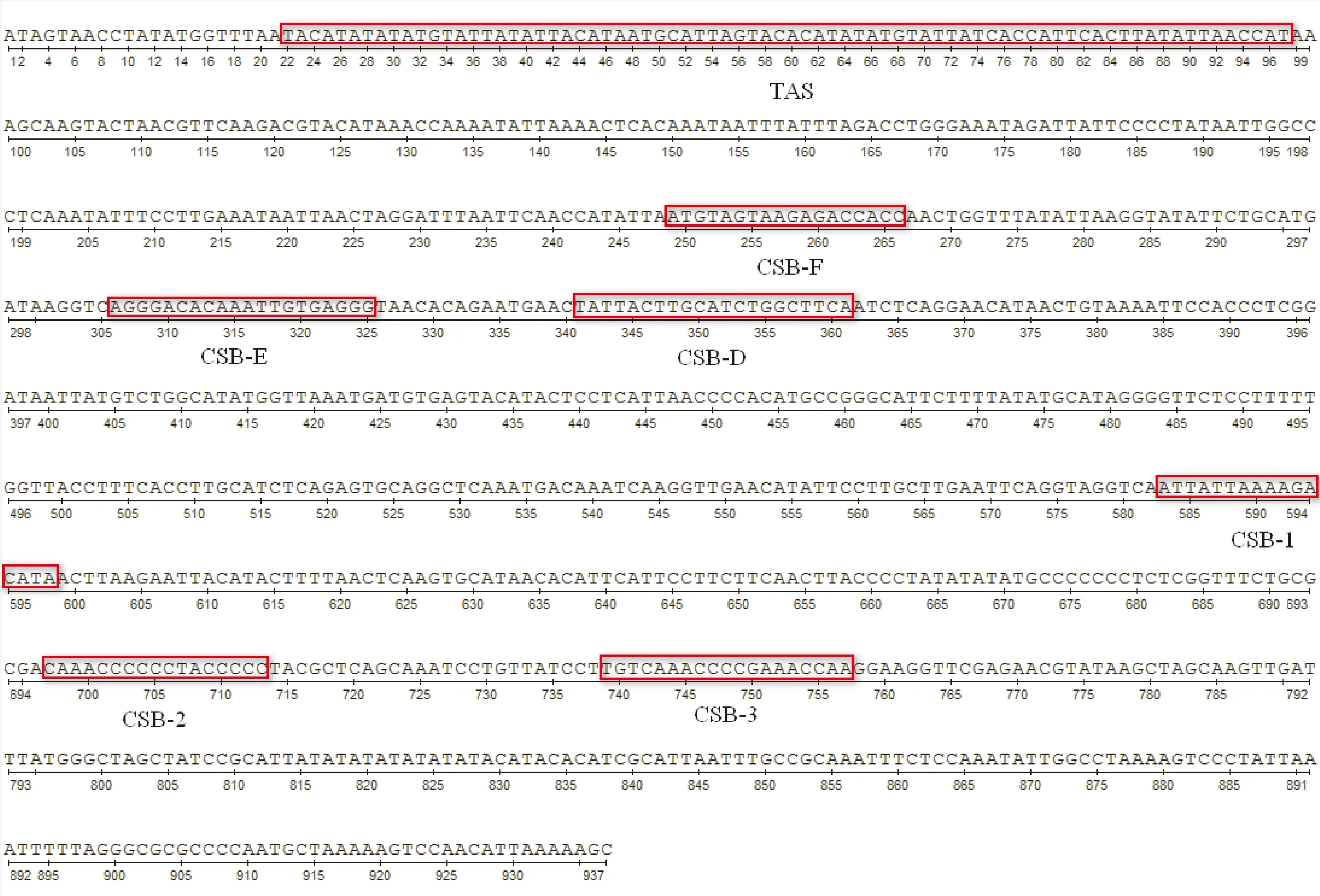
图7 钱塘江三角鲂线粒体控制区序列Fig.7 Control region sequences of Megalobrama terminalis from Qiantang River
2.4 蛋白编码基因
钱塘江三角鲂线粒体包含13个蛋白编码基因,基因长度从165 bp到1 836 bp不等,基因总长度达到11 422 bp,占基因组总长度的68.72%,其中NAD6基因编码于L链上,而其他12个蛋白基因则编码于H链上(表2)。13个蛋白基因中,COX1基因的起始密码子是GTG,而其他12个基因的起始密码子则都为ATG;终止密码子最多的是TAA,共有6个,ATP6、COX3和NAD4的终止密码子为不完全的TA-,剩余基因为不完全的终止密码子T--。此外,蛋白编码基因之间存在重叠现象,例如ATP6基因和ATP8基因之间有7 bp重叠,NAD4L基因和NAD4基因之间也有7 bp重叠,NAD5与NAD6之间则有4 bp重叠。
钱塘江三角鲂线粒体中所有蛋白编码基因的A+T含量均高于50%(表3),A+T总含量达到了55.86%,表明蛋白编码基因具有AT偏好性。由于钱塘江三角鲂线粒体的A+T含量为55.97%,可见非蛋白编码区也具有AT偏好性。密码子使用偏好性分析表明,在13个蛋白编码基因中存在32个偏好密码子(表4),其中第3位点为A或T(U)的密码子中相对密码子使用频率(relative synonymous codon usage,RSCU)大于1的密码子有65.63%,第3位点为C或G的密码子中RSCU大于1的密码子只有34.38%,说明第3位点为A或T(U)的密码子普遍具有较高的使用频率。而在钱塘江三角鲂线粒体中,密码子第3位碱基为A或T(U)的密码子出现的总概率也达到了62.50%,而RSCU也均大于1(除了UUU(F)、CUU(L)、CCU(P)、ACU(T)、GCU(A)、CAU(H)、GAU(D)、UGU(C)、CGU(R)、AGU(S)、AGA(*)、GGU(G)等密码子之外),以上结果表明,线粒体密码子第3位点对A、T(U)的偏好性与蛋白编码基因密码子第3位点对A、T(U)偏向性一致。

表3 钱塘江三角鲂13个蛋白编码基因碱基组成Table 3 Nucleotide composition of thirteen protein-coding genes of Megalobrama terminalis from Qiantang River

表4 钱塘江三角鲂13个蛋白编码基因密码子使用频率Table 4 Total codon average usage in the thirteen protein-coding genes of Megalobrama terminalis from Qiantang River
2.5 不同流域三角鲂线粒体基因组比较
基于线粒体基因组全序列的测定,比较了珠江流域[4](JX242528)以及黑龙江流域[5](MH289765)的三角鲂和采自钱塘江流域的2个三角鲂样本的线粒体基因组(MN725725、MH289767)的一致性和遗传距离。2个钱塘江三角鲂样本均采集自国家级钱塘江三角鲂原种场,一个为2011年保存的样品(MN725725),一个为2017年保存的样品[5](MH289767)。经BLAST

A,RepeatMasker软件在TAS区前端标记的串联重复序列;B,Mfold软件预测的串联重复序列的二级结构。A, The tandem repeat sequence marked by the RepeatMasker in the front of the TAS area; B, The secondary structure of the tandem repeat sequence predicted by the Mfold.图8 钱塘江三角鲂线粒体终止结合序列区中的重复序列Fig.8 Repeat sequences in terminal associated sequences of Megalobrama terminalis from Qiantang River
比较(图9),2个钱塘江三角鲂样本序列一致性达到99.98%,与黑龙江流域三角鲂(MH289765)一致性为99.76%,与珠江流域三角鲂[4](JX242528)一致性达到了99.87%,与广东鲂[4](JX242530)一致性为96.06%。基于线粒体基因组序列间遗传距离分析(表5)也表明,采自钱塘江流域的2个三角鲂样本的线粒体基因组具有更近的遗传距离,钱塘江三角鲂(MN725725、MH289767)与珠江流域三角鲂[4](JX242528)之间的遗传距离要比钱塘江三角鲂(MN725725、MH289767)与黑龙江流域的三角鲂(MH289765)之间遗传距离更近。此外,基于BLAST比较圆形图可发现,2个钱塘江三角鲂样本线粒体基因组(MN725725、MH289767)差异碱基分别位于NAD1,tRNA-Lys和NAD5基因中。

表5 基于线粒体基因组全序列分析不同流域三角鲂相对遗传距离Table 5 Analysis of pairwise distances of Megalobrama terminalis in different basin based on complete mitochondrial genome
2.6 线粒体基因组系统进化分析

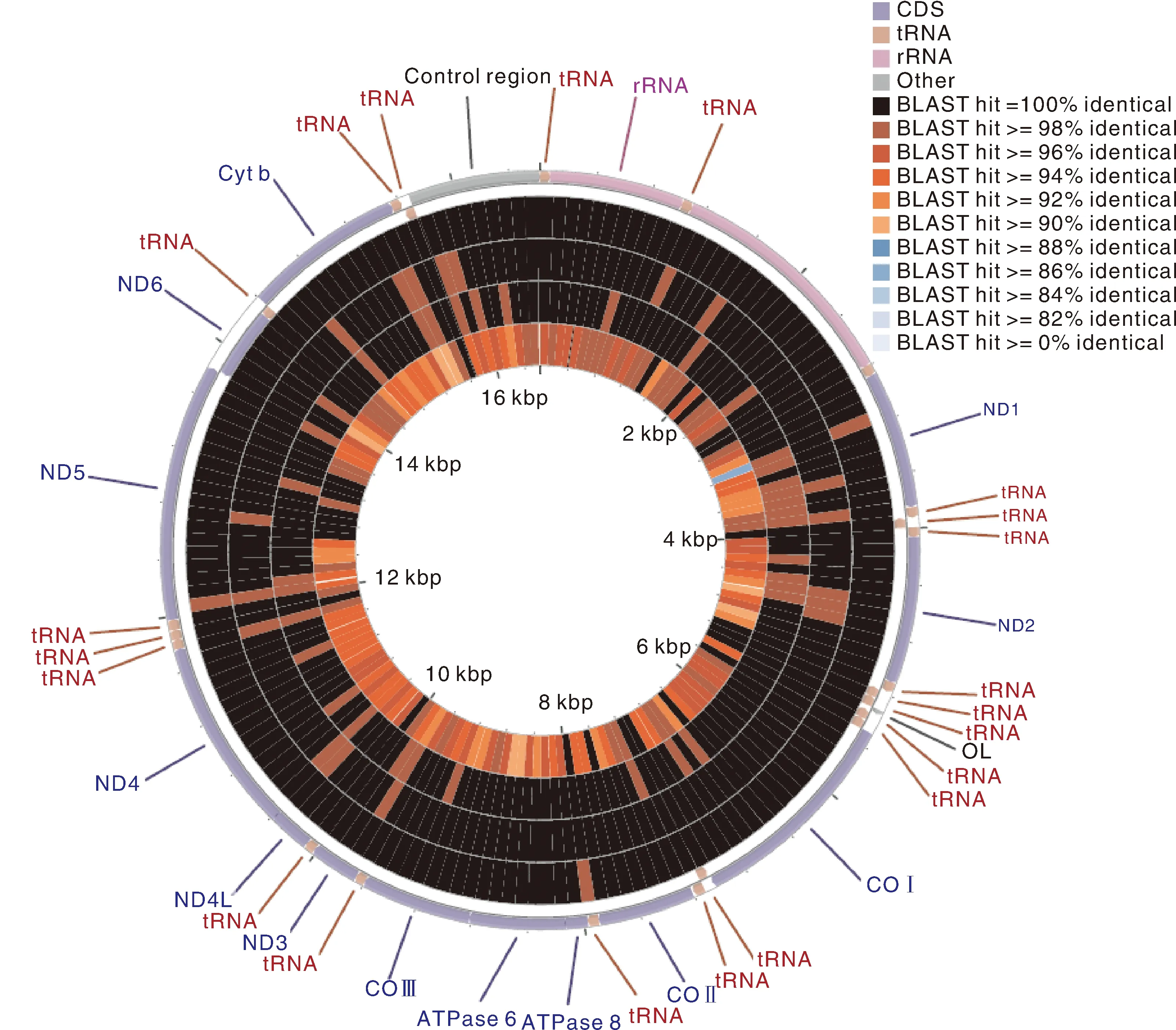
圆环由外及里依次代表钱塘江三角鲂MN725725,钱塘江三角鲂MH289767,珠江三角鲂JX242528,黑龙江三角鲂MH289765,广东鲂JX242530。The circle from outside to inside, represented Megalobrama terminalis from Qiantang River MN725725, Megalobrama terminalis from Qiantang River MH289767, Megalobrama terminalis from Pearl River JX242528, Megalobrama terminalis from Heilong River MH289765, and Megalobrama hoffmanni JX242530.图9 基于BLAST的线粒体基因组序列一致性圆形图Fig.9 Pie chart of the nucleotide percent identity of complete mitochondrial genomes determined by BLAST

续表5 Continued Table 5
3 讨论
本研究测定了钱塘江三角鲂线粒体全长,其序列长度达到16 621 bp,A+T含量为55.97%,碱基含量呈现出AT偏好性。线粒体碱基中G的含量为16.17%,与其他鲂属鱼类[4-5,29]以及太平洋鳕(GadusmacrocephalusTilesius)(16.9%)[30]、黄条鰤(16%)[34]等其他鱼类线粒体中碱基G的含量接近;碱基A、C、T的含量分别为31.23%、27.87%、24.73%,与其他鱼类以及哺乳动物的碱基组成类似[1,28]。这种碱基的偏好性,可能是自然突变以及选择压力等原因造成的[35]。
钱塘江三角鲂线粒体具有标准数量的22个tRNA,其长度从69 bp到76 bp不等,除了tRNA-Ser(AGY)二级结构由于缺失D臂而不能形成典型的三叶草结构,其他tRNA均可形成典型的三叶草结构。tRNA二级结构一般采用tRNA scan-SE软件[9]和RNAstructure软件[36]进行预测,但tRNA scan-SE软件[9]和RNAstructure软件[36]预测并不十分准确,如太平洋鳕[30]、黄条鰤[34]和兰州鲇(SiluruslanzhouensisChen)[37]中关于tRNA-Ser(AGY)二级结构预测并不符合多数鱼类tRNA-Ser(AGY)二级结构的认识[21]。目前并没有关于鲂属鱼类线粒体tRNA-Ser(AGY)二级结构的分析,因此本研究结合mitotRNAdb数据库[20]上发布的斑马鱼tRNA-Ser(AGY)二级结构,对tRNA scan-SE软件[9]预测结果进行了修正,从而确定了钱塘江三角鲂线粒体tRNA-Ser(AGY)二级结构。此外,与人线粒体tRNA-Leu(UUR)序列比较,本研究预测了钱塘江三角鲂线粒体基因tRNA-Leu(UUR)D臂上存在一个Motif(5′-TGGCAGAGCATGG-3′),其可能参与线粒体的转录与终止,这对于进一步研究三角鲂线粒体功能具有一定意义。
钱塘江三角鲂线粒体基因组中12S rRNA和16S rRNA均为单拷贝且基因内部无间隔区,且存在串联重复序列,以及序列插入与缺失等现象,这些均符合后生动物的特征[34,38],也是物种进化的象征[34]。RNA由于其独特的化学特性和进化地位,其二级结构与其功能息息相关,因此RNA二级结构在RNA的功能和系统学研究中被广泛应用,而在其中又以rRNA基因最为常见[39]。由于生物序列复杂多样,rRNA结构预测仍是生物信息学中的一道难题,目前关于鱼类12S rRNA和16S rRNA二级结构分析的文献也相对较少。毛明光等[30]、史宝等[34]对太平洋鳕、黄条鰤线粒体基因组中12S rRNA和16S rRNA仅仅从碱基组成上进行了简单分析,连总强等[37]则参照黑尾地鸦(PodoceshendersoniHume)12S rRNA 和麦穗鱼(PseudorasboraparvaTemminck & Schlegel)16S rRNA基因二级结构绘制了兰州鲇的12S rRNA和16S rRNA二级结构。赖瑞芳等[4]和Hu等[5]关于鲂属鱼类12S rRNA和16S rRNA也是从碱基组成上进行了简单分析,而本研究则利用RNAcentral网站[8]的Auto Traveler软件,基于鲤鱼线粒体12S rRNA(b.16.m.C.carpio)文件和人线粒体16S rRNA(mHS_LSU_3D)文件,对钱塘江三角鲂线粒体基因组中12S rRNA和16S rRNA二级结构进行了预测,这为进一步研究三角鲂rRNA结构与功能提供了基础资料。
钱塘江三角鲂D-loop区的前端和后端共发现了2个串联重复序列。关于串联重复序列形成的机制观点众多[40],如重组和转座、不等交换或基因转换、滑链以及非正常延长等。由于脊椎动物线粒体DNA中一般不发生重组现象[41],蔡珊珊等[40]认为,滑链错配是串联重复序列形成的主要原因,这在核基因中同样存在[42]。D-loop区中的TAS区是鱼类线粒体中变异积累最多的区域,重复序列也多发生在TAS区。这在钱塘江三角鲂,康氏似鲹[33],黄条鰤[34]以及黄鲫(SetipinnatenuifilisValenciennes)[40]等鱼类中均有发现,且在康氏似鲹[33]D-loop区内存在多个TAS区。钱塘江三角鲂线粒体D-loop区的CSB区的关键序列CSB-F、E、D,与其他鱼类[4-5,29,33-34,40]CSB区的关键序列也比较类似,且CSB-E关键序列中含有GTGGG-box[31]的类似序列GAGGG。CSB-D被认为可能与H-链的复制[43]和D-loop的起始[44]有关,还可能与线粒体的代谢有关[31]。因此,CSB区被认为是整个D-loop区最关键的部分[32]。本文研究的钱塘江三角鲂具有哺乳动物上所描述的CSB1特征序列(GACATA)[32],这与鲹科鱼类和鲈形目鱼类有所差别[33]。此外,钱塘江三角鲂线粒体D-loop区中不仅发现了CSB1,还发现了CSB2和CSB3这2个保守序列,这在康氏似鲹[33]、黄条鰤[34]以及黄鲫[40]等鱼类中均有发现,而CSB2和CSB3在哺乳动物的D-loop区中却不是普遍存在的[28,32],这也表现出了鱼类与哺乳动物的差异性。
20世纪50年代以前,鲂属一直被认为在我国境内只有1个种,鲂,通常称为平胸鳊,拉丁名为Megalobramaterminalis。一直到1955年,易伯鲁撰文表示,鲂属鱼类在我国境内至少有2个种,三角鲂和团头鲂,三角鲂沿用原来鲂的拉丁名,而团头鲂则使用了新的拉丁名Megalobramaamblycephala。后来,成庆泰等[2]将鲂属鱼类划分为3种,团头鲂、鲂和广东鲂。罗云林[45]基于鲂属鱼类分类的已有研究,将鲂属修订为4种,三角鲂、团头鲂、鲂和厚颌鲂。现在普遍认为鲂属鱼类为4个种[46],分别是三角鲂、团头鲂、广东鲂和厚颌鲂,其中三角鲂为罗云林[45]所指的鲂,广东鲂则为罗云林[45]所指的三角鲂,团头鲂、厚颌鲂与罗云林[45]的分类相同。基于线粒体基因组的BLAST、遗传距离和系统进化树分析,赖瑞芳等[4]文中所称的广东鲂(文中用拉丁名Megalobramaskolkovii表示,GenBank号为JX242528)应该是现在普遍认为的珠江流域三角鲂,三角鲂(文中用拉丁名Megalobramaterminalis表示,GenBank号为JX242530)应该是现在普遍认为的广东鲂。通过不同流域三角鲂线粒体基因组比较来看,钱塘江流域三角鲂在亲缘关系上与珠江流域三角鲂较近,而与黑龙江流域三角鲂较远。这在一定程度上也反映出,地理位置的远近与亲缘关系的远近有一定相关性。基于线粒体基因组系统进化树分析上的鳊属、鲂属鱼类亲缘关系比较发现,长春鳊与鲂属鱼类亲缘关系较远,但与基于形态分析上的亲缘关系比较稍有出入[47]。这可能是因为基于形态差异的系统进化分析,受到干扰的因素较多,误差较大;而基于碱基差异的系统进化分析,由于碱基的保守性等原因,受到干扰的因素相对较小,因而结果相对准确。基于线粒体基因组系统进化树分析上的鲂属鱼类的亲缘关系分析结果,与其他学者基于线粒体基因组系统进化树分析研究结果一致[4-5],也与基于线粒体细胞色素b构建的鲂属鱼类亲缘关系分析结果一致[46],而与基于微卫星构建的鲂属鱼类亲缘关系分析结果则有所差别[48]。这种分析结果差异的产生可能是由于核基因组和线粒体基因组的差异性所造成。Hu等[5]基于线粒体基因组构建的系统进化树比较了黑龙江流域三角鲂和钱塘江流域三角鲂的亲缘关系,与本研究结果一致,而本研究则基于赖瑞芳等[4]研究结果进一步比较了钱塘江流域三角鲂和黑龙江流域、珠江流域三角鲂的亲缘关系,补充了对三角鲂不同地理群体间亲缘关系的认识。此外,基于同一群体的不同个体间,单核苷酸多态性是比较常见的。对不同年份保存的钱塘江三角鲂样品的BLAST分析表明,2个钱塘江三角鲂线粒体基因组的单核苷酸差异位点有3个,分别位于NAD1,tRNA-Lys和NAD5基因中。
三角鲂的自然分布,一般认为是长江、黄河和黑龙江等水系[2],珠江流域一般不存在自然分布,尽管有报道在珠江流域采集到了三角鲂[4,47-48],这些采集到的三角鲂的原始种很可能来自于钱塘江水系或长江等其他水系。从基于线粒体基因组构建的系统进化树分析结果上看,珠江流域三角鲂和钱塘江流域三角鲂在系统进化树上形成分支的可靠性较低,SH-aLRT检验值38.4%小于80%,ultrafast bootstrap校验值65%也小于95%[49],这也表明珠江流域三角鲂很可能与钱塘江流域三角鲂属于同一个进化分支。此外,从基于线粒体基因组构建的系统进化树分析结果上还发现,在鲂属进化分支中,厚颌鲂处于钱塘江流域三角鲂和黑龙江流域三角鲂进化分支之间,这与Hu等[5]的研究结果一致,究其原因还有待进一步研究。
