基于高密度SNP标记的苹果属15种植物资源的亲缘关系与遗传结构分析
2020-09-11高源王大江王昆丛佩华李连文朴继成
高源,王大江,王昆,丛佩华,李连文,朴继成
(中国农业科学院果树研究所/农业农村部园艺作物种质资源利用重点实验室,辽宁兴城 125100)
0 引言
【研究意义】苹果属(MalusMill.)为蔷薇科(Rosaceae)植物,世界苹果属植物资源约35个种[1-2],主要分布于北温带,横跨欧亚大陆和北美洲;而原产中国的有27个种,其中野生近缘种21种、栽培种6种[3]。研究分析苹果属植物不同种的遗传结构,探讨其遗传多样性,是揭示物种的进化历史[4-5]、分析其进化潜力和未来命运[6]、探讨物种稀有或濒危原因[7-8]的重要方法,对制订种质资源保护策略、指导核心种质资源筛选和相关优异基因挖掘具有重要意义。【前人研究进展】分子标记是揭示不同品种间遗传多样性和亲缘关系的有效手段[9],RAPD[10]、AFLP[11]等分子标记曾被用于苹果的遗传多样性分析,SSR分子标记被认为是动植物中最重要的标记之一[12],在苹果遗传多样性研究中应用最为广泛[13]。SNP即单核苷酸多态性,是由单个核苷酸变异引起的 DNA序列多态性,SNP标记是目前所有DNA分子标记中多样性最为丰富的标记。研究SNP最彻底、最精确的方法即为直接测定某特定区域的核苷酸序列,与参照基因组中对应区域的核苷酸序列进行比较,从而检测出具有多态性的单个核苷酸变异。SNP基因型鉴定已经被应用到许多作物的遗传结构分析、遗传多样性评价和遗传连锁图谱的构建[14-15]。CHAGNE等[16]利用EST-SNP标记的方法对苹果基因进行了研究。现代测序技术的发展,苹果全基因组测序的完成,对于SNP在苹果研究中的应用具有重要的推动作用。MICHELETTI等[17]对 27份苹果属种质进行测序,并筛选出237个SNP标记研究 260份苹果品种的遗传多样性并构建遗传连锁图谱。常源升等[18]、SUN等[19]和孙瑞[20]分别将SNP分子标记的方法用于苹果果形和果实品质相关基因的主基因分析与QTL定位分析,刘更森[21]利用SNP标记构建了苹果遗传图谱。随着苹果参考基因组的发表[22-24],结合现代测序技术,有利于在全基因组范围内寻找多态的SNP分子标记用于苹果属植物种质资源遗传多样性研究,对于其保存和利用具有重要意义[25]。SLAF-seq(Specific Locus Amplified Fragment Sequencing)是一种高通量的简化基因组深度测序技术,通过生物信息学方法设计最佳试验方案,在测序获得海量特异性长度的DNA片段(SLAF标签)基础上,在全基因组范围开发出大量特异性SNP标记。其已经被应用到多种作物,如水稻[26]、甘薯[27]、金花茶[28]和葡萄[29]等的SNP标记开发中,陶红霞[30]基于SLAF技术开发SNP构建苹果遗传连锁图谱。【本研究切入点】随着近些年国家苹果资源圃苹果属植物种质资源收集和保存工作的不断深入,国家苹果资源圃保存了大量待鉴定的苹果属植物种质资源,可在 SLAF-seq基础上从全基因组开发 SNP标记研究苹果属植物不同种的亲缘关系和遗传结构。【拟解决的关键问题】本研究在对国家苹果种质资源圃保存的15个种的427份苹果属植物种质资源进行高通量简化基因组测序基础上,开发SNP标记,解析15种苹果属植物种内和种间的亲缘关系和遗传结构,探讨不同种间的系统演化关系,为不同种苹果属植物的鉴定评价以及进一步收集和保存提供依据。
1 材料与方法
1.1 材料
427份苹果种质材料中有25份取自国家果树种质公主岭寒地果树圃(吉林省公主岭),1份滇池海棠和1份沧江海棠取自国家果树种质云南特有果树及砧木圃(云南省昆明),其余400份材料均取自国家果树种质兴城梨、苹果圃(辽宁省兴城市)。427份种质分属于苹果属的15个种,供试各种名称及种质来源见表1。于2017年春季采集健康幼嫩叶片,叶片经硅胶干燥之后备用。
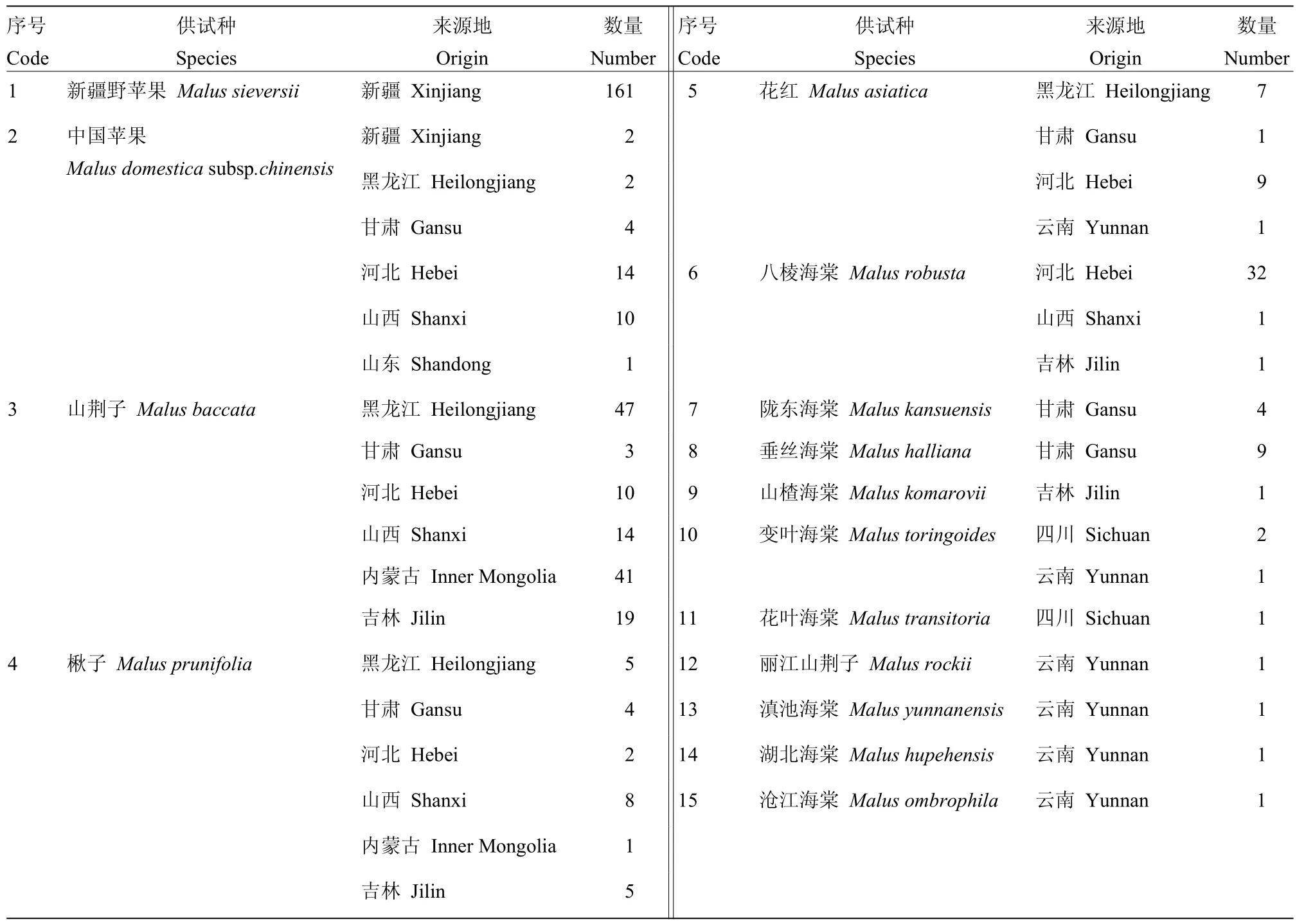
表1 用于SLAF测序分析的15个种苹果属植物种质资源Table 1 Fifteen species of Malus Mill.germplasm resources for SLAF-seq analysis
1.2 基因组DNA的提取
采用德国QIAGEN的DNeasy Plant Mini Kit提取供试材料春季嫩叶的基因组DNA。分别用1%的琼脂糖凝胶电泳和超微量紫外分光光度计(美国DeNovix,DS-11 型)检测其浓度和纯度,对照 λDNA(40 ng·μL-1)将提取基因组的DNA浓度调整到100 ng·μL-1,-20℃保存备用。
1.3 酶切预测
以 2010年已经发表的苹果基因组作为参考基因组进行酶切预测。参考基因组信息:苹果(Malus pumilaMill.)基因组[22](http://www.ncbi.nlm.nih.gov/genome/?term=apple),组装出的基因组大小为1 874.77 Mb,GC含量为45.32%。利用SLAF-predict软件,通过苹果基因组进行方案预测,确定酶切组合并进行酶切。
1.4 测序及数据开发
将获得酶切片段的3′端进行加A处理,在加有polyA的酶切片段上连接测序接头,经过PCR扩增和切胶回收目的片段,构建文库。在 Illumina HiSeqTM2500(美国Illumina公司,HiSeq 2500型)上对检验合格的文库进行测序,测序结果经过去接头、低质量阅读框和污染处理而获得干净序列即SLAF标签。评估测序获得序列的GC含量和Q30指标,检验测序质量。
在不同样品间有差异的 SLAF标签即为多态性SLAF标签。通过BWA软件[31]将SLAF标签与苹果参考基因组[22]以及在不同样品间进行比对,将其定位到参考基因组上获得多态性的 SLAF标签。利用GATK[32]和SAMtools[33]两种方法在多态性SLAF中开发 SNP,筛选两种方法共同得到的 SNP作为开发的SNP标记数据集。根据完整度>0.94,次要等位基因频率(MAF)>0.05过滤[34],筛选多态性的SNP,用于进一步的数据统计和分析。
1.5 群体遗传结构和亲缘关系分析
基于筛选的多态性 SNP,使用 MEGA 7[35]的 NJ(neighbor-joining)算法[36],构建苹果属不同种的系统进化树。不同种间的遗传距离大小用系统进化树的分支长度体现,长度越短即代表两份种质之间的亲缘关系越近。利用Admixture软件[37]进行群体遗传结构分析,假设样品的分群数(K)为 1—15进行聚类,根据交叉验证错误率确定分群数。
2 结果
2.1 建库和测序质量评估
利用 SLAF-predict软件,参照2010年苹果基因组进行酶切预测,确定选择RsaI+HaeIII酶组合进行酶切,筛选SLAF标签长度范围为314—414 bp,共预测到151 808个SLAF标签,SLAF标签在基因组上基本分布均匀。以水稻的测序数据(http://rapdb.dna.affrc.go.jp/)为对照进一步评估酶切的有效性和酶切效率,将水稻的测序数据与苹果参考基因组进行比对,双端比对效率为 95.19%,酶切效率为 92.79%,酶切反应正常,SLAF建库正常。
对所有供试苹果属植物种质测序共获得1 276.7 Mb的读长数据,各种质样品获得的读长数目在 1 189 223—1 968 102 bp,测序平均Q30为91.58%,所有测序样品的Q30值均在80%以上;平均GC含量为40.04%,GC含量普遍较低。用于评估试验建库准确性的水稻测序获得0.45 Mb数据量。测序结果的碱基错误率低,测序数据达到要求。
2.2 SLAF标签与多态性SNP标记筛选
对427份苹果属植物种质测序获得586 454个SLAF标签,平均测序深度为 6.42 X。通过 BWA软件比对获得463 612个多态性SLAF标签,开发5 896 021个群体SNP,根据完整度>0.94、MAF>0.05过滤,共得到46 460个多态性SNP位点,用于后续的群体结构分析。根据筛选的多态性 SNP在染色体上的分布,绘制多态性的 SNP在染色体上的分布图(图1)。
2.3 基于SNP标记的苹果属植物亲缘关系分析
以每个种作为一个种群,基于每个种群的 SNP标记用 MEGA7计算苹果属各种间的遗传距离(表2)。苹果属植物两两种间的遗传距离为0—1.000,其中花叶海棠与变叶海棠、丽江山荆子、滇池海棠、湖北海棠、沧江海棠、陇东海棠和山楂海棠间的遗传距离均为1.000;变叶海棠与湖北海棠、滇池海棠、沧江海棠、陇东海棠、丽江山荆子和山楂海棠间的遗传距离均为0.000;丽江山荆子与滇池海棠、湖北海棠、沧江海棠、陇东海棠和山楂海棠间的遗传距离均为0.000;滇池海棠与湖北海棠、沧江海棠,陇东海棠和山楂海棠间的遗传距离均为0.000,湖北海棠与沧江海棠、陇东海棠和山楂海棠间的遗传距离均为0.000;沧江海棠与陇东海棠和山楂海棠,陇东海棠和山楂海棠间的遗传距离均为0.000。以种间遗传距离做15个种的系统发育树(图2),15个种明显分为 4个类群,类群Ⅰ为陇东海棠、山楂海棠、滇池海棠、沧江海棠、湖北海棠、丽江山荆子和变叶海棠,类群Ⅱ为山荆子,类群Ⅲ为垂丝海棠、楸子、花红和八棱海棠,类群Ⅳ为中国苹果、新疆野苹果和花叶海棠。
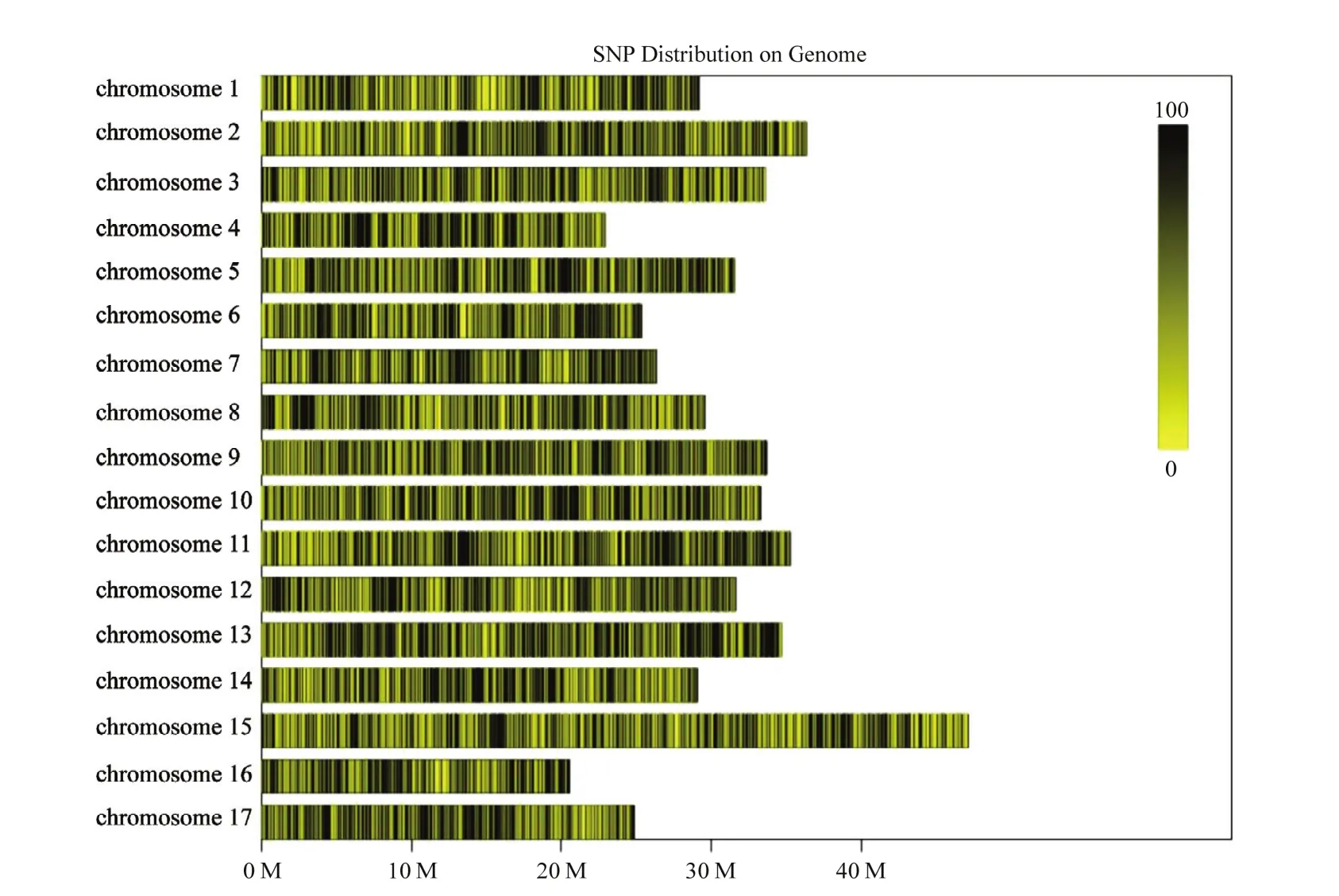
图1 多态性的SNP在染色体上的分布Fig.1 The distribution of polymorphic SNP in 17 chromosomes
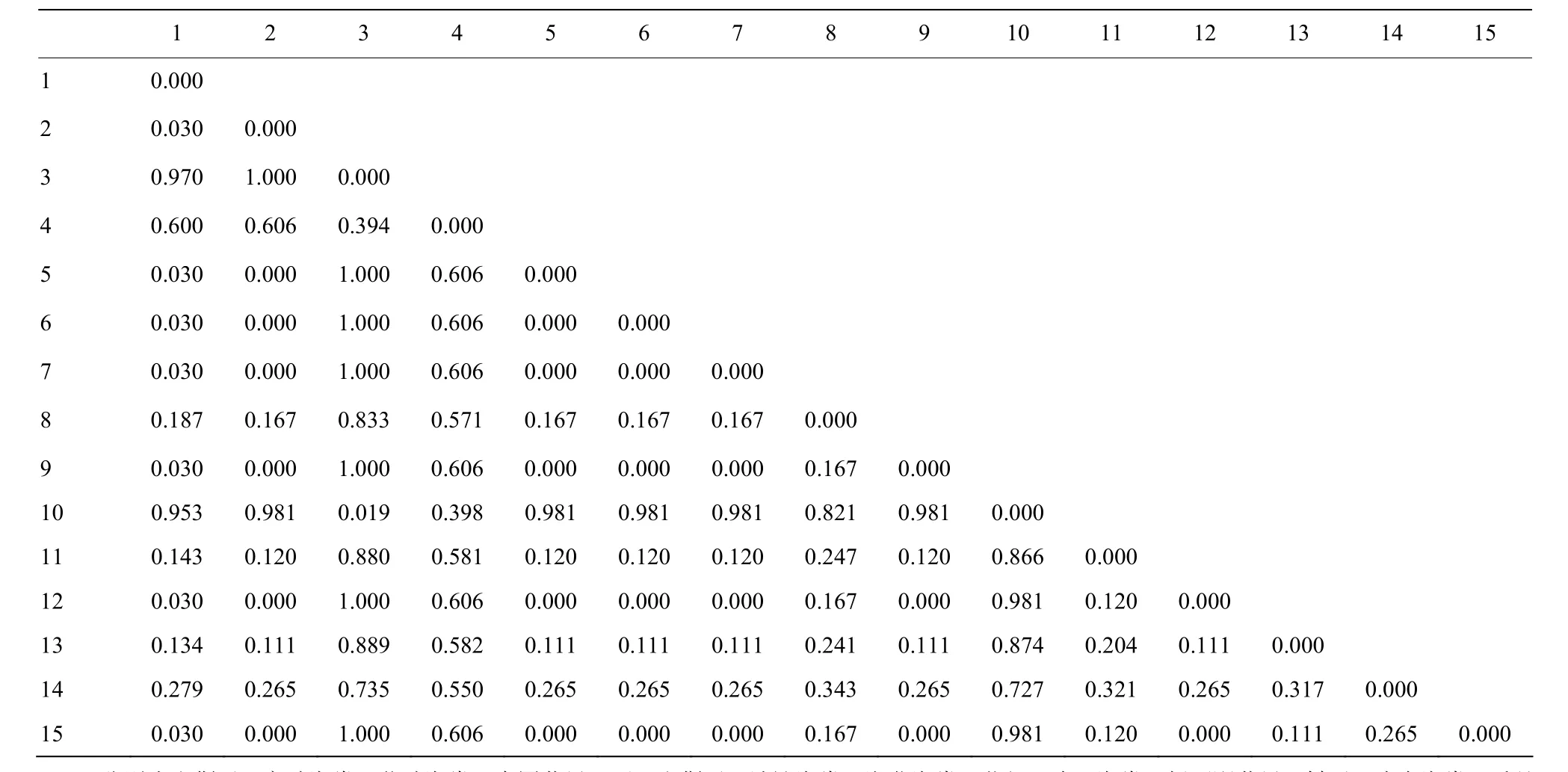
表2 基于SNP的苹果属植物15个种间的遗传距离Table 2 Genetic distance of 15 species of Malus Mill.based on SNP
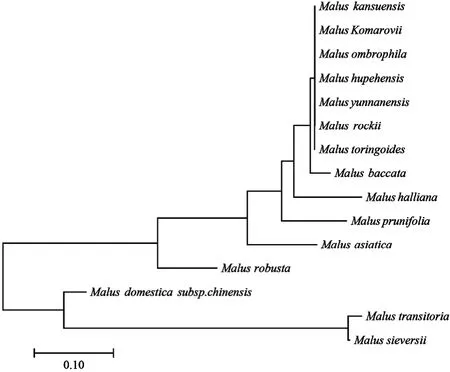
图2 基于SNP位点的苹果属15个种的进化树Fig.2 Polygenetic tree of 15 species of Malus Mill.based on SNP
2.4 基于SNP标记的苹果属植物的遗传结构分析
群体遗传结构分析能够获得个体的血统来源及其组成信息,是遗传关系分析的一种重要手段。运用Admixture软件,分析 427份苹果属植物种质基于多态性SNP的遗传结构。分别假设427份种质的分群数(K值)为1—15(图3),进行聚类。根据交叉验证错误率,当K=5的时候,交叉验证错误率有明显降落,应该为确定分群数的一个关键点,首先确定所有供试苹果属植物的分群数为5,来自于5个可能的原始祖先。随着进一步的分化,当K=14时,交叉验证错误率最小,因此确定最佳分群数为14,反映了这些苹果属植物种质分化后可能来自于 14个祖先。从 K=1—14的群体遗传结构分析见图4。

图3 每个K值对应的交叉验证错误率Fig.3 Cross validation error rates corresponding to every K values
当确定分群数为5时,类群Ⅰ包含25份种质,有3份变叶海棠、3份垂丝海棠、1份丽江山荆子、1分新疆野苹果和17份山荆子,其中有7份山荆子的Q值为1.000,这7份山荆子代表该类群的基因库(蓝色基因库)。类群Ⅱ包含190份种质,有1份八棱海棠、1份垂丝海棠、4份花红、5份楸子、1份山荆子、20份中国苹果和158份新疆野苹果,其中有63份新疆野苹果和1份中国苹果(新疆)的Q值为1.000,这63份新疆野苹果和1份中国苹果代表该类群体的基因库(绿色基因库)。类群Ⅲ包含55份种质,有3份八棱海棠、1份沧江海棠、2份垂丝海棠、1份滇池海棠、12份花红、1份花叶海棠、4份陇东海棠、16份楸子、4份山定子、1份山楂海棠和10份中国苹果,其中有1份八棱海棠、1份花红、1份山荆子、1份中国苹果和1份楸子的Q值为1.000,这5份材料代表了该类群的基因库(淡绿色基因库)。类群Ⅳ包含119份种质,有2份八棱海棠、3份垂丝海棠、1份湖北海棠、2份楸子、2份新疆野苹果、2份中国苹果和107份山荆子,其中有26份山荆子的Q值为1.000,26份山荆子代表了该类群的基因库(黄色基因库)。类群Ⅴ包括39份种质,有28份八棱海棠、3份花红、2份楸子、4份山定子和1份中国苹果,其中16份八棱海棠和1份楸子的Q值为1.000,该17份种质代表了该类群的基因库(浅黄色基因库)。
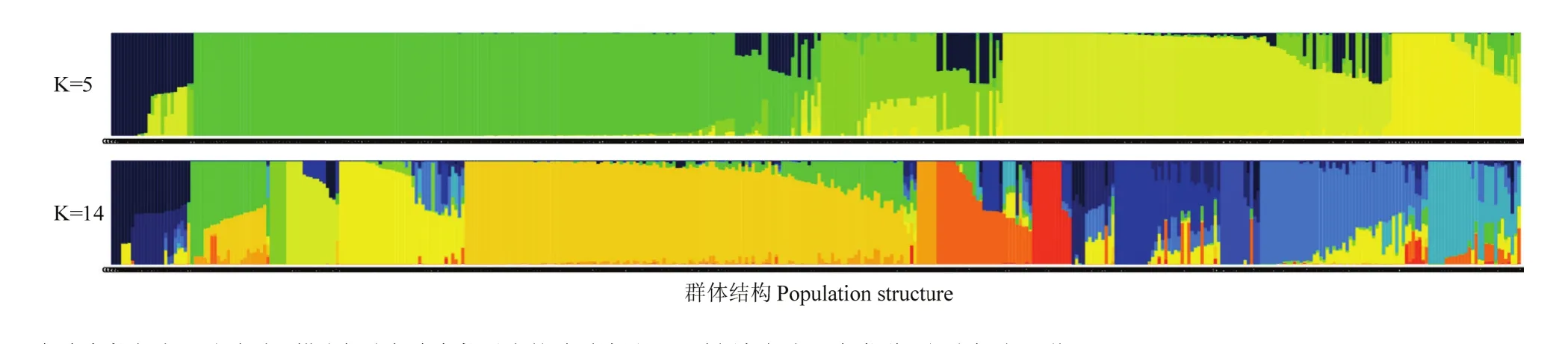
图4 427份15种苹果属植物种质群体遗传结构(K=5和K=14)Fig.4 The genetic structure of 427 accessions of 15 species of Malus Mill.(K=5 and K=14)
当确定分群数为14时,类群1包含24份种质,有14份八棱海棠、3份花红、1份楸子、1份山荆子和5份中国苹果,其中有3份中国苹果(绵苹果)的Q值为0.9999,其代表了该类群的基因库(藏蓝色基因库)。类群2包含37份种质,有1份楸子、1份中国苹果和35份新疆野苹果,其中有2份新疆野苹果的Q值为0.9999,其代表了该类群的基因库(绿色基因库)。类群3包含5份种质,全部为山荆子,且全部为内蒙古收集的山荆子,其Q值全部为0.9999,其代表了该类群的基因库(浅绿色基因库)。类群4包含16份种质,有10份中国苹果、2份花红、2份楸子、1份八棱海棠和1份垂丝海棠,其中有5份中国苹果(2份绵苹果和3份槟子)的Q值为0.9999,其代表了该类群的基因库(浅黄色基因库)。类群5包含38份种质,有1份中国苹果,其余全部为山荆子,其中有6份山荆子的Q值为0.9999,此6份山荆子全部为内蒙古收集到的山荆子,其代表了该类群的基因库(黄色基因库)。类群6包含124份种质,除4份中国苹果、1份山荆子和2份楸子外,其余全部为新疆野苹果,且有11份新疆野苹果的Q值为0.9999,其代表了该类群的基因库(深黄色基因库)。类群7包含了6份种质,全部为新疆野苹果,且Q值全部为0.9999,该类群为由新疆野苹果全部代表的类群(土黄色基因库)。类群8包含28份种质,有2份花红、4份山荆子、8份中国苹果、3份垂丝海棠、8份楸子和3份八棱海棠,其中有1份中国苹果、1份山荆子、1份花红、1份楸子和2份八棱海棠的Q值为0.9999,其代表了该类群基因库(橘色基因库)。类群9包含12份种质,有4份陇东海棠、1份滇池海棠、1份沧江海棠、1份山楂海棠、1份花叶海棠、3份变叶海棠和1份楸子,其中除 3份变叶海棠外的 9份种质的 Q值全部为0.9999,代表了该类群的基因库(红色基因库)。类群10包含13份种质,有4份八棱海棠和9份山荆子,其中占比最重的为深蓝色基因库,但无纯深蓝色基因库的种质,全部为多种基因库混杂的种质。类群 11包含32份种质,有1份丽江山荆子、1份花红、1份中国苹果、4份垂丝海棠、2份新疆野苹果和23份山荆子,其中有9份山荆子和1份垂丝海棠的Q值为0.9999,9份山荆子来源于黑龙江和内蒙古,其代表了该类群的基因库(蓝色基因库)。类群12包含12份种质,由 1份楸子和 11份八棱海棠组成,其中有 7份八棱海棠的Q值为0.9999,其代表了该类群的基因库(浅蓝色基因库)。类群13包含51份种质,除1份垂丝海棠、1份湖北海棠、1份八棱海棠、1份新疆野苹果和3份楸子外,其余全部为山荆子,其中有5份山荆子的Q值为0.9999,其代表了该类群的基因库(天蓝色基因库)。类群 14包含 28份种质,由 10份花红、9份山荆子、3份中国苹果和6份楸子组成,其中 2份楸子、2份花红和 1份中国苹果的Q值≥0.9999,其代表了该类群的基因库(湖蓝色基因库)。只由一个种来代表一个基因库的类群有类群 1、类群2、类群3、类群4、类群5、类群6、类群11、类群12、类群13,代表种分别为中国苹果(绵苹果)、新疆野苹果、山荆子(内蒙古)、中国苹果(绵苹果和槟子)、山荆子(内蒙古)、新疆野苹果、山荆子(黑龙江和内蒙古)、八棱海棠、山荆子(黑龙江)。
3 讨论
SNP变异通常由单个核苷酸碱基的替换、较小片段的插入或缺失引起[38],其类型有单个碱基的转换、颠换、掺入和缺失以及小片段的掺入缺失(InDel)。大多数的SNP并不直接体现在表型上,位于基因的非编码区,但却能够体现群体间的遗传和生物进化关系,可作为遗传标记而应用于动植物的群体遗传关系和进化研究[39-40]。在动植物研究的诸多方面均有应用,诸如果蝇和小鼠[41]等动物以及拟南芥[42]、水稻[43]和小麦[44]等植物。SLAF测序技术的发展促进了SNP标记在多种植物的种质资源鉴定、系统发育、遗传进化以及性状关联分析中的研究,诸如玉米[45]、小麦[46]、棉花[47]、茶树[48]、大豆[49]和葡萄[50]等。本研究在 SLAF测序的基础之上开发了46 460个多态性的SNP标记,分析了15种苹果属植物的亲缘关系和遗传结构,探讨不同种间和种内的系统演化关系。
基于种群间遗传距离的系统发育分析以及群体遗传结构分析均表明,15种苹果属植物分为4个基本的类群,基于种间遗传距离进行系统发育分析时,花叶海棠与新疆野苹果的遗传距离只有0.019,与群体遗传结构的分析结果有所偏差,有可能是供试的15个种在以种作为种群分析时,各种群内个体数量差异较大造成的分析结果有所偏差。
苹果属植物的遗传结构分析中,在K=5和K=14两种分群情况下,陇东海棠、变叶海棠、花叶海棠、滇池海棠、沧江海棠和山楂海棠均在同一类群中,基因来源和背景相似。6个野生种中,除山楂海棠主要分布在中国吉林省长白山地区,其余5个种在中国的西南地区均有野生分布。陇东海棠、变叶海棠、花叶海棠和山楂海棠叶片均有裂刻,只是裂刻的深浅不一。这6个苹果属植物的野生种可能具有相似的起源种,但在演化过程中逐渐出现分化。
当K=5时,类群2和类群3的中国苹果中均有来自于山荆子代表的纯基因库(蓝色基因库)基因,两个类群中各有1份中国苹果种质具有该类群的纯基因库,类群3的中国苹果含有极少量的新疆野苹果基因。此外,八棱海棠具有纯的基因库,其在遗传结构中的作用被突显出来。新疆野苹果和山荆子都有比较纯的基因库,中国苹果的基因并不全来自于新疆野苹果,而有山荆子基因的加入,并与栽培种的关系密切。
当K=14时的群体遗传结构分析表明,中国原产苹果属植物有9个比较纯的同源基因库,其中有2个为中国苹果的同源基因库,2个为新疆野苹果的同源基因库,3个为山荆子的同源基因库,中国苹果中的部分绵苹果和槟子、新疆野苹果、内蒙古和黑龙江的山荆子、八棱海棠代表了较为原始的基因来源。部分中国苹果的种质中有新疆野苹果和山荆子的基因背景,再次证明部分中国苹果在起源演化过程中有新疆野苹果和山荆子的参与,这与 DUAN等[51]的结论一致。但中国苹果中还有一部分绵苹果(主要指彩苹)和槟子可以独立代表类群基因库,其基因库中并没有新疆野苹果的参与,其所在类群与山荆子以及苹果属植物的栽培种花红、楸子和八棱海棠密切相关。花红、楸子和八棱海棠与山荆子的亲缘关系更近,与苹果属植物其他野生种之间的亲缘关系也近于与新疆野苹果的亲缘关系。而根据李育农[3]的理论,中国苹果起源于新疆野苹果,在中国有近2 000年的栽培历史,其作为“西洋苹果”(Malus domesticaBorkh.)引入中国之前的中国特有种,与“西洋苹果”有着不同的起源和演化过程。分析此种情况产生的原因,一是按照CAO等[52]的理论,人类活动可以造成SNP多态性的迅速降低。如果苹果属植物栽培种中国苹果起源于新疆野苹果,在从新疆野苹果向中国苹果的演化过程中,由于人为参与活动而使SNP多态性迅速降低,取而代之的是与其地理位置相近的苹果属其他种对其造成的影响;二是中国苹果中部分类型本身可能也是一个起源种,其起源演化过程与新疆野苹果可能并不相关。此次供试的中国苹果大多数来源于中国的华北地区,来源于废弃果园旁、靠近果园的山坡或者农家院,与新疆野苹果原始集中分布区相距甚远。即使是人类活动和鸟兽等对新疆野苹果向中国苹果的驯化过程有一定影响,也不足以使新疆野苹果基因在部分供试的中国苹果中几乎完全消失。因此,中国苹果与新疆野苹果的起源演化关系有待于进一步考究。随着测序技术的发展,全基因重测序成本的降低,全基因组范围零缺失的SNP标记挖掘将为相关研究提供更多的证据。
4 结论
利用SLAF技术快速挖掘覆盖全基因组的46 460个多态性SNP标记,对427份15种中国原产苹果属植物种内和种间的亲缘关系和遗传结构进行研究。15种苹果属植物分为4个基本的类群,一是山荆子类群,二是新疆野苹果和少数中国苹果类群,三是变叶海棠、花叶海棠、陇东海棠、山楂海棠、滇池海棠和沧江海棠类群,四是4个苹果属植物栽培种中国苹果、八棱海棠、花红和楸子类群。中国原产苹果属植物栽培种中国苹果中部分种质的起源演化过程有山荆子和新疆野苹果的参与,中国苹果与其他栽培种的亲缘关系密切,其与新疆野苹果的起源演化关系有待进一步考究。
