Streptomyces yanglingensis KM-1-2遗传转化体系的建立及 km790的过表达
2020-09-10武立清黄丽丽
武立清,孙 玲,黄丽丽,颜 霞
(1.西北农林科技大学 生命科学学院,陕西杨凌 712100;2.西北农林科技大学,旱区作物逆境生物学国家重点实验室,陕西杨凌 712100; 3.西北农林科技大学 植物保护学院,陕西杨凌 712100)
据统计,目前所发现的抗生素约2/3直接或间接来源于微生物,其中链霉菌是主要的生物活性物质生产者[1-2]。链霉菌是一类好氧、高GC含量的革兰氏阳性菌,能够产生种类繁多的次级代谢产物,在医药、农业以及畜牧业中体现出重要的应用价值。
杨凌链霉菌KM-1-2是从银杏种子中分离到的链霉菌(Streptomyces)新种[3]。全基因组测序结果表明,KM-1-2基因组大小约为5.78 Mb,GC含量达72.8%,预测得到5 299个编码基因,占基因组全长的88.67%。根据antiSMASH在线分析结果,KM-1-2基因组共有21个次级代谢产物生物合成基因簇,包括非核糖体多肽合成酶(Non-ribosomal Peptide Synthetase, NRPS)、聚酮合酶(Polyketide synthase, PKS)、萜烯类(Terpene)、铁载体(Siderophore)及四氢嘧啶(Ectoine)等多种类型。其中NRPS型基因簇Cluster 4上存在1个SARP家族转录调控因子km790,SARP家族转录调控因子包括1个保守的DNA结合结构域(DNA-binding domain, DBD)和1个毗邻的细菌转录激活结构域(bacterial transcriptional activation domain, BTAD),通常被认为在抗生素合成途径中发挥正调控的作用[4-7]。在链霉菌抗生素合成的过程中,SARPs通过招募RNA聚合酶(RNA polymerase,RNAP)到目的基因的启动子上形成1个DNA-SARP-RNAP激活转录复合物[8],从而促进目标抗生素的合成。为了进一步研究菌株KM-1-2的次级代谢产物生物合成基因簇,必须先建立简单高效的遗传转化体系。
链霉菌中外源质粒或基因导入的方法主要有包括PEG介导的原生质体法、接合转移法和电转化法[9-11]。原生质体法是一种常规的基因转移方法,但由于很多链霉菌都具有限制修饰系统[12],所以外源遗传物质难以转化进入,而接合转移操作简便,能够避开胞外核酸酶,在一定程度上可以克服链霉菌对外源遗传物质的限制[13]。Mazodier等[14]于1989年首次建立大肠杆菌-链霉菌的接合转移法;而后随着大量穿梭质粒成功的构建[15],接合转移法作为一种高效的基因转移法被广泛用于链霉菌遗传转化体系的建立。如:Azalomycin F产生菌—链霉菌21172613(Streptomycessp. 21172613)[16]、Toyocamycin产生菌—淀粉产色链霉菌1628(Streptomycesdiastatochromogenes1628)[17]、Xinaomycins产生菌—诺尔斯链霉菌(Streptomycesnoursei)[18]以及谷氨酰胺转氨酶产生菌(Streptomycesmobaraensis)[19]接合转移体系的构建。
本研究首次通过接合转移的方法将外源质粒导入杨凌链霉菌KM-1-2中,建立并优化该菌的遗传转化体系,同时过表达SARP家族转录调控因子km790,并检测过表达菌株的生长状态及对病原真菌的拮抗性,为后续研究KM-1-2的次级代谢产物的合成及其调控奠定基础。
1 材料与方法
1.1 材 料
1.1.1 菌株与质粒菌株 链霉菌(Streptomycesyanglingensis)KM-1-2、大肠杆菌(Escherichiacoli)S17-1、大肠杆菌(Escherichiacoli)DH5α、苹果腐烂病菌(Valsamali)03-8、黄瓜灰霉病菌(Botrytiscinerea)、番茄灰霉病菌(Botrytiscinerea)及葡萄灰霉病菌(Botrytiscinerea)均来自西北农林科技大学果树病害研究室。
质粒:pSET152为大肠杆菌-链霉菌穿梭整合型质粒,具有安普霉素抗性基因acc(3)Ⅳ、接合转移起点OriT、pUC18复制子、噬菌体φC31整合酶(int)基因和attP整合位点[15];pSET152-ermEp*为带有红霉素抗性基因启动子的过表达型质粒,由西北农林科技大学生命科学学院贾良辉老师馈赠;pKC1139为温敏穿梭型质粒,具有安普霉素抗性基因acc(3)Ⅳ,在温度高于39 ℃时不能自主复制,常用于链霉菌的基因敲除[15]。
1.1.2 培养基和试剂培养基 链霉菌KM-1-2产孢培养基为黄豆粉培养基;孢子预萌发培养基为2×YT;接合转移培养基分别采用高氏Ⅰ号、MS、2CMY及黄豆粉培养基;液体培养基为胰蛋白胨大豆肉汤培养基(TSB);发酵培养基为SPY,具体配方参见链霉菌遗传操作手册[20];大肠杆菌培养基为LB;病原真菌采用PDA培养基。
抗生素:安普霉素(Apramycin)、卡那霉素(Kanamycin)、红霉素(Erythromycin)、潮霉素(Hygromycin)、萘啶酮酸(Nalidixicacid)均购自上海生工。
1.2 方 法
1.2.1 质粒及链霉菌基因组提取 大肠杆菌质粒提取及转化参照分子生物学指南[21],链霉菌基因组DNA和质粒的提取参照链霉菌遗传操作手册[20]。
1.2.2 抗生素耐受性测定 分别配制含安普霉素、卡那霉素、红霉素、潮霉素以及萘啶酮酸不同质量浓度梯度(0、0.5、1.0、2.0、5.0、10.0、25.0、50.0 mg/L)的黄豆粉培养基平板,划线接种链霉菌KM-1-2,28 ℃培养6 d后观察菌株的生长 情况。
1.2.3 接合转移 将整合型质粒pSET152通过热激法转入大肠杆菌S17-1中,得到供体菌大肠杆菌S17-1/pSET152。挑取单克隆接种至LB液体培养基(含Apr 50 mg/L),37 ℃、200 r/min培养12~14 h后按体积比1∶100转接于LB液体培养基(含Apr 50 mg/L)中。37 ℃、200 r/min培养至OD600为0.4~0.6时离心收集菌体,用等体积不含抗生素的LB液体培养基洗涤菌体3次,最后将菌体悬浮于0.5 mL的LB培养基中。同时,刮取生长7 d的KM-1-2孢子,放入10 mL蒸馏水中涡旋振荡并过滤,2 739 g离心10 min收集孢子,并悬浮于0.5 mL的2×YT培养基中。45~55 ℃热激10 min后于28 ℃、180 r/min预萌发2~3 h。将备用的大肠杆菌S17-1和预萌发好的KM-1-2孢子悬液按体积比为1∶1的比例混合,涂布于接合转移培养基上,28 ℃培养 16~20 h后覆盖萘啶酮酸(质量浓度25 mg/L)和安普霉素(质量浓度3 mg/L),然后于28 ℃继续培养至长出接合子。本试验中供体菌大肠杆菌S17-1的数量密度保持为108CFU/mL,受体菌KM-1-2孢子数密度为107~109CFU/mL,最后以肉眼可分辨的转化子为准。接合效率=接合子数/孢子量,取3次重复的平均值。
温敏型穿梭质粒pKC1139的接合转移方法同上。
1.2.4 接合子的PCR验证 将接合子转接在含有安普霉素(Apr 3 mg/L)的黄豆粉培养基上,挑取单克隆接种于TSB培养基中,28 ℃、180 r/min培养48 h。
pSET152型接合子的验证需提取链霉菌基因组DNA作为模板。根据安普霉素抗性基因设计验证引物,以链霉菌KM-1-2基因组DNA为阴性对照,质粒pSET152为阳性对照,扩增Apr抗性基因,片段大小为750 bp。PCR反应体系: 5 μL的2×TaqMasterMix,10 μmol/L的正反引物各1 μL,1 μL的DNA模板,1 μL的DMSO,ddH2O补至10 μL。PCR扩增程序:95 ℃ 5 min;94 ℃ 30 s,64 ℃ 30 s,72 ℃ 30 s,30个循环;72 ℃ 10 min。引物由上海生工合成,序列如下:AMF,5′-GGTTCATGTGCAGCTCCATCAGC-3′;AMR,5′-ATGAGCTCAGCCAATC- GACTGG-3′。
pKC1139型接合子的验证需提取链霉菌质粒作为模板。根据pKC1139载体上的序列设计验证引物,以链霉菌KM-1-2的质粒为阴性对照,质粒pKC1139为阳性对照,扩增片段大小为898 bp。PCR反应体系:5 μL的2×TaqMasterMix,10 μmol/L的正反引物各1 μL,1 μL的DNA模板,1 μL的DMSO,ddH2O补至10 μL。PCR扩增程序为:94 ℃ 2 min;94 ℃ 30 s,56 ℃ 30 s, 72 ℃ 30 s,34个循环;72 ℃ 2 min。引物由上海生工合成,序列如下:pKCF,5′-AAGGCGATTAAGTTGGGTA-3′;pKCR,5′-GTGGCGATAAGTCGTGTCT-3′。
1.2.5 过表达菌株Skm790的构建 根据链霉菌KM-1-2中km790基因序列设计引物,并在引物5′端引入pSET152-ermEp*载体双酶切末端同源序列及酶切位点,引物序列如下:km790F,5′-TCGTGCCGGTTGGTAGGATCCCCACAGGAGGACGTGACATGGAGTTCCGGATACT-CGGCCCG-3′;km790R,5′-AGCTTGGGCTGCAGGTCGACTCTAGATCAGCCGGCCGCCG-CCAC-3′,由上海生工合成。模板为链霉菌KM-1-2基因组DNA,PCR反应体系:25 μL的2×Phanta Max Buffer,1 μL的10 mmol/L dNTP Mix,10 μmol/L的正反引物各2 μL,1 μL基因组DNA模板,1 μL 的Phanta Max Super-Fidelity DNA Polymerase,ddH2O补至50 μL。PCR扩增程序:95 ℃ 3 min;95 ℃ 15 s,58 ℃ 30 s, 72 ℃ 1 min,35个循环;72 ℃ 5 min。
PCR扩增产物由1%的琼脂糖凝胶电泳检测,回收后与经BamHⅠ和XbaⅠ双酶切后的pSET152-ermEp*载体回收产物通过一步克隆试剂盒(ClonExpress Ⅱ One Step Cloning Kit)(Vazyme)进行连接,获得的重组质粒转入大肠杆菌DH5α。挑取单克隆提取质粒,PCR检测后测序验证,得到阳性重组质粒pSkm790。将重组质粒pSkm790转入大肠杆菌S17-1中,与野生型链霉菌KM-1-2进行接合转移获得接合子。提取基因组DNA,用Apr抗性引物AMF/AMR进行PCR验证,以野生型链霉菌KM-1-2为阴性对照,质粒pSkm790为阳性对照。
1.2.6 过表达菌株Skm790表型及活性检测 挑取野生型菌株KM-1-2与过表达菌株Skm790的单菌落分别接种于TSB液体培养基中,28 ℃、180 r/min进行培养,每隔24 h取5 mL菌液,重复3次。烘干后测量菌丝体干质量,以培养时间为横坐标,菌丝体干质量为纵坐标绘制生长曲线。此外,将链霉菌KM-1-2和Skm790分别接种于黄豆粉培养基上,28 ℃恒温培养,观察二者的产孢情况。
采用皿内对峙的方法,检测野生型菌株KM-1-2与过表达菌株Skm790对病原真菌的拮抗性。将病原真菌接种于PDA培养基上,25 ℃培养 3 d,链霉菌KM-1-2和Skm790分别接种于黄豆粉培养基上,28 ℃培养7 d,用打孔器分别打取菌饼。然后将病原真菌的菌饼放置在PDA平板中央,距病原真菌菌饼2.5 cm处对称接种野生型菌株KM-1-2和过表达菌株Skm790,25 ℃培养并观察拮抗效果。
1.2.7 菌株发酵及代谢产物分析 将生长7 d的野生型菌株KM-1-2和过表达菌株Skm790分别接种于TSB种子培养基中,28 ℃、180 r/min培养48 h。按体积比为1∶10的比例将种子培养液转接于SPY发酵培养基中,28 ℃、180 r/min培养7 d。将发酵液用乙酸乙酯按体积比为1∶1的比例萃取3次,收集上层有机相旋蒸浓缩,浓缩后的产物用1 mL甲醇溶解后用于后续HPLC分析。
HPLC分析条件:利用C18反向层析柱,采取梯度洗脱的方法,流动相为水(A)和甲醇(B)。整个洗脱过程如下:0~5 min,10% B;5~35 min,10%~100% B;35~45 min,100% B;45~50 min,100%~10% B;柱温25 ℃,进样量10 μL,流速1 mL/min,检测波长210 nm。
2 结果与分析
2.1 KM-1-2遗传转化体系的建立和优化
2.1.1 KM-1-2的抗生素耐受性 链霉菌KM-1-2的抗生素耐受性检测结果如表1所示,其对安普霉素和卡那霉素比较敏感,在Apr 1 mg/L、Kan 5 mg/L的培养基上生长极慢,在Apr 2 mg/L、Kan 10 mg/L时不能生长,对红霉素、潮霉素和萘啶酮酸不敏感,在质量浓度为25 mg/L时仍可生长。因此,接合转移中可用安普霉素或卡那霉素作为抗性筛选标记,用于筛选接合子,用25 mg/L的萘啶酮酸抑制供体菌大肠杆菌的 生长。

表1 KM-1-2对不同抗生素的敏感性Table 1 Sensitivity of KM-1-2 to antibiotics
2.1.2 接合转移培养基的选择 选取高氏Ⅰ号、MS、2CMY及黄豆粉培养基作为接合转移培养基,结果如表2所示,高氏Ⅰ号和MS培养基上均未长出接合子。黄豆粉培养基上接合子长势良好,但大肠杆菌生长速率较快,二者混合生长,难以挑出单菌落。而2CMY培养基上接合子数量多且大肠杆菌生长速率较慢,因此选择2CMY作为接合转移最适培养基。
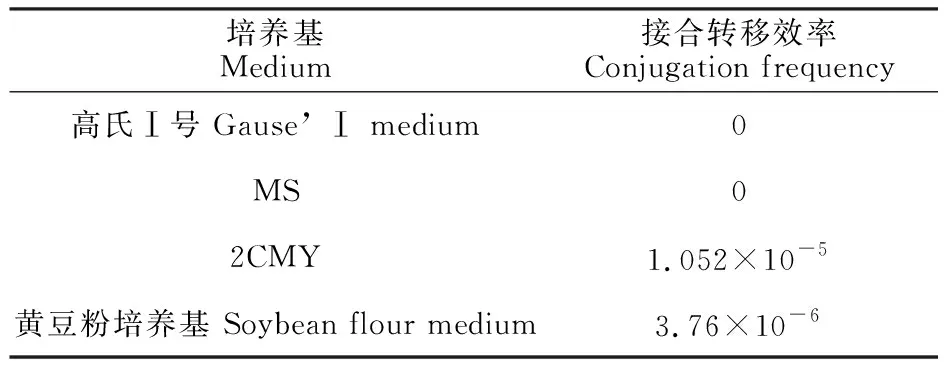
表2 KM-1-2接合转移培养基的选择Table 2 Selection of KM-1-2 conjugation transfer medium
2.1.3 热激温度对接合转移效率的影响 选取45、50、55 ℃ 3个热激温度,分别处理10 min,然后于28 ℃、180 r/min预萌发2.5 h。结果如表3所示,在50 ℃,热激10 min的条件下,接合转移效率最高,达1.052×10-5。温度为45 ℃时,接合效率为5.16×10-6,而当热激温度为55 ℃时,没有接合子出现。因此,50 ℃热激10 min后,于28 ℃、180 r/min预萌发2.5 h为孢子预萌发的最适条件。
2.1.4 接合转移时间对接合转移效率的影响 选取17~18、18~19、19~20 h 3个时间段覆盖抗生素,其中萘啶酮酸的质量浓度为25 mg/L,安普霉素的质量浓度为3 mg/L。结果如表4所示,在17~18 h覆盖抗生素时,接合子较少。在18~19 h覆盖抗生素,接合转移效率最高,达到 1.052×10-5。而在19 h后再覆盖抗生素则得不到接合子。因此,18~19 h,Nal 25 mg/L,Apr 3 mg/L为链霉菌KM-1-2抗生素覆盖的最佳 条件。
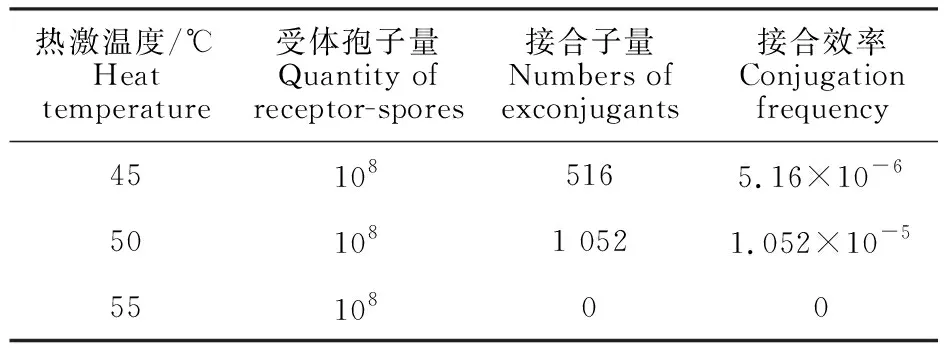
表3 不同热激温度下KM-1-2接合转移效率Table 3 Conjugation frequency of KM-1-2 at differentheat shock temperatures
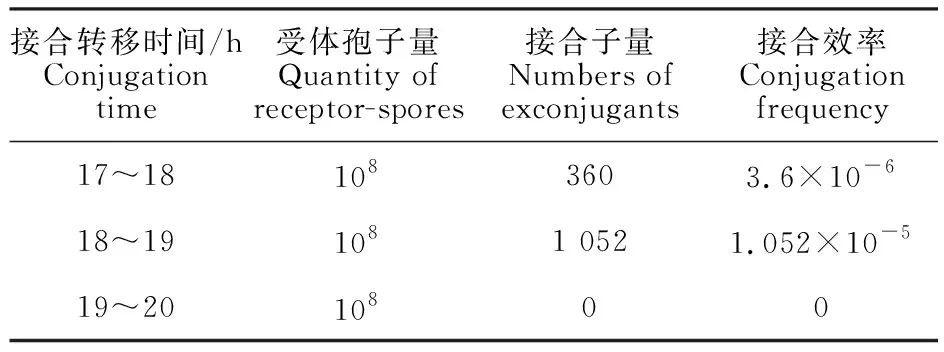
表4 不同接合转移时间下KM-1-2接合转移效率Table 4 Conjugation frequency of KM-1-2at differentconjugation transfer times
2.1.5 接合子的PCR鉴定及稳定性检测 pSET152是基因整合型质粒,在转入链霉菌KM-1-2后,整合在该菌株的基因组上。随机挑取接合子,参照CTAB法提取基因组DNA,利用安普霉素抗性基因引物AMF/AMR进行PCR鉴定。结果表明,阳性对照pSET152和接合转化子均可以扩增出750 bp的Apr基因片段,而野生型链霉菌KM-1-2未扩增出目的片段,由此证明质粒pSET152成功整合在链霉菌KM-1-2的基因 组上。
pKC1139质粒转入链霉菌KM-1-2后,采用碱裂解法提取质粒,利用质粒上的序列引物pKCF/pKCR进行PCR鉴定。结果表明,阳性对照pKC1139和接合转化子均扩增出898 bp的目的片段,而野生型链霉菌KM-1-2未能扩增出该片段,由此证明质粒pKC1139成功转入链霉菌KM-1-2中。
为了检测质粒在接合子中的遗传稳定性,将验证正确后的接合子在含有安普霉素和不含安普霉素的两类平板上进行抗性交替传代,传代10次后接种于TSB培养基中(Apr 3mg/L,Nal 25 mg/L)培养,结果显示接合子均可以正常生长。提取基因组DNA或质粒经PCR鉴定后,与之前的验证结果一致。表明质粒pSET152/pKC1139均可以在KM-1-2中稳定遗传。
2.2 基因 km790的过表达
2.2.1 基因km790的生物信息学 通过antiSMASH分析链霉菌KM-1-2的全基因组,序列分析表明Cluster 4预测为NRPS型基因簇,该基因簇上存在1个SARP家族转录调控因子km790。基因km790编码1个含835个氨基酸的SARP家族蛋白,结果如图1所示。该蛋白存在1个结合DNA的特征性结构—螺旋-转角-螺旋(Helix-turn-helix, HTH)和一个细菌转录激活结构域(Bacterial Transcriptional Activation domain, BTA),二者共同作用促进基因簇的表达。
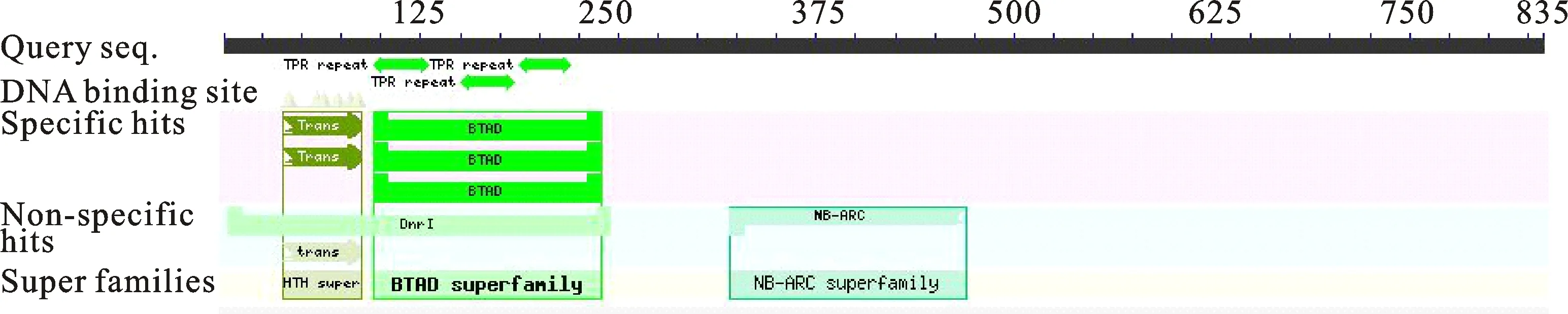
图1 km790编码蛋白的功能结构域预测Fig.1 Functional domain prediction of km790 encoded protein
2.2.2 过表达菌株Skm790的构建 通过接合转移筛选过表达菌株Skm790,提取基因组DNA,利用Apr抗性基因引物AMF/AMR进行鉴定。结果如图2所示,阳性对照pSkm790和过表达接合子Skm790均能扩增出750 bp的目的片段,而野生型链霉菌KM-1-2未能扩增出该片段,表明菌株Skm790过表达成功。

W. 野生型链霉菌KM-1-2;M. DNA Maker;P. 质粒pSkm790;S. Skm790接合子
2.2.3 过表达菌株Skm790的表型及活性分析 野生型菌株KM-1-2和过表达菌株Skm790的生长曲线如图3所示。0~24 h处于菌体生长的迟缓期,24 h后菌体生长迅速,进入对数生长期,于96 h时生长量达到最大值,而当培养至120 h时,菌体生长变缓,甚至有衰亡的趋势。从两株菌的生长曲线可以看出,与野生型菌株KM-1-2相比,过表达菌株Skm790生长速率加快,菌体量 增加。
将野生型链霉菌KM-1-2和过表达菌株Skm790同时划线接种于黄豆粉培养基上,在 28 ℃恒温培养箱中培养,观察野生型菌株和过表达菌株的生长及形态分化,结果如图4所示。在 28 ℃培养4 d时,野生型链霉菌KM-1-2在黄豆粉平板及光学显微镜下可观察到白色的气生菌丝,而过表达菌株Skm790在产生气生菌丝的同时可观察到孢子链和孢子的产生。表明km790的过表达可以促进孢子分化,使菌株的产孢时间提前。
链霉菌KM-1-2和过表达菌株Skm790对病原真菌的拮抗效果如图5所示。结果表明,Skm790对病原真菌苹果腐烂病菌、黄瓜灰霉病菌、番茄灰霉病菌以及葡萄灰霉病菌的抑菌圈均大于KM-1-2,抑制效果明显强于野生型 KM-1-2。
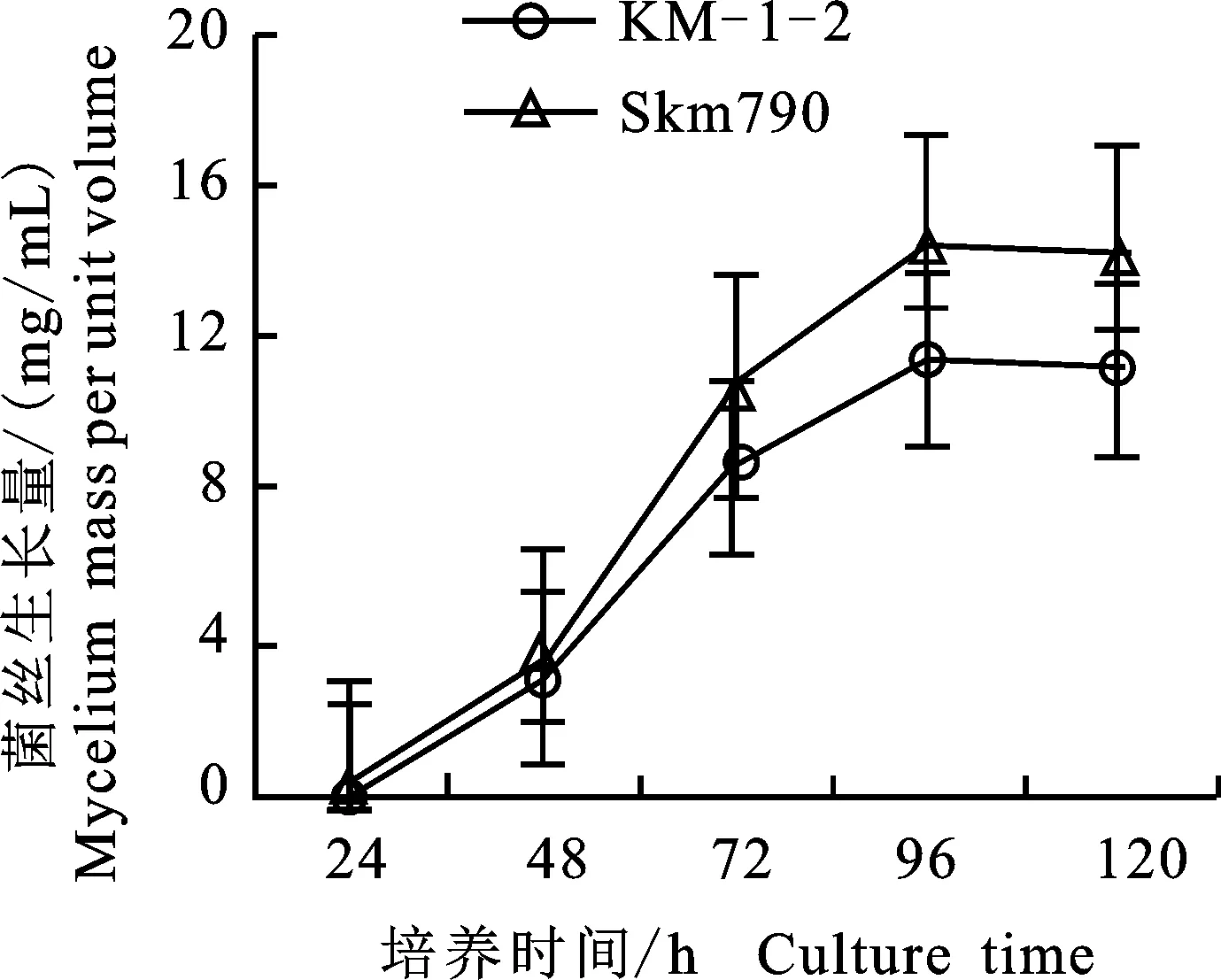
图3 链霉菌KM-1-2和Skm790的生长曲线Fig.3 Growth curve of KM-1-2 and Skm790
2.2.4 次级代谢产物分析 检测波长为210 nm时,野生型链霉菌KM-1-2和过表达菌株Skm790的HPLC分析结果如图6所示。结果显示,在出峰时间为27.15和31.18 min左右时,Skm790对应的吸收峰显著增强,命名为X和Y。二者的紫外扫描结果见图7,结果显示,X对应的物质在(225±1) nm和(277±1) nm处有典型吸收峰,而Y对应的物质在(218±1) nm和(278±1) nm处存在典型吸收峰。HPLC结果表明,基因km790对X和Y对应物质的合成起正调控作用,而本研究表明过表达菌株Skm790对病原真菌的拮抗性显著大于野生型链霉菌KM-1-2,推测X或Y对应的物质可能与菌株的拮抗性相关。
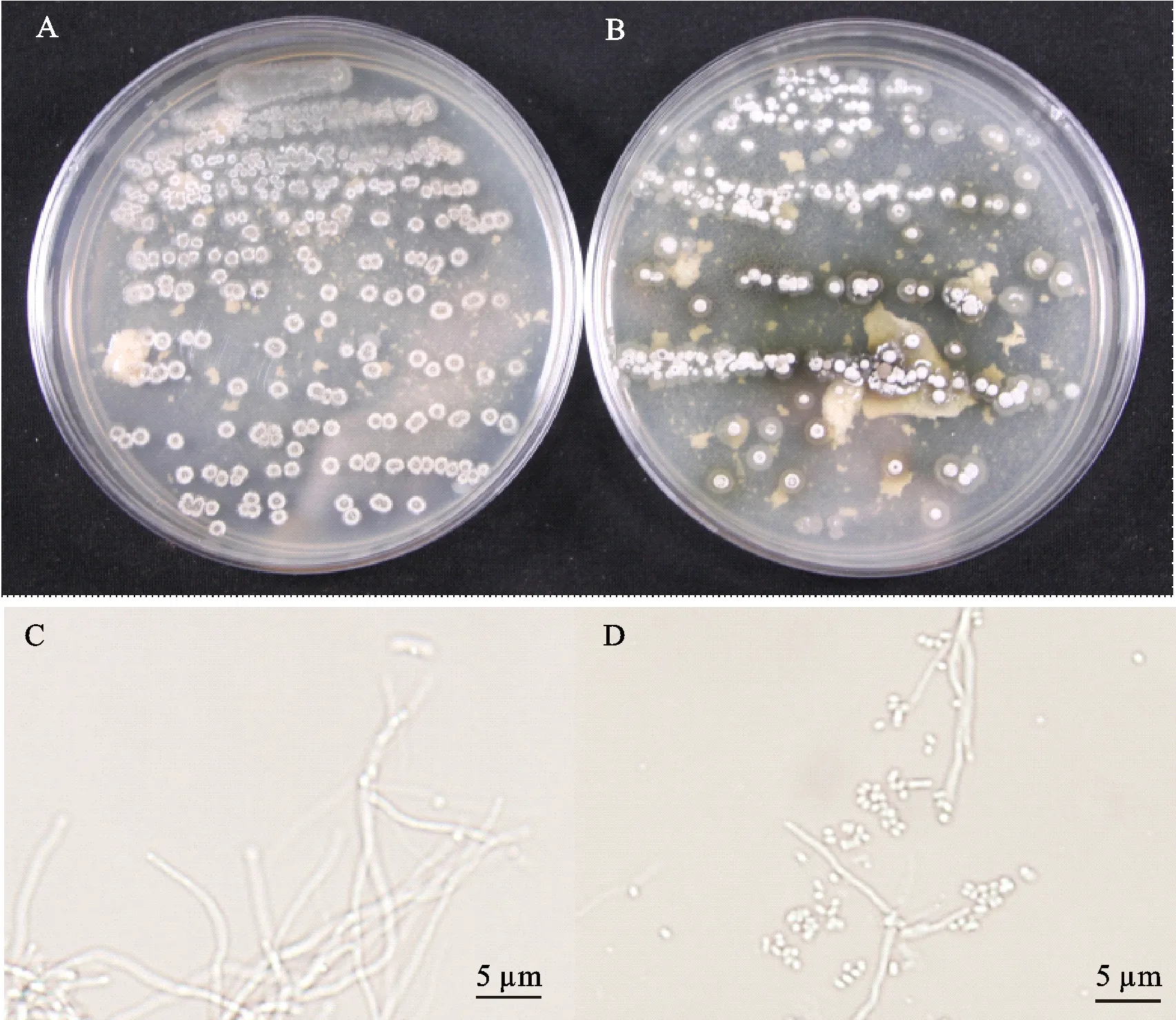
链霉菌KM-1-2在黄豆粉平板上的生长状态(A)及在光学显微镜下的形态观察(C);过表达菌株Skm790在黄豆粉平板上的生长状态(B)及在光学显微镜下的形态观察(D)
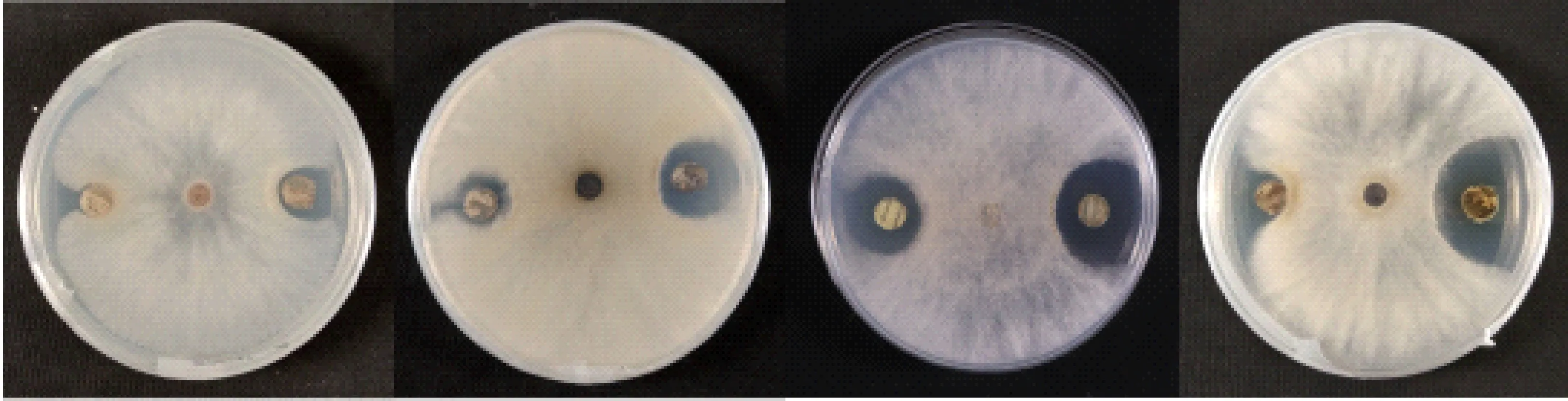
病原真菌从左向右依次为苹果腐烂病菌、黄瓜灰霉病菌、番茄灰霉病菌、葡萄灰霉病菌;左侧菌饼为野生型链霉菌KM-1-2,右侧菌饼为过表达菌株Skm790
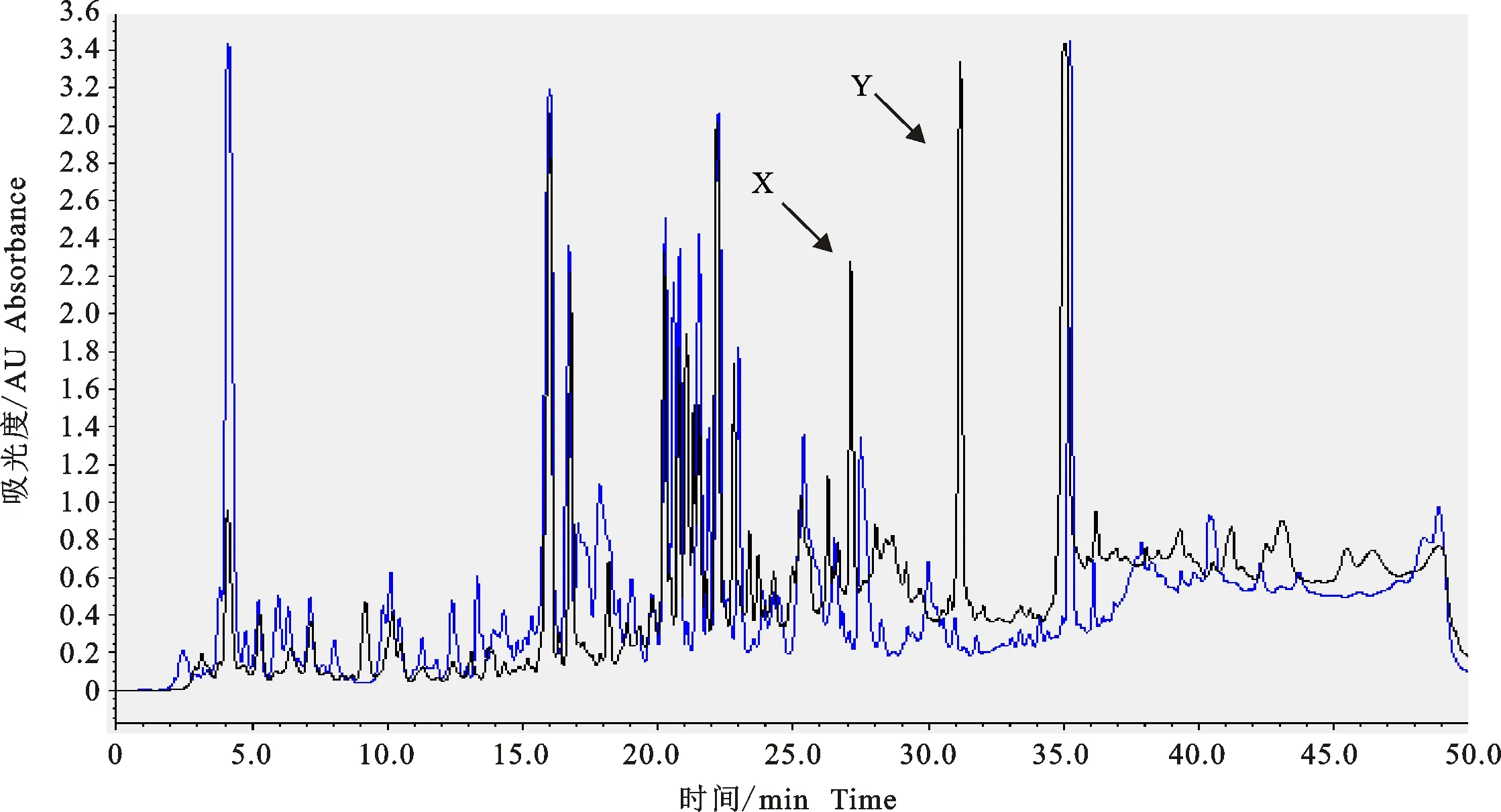
蓝色曲线代表野生型链霉菌KM-1-2,黑色曲线代表过表达菌株Skm790
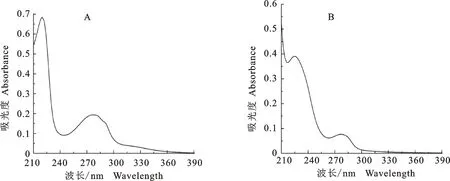
图7 X(A)和Y(B)的紫外光谱Fig.7 UV spectrum of X(A) and Y(B)
3 讨 论
本研究以杨凌链霉菌KM-1-2为出发菌株,通过接合转移的方法建立其遗传转化体系。由于链霉菌是一个高度分化的菌属,不同菌种之间差异很大,适用于一种链霉菌的转化方法不一定适用于其他链霉菌[22-23],而且由于遗传背景的不同,即使同一种链霉菌的不同菌株,转化效率也存在很大差别[24]。因此,本试验在链霉菌KM-1-2抗生素耐受性检测、热激温度、接合转移培养基以及接合转移时间等选择的基础上,探索该菌株的最佳转化条件。
链霉菌KM-1-2在黄豆粉培养基上生长迅速且产孢量丰富,所以以黄豆粉培养基作为该菌最适产孢培养基。在接合转移时,大肠杆菌在黄豆粉培养基上生长速度较快,得到的转化子易被污染,而在2CMY培养基上接合子数量多且大肠杆菌生长速度缓慢,因此选择2CMY作为最适接合转移培养基。研究表明,适当的热激处理可降低细胞内限制酶的活性[25-26],提高接合转移效率,随着热激温度的升高,接合转移效率也随之升高,但温度达到55 ℃时,孢子会丧失活性,因此确定最适热激温度为50 ℃。此外,抗生素覆盖时间对接合转移效率也有显著影响,时间少于17 h,接合转移不完全,覆盖抗生素时极易刮断基内菌丝,而时间长于19 h,大肠杆菌大量生长覆盖培养基,难以挑出转化子。综合以上因素,适合杨凌链霉菌KM-1-2的遗传转化体系为:以2CMY为最适接合转移培养基,孢子于50 ℃热激10 min,抗生素覆盖时间介于18~19 h,其中萘啶酮酸质量浓度为25 mg/L,安普霉素为3 mg/L,此时接合转移效率最高,达1.052×10-5。
SARPs编码基因是不同链霉菌中一种重要的次级代谢产物调控基因,在trioxacarcin、indigoidine、pristinamycin、tacrolimus和chromomycins等的合成中,SARPs作为激活子促进这些次级代谢产物的生物合成[27-31]。本试验利用该转化体系,成功将KM-1-2基因组中SARP家族转录调控因子km790过表达,获得的过表达菌株Skm790生长速率加快,孢子产生时间提前,通过对部分病原真菌的拮抗性检测,发现其抗病性增强。代谢图谱检测分析结果表明,与野生型链霉菌KM-1-2相比,过表达菌株Skm790在保留时间为27.15和31.18 min左右时对应的吸收峰显著增强。推测该吸收峰对应的物质可能与菌株的拮抗性有关,但其确切的功能还需对菌株进行大量发酵,并对该物质分离纯化进行鉴定,从而进一步研究其抗病机制。
杨凌链霉菌KM-1-2遗传转化体系的建立及优化是对次级代谢产物基因簇进行研究的必要前提,为进一步对菌株资源的开发利用奠定基础。
