联合GF-3 PolSAR 数据与Landsat-8 OLI 数据的森林地上生物量估测
2020-08-31潘婧靓邢艳秋黄佳鹏汪献义
潘婧靓,邢艳秋,黄佳鹏,汪献义
(东北林业大学 森林作业与环境研究中心,黑龙江 哈尔滨 150040)
森林地上生物量是森林生态系统长期生产代谢过程的积累,是森林固碳能力的重要标志[1]。准确估测森林地上生物量及其变化对森林资源的管理与经营具有重要的指示作用,同时对研究全球碳循环、气候变化也具有重要意义。
遥感技术以其宏观、快速、动态、可重复等特点被广泛用于区域森林地上生物量估算,已成为目前森林地上生物量估测的主要方法[2]。合成孔径雷达(Synthetic aperture radar, SAR)作为近年来迅速发展的主动遥感技术,对云雾、森林、土壤等具有一定穿透能力,可以全天时全天候工作,其侧视成像、多极化能力可以较好地反映地表植被散射机制[3]。L、P 波段SAR 数据的后向散射系数与森林地上生物量存在较好的相关性,但是目前星载L、P 波段SAR 数据的时空覆盖范围有限,而C 波段SAR 数据来源丰富且极化方式多样[4],在大尺度森林地上生物量估测上具有较大的应用前景。Michele 等[5]和 Chand 等[6]研究表明 C 波段SAR 与森林结构参数存在良好的相关关系。然而C 波段SAR 在林分密集处穿透性不够,在林分稀疏处受到土壤条件影响较大[4]。李明泽等[1]研究发现C 波段SAR 数据的后向散射系数与森林生物量的相关性较差,而结合全极化C 波段SAR 数据的极化分解参数和Landsat-5 TM 光学数据构建回归模型反演森林生物量,效果较好。该研究表明借助极化分解的手段对SAR 的参数进行组合优化,可以改善C 波段SAR 数据后向散射系数估测森林地上生物量的不足,且利用不同遥感数据源所含信息的互补性,能够较准确地反演森林生物量。然而统计回归模型容易受到遥感数据多重共线性的影响,在样本数较少时易发生过学习现象[7]。
为解决以上问题,神经网络[8]、随机森林[9]、KNN 方法[10]等一些非参数方法被用于森林生物量的估测。KNN 方法作为一种典型的非参数方法,能够克服传统统计回归方法对自变量间非共线性限定的缺陷[10],在样本数量较少的情况下易于估算缺失值。李春梅等[11]和Shao 等[4]结合Landsat-8 OLI 数据和SAR 数据使用KNN 方法估测森林地上生物量,其研究结果表明,KNN 方法估测森林地上生物量的精度优于统计回归模型的估测精度。然而多源数据遥感的结合容易引起数据冗余和维度灾难,影响森林地上生物量的估测精度,所以多源遥感数据特征选择方法的确定就显得尤为重要。常用的特征选择方法是依据遥感特征与森林地上生物量的相关程度进行特征筛选[12],而韩宗涛等[13]研究表明“最优”特征组合并非是遥感特征与森林地上生物量相关性由强到弱排序的结果,森林地上生物量与遥感特征之间的关系是复杂多样的,往往难以用简单的线性关系来描述。
综上所述,本研究以广西壮族自治区南宁市高峰林场为研究区,结合高分三号(Gaofen-3,GF-3)全 极 化(Polarimetric synthetic aperture radar,PolSAR)数据的后向散射系数和极化分解参数,Landsat-8 OLI 数据的光谱信息、植被指数、纹理和样地调查数据,提出基于序列前向选择方法的K最近邻法(K-nearest neighbor based on sequence forward feature selection,KNN-SFS),在像素级水平上对森林地上生物量进行估测研究。
1 研究区概况与研究方法
1.1 研究区概况
本研究选取广西南宁市高峰林场的界牌分场和部分东升分场为研究区(图1),地理范围为108°19′ ~ 108°26′E,22°56′ ~ 23°1′N, 总 面 积为42.96 km2。高峰林场是广西规模最大的国有林场,森林经营面积超过934 km2,森林蓄积量超过5.30×106m3,森林覆盖率达85.5%。该区域属于亚热带季风气候,年平均气温21.6 ℃,年均降水量达1 304.2 mm。主要乔木树种有桉树Eucalyptus robustaSmith、杉木Cunninghamia lanceolata、马尾松Pinus massonianaLamb。
1.2 研究数据
1.2.1 样地调查数据
2017 年12 月对研究区进行了野外数据采集,根据林分的密度不同,布设了20 m×20 m 的样地。利用全站仪对样地中心以及样地内所有活立木进行定位,定位精度约0.5 m。利用超声波测高仪对样地内所有单木进行3 次树高测量,取3 次测量的平均值作为单木树高的实测值。同时利用胸径尺对样地内的所有单木进行3 次胸径测量,取平均值作为胸径的实测值。最终在研究区采集的地面样地共33 块(图1)。

图1 研究区地理位置及野外调查样地点分布Fig. 1 Location of the study area and filed sample plots distribution
将样地每木检尺数据代入杜虎等[14]构建的广西主要树种的异速生长模型中,计算每个样地内的单木地上生物量并求和得到每个样地内的总地上生物量,最后将总地上生物量除以样方面积得到单位面积森林地上生物量(t·hm-2)。具体模型见表1,表中D为胸径,WL为叶生物量,WB为枝生物量,WS为树干生物量,森林地上生物量为叶生物量、枝生物量、树干生物量之和。
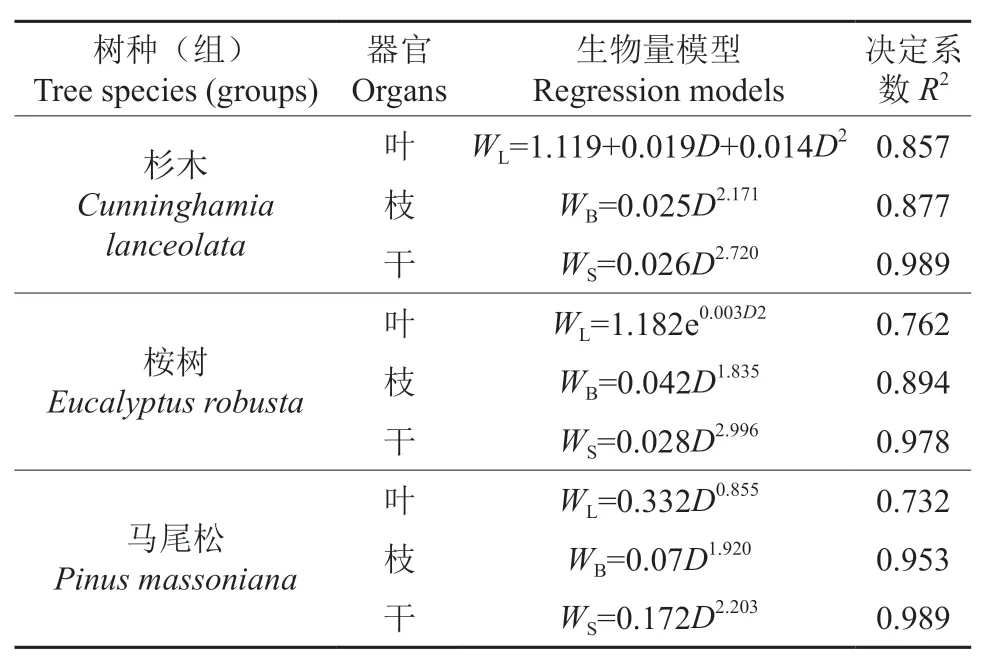
表1 研究区树种(组)生物量估算模型Table 1 The biomass regression models for trees species (groups)
1.2.2 GF-3 PolSAR 数据
GF-3 卫星是我国于2016 年8 月10 日发射的首颗高分辨率C 波段多极化SAR 遥感卫星,具有聚束模式、方位多波束模式、条带模式、扫描模式、四极化模式、波模式等12 种成像模式,最高分辨率可达1 m,极化方式分单极化、双极化和全极化[15-16]。本研究使用一景GF-3 的全极化8 m 分辨率QPSI 影像,数据获取时间为2017 年10 月,距离向分辨率为2.25 m,方位向分辨率为4.72 m,数据产品参数见表2。
根据GF-3 PolSAR 影像的成像原理以及本研究的应用需求,对影像的预处理工作(图2)包括辐射定标、极化滤波、正射校正和地理编码。为了与Landsat-8 OLI 数据分辨率一致,对GF-3 PolSAR影像进行重采样,重采样后像元大小为30 m×30 m。
1.2.3 Landsat-8 OLI 数据
本研究使用的一景Landsat-8 OLI 影像数据来源于美国地质调查局,成像时间为2017 年10月24 日,影像无云层覆盖,产品级别Level 1T,即已进行了基于地形的几何校正。本研究所用7个波段分别为:海岸波段(0.433 ~0.453 μm)、蓝光波段(0.450 ~0.515 μm)、绿光波段(0.525 ~0.600 μm)、 红 光 波 段(0.630 ~ 0.680 μm)、近红外波段(0.845 ~0.885 μm)、短波红外波段一(1.560 ~1.660 μm)、短波红外波段二(2.100 ~2.300 μm),空间分辨率为30 m。在ENVI 5.3 中对Landsat-8 OLI 数据进行了辐射标定、大气校正及重采样等预处理操作。
1.3 研究方法
本研究先分别提取GF-3 PolSAR 数据和Landsat-8 OLI 数据的特征,然后分别基于GF-3 PolSAR 数据、Landsat-8 OLI 数据以及两种数据联合的3 种数据来源,使用KNN-SFS 方法对森林地上生物量进行估测。

表2 GF-3 PolSAR 数据产品参数Table 2 Parameters of GF-3 PolSAR data

图2 GF-3 PolSAR 数据预处理Fig. 2 GF-3 PolSAR data preprocess
1.3.1 GF-3 PolSAR 数据特征提取
GF-3 PolSAR 数据提取的特征包括4 种后向散射系数和极化分解参数。研究区设置在林区,地物目标为具有普遍的时变效应的分布式地物,基本都是非相干目标,因此主要选取非相干极化目标分解方法进行研究[1],该方法可以根据地物的不同散射机制更加有效地提取目标地物的主要散射特征[17]。为了探究不同极化分解方法所得参数对估测森林地上生物量的敏感程度,使用7 种主要的非相干极化分解方法和1 种可以表征分布式目标的相干极化分解方法提取GF-3 PolSAR 数据的极化分解参数。非相干极化分解方法包括:Yamaguchi4 分解、Yamaguchi3 分解、VanZyl3 分解、Freeman3 分解、Freeman2 分解、Neumann 分解、H/A/Alpha 分解,1 种相干极化分解方法为TSVM分解。
1.3.2 Landsat-8 OLI 数据特征提取
由Landsat-8 OLI 遥感数据提取的特征包括7 个波段光谱值、植被指数和纹理信息。本研究提取的4 个植被指数是基于波段光谱值计算得到,包括归一化植被指数(NDVI)、比值植被指数(RVI)、土壤调节植被指数(SAVI)和差值植被指数(DVI),其计算公式如式(1)~(4)所示:
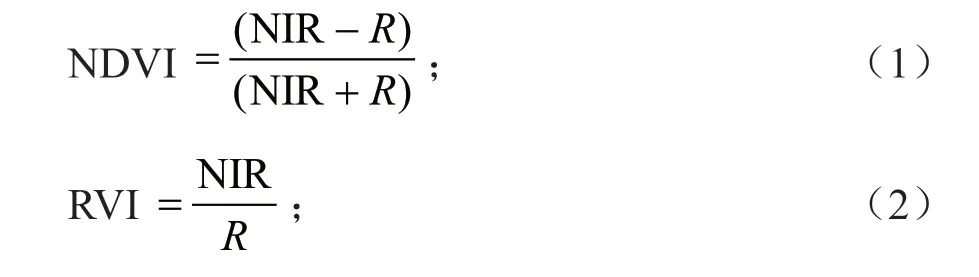
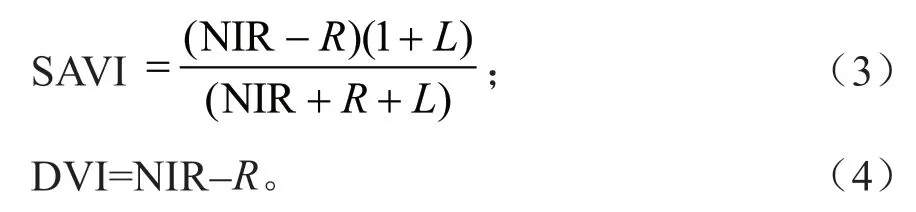
式中:NIR 表示近红外波段;R表示红光波段;L表示土壤调节系数,本研究取L=0.5。纹理基于灰度共生矩阵计算得到,本研究选择窗口为5×5,提取8 种纹理特征,即均值、方差、均一性、对比度、相异性、熵、二阶矩、相关性[11]。
1.3.3 KNN-SFS 方法森林地上生物量估测
本研究选择KNN 法作为估测森林地上生物量的方法,该方法通过结合遥感数据提取的特征和实测样地点数据估测森林地上生物量,待估像元的生物量由距离它最近的K个样地的生物量加权求得。基于序列前向选择方法(Sequential forward selection,SFS)来进行特征选择,其基本原理为先设特征子集为空集,每次选择一个特征加入特征子集,计算评价函数,最后根据评价函数来选择最优的特征子集[18]。KNN-SFS 方法具体步骤如下(设特征数为m):
1)以样地点坐标提取样地点的遥感特征作为训练数据,特征向量为Sm=[X1,X2,...,Xm];
2)初始化最优特征子集Pk为空;
3)依次选取Sm中的一个特征分别与Pk组合,使用KNN 法估测森林地上生物量,选出RMSE 最小的特征加入Pk,并从Sm中剔除;
4)迭代直到所有特征都被选入Fs,对比每轮迭代中的RMSE,选出最优解RMSE0,并找到RMSE0对应的特征组合作为最优特征组合。
当距离度量标准确定时,KNN 估测结果受K取值影响。K值越大,估测结果越容易向平均值的方向平衡并呈集中分布趋势,此时估测结果难以维持原有的森林参数统计分布特征[10],所以K取值不宜过大,本研究设置K的取值范围为1 ~10。研究采用留一法交叉检验方法,森林地上生物量KNN 估测结果采用均方根误差(Root Mean Square Error,RMSE)和决定系数R2进行评价。
2 结果与分析
基于33 个样地中心点对应的遥感影像像元对GF-3 PolSAR 数据进行特征提取,获得后向散射系数和极化分解参数共47 个特征;对Landsat-8 OLI数据进行特征提取,获得波段光谱值、纹理和植被指数共67 个特征。然后分别针对3 种数据源(GF-3 PolSAR 数据、Landsat-8 OLI 数据、GF-3 PolSAR 数据及Landsat-8 OLI 数据)基于KNNSFS 方法对特征进行选择并对森林地上生物量进行估测,以样地点实测生物量为验证数据,采用留一法交叉验证,得到3 种数据源的森林地上生物量估测精度(表3)。
从表3 可以看出,随着K值的增大,3 种数据源估测的森林地上生物量与实测样地森林地上生物量之间的均方根误差先减小后增大,R2先增大后减小,即估测精度先增大后减小;当K值较小时,多源遥感数据的估测精度要显著优于单一来源遥感数据的估测精度,而当K值较大时,多源遥感数据的精度与单一来源遥感数据的精度差距较小。这是因为随着K值的增大,KNN 方法估测森林地上生物量的结果会向平均值的方向平衡,标准差会逐渐减小,估测值呈集中分布趋势,导致精度降低。
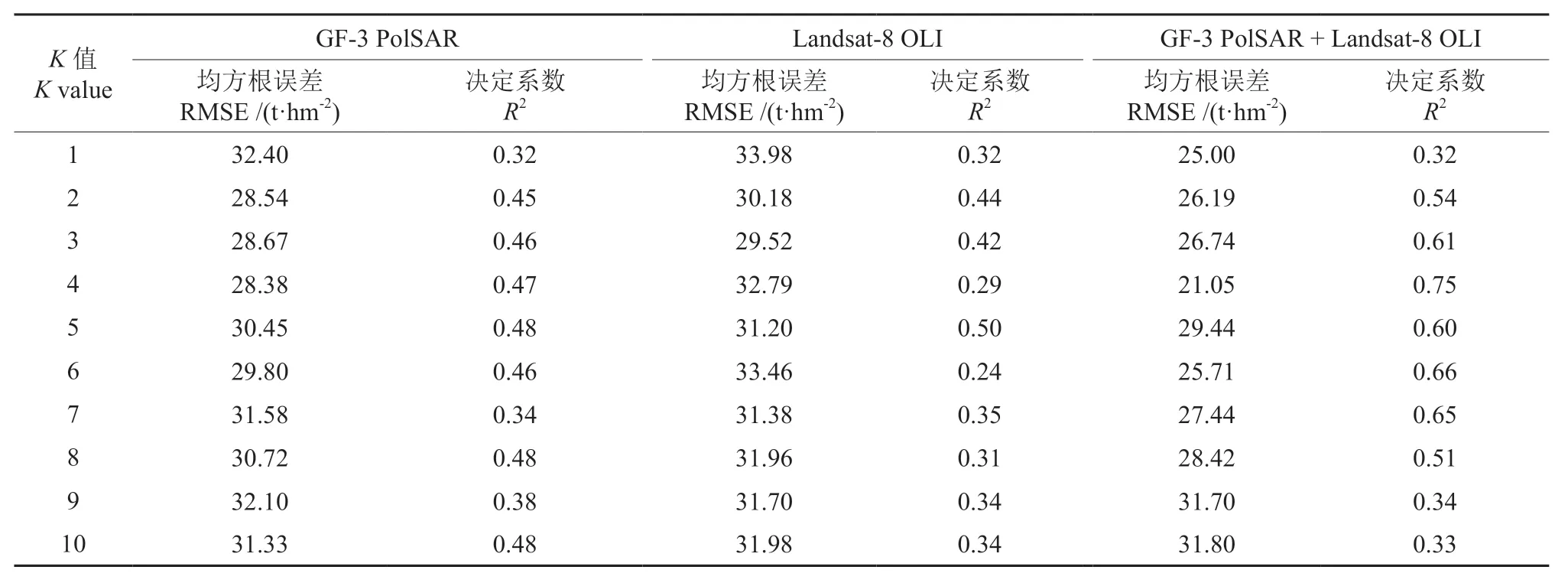
表3 不同数据源估测精度对比Table 3 The comparison of different data source’s estimation accuracy
当K=4 时, 联 合 GF-3 PolSAR 数 据 和Landsat-8 OLI 数据估测的森林地上生物量与实测样地森林地上生物量的均方根误差最小,精度最优,估测精度为 RMSE=21.05 t·hm-2,R2=0.75;当K=4 时,使用GF-3 PolSAR 数据估测精度最优,估测的森林地上生物量与实测样地森林地上生物量的均方根误差为28.38 t·hm-2,R2=0.47;当K=3时,使用Landsat-8 OLI 数据估测精度最优,为RMSE=29.52 t·hm-2,R2=0.42。3 种数据源估测精度最优时的森林地上生物量估测值与样地实测值的散点图见图3,最优特征组合见表4。
从图3 可以看出,当估测精度最优时,联合GF-3 PolSAR 数据和Landsat-8 OLI 数据估测森林地上生物量的精度高于单独使用GF-3 PolSAR 数据或Landsat-8 OLI 数据的估测精度。当K=4 时,联合两种数据源估测森林地上生物量与实测森林地上生物量的相关关系达到0.864,模型预测效果较好。图3 也反映出使用KNN-SFS 方法估测时,在高生物量区会低估实际生物量,在低生物量区会高估实际生物量,这与KNN 方法的聚类性质有关,因为KNN 方法实质上是一种反距离加权平均,所以它估测的生物量是在实测样地生物量的范围内的。
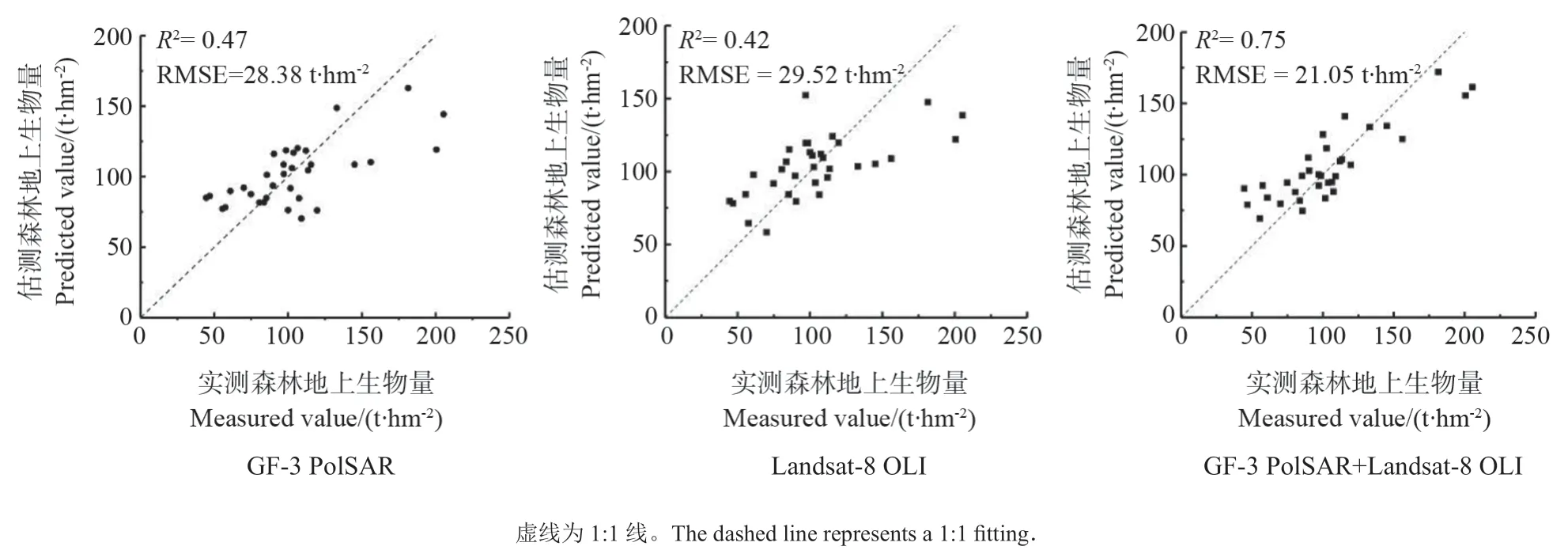
图3 KNN-SFS 估测森林地上生物量交叉验证结果Fig. 3 The cross-validation results of estimating forest above-ground biomass using KNN-SFS
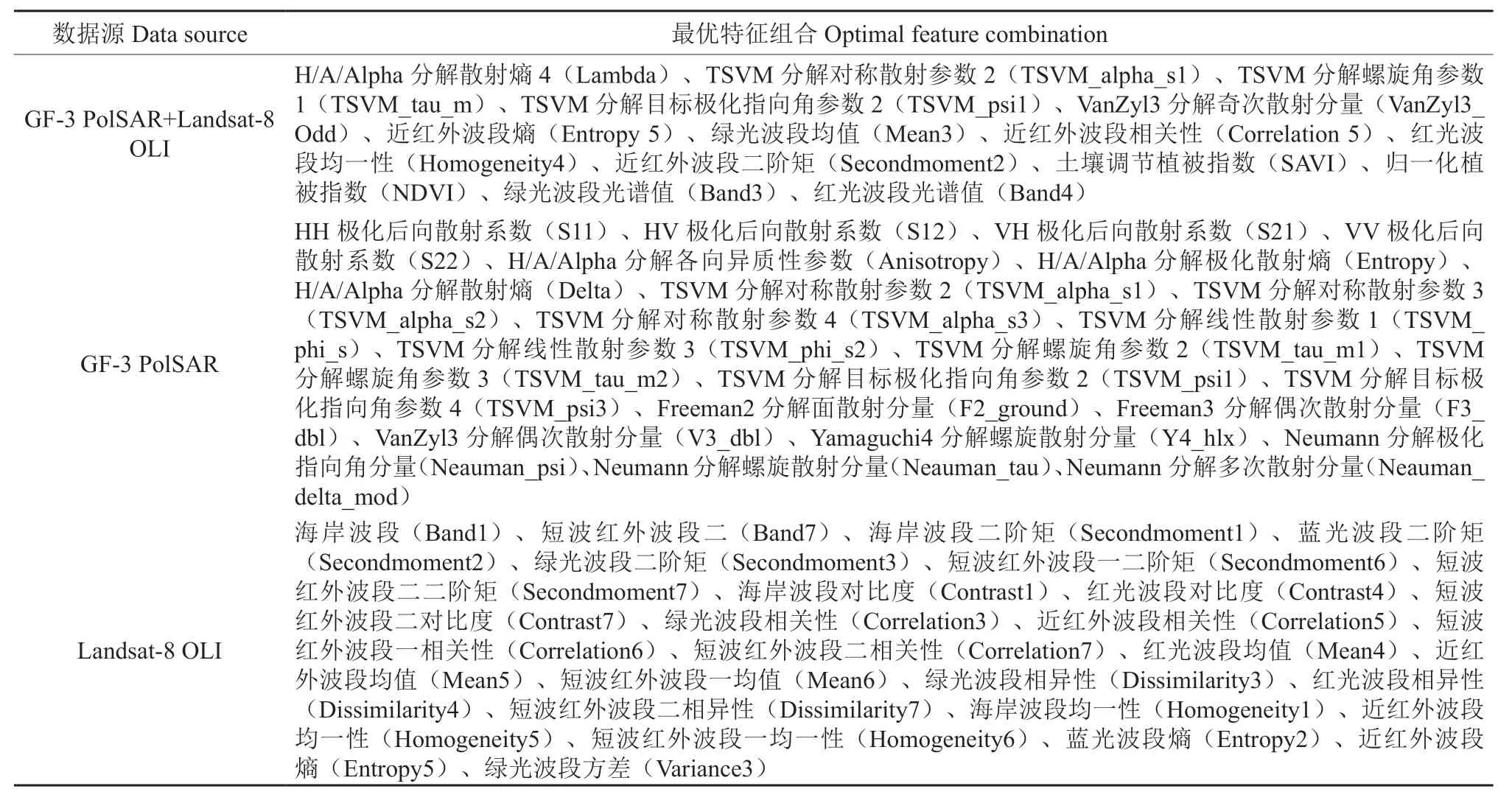
表4 3 种数据源最优特征组合Table 4 Optimal feature combination of three data sources
GF-3 PolSAR 数据筛选出的最优特征组合包含23 个特征,Landsat-8 OLI 数据筛选出的最优特征组合包含26 个特征,均较联合两种数据筛选出的最优特征组合特征数(14 个)多,表明联合多源数据的结合在一定程度上可以充分利用不同传感器的优点,优化特征组合,减少估测森林地上生物量所需的特征数,降低特征冗余度。从表4 可以看出,联合GF-3 PolSAR 数据和Landsat-8 OLI 数据估测森林地上生物量的最优特征组合主要来自H/A/Alpha 分解、TSVM 分解、VanZyl3 分解、纹理、植被指数和波段光谱值,包含了除了后向散射系数以外的所有特征类型;GF-3 PolSAR 数据估测森林地上生物量的最优特征组合主要来自后向散射系数、H/A/Alpha分解、TSVM分解、Freeman2分解、Freeman3 分解、VanZyl3 分解、Yamaguchi4 分解和Neumann 分解,只有Yamaguchi3 分解没有特征参数选入;Landsat-8 OLI 数据估测森林地上生物量的最优特征组合包括了部分波段光谱值和所有纹理类型,没有植被指数选入。从参数入选的角度来看,H/A/Alpha 分解、TSVM 分解、VanZyl3分解对森林地上生物量比较敏感,纹理、植被指数和光谱值均对提高估测精度起到了一定的作用。
结合表4 中的多源数据估测森林地上生物量的14 个最优特征,利用KNN 估测模型,生成研究区森林地上生物量分布图(图4)。
3 结论与讨论
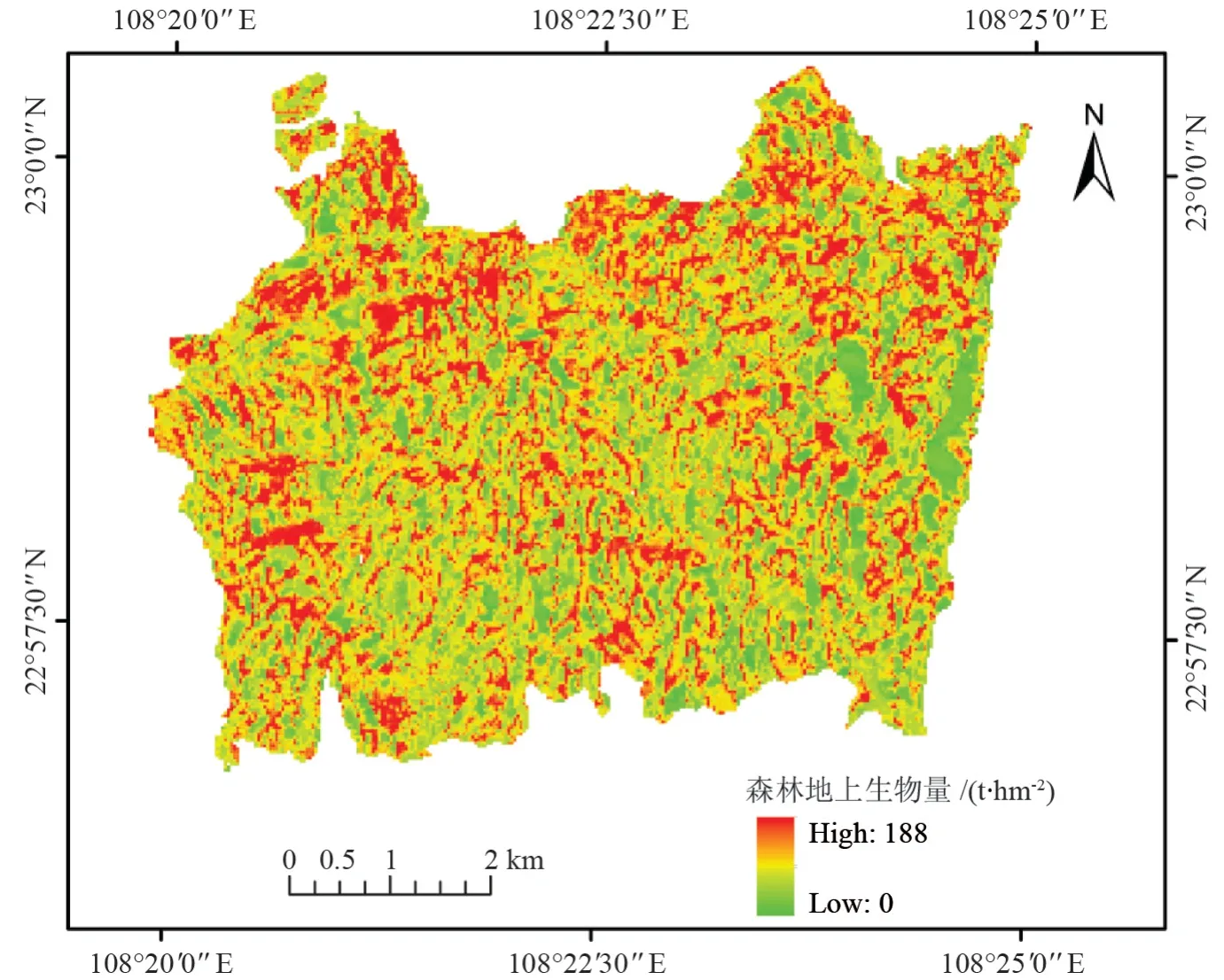
图4 研究区森林地上生物量分布Fig. 4 Distribution of forest aboveground biomass of the study area
本研究以广西南宁市高峰林场为研究区,基于GF-3 PolSAR 数据、Landsat-8 OLI 数据和野外实测样地数据,利用KNN-SFS 方法估测森林地上生物量。研究结果发现,联合GF-3 PolSAR 数据和Landsat-8 OLI 数据估测森林地上生物量的精度优于单独使用GF-3 PolSAR 数据或Landsat-8 OLI数据的估测精度。基于GF-3 PolSAR 数据特征估测森林地上生物量的精度为RMSE=28.38 t·hm-2、R2=0.47,基于Landsat-8 OLI 数据特征估测森林 地 上 生 物 量 的 精 度 为 RMSE=29.52 t·hm-2、R2=0.42,均低于联合这两种数据特征的估测精度(RMSE=21.05 t·hm-2,R2=0.75)。这是因为单一来源遥感数据在光谱和辐射分辨率上存在一定的局限性,在植被覆盖相对密集的地区,Landsat-8 OLI 数据由于波长较短,估测森林地上生物量时容易受到饱和现象的影响,而GF-3 PolSAR 数据在林分密集处穿透性不够,在林分稀疏处受到土壤条件影响较大,影响了估测森林地上生物量的精度。为了提高遥感数据估测森林地上生物量的能力,应该结合多源遥感数据,利用每种传感器的优点:Landsat-8 OLI 数据光谱信息丰富,其不同光谱波段组成的植被指数能够较为准确地反映植被生长状况,而GF-3 PolSAR 数据具有穿透顶层树冠的能力,不仅与叶生物量有关,也与树枝生物量和树干生物量有关,且GF-3 PolSAR 数据可以穿透云雾,能够减少大气对定量估测的影响,在森林地上生物量估测中有一定的优势,因此结合GF-3 PolSAR 数据和Landsat-8 OLI 数据能够提高估测生物量的精度。
本研究联合GF-3 PolSAR 数据和Landsat-8 OLI 数据,使用KNN-SFS 方法估测生物量,估测精度为 RMSE=21.05 t·hm-2,R2=0.75。韩 宗涛等[13]联合P 波段机载SAR 数据和Landsat-8 OLI数据,利用KNN-FIFS 方法估测大兴安岭根河森林保护区的森林地上生物量,估测精度为RMSE=22.74 t·hm-2,R2=0.77,与本研究的精度相近,证明了结合GF-3 PolSAR 数据和Landsat-8 OLI 数据使用KNN-SFS 方法估测森林地上生物量的精度较高,方法可行,为生物量估测提供了一种新的遥感研究方法。本研究在使用SFS 方法进行特征选择时,发现H/A/Alpha 分解、TSVM 分解、VanZyl3 分解等极化分解方法对森林地上生物量比较敏感,但是其物理作用机制尚不明确,今后可以探究不同极化分解方法对于森林地上生物量的作用机制,以筛选出更加适合反演森林地上生物量的极化分解方法及参数。
