不同恢复方式下二郎山公路创面土壤酶活性与土壤肥力变化特征
2020-08-31郝云庆易津鑫郑文丽李文俊吴军佑李伟
郝云庆, 易津鑫, 郑文丽, 李文俊, 吴军佑, 李伟
1. 成都信息工程大学资源环境学院,四川 成都 610225;
2. 中国科学院水利部成都山地灾害与环境研究所山地表生过程与生态调控重点实验室,四川 成都 610041
从20 世纪80 年代以来,我国经济的高速增长,极大地刺激了资源能源的开发、交通体系的完善。施工不仅破坏了原生植被,还对土壤生态系统造成不同程度的影响,引起诸如水土流失等后续生态问题。土壤酶作为森林生态系统的重要组成部分,主要来源包括植物和土壤微生物的分泌物,发挥着调节土壤生化过程,参与土壤中有机质分解、养分循环和能量流动过程的重要作用[1-4],它对土壤中氮磷化合物的活化和转移过程以及提高土壤肥力方面起着重要作用[5]。土壤酶活性作为土壤质量和生态系统功能变化的生物活性指标,可反映土壤中各种生化过程的强度和方向[6]。
对于裸露土地的生态恢复问题不同的学者研究的侧重点不同,在人工恢复和自然恢复方面都有所收获。碳、氮、磷、钾、有机质等是土壤养分的主要组成部分[7],酶是土壤养分吸收及转化的催化剂[8],二者相互作用一定程度上共同决定着土壤生态系统的健康发展。因此,土壤养分与土壤酶活性综合研究可较客观地反映土壤功能和肥力的变化[9],不同土地利用方式和人工措施下土壤养分与酶活性变化量及其互作关系也成为评价土壤生态功能恢复的研究热点[10]。李海林[11]、方航空[12]等人研究了人工植被恢复下土壤养分与酶活性变化关系多基于不同植被类型间的比较,郭曼[13]、杨宁[14]等人从草地、灌草、灌木到纯林等不同植被演替阶段分析了土壤养分与酶活性存在的协同恢复关系,也有研究表明,土壤酶系统变异性大[15-16],不同种类酶活性对生态措施和土壤养分变化响应显著程度不同[11]。
中国西部地区属多山地形,该地区进行高速公路建设基本是挖山打隧道,严重扰动了土壤环境[17],土壤环境改变的同时会影响土壤中酶促反应发生变化[18]。本研究以二郎山公路创面土壤酶活性与土壤肥力在人工恢复和自然恢复方式下的变化特征为研究对象,通过分析5 种土壤酶活性变化及与土壤理化性质的相关性,揭示不同恢复方式下不同土壤肥力指标对土壤酶活性的响应机制,为土壤创面退化生态系统的恢复与重建提供参考。
1 研究区概况
二郎山位于四川省天全县、泸定县境内,主峰海拔3 437 m,海拔高差悬殊,是青衣江、大渡河的分水岭,为自然地理的分界线。其东西坡气候差异明显,山地气候特征显著。东坡年降雨约1 900 mm,多集中于8—10 月;西坡年降雨仅900~1 000 mm,多风少雨少冰雪霜冻。土壤复杂多样,自然土壤主要为水稻土、潮土、紫色土、黄壤、高山寒漠土。生物种群复杂,物种丰富,据不完全统计,共有维管束植物68 科380 属1 500 余种,其中国家重点保护珍稀树种便有18 种。山麓多亚热带植被,随海拔上升依次出现暖温带、寒温带植被乃至高山灌丛草甸。
2 研究方法
2.1 样品采集
于2018 年4 月中旬,采用典型抽样法对四川二郎山东西坡进行样地布点,选取G318 路面边坡植被遭破坏后人工恢复(见图1)和自然恢复采样点以及东西坡面土壤背景值共10 个采样点(见图2),在每个采样点采用蛇形法随机布设3 个1 m×1 m 样方采集0~10 cm、10~20 cm 各2 kg 土样,共计60 份样品,带回实验室后,避免阳光直射,自然风干,去除石子、树枝等,充分混匀后采用四分法收集土壤,一份存留、一份研磨后分别过2 mm、0.25 mm、0.149 mm 筛,以用于土壤养分各要素的测定分析。

图 1 人工恢复措施示意图Fig. 1 Schematic diagram of artificial restoration modes
2.2 测定项目与方法
土壤脲酶活性用苯酚钠-次氯酸钠比色法测定,以24 h 后1 g 土壤中NH3-N 的毫克数表示土壤脲酶活性;土壤蔗糖酶活性用3-氨基-5-硝基水杨酸比色法测定,以24 h 1 g 干土生成葡萄糖毫克数表示;土壤磷酸酶活性用磷酸苯二钠比色法测定,以每克土壤的酚毫克数表示;过氧化氢酶活性用高锰酸钾滴定法测定,以每克干土1 h 内消耗的0.1 mol/L KMnO4体积数(以mL 计)表示;土壤蛋白酶活性用茚三酮比色法测定,以24 h 后1 g 土壤中甘氨酸的毫克数表示[20]。
图 2 采样点布设示意图Fig. 2 Schematic diagram of sampling points
2.3 数据处理
采用Excel 和SPSS 21.0 统计软件进行数据处理及描述性分析。采用RDA 分析对研究区土壤酶活性影响因子进行排序,分析得出主控影响因素并对其进行排序。
3 结果与分析
3.1 不同恢复方式下土壤酶活性变异特征
通过对不同恢复方式下5 种壤酶活性的变异特征分析发现,除了脲酶活性外,其余4 种土壤酶活性的变异系数较小,各指标均变化较稳定。峰度是描述总体中所有取值分布形态陡缓程度的统计量[21],除了脲酶活性的峰度值为正数外,其余土壤酶活性峰度值都出现负值,表示该总体数据分布为平顶峰,与正态分布相比较为平坦。人工恢复方式下的脲酶活性偏度值为1.4,总体的偏度值都较小都在1 以下,因此整体数据出现的极端值也不会特别大,只有脲酶活性这一指标值会出现极值情况(见表1)。

表 1 不同恢复方式下5 种酶活性的变异特征Tab. 1 Variation characteristics of enzyme activities under different restoration modes
在自然恢复和人工恢复下,同种土壤酶活性变化有所不同(见图3)。从图3a 看出不同恢复方式下脲酶活性变化差异较大,最大差值为4.295 mg·g-1·24 h-1(点1),其中除了采样点5 和8 外,自然恢复方式下下脲酶活性值高于人工恢复值,采样点5 相差1.06 mg·g-1·24 h-1,点8 相差2.86 mg·g-1·24 h-1,说明在自然恢复方式下对于大部分采样点的脲酶活性值产生正反馈作用。从图3b 看出两种恢复方式下,蛋白酶活性的变化差异不大,均在1 mg·g-1·24 h-1以下,最大差值仅为0.77 mg·g-1·24 h-1,说明无论何种恢复方式下对土壤蛋白酶活性的影响不大。从图3c可知,除了采样点2 和5 外,人工恢复下磷酸酶活性均小于自然恢复状态下的磷酸酶活性,除采样点1 外1.662 mg·g-1·24 h-1,其余采样点在两种恢复方式作用下,差异均小于1 mg·g-1·24 h-1。从图3d 看出不同恢复方式下,各采样点的蔗糖酶活性变化差异较大,最大差异为采样点5 相差6.867 mg·g-1·24 h-1,最小差异 为 采样点1 相 差0.352 mg·g-1·24 h-1,因此在两种恢复方式作用下蔗糖酶活性没有明显的变化规律。从图3e 看出,两种恢复方式作用下,过氧化氢酶活性差值变异较小,最大差值仅为0.121 ml·g-1·24 h-1,最小差值为0.004 ml·g-1·24 h-1,相比前四种土壤酶活性,过氧化氢酶活性受不同恢复方式的影响较小。
3.2 土壤酶活性与土壤肥力相关性分析
通过SPSS 相关性分析得到土壤酶活性与主要肥力因子指标间的相关系数(见表2),结果表明在人工恢复方式下,蛋白酶活性与土壤全氮、全钾之间相关关系极显著(p<0.01),与铵态氮相关关系显著(p<0.05);磷酸酶活性、脲酶活性与土壤各肥力因子指标相关性不显著(p>0.05);蔗糖酶活性与土壤有机质间相关关系极显著(p<0.01);过氧化氢酶活性与土壤全氮、全钾之间相关关系极显著(p<0.01),与铵态氮、全磷之间相关关系显著(p<0.05);并且,从表格中还发现,蛋白酶活性与磷酸酶活性、过氧化氢酶活性三者之间相关关系极显著(p<0.01)。
在自然恢复方式下脲酶活性与土壤有机碳、有机质之间相关关系极显著(p<0.01),与全氮相关关系显著(p<0.05);蛋白酶活性与有机碳之间相关关系极显著(p<0.01);磷酸酶活性与土壤有机碳、无机磷之间相关关系显著(p<0.05);蔗糖酶活性与土壤各肥力因子指标相关性不显著(p>0.05);过氧化氢酶活性与土壤有机碳、铵态氮、全磷之间相关关系显著(p<0.05);脲酶活性与蛋白酶活性之间相关关系极显著(p<0.01),与蔗糖酶活性、过氧化氢酶活性之间相关关系显著(p<0.05);蛋白酶活性与磷酸酶活性、过氧化氢酶活性之间相关关系显著(p<0.05);磷酸酶活性与过氧化氢酶活性之间相关关系极显著(p<0.01)。
结果说明,无论在何种恢复方式下,表现土壤肥力的各个指标与土壤这5 种酶活性之间都有一定的相关关系,如果用土壤酶活性来衡量土壤肥力时需综合考虑各指标之间的相关关系分析。
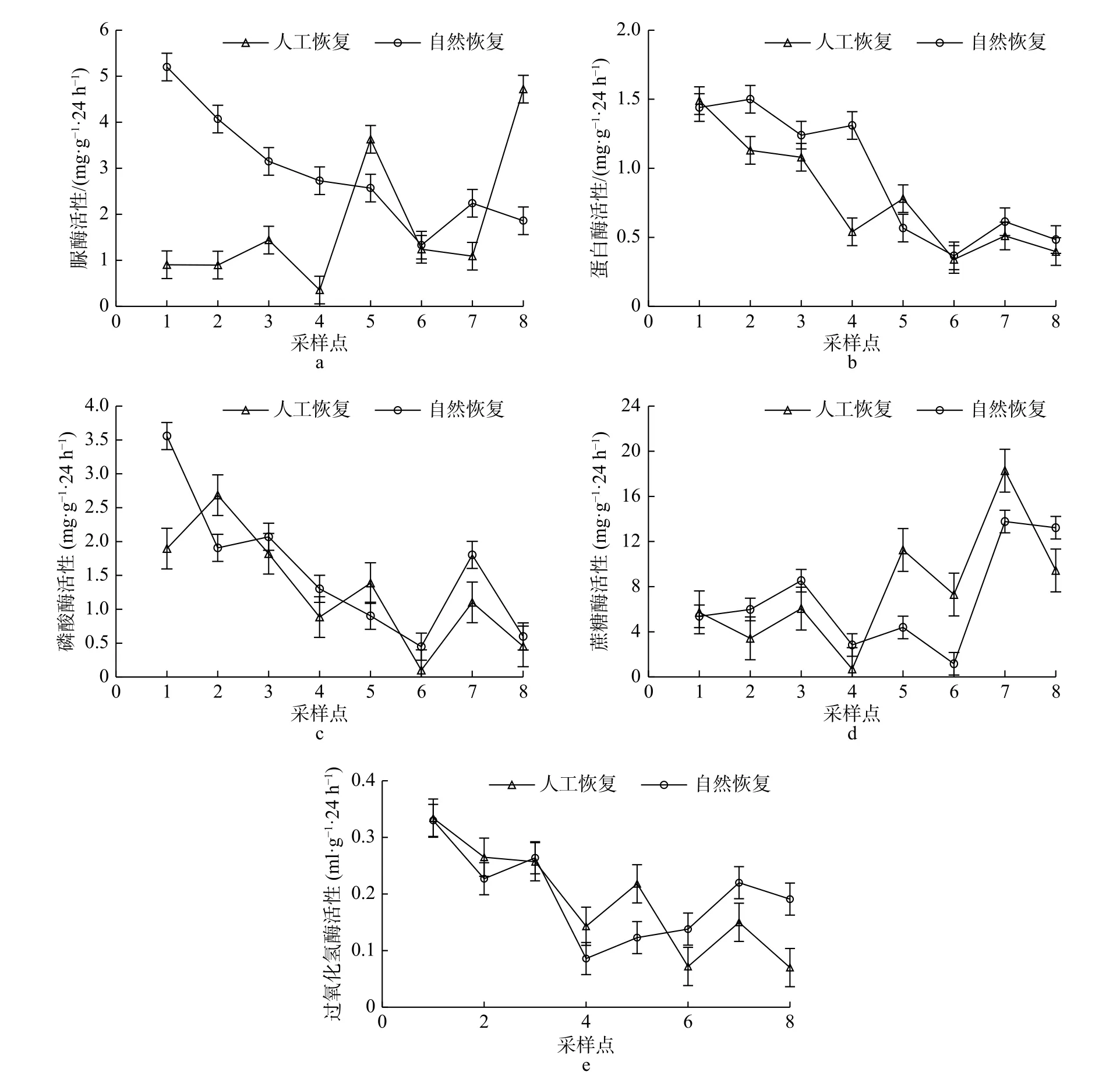
图 3 不同恢复方式下5 种土壤酶活性的变化特征Fig. 3 Variation characteristics of soil enzyme activities under different restoration modes
3.3 土壤酶活性与土壤肥力的冗余分析及排序
以土壤酶活性作为响应变量,土壤各肥力指标作为环境解释变量,并结合不同采样点,分别对不同恢复方式下土壤的酶活性进行冗余分析。结果表明:无论何种恢复方式下,RDA 排序图(见图4)显示土壤酶活性受不同土壤肥力指标的影响效果存在明显差异,对土壤酶活性的影响因子进行去趋势分析,同时采用Monte Carlo 检验来检测土壤肥力因子变量和土壤酶活性之间是否存在统计学意义上的显著相关性。
从图4 中各主要肥力因子的连线长度可以看出,在人工恢复方式下(见图4a),有机质的箭头连线最长,由此可知,土壤有机质与5 种土壤酶活性的相关性最大,并且与脲酶活性和蔗糖酶活性的相关性最为明显。总体来看,8 种土壤肥力因子分别在冗余分析图的第三、第四象限,说明这8 个环境因子与土壤酶活性存在负相关性较正相关性明显;两者连线夹角较小的有:蔗糖酶活性与有机质因子、蛋白酶活性与全钾因子、磷酸酶活性与铵态氮因子、过氧化氢酶活性与无机磷和铵态氮因子,说明以上酶活性受相对应的因子影响最大;在自然恢复方式下(见图4b),全钾、硝态氮因子与土壤酶活性的相关关系较小外,其余影响因子相关关系较大,全钾在RDA 排序图的第三象限,说明此环境因子与土壤酶活性存在负相关性,其余环境因子(有机碳、全氮、铵态氮、铵态氮、全磷、无机磷、有机质)都分布在第一和第二象限,与土壤酶活性存在正相关性。两者连线夹角较小的有:蛋白酶活性与有机质因子、过氧化氢酶活性与硝态氮因子、蔗糖酶活性与铵态氮因子、磷酸酶活性与有机碳因子、脲酶活性与有机质因子,说明以上酶活性受相对应的因子影响最大。

表 2 土壤酶活性与主要养分相关关系分析Tab. 2 Correlation coefficient between soil enzyme activity and main fertility factors under different restoration modes

图 4 不同恢复方式下土壤酶活性与土壤肥力指标的冗余分析排序图Fig. 4 Redundant analysis sequencing diagram of soil enzyme activity and soil fertility index under different restoration modes
从影响因子的RDA 排序结果来看(见表3):人工恢复方式下,第一、二、三排序轴分别能够解释所有信息的91.1%、6.6%、2.2%,累计解释信息量99.9%;自然恢复方式下,第一、二、三排序轴分别能够解释所有信息的88.6%、10.8%、0.4%,累计解释信息量99.8%;因此,无论何种恢复方式下,前三个排序轴已能够很好地解释肥力因子与土壤酶活性的关系,并且主要是由第一排序轴的解释量来决定。从表3 的RDA 分析结果显示,人工恢复方式下,在第一排序轴上土壤肥力因子与土壤酶活性的显著性排序为:有机质>全钾>全氮>铵态氮>硝态氮>无机磷>全磷>有机碳。从典范特征值分析结果显示,仅看第一排序轴的值可解释肥力因子与土壤酶活性关系91.1%的信息量,比总的解释信息量仅少8.9%,说明第一排序轴与肥力因子之间的线性结合能最大程度地反映肥力因子与酶活性的关系,并且排序的结果是可靠的。自然恢复方式下,在第一排序轴上土壤肥力因子与土壤酶活性的显著性排序为:铵态氮>无机磷>全氮>全磷>硝态氮>有机质>全钾>有机碳。从典范特征值分析结果显示,仅选择仅看第一排序轴的值可解释肥力因子与土壤酶活性关系的88.6%的信息量,比总的解释信息量仅少11.4%,说明第一排序轴与肥力因子之间的线性结合能最大程度地反映肥力因子与酶活性的关系,排序结果可靠。

表 3 不同恢复方式下土壤酶活性RDA 排序前三轴与主要肥力因子的相关系数Tab. 3 Correlation coefficient between the first three axes of soil enzyme activity RDA ranking and main fertility factors under different restoration modes
4 讨论
通过人工恢复对创面进行栅格固定及任其在自然恢复方式生长植被,而植被的逐渐恢复使得返回土壤的枯落物、根系残体及其根系分泌物逐渐增加,从而影响土壤肥力及土壤酶活性强度[13]。并且土壤酶在土壤养分循环以及植物生长所需养分的供给过程中扮演着重要角色,土壤酶活性的变化在一定程度上反映土壤和植被演替的规律,演替年限越长,土壤酶活性越高。本研究中除土壤过氧化氢酶活性受恢复方式的影响较小外,其余的脲酶活性、蔗糖酶活性、磷酸酶活性和蛋白酶活性受不同恢复方式的影响较显著,说明可能不同恢复方式下土壤肥力及其他生物理化性质对土壤酶活性产生了直接影响。并且相关研究表明土壤肥力在一定程度上受土壤酶活性影响[22],土壤肥力水平与酶活性之间相关关系显著[23]。本研究种不同恢复方式下土壤养分与酶活性有着不同程度的相关关系。进一步进行相关性分析及冗余分析则显示,土壤总有机碳、全氮、有机碳、氨氮、全钾等含量是与土壤酶活性相关性较强的因子。这是因为这5 种酶的功能都促进土壤碳氮磷养分转化,如蔗糖酶、过氧化氢酶是土壤有机质分解的重要驱动力[24],脲酶主要驱动土壤氮素转化,磷酸酶则对土壤碳氮磷转化都有促进作用[10,25]。本研究结果表明,无论在何种恢复方式下,土壤肥力的各个指标与土壤5 种酶活性之间都有显著的相关关系,土壤酶活性在一定程度上可指示土壤肥力的结果与杨瑞[11]等人研究结果[26]相似。总之,土壤养分元素和酶相互作用相互影响,调节着土壤创面的生态环境,促进植被生长,土壤发育。
5 结论
在自然恢复和人工恢复下,同种土壤酶活性变化有所不同。不同恢复方式下脲酶活性变化差异较大,最大差值为4.295 mg·g-1·24 h-1,在自然恢复方式下对于大部分采样点的脲酶活性值产生正反馈作用;蛋白酶活性的变化差异不大,均在1 mg·g-1·24 h-1以下,最大差值仅为0.77 mg·g-1·24 h-1,无论何种恢复方式下对土壤蛋白酶活性的影响不大。人工恢复下磷酸酶活性均小于自然恢复状态下的磷酸酶活性;蔗糖酶活性变化差异较大,最大差值为6.867 mg·g-1·24 h-1,最小差值为0.352 mg·g-1·24 h-1,在两种恢复方式作用下蔗糖酶活性没有明显的变化规律;相比前四种土壤酶活性,过氧化氢酶活性受不同恢复方式的影响较小,最大差值仅为0.121 ml·g-1·24 h-1,最小差值为0.004 ml·g-1·24 h-1。
无论在何种恢复方式下,表现土壤肥力的各个指标与土壤这5 种酶活性之间在0.05 级别和0.01 级别(双尾)都存在相关性显著,有的存在负相关性,有的存在正相关性。
从RDA 排序图来看,在人工恢复方式下,8 种土壤肥力因子与土壤酶活性存在负相关性较正相关性明显;在自然恢复方式下,全钾、硝态氮因子与土壤酶活性的相关关系较小外,其余影响因子相关关系较大,全钾环境因子与土壤酶活性存在负相关性,其余环境因子(有机碳、全氮、铵态氮、铵态氮、全磷、无机磷、有机质)与土壤酶活性存在正相关性。
从影响因子的RDA 排序结果来看:人工恢复方式下,土壤肥力因子与土壤酶活性的显著性排序为:有机质>全钾>全氮>铵态氮>硝态氮>无机磷>全磷>有机碳;自然恢复方式下,显著性排序为:铵态氮>无机磷>全氮>全磷>硝态氮>有机质>全钾>有机碳,第一排序轴分别能够解释人工恢复与自然恢复所有信息的91.1%、88.6%。
