MALDI-TOF MS对新生隐球菌和格特隐球菌的快速鉴定与分析
2020-07-28吴思颖肖玉玲
吴思颖,康 梅,刘 雅,肖玉玲,何 超,谢 轶,马 莹
(四川大学华西医院实验医学科,四川成都 610041)
隐球菌脑膜炎是由隐球菌引起的中枢神经系统感染,主要累及免疫功能受损人群,如人类免疫缺陷病毒(HIV)感染者和器官移植患者,也可引起免疫功能正常者的感染。中枢神经系统隐球菌病预后较差,病死率高,即使经过标准的抗真菌治疗,病死率仍可高达60%[1]。因此早期诊断和治疗尤为重要。常见的致病隐球菌主要包括新生隐球菌和格特隐球菌,主要包括8种基因型:新生隐球菌基因型VNⅠ~Ⅳ,格特隐球菌基因型VGⅠ~Ⅳ[2]。隐球菌的传统鉴定方法主要依靠生化试验,但是该方法耗时长,缺乏及时性。分子生物学鉴定和分型方法成本较高且操作复杂,不适用于实验室常规检测。基质辅助激光解析电离飞行时间质谱(MALDI-TOF MS)是分析非挥发性生物大分子的一种新型软电离质谱技术,可用于病原微生物的鉴定[3]。由于其具有快速、灵敏、准确、高通量及自动化等特点,MALDI-TOF MS已成为微生物鉴定领域非常重要的技术工具和手段。本研究应用MALDI-TOF MS对常见的致病隐球菌进行快速鉴定并进一步对隐球菌不同基因型进行聚类分析,现报道如下。
1 资料与方法
1.1一般资料 收集2017年6月至2019年11月本院各科室分离的44株隐球菌临床株。
1.2仪器与试剂 MALDI-TOF MS(Bruker Daltonik GmbH,德国),API 20C(bioMérieux,法国),PCR扩增仪(ABI,美国),限制性内切酶Sau96Ⅰ、HhaⅠ(New England Biolabs,美国),Gel Doc XR 凝胶成像系统(BIO-RAD,美国)。沙氏琼脂平板(郑州安图),α-氰基-4-羟基肉桂酸(HCCA,Bruker Daltonik GmbH,德国),无水乙醇(成都科龙),三氟乙酸(MERCK公司,德国),乙腈(Tedia公司,美国),甲酸(Anaqua Chemicals Supply,美国)。
1.3方法
1.3.1隐球菌基因分型 利用URA5基因PCR产物限制性片段长度多态性(URA5-RFLP)方法对44株隐球菌临床株进行基因分型。PCR扩增引物:URA5(5′-ATG TCC TCC CAA GCC CTC GAC TCC G-3′),SJ01(5′-TTA AGA CCT CTG AAC ACC GTA CTC-3′)。PCR扩增条件:94 ℃预变性2 min,94 ℃变性45 s,61 ℃退火1 min,72 ℃延伸2 min,35个循环,最后72 ℃延伸10 min。使用Sau96Ⅰ(10 U/μL)和HhaⅠ(20 U/μL)这两种限制性内切酶对上述PCR产物进行双酶切。将酶切体系于37 ℃水浴3 h,得到酶切产物。将临床株和标准菌株的酶切产物10 μL于2%的琼脂糖凝胶中100 V电泳2.5 h, 以DL2000作为分子量标记物,在Gel Doc XR凝胶成像系统中观察电泳结果,比较隐球菌临床株与8株不同基因型标准菌株的酶切谱型,得出隐球菌临床株的基因型。隐球菌基因分型标准菌株如下:WM148(VNⅠ),WM626(VNⅡ),WM628(VNⅢ),WM629(VNⅣ),WM179(VGⅠ),WM178(VGⅡ),WM161(VGⅢ)和WM779(VGⅣ)。
1.3.2MALDI-TOF MS鉴定 MALDI-TOF MS系统采用线性正性模式,采集相对分子质量为(2~20)×103的图谱。采用甲酸提取法,取待测菌株,加入300 μL水、900 μL乙醇,混匀,加入30 μL 70%甲酸溶液,混匀后再加入30 μL乙腈混匀,离心取1 μL上清液在MALDI靶板上点样,晾干后添加1 μL HCCA,晾干。每个样本置于靶板上的6个不同位点,即每个样本生成6个合成的图谱(MSP)。将待鉴定隐球菌的质谱数据与数据库中参考菌株的数据进行比对,根据匹配程度得到相应分值,最终得到鉴定结果。得分≥2.0表示鉴定到种水平,1.7~<2.0表示鉴定到属水平,<1.7表示不可信的鉴定结果。
1.3.3聚类分析 使用MALDI Biotype软件对44株隐球菌临床株和8株基因分型标准菌株进行聚类分析,构建MSP聚类分析树状图。
2 结 果
2.1隐球菌基因分型结果 44株隐球菌临床株URA5-RFLP分型结果显示,42株为新生隐球菌VNⅠ型,1株为格特隐球菌VGⅠ型,1株为格特隐球菌VGⅡ型。隐球菌基因分型结果见图1。
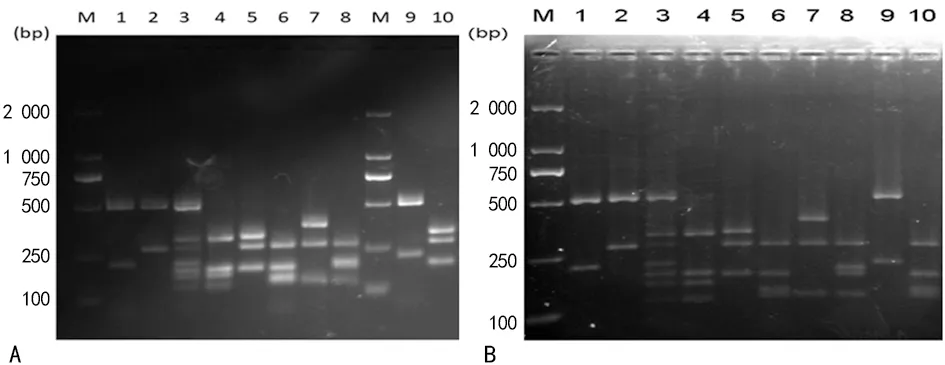
注:A和B为隐球菌基因分型标准菌株和部分临床株酶切产物电泳图;图A中10为格特隐球菌VGⅠ型(临床株),9为新生隐球菌VNⅠ型(临床株);图B中10为格特隐球菌VGⅡ型(临床株),9为新生隐球菌VNⅠ型(临床株);M为标记物;1~4分别为新生隐球菌基因型标准菌株WM148(VNⅠ)、WM626(VNⅡ)、WM628(VNⅢ)、WM629(VNⅣ);5~8分别为格特隐球菌基因型标准菌株WM179(VGⅠ)、WM178(VGⅡ)、WM161(VGⅢ)、WM779(VGⅣ)。图1 隐球菌基因分型标准菌株和部分临床株URA5-RFLP分型结果
2.2MALDI-TOF MS鉴定结果 MALDI-TOF MS对隐球菌标准菌株和临床株的鉴定结果与分子生物学鉴定结果完全一致。42株隐球菌临床株鉴定为新生隐球菌,另外2株鉴定为格特隐球菌,鉴定结果得分为1.975~2.346分。标准菌株的鉴定得分如下,WM148:2.109分;WM626:2.059分;WM628:2.134分;WM629:2.045分;WM179:2.041分;WM178:2.210分;WM161:2.051分;WM779:1.969。
2.3新生隐球菌和格特隐球菌以及不同基因型之间质谱图的特点 新生隐球菌和格特隐球菌之间,以及新生隐球菌和格特隐球菌不同基因型之间,特征蛋白的质量和峰的信号强度存在差异。新生隐球菌VNⅡ型标准菌株WM626在8 000~1 000 m/z有2个明显的质谱峰,而新生隐球菌其他基因型却不明显。格特隐球菌VGⅠ型标准菌株WM179在5 000~6 000 m/z有1个明显的质谱峰。所有新生隐球菌临床株的质谱图与VNⅠ型标准菌株WM148更接近。格特隐球菌VGⅠ型临床株(F14)的质谱图与VGⅠ型标准菌株WM179更相似。格特隐球菌VGⅡ型临床株(F44)的质谱图与VGⅡ型标准菌株WM178更接近。
2.4聚类分析 对所有隐球菌临床株和基因分型标准菌株进行聚类分析,得到MSP聚类分析树状图。从图中可以看到,新生隐球菌和格特隐球菌分别位于两个不同的集群,且新生隐球菌和格特隐球菌不同基因型被区分开。绝大多数新生隐球菌VNⅠ型临床株与VNⅠ型标准菌株WM148位于同一集群,格特隐球菌VGⅠ型和VGⅡ型临床株分别与VGⅠ型标准菌株WM179和VGⅡ型标准菌株WM178位于同一集群。见图2。
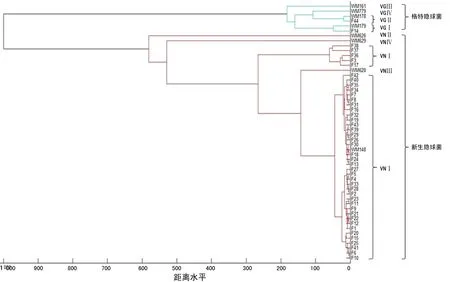
图2 44株隐球菌临床株和8株基因分型标准菌株MSP聚类分析树状图
3 讨 论
临床实验室中隐球菌的传统鉴定方法主要依靠生化试验和形态学检查,但均不能区分新生隐球菌和格特隐球菌,而两者所致感染在临床表现、体外药敏试验和治疗预后等方面均存在明显差异[4]。分子生物学分型方法如限制性片段长度多态性(RFLP)、扩增片段长度多态性(AFLP)和多位点或全基因组序列分析等,由于价格昂贵,操作较烦琐,往往局限在参考实验室,目前未在临床常规使用[5]。MALDI-TOF MS具有经济、操作简单的特点,可快速为临床提供检测结果,使患者得到即时的治疗。本研究应用MALDI-TOF MS对隐球菌临床株和标准菌株进行快速鉴定,结果显示44株隐球菌临床株和8株基因分型标准菌株的鉴定结果与分子生物学鉴定结果一致,可准确区分新生隐球菌和格特隐球菌。刘涛华等[6]对20株隐球菌的研究发现,MALDI-TOF MS可作为隐球菌属分类鉴别中有效的辅助手段,相比于多位点序列分析技术,MALDI-TOF MS更为快捷。ZHANG等[7]研究发现,MALDI-TOF MS对新生隐球菌的鉴定具有100%的准确性,但唯一1株格特隐球菌被错误鉴定为新生隐球菌。由于国内的隐球菌感染主要以新生隐球菌为主,格特隐球菌属于散发病例,因此还需收集更多的格特隐球菌以评价MALDI-TOF MS对格特隐球菌的鉴定能力。黄声雷等[8]对两种MALDI-TOF MS系统进行了评价,对于常见的非白色念珠菌,Autoflex MALDI-TOF MS系统鉴定准确性优于Vitek MALDI-TOF MS系统。对于隐球菌的鉴定,Autoflex MALDI-TOF MS系统是否优于Vitek MALDI-TOF MS系统,尚需进一步研究。
MALDI-TOF MS不仅能用于病原微生物的鉴定,而且还具有分型能力,可进一步应用于病原微生物的分型。例如MALDI-TOF MS应用于弗劳地枸橼酸杆菌和黏质沙雷菌的分型,其结果与全基因组序列分析具有较好的一致性[9]。基于MALDI-TOF MS的聚类分析可对哈维弧菌进行溯源性分析[10]。MALDI-TOF MS也可用于真菌的分型。CHAO等[11]利用MALDI-TOF MS聚类分析可区分皱褶假丝酵母菌复合体中的发酵假丝酵母菌和季也蒙假丝酵母菌。FIRACATIVE等[12]研究发现,新生隐球菌和格特隐球菌不同基因型之间的质谱峰图存在差异,聚类分析发现新生隐球菌与格特隐球菌不同基因型分别位于不同的集群,相同基因型则位于同一集群。POSTERARO等[13]运用MALDI-TOF MS对82株隐球菌(72株新生隐球菌、10株格特隐球菌)进行聚类分析,其中81株(98.8%)被准确归于相应的基因型,仅1株VGⅡ型格特隐球菌被错误地归于VGⅠ集群。SIQUEIRA等[14]纳入隐球菌不同基因型建立数据库,进一步评估该数据库用于区分新生隐球菌和格特隐球菌不同基因型的能力,结果显示该数据库可对所有新生隐球菌和格特隐球菌基因型进行正确鉴定。本研究中隐球菌临床株和不同基因型的标准菌株聚类分析发现,绝大多数VNⅠ型临床株与VNⅠ型标准菌株位于同一集群,仅少数VNⅠ型临床株位于另一集群。推测基于MALDI-TOF MS的聚类分析可能会受到样品蛋白制备以及菌株蛋白表达量的影响。在进行分型或亚种水平鉴定时,需要识别更多的具有重现性的质谱峰,需要设置更多的平行组。此外,对未知病原菌的分型鉴定必须要有足够多的已知菌株的数据库,方可实现最佳匹配,提高其鉴定分型能力[15]。本研究中44株经过分子生物学确认基因型的隐球菌已经全部纳入数据库中。由于本研究中所有新生隐球菌临床株均为VNⅠ型,且只有2株格特隐球菌,应该纳入更多隐球菌基因型以评价MALDI-TOF MS对隐球菌的分型能力。
4 结 论
综上所述,MALDI-TOF MS作为一种快速的鉴定方法可以准确区分新生隐球菌和格特隐球菌,且可作为一种潜在的分型方法对新生隐球菌和格特隐球菌不同基因型进行区分。
