基于简化基因组测序的中国明对虾3个选育世代遗传多样性分析*
2020-07-26王凤娇孟宪红
王凤娇 孟宪红 傅 强 栾 生 隋 娟
基于简化基因组测序的中国明对虾3个选育世代遗传多样性分析*
王凤娇1,2,3孟宪红1,2①傅 强1,2栾 生1,2隋 娟1,2
(1. 中国水产科学研究院黄海水产研究所 农业农村部海洋渔业可持续发展重点实验室 青岛 266071;2. 青岛海洋科学与技术试点国家实验室海洋渔业科学与食物产出过程功能实验室 青岛 266071;3. 上海海洋大学水产与生命学院 上海 201306)
本研究基于简化基因组测序(2b-RAD)技术,对中国明对虾() “黄海2号” 2015~2017年3个连续选育世代(G9~G11)的亲本群体、共649个个体进行了简化基因组测序,并对这3个亲本群体进行了遗传结构和遗传多样性分析。实验在3个选育世代中共获得66985个SNP位点。遗传分析的结果显示,G9~G11平均核苷酸多样性(P)分别为0.1439、0.1587和0.1674,平均观测杂合度(H)分别为0.1388、0.1515和0.1609,多态信息含量(PIC)分别为0.1241、0.1360和0.1430。G9~G11亲本群体的遗传多样性整体呈现一定的上升趋势,但差异不显著。检验显示, 3个世代总的st值为0.0061,G9~G11相邻世代群体间遗传分化程度较弱(G9~G10为0.0029, G10~G11为0.0026),表明相邻世代的遗传距离逐渐减小。3个世代间基因交流充分,基因流为62.91~94.63。本研究表明,人工定向选育工作的推进对中国明对虾选育群体遗传多样性和遗传结构产生了一定的影响:在固定的选择压力下(4%~5%),亲本群体的遗传多样性并无降低的趋势,中国明对虾选育群体遗传分化小,遗传结构趋向稳定。研究结果为进一步制定中国明对虾选育计划提供基础遗传数据和科学的理论指导。
中国明对虾;选育群体;2b-RAD;SNP;遗传多样性
中国明对虾()是中国近海特有种,主要分布在黄海、渤海海区的山东、河北、辽宁、天津及江苏近海和朝鲜半岛西海岸(王清印, 2014)。其适应能力强、生长快、耐低温、品质好、营养价值高,是我国重要的海水养殖经济物种之一。20世纪80年代,中国明对虾养殖技术得到突破,中国明对虾的养殖产量迅速增加,1991年总产量达20万t,成为当时我国最重要的对虾养殖品种(李健等, 2015)。1993年开始暴发的白斑综合症病毒(White spot syndrome virus, WSSV)对中国明对虾产业造成了沉重的打击。随之而来的养殖环境恶化,种质资源衰退等问题,均严重影响了整个养殖业的发展。为了保护中国明对虾的种质资源,开展优良群体的选育工作,培育高产、抗逆的中国明对虾新品种已成为必然。
利用传统数量遗传学方法在对虾选择育种中进行遗传参数评估和遗传解析已经取得了很大的进展(栾生等, 2013; 何玉英等, 2011)。随着现代生物技术的发展,相继涌现出RFLP、AFLP、SSR和SNP等多种分子标记技术,这些分子标记技术为群体的遗传结构评估提供新的途径。SNP标记作为遗传学研究中最为流行的分子标记,与传统分子标记相比,具有在基因组中分布广泛、密度高、稳定遗传等优势,是目前水产动物高密度遗传图谱构建、群体遗传学研究和种质资源保护利用等领域的理想标记(徐安毕等, 2007; Li, 2003; 黄映萍, 2010; 刘安然等, 2019)。然而,水产物种的基因组研究背景相对滞后,基因组的杂合度和高复杂性也限制了高通量测序的应用。近年来,简化基因组测序的发展为水产物种提供了新的手段。2b-RAD (Wang, 2012)技术是近年来在非模式生物中进行SNP开发最为有效、经济的简化基因组测序方法之一。该技术由于具有酶切DNA片段大小一致、文库构建简单快速、标签密度易于调节、成本低廉、准确性高等优点得到广泛的应用。
本研究的实验对象中国明对虾“黄海2号”是采用群体、家系与多性状复合育种技术,经连续选育 13代获得的新品种。该品种生长速度快、具有明显的抗病性(李旭鹏等, 2018)。根据多性状复合育种技术制定的配种方案,该群体获得了可观的遗传进展(Sui, 2018)。但累代人工选育是否会降低该选育群体的遗传多样性、影响中国明对虾的选育效果,尚缺少实际检测数据的支撑。目前,虽然已有学者通过RAPD分析(石拓等, 2001)、线粒体DNA序列分析(张辉等, 2010)、AFLP分析(安丽等, 2008)和SSR分析(张天时等, 2005)对中国明对虾养殖群体进行了遗传多样性研究,吴莹莹等(2013)利用非探针HRM开发的39个SNP,分析了中国明对虾群体的遗传多样性。但这些研究的标记数目较低,且尚缺少中国明对虾人工选育群体的分析报道。本研究利用2b-RAD简化基因组技术,对中国明对虾“黄海2号” G9~G113个连续选育世代的亲本群体进行高通量SNP开发,并基于这些标记分析了遗传多样性参数、群体间遗传分化和遗传距离,从分子水平揭示不同世代的群体遗传结构,了解人工选育方案对其遗传结构和遗传多样性的影响,从而为选育工作提供可靠的理论基础,确保选育工作顺利进行。
1 材料与方法
1.1 实验材料
实验所用中国明对虾“黄海2号”取自中国水产科学研究院黄海水产研究所水产遗传育种中心。2015~ 2017年(G9~G11)该育种群体按照多性状复合育种的配种方案进行家系构建、选育。选取在传代过程中用于构建核心家系的亲本,置于–80℃超低温冰柜内保存备用。该亲本群体包括2015年137尾、2016年244尾和2017年268尾,共计649尾。
1.2 实验方法
1.2.1 DNA提取和检测 采用TIANamp Marine Animals DNA Kit (天根生化科技有限公司,北京)提取中国明对虾肌肉组织基因组DNA,通过琼脂糖检测和NanoDrop 2000紫外分光光度计(Thermo Fisher Scientific)对抽提的DNA质量和浓度进行检测。DNA产物置于–20℃保存备用。
1.2.2 2b-RAD文库构建和测序质量分析 利用2b-RAD标签串联技术进行文库构建,文库的构建过程参考Wang等(2012)。选用的IIB型限制性内切酶为XI,为了得到尽可能多的标记,所有样品均采用标准型5′-NNN-3′接头建库。文库质控合格后在Illumina Hiseq X Ten平台进行Paired-end测序(由 青岛欧易生物科技有限公司完成)。原始数据下机后,按照以下条件进行过滤:剔除不含有XI酶切识别位点的序列;剔除低质量序列(即大于10个碱基的质量分数小于20的序列);剔除有10个以上连续相同碱基的序列。由此获得可用于后续标记筛查、分型的高质量序列。
1.2.3 全基因组SNP筛查和分型 由于中国明对虾尚未有发表的全基因组序列作为参考,本研究借用课题组前期构建的中国明对虾高密度遗传连锁图谱数据(标记密度为0.41 cM, 尚未发表),利用该作图群体2个亲本构建的已有Reference (命名为Family- Reference,该参考标签库可靠性已在图谱构建中得到验证)进行后续的标记筛查、分型。该Reference是在核心群体中选取2个性状差异大的雌雄代表性个体进行测序标签(Tag)聚类,构建的RAD分型参考标签库。全基因组标记分型参照RAD-typing分型策略(Fu, 2013)。分型过程是将本研究所有测序个体(作图群体的2个亲本)的高质量reads,利用SOAP软件(Li, 2009) (参数设置为:–M4–v2–r0)比对到该Reference上,筛查标记位点及分型信息。对于共显性的SNP标记,利用最大似然法ML得到每个个体在每个位点的基因型。为了保证SNP位点分型的准确性和严谨性,进行以下条件的过滤:在个体内标签深度500以上的不进行分型。只留取标签内最多有 3个SNP的标签位点;标记在80%的个体中可以分型;最小等位基因频率MAF≥0.01。
1.3 数据统计与分析
利用Genepop软件(Version 1.0.5) (Rousset, 2008)对SNP位点的分型结果进行处理,计算其有效等位基因数(N)、期望杂合度(H)、观测杂合度(H)、多态信息含量(PIC)、最小等位基因频率(MAF)和等位基因频率等遗传参数,并计算世代间的遗传分化系数(st)、近交系数(is)、群体间的遗传距离(DR)和基因流(N)。利用vcftools软件(Version 0.1.14) (Danecek, 2011)计算核苷酸多样性(P)和转换/颠换(s/v)的比值。使用Admixture软件(Version 1.3.0) (Alexander, 2009)分析中国明对虾的群体结构。
2 结果
2.1 基因组DNA的提取及文库的构建
提取的基因组DNA用1%的琼脂糖凝胶电泳检测,图1为部分基因组DNA的琼脂糖凝胶电泳检测结果,提取的基因组DNA无明显拖带现象,完整性较好。用NanoDrop 2000紫外分光光度计检测显示,OD260 nm/OD280 nm在1.8~2.0之间,表明提取的基因组DNA质量较高,可用于后续的建库和测序。

图1 基因组DNA琼脂糖凝胶电泳结果
DNA质检合格后,为了保证每个文库的质量,对构建的每个文库进行聚丙烯酰胺凝胶电泳检测,可直观的显示文库构建是否成功。图2为聚丙烯酰胺凝胶电泳检测文库质量。
2.2 2b-RAD测序及SNP分型结果
本研究基于2b-RAD测序技术构建中国明对虾649个样品的标签测序文库,测序获得1217161378条原始Reads,平均每个样本测序Reads数为28938658。质量控制后,平均每个样本中获得含XI酶切位点的高质量Reads占测序原始Reads的74.86%。引用的高质量参考(Reference)共包含286345条共显性标签序列。在对相同的Reads聚类得到特异性标签后,平均每个样本获得特异性标签数目为343262,样本的平均测序深度为43.08×。3个选育世代获得66985个高质量的SNP位点,用于后续群体遗传分析。
2.3 中国明对虾SNP突变类型分析
统计SNP分型结果中各位点的突变类型特点。如图2所示,C/T转换类型最多,占所有碱基突变类型的39%,A/G转换类型占14%。G/C颠换类型最少,占所有碱基突变类型的6%,A/C与A/T颠换类型均占14%,G/T颠换类型占8%。转换与颠换(s/v)之比为1.402。

图2 目的片段PCR产物结果
2.4 不同世代的SNP遗传多样性分析
由于本次分型只统计了二等位基因型,因此,每个SNP位点观测等位基因数目(N)均为2。位点的平均分型率为92.47%。结果显示(表1),G9~G11世代有效等位基因(N)平均分别为1.2069、1.2324和1.2475;平均期望杂合度(H)分别为0.1434、0.1584和0.1671;测得平均观测杂合度(H)分别为0.1388、0.1515和0.1609;核苷酸多样性(P)分别为0.1439、0.1587和0.1674。G9~G11选育世代多态信息含量(PIC)的均值分别为0.1241、0.1360和0.1430;最小等位基因频率(MAF)分别为0.1074、0.1038和0.1146。近交系数分别为0.1141、0.0701和0.0532。
表1 不同世代SNP位点的遗传多态性参数分析*
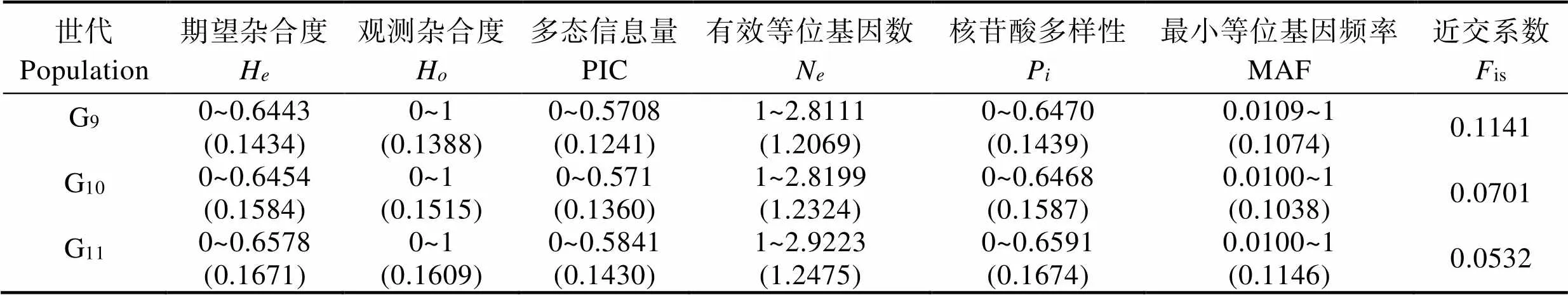
Tab.1 Analysis of genetic polymorphism parameters of SNP loci in different generations
*:括号中的数据为平均值
*: Data in brackets are means
2.5 世代间等位基因频率变化
G9~G11所有位点的平均等位基因频率分别为86.92%、86.31%和85.87%。3个世代选育群体野生型等位基因频率整体呈现一定的下滑趋势,但差异并不显著。从每个位点统计3代等位基因的变化规律,其中,逐代下降的SNP数量为20348,占30.38%;逐代上升的SNP数量为8558,占12.78% (表2),逐代下降的位点数是逐代上升位点的2.4倍。从逐代下降的位点中,筛选出50个变化程度高的位点,变化程度在24.43%~29.76%之间,平均值为27.56%,从逐代上升的位点中,筛选了50个变化程度高的位点,变化程度在27.16%~30.45%之间,平均值为28.79%,二者之间也无显著性差异(=0.552),图3和图4表示选取的50个具有代表性的等位基因频率变化情况。
表2 G9~G11世代SNP位点的基因频率变化规律

Tab.2 Gene frequency variation of SNP loci in G9~G11 generations
2.6 世代间遗传变异及分化
群体间总的遗传分化系数为0.0061。随着选育世代的增加,遗传分化系数在相邻世代之间相继减小,G9与G10间st值为0.0029,G10与G11间st值为0.0026,两两比较所得st值均小于0.05,3个选育世代群体间遗传分化较弱。从图6可看出,3个亚群的波形一致性很高,这再次说明3个选育世代遗传分化很弱,遗传结构基本稳定。选育世代间的遗传距离(DR)范围为0.0026~0.0040,G9与G10间的遗传距离为0.0029,G10与G11间的遗传距离为0.0026,相邻世代的遗传距离也逐步减小。G9与G10间的基因流(N)为86.83,G10与G11间的基因流(N)为94.63。
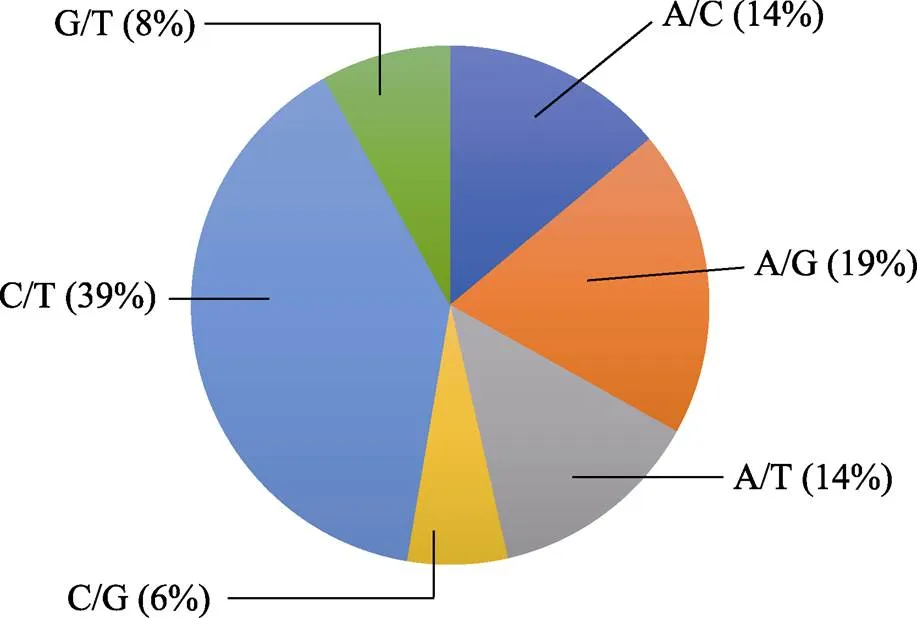
图3 SNP位点碱基突变类型及其比例
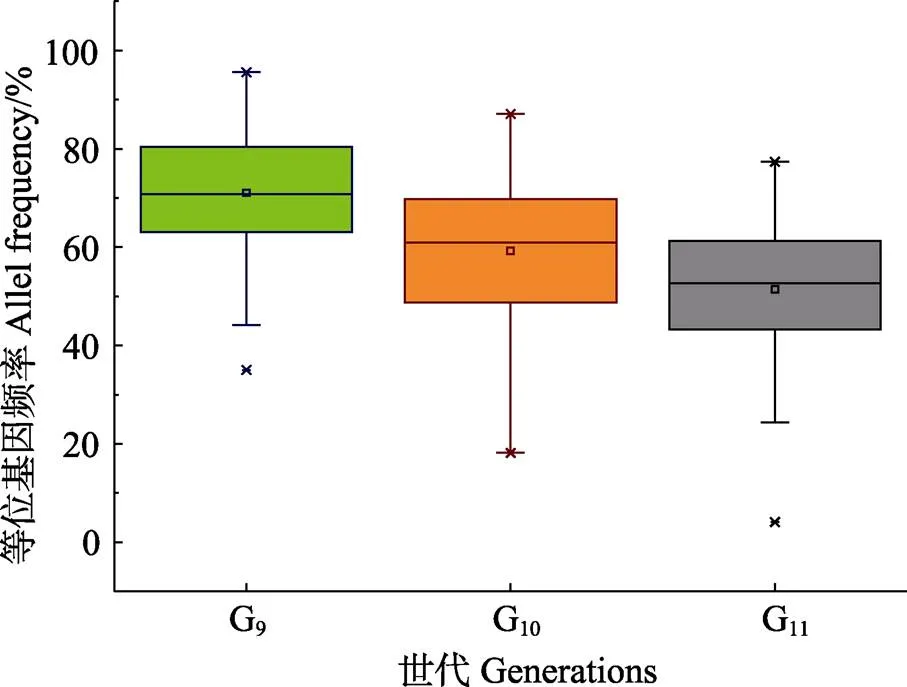
图4 G9~G11 50个逐代下降SNP位点的基因频率变化

图5 G9~G11 50个逐代上升SNP位点的基因频率变化

图6 中国明对虾样品聚类结果
表3 中国明对虾G9~G11世代遗传分化系数(st)和遗传距离(DR)
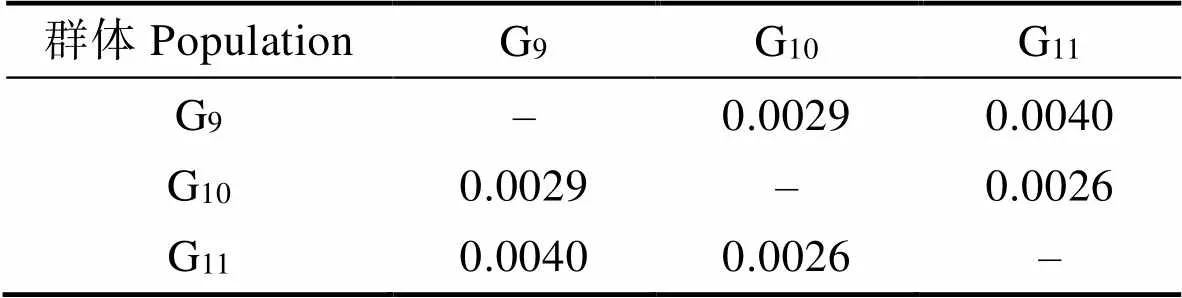
Tab.3 Genetic differentiation coefficient (Fst) and genetic distance (DR) in G9~G11F. chinensis
注:下三角为群体间遗传分化系数(st),上三角为群体间遗传距离群体(DR)
Note: The lower triangle is the genetic differentiation coefficient (st) between groups, and the upper triangle is the genetic distance group between groups (DR)
3 讨论
3.1 高通量测序及SNP突变类型分析
由于中国明对虾没有已发表的参考基因组序列,依据RAD-typing的分型策略(Fu, 2013),需要从测序数据中提取含有酶切识别位点的Reads,使用ustacks(version 1.34)软件进行聚类,从头构建一个供分型使用的参考标签库(Reference)。本研究在选择参考标签库的过程中,比较了使用2种参考序列(Reference)对开发SNP标记的影响,即分别利用中国明对虾高密度连锁图谱作图群体2个亲本构建的family-Reference以及从选育群体中随机选取4个测序良好的个体构建的progeny-Reference,使用相同的分型标准对3代选育群体进行分型。对于2种Reference分型得到的标记进行比对,在允许2个错配的条件下,共比对到60176个标记,即标记共有率为92.45%,2种Reference的基因组覆盖度和代表性均良好。分型后,分别得到66304和66985个SNP,2次分型得到的标记数均无显著差异。考虑到遗传图谱的前期工作基础,最终确定了family-Reference作为参考序列(待发表数据)。经过2b-RAD测序后,每个样本得到的原始Reads从29119715~89664272不等。质量过滤后,平均每个样本中获得含有XI酶切位点的高质量Reads占测序原始Reads的74.86%,平均每个个体的测序深度为43.08×。说明了本次构建的文库质量较高,测序深度达到了高准确度下的分型标准(Jiao, 2014)。
本研究中选取的每个世代的测序样本数都不一致(G9选取137尾、G10选取244尾、G11选取268尾),是由于当年需要构建的家系数目不同。理论上,不同的样本数会造成统计上严谨度的不同,另外,个体数量少的G9世代,也有137个样本提供分型,因此认为,虽然样品数量不同但并不会对实验结果造成影响。不同世代样本数不同,在遗传多样性分析中也有报道,如Zhang等(2018)曾经利用类似的方法对3个世代连续选择的栉孔扇贝()进行分析,从G1世代抽取76只,G2世代抽取107只,G3世代356只,2b-RAD分型得到的18450个标记进行群体遗传学分析,也发现样本量不同对结果几乎无影响。
SNP碱基替换类型分为转换(s)与颠换(v)两类,转换发生在A与G或C与T之间,颠换有4种,发生在A与C、A与T、G与C和G与T之间。理论上发生转换的概率与发生颠换概率(s/v)比值为0.5,但实际上s/v值往往大于0.5,这种差异性被称为“转换性偏差”(Gojobori, 1982; Li, 1984; Collins, 1994)。本研究验证的SNP多态性类型存在明显的转换型偏差现象,s/v=1.402>0.5。不同物种间转换/颠换比值存在很大的差距。马氏珠母贝()血细胞转录组中,转换与颠换的比值为0.5,与理论比率相符(王忠良等, 2018)。凡纳滨对虾()转录组中,转换和颠换的比率为2.0(于洋, 2014),而鲍中碱基的转换大大多于碱基的颠换,转换与颠换比为2.33 (王鹭骁等, 2006)。产生这种差异的原因可能与不同物种在进化中承受的选择压力有关(Zhao, 2002)。
3.2 群体遗传多样性
人工定向选育是一个复杂的过程,累代的人工选择压力及选择环境势必会造成群体遗传水平的波动,而遗传多样性是生物多样性的基础,对物种的生存、繁衍和进化至关重要(张荣良等, 2016)。人工选择育种的目的是在保持选育群体具有一定遗传多样性的基础上,筛选获得具有目标性状的选育新品种或新品系(张荣良等, 2016)。因此,在水产良种培育过程中维持群体的遗传多样性,是种质资源利用的前提条件。本研究中的3个选育世代SNP标记从整体水平上,野生型的平均等位基因频率呈现一定的下降趋势,但并不显著,分别为86.92%、86.31%和85.87%。在人工选育的背景下,本研究显示群体遗传多样性没有明显变化,推测原因为人工选择只定向改变少数位点的基因频率。本研究进一步从每个SNP位点角度统计等位基因变化规律,并从其中选取50个变化程度大的连续下降和连续上升的位点(图4和图5),二者等位基因频率变化程度分别为27.56%和28.79%,无显著性差异(=0.552)。这些位点的变化是否与性状存在遗传上的相关性还需进一步验证。
多态信息含量(PIC)和杂合度是2个较好的衡量基因多态性的指标。当PIC<0.25时,为低度多态位点;0.25
本研究发现,3个世代亲本的遗传参数有上升的趋势(但不显著)。分析原因可能是与本研究所选取实验样品有关。本研究所用的样品并不是在选育群体中随机抽选的个体,而是用于构建G9~G11代核心家系的亲本作为样品,即这些个体是依据育种方案挑选出的性状优良个体,代表的是下一代群体的遗传多样性。选育世代亲本遗传多样性高低,是下一代遗传多样性维持的重要基础,而本研究亲本的遗传多样性没有下降,进一步说明我们的育种方案是科学的,在保证遗传进展的同时,遗传多样性不会降低。
3.3 世代间的遗传变化
种群的遗传分化指数(st)是衡量群体间遗传分化程度的重要参数,根据Wright (1978)对遗传分化指数的界定,st值介于0~0.05之间,表示群体遗传分化较弱。在本研究中,群体总遗传分化指数为0.0061以及世代间st值均小于0.05 (表2),说明3年选育世代遗传分化程度较弱,群体间的遗传结构差异不显著。随着选育世代的增加,相邻世代间遗传距离呈减小趋势,说明随着选育时间的推进,选育群体的遗传背景趋于一致,遗传结构趋向稳定,也体现了人工定向选择的效应。基因流强弱对种群遗传分化也具有重要影响,基因流N<1,表明由于遗传漂变发生了分化;N>1,表明遗传分化较小;N>4,表明群体间的基因交流更充分,遗传分化更小。3个选育世代亲本的基因流值为62.91~94.63,3个选育世代间基因交流非常充分,群体间的遗传分化很小。由于中国明对虾是一年生物种,遗传变异程度低,对固定性状的人工选择并没有在整个基因组上产生明显的影响,因此遗传分化程度低。
本研究采用2b-RAD技术对连续选育的中国明对虾“黄海2号”G9~G113个世代共计649个亲本进行了简化基因组测序,共获得66985个SNP用于群体遗传多样性和遗传结构变化的评估,研究表明,随着人工定向选育工作的推进,对中国明对虾选育群体遗传多样性和遗传结构产生了一定的影响,但中国明对虾选育群体遗传分化相当小,遗传结构趋向稳定,亲本的遗传多样性并无降低的趋势。研究结果为下一步制定中国明对虾选育计划提供基础数据和参考。
Alexander DH, Novembre J, Lange K. Fast model-based estimation of ancestry in unrelated individuals. Genome Research, 2009, 19(9): 1655–1664
An L, Liu P, Li J,AFLP analysis on selected populations of shrimpnamed "Yellow Sea 1"Periodical of Ocean University of China (Natural Science), 2008, 38(6): 921–926 [安丽, 刘萍, 李健, 等. “黄海1号”中国对虾不同世代间的AFLP分析. 中国海洋大学学报(自然科学版), 2008, 38(6): 921–926]
Botstein D, White RL, Skolnick M,. Construction of a genetic linkage map in man using restriction fragment length polymorphisms. American Journal of Human Genetics, 1980, 32(3): 314–331
Collins DW, Jukes TH. Rates of transition and transversion in coding sequences since the human-rodent divergence. Genomics, 1994, 20(3): 386–396
Danecek V, Silhavy P. The fault-tolerant control system based on majority voting with kalman filter. International Conference on Telecommunications and Signal Processing, 2011
Fu XT, Dou JZ, Mao JX,. RAD typing: An integrated package for accurate de novo codominant and dominant RAD genotyping in mapping populations. PLoS ONE, 2013, 8(11): e79960
Gojobori T, Li WH, Graur D,. Patterns of nucleotide in pseudogenes and functional genes. Journal of Molecular Evolution, 1982, 18(5): 360–369
He YY, Wang QY, Tan LY,.Estimates of heritability and genetic correlations for growth traits in Chinese shrimp.Journal of Anhui Agricultural Science, 2011, 39(17): 10499–10502 [何玉英, 王清印, 谭乐义, 等. 中国对虾生长性状的遗传力和遗传相关估计. 安徽农业科学, 2011, 39(17): 10499–10502]
Huang YP. Progress of studies on molecular markers. Journal of the Graduate Sun Yat-Sen University: Natural Science, Medicine, 2010, 31(2): 27–36 [黄映萍. DNA分子标记研究进展. 中山大学研究生学刊: 自然科学、医学版, 2010, 31(2): 27–36]
Jiao WQ, Fu XT, Dou JZ,.High-resolution linkage and quantitative trait locus mapping aided by genome survey sequencing: Building up an integrative genomic framework for a bivalve mollusc. DNA Research, 2014, 21(1): 85–101
Li G, Hubert S, Bucklin K,.Characterization of 79 microsatellite DNA markers in the Pacific oyster. Molecular Ecology, 2003, 3(2): 228–232
Li J, He YY, Wang QY,. Selective breeding of fast-growing andammonia toxicity-resistant Chinese shrimp (). Progress in Fishery Sciences, 2015, 36(1): 61–66 [李健, 何玉英, 王清印, 等. 中国对虾() “黄海3号”新品种的培育. 渔业科学进展, 2015, 36(1): 61–66]
Li R, Yu C, Li Y,. SOAP2: An improved ultrafast tool for short read alignment. Bioinformatics, 2009, 25(15): 1966– 1967
Li WH, Wu CI, Luo CC. Nonrandomness of point mutation as reflected in nucleotide substitutions in pseudogenes and its evolutionary implications. Journal of Molecular Evolution, 1984, 21(1): 58–71
Li XP, Kong J, Meng XH,. Effect of white spot syndrome virus infection on bantam and candidate target genes in the Chinese shrimp. Progress in Fishery Sciences, 2018, 39(6): 106–113 [李旭鹏, 孔杰, 孟宪红, 等. WSSV感染对中国明对虾bantam及其候选靶基因的影响. 渔业科学进展, 2018, 39(6): 106–113]
Luan S, Jin W, Kong J,.Genetic evaluation and investment appraisal of multi-trait selection breeding program in Chinese shrimpActa Oceanologica Sinica, 2013, 35(2): 133–142 [栾生, 金武, 孔杰, 等. 中国对虾()多性状复合育种方案的遗传和经济评估. 海洋学报, 2013, 35(2): 133–142
Liu AR, Liao MJ, Li B,. Validation of SNPs associated with important economic traits of sea cucumber (). Progress in Fishery Sciences, 2019, 40(1): 101– 109 [刘安然, 廖梅杰, 李彬, 等. 刺参()重要经济性状相关SNP标记的验证分析. 渔业科学进展, 2019, 40(1): 101–109]
Liu P, Meng XH, Kong J,.Constructing fractional genomic libraries and screening microsatellites DNA ofHigh Technology Letters, 2004, 14(2): 87–90 [刘萍, 孟宪红, 孔杰, 等. 中国对虾部分基因组文库构建和微卫星DNA序列的筛选. 高技术通讯, 2004, 14(2): 87–90]
Ma DM, Su HH, Zhu HP,. Genetic diversity and genetic structure analysis of different selective breeding generations inusing microsatellite markers. Acta Hydrobiologica Sinica, 2018, 42(5): 887–895 [马冬梅, 苏换换, 朱华平, 等. 华南鲤选育群体不同世代遗传多样性与遗传结构的微卫星分析. 水生生物学报, 2018, 42(5): 887–895]
Rousset F. Genepop’007: A complete re-implementation of the genepop software for Windows and Linux. Molecular Ecology Resources, 2008, 8(1): 103–106
Shi T, Zhuang ZM, Kong J,. RAPD analysis of genetic diversity of Chinese shrimp (). Progress in Natural Science: National Key Laboratory Newsletter, 2001, 11(4): 360–364 [石拓, 庄志猛, 孔杰, 等. 中国对虾遗传多样性的RAPD分析. 自然科学进展: 国家重点实验室通讯, 2001, 11(4): 360–364]
Sui J, Luan S, Yang GL,. Genetic diversity and population structure of a giant freshwater prawn () breeding nucles in China. Aquaculture Research, 2018, 49(6): 2175–2183
Wang LX, Ke CH, Wang ZY,. Sequence analysis of mitochondrial 16S rRNA gene and molecular phylogeny of eight species of abalones in genus. Journal of Fisheries Science of China, 2006, 13(2): 167–173 [王鹭骁, 柯才焕, 王志勇, 等. 中国沿岸几种鲍线粒体16S rRNA基因片段序列比较及鲍属系统发育. 中国水产科学, 2006, 13(2): 167–173]
Wang QY. From wild to domestication: The development of Chinese shrimpfarming industry in China. In: Progress of mariculture science and technology in China. Beijing: China Ocean Press, 2014, 149–180 [王清印. 中国海水养殖科技进展: 从野生到家养–中国对虾养殖发展评述. 北京: 海洋出版社, 2014, 149–180]
Wang S, Meyer E, Mckay JK,. 2b-RAD: A simple and flexible method for genome-wide genotyping. Nature Methods, 2012, 9(8): 808–810
Wang ZL, Ding Y, Xu YH,. SNP discovery and functional annotation in transcriptome datasets from hemocytes of. Oceanologia et Limnologia Sinica, 2018, 49(2): 403–412 [王忠良, 丁燏, 许尤厚, 等. 马氏珠母贝()血细胞转录组测序数据中SNP标记的开发及其功能注释分析. 海洋与湖沼, 2018, 49(2): 403–412]
Wright S. Variability within and among natural populations. Chicago: University of Chicago Press, 1978, 121–124
Wu YY, Meng XH, Kong J,. Application of unlabeled probe by HRM in development of EST-SNPs of. Progress in Fishery Sciences, 2013, 34(1): 111–118 [吴莹莹, 孟宪红, 孔杰, 等. 非标记探针HRM法在中国对虾EST-SNP筛选中的应用. 渔业科学进展, 2013, 34(1): 111–118]
Xu AB, Wang WQ. Novel molecular marker method established by combining several molecular labeling methods. Bulletin of Biology, 2007, 42(1): 26–28 [徐安毕, 王文泉. 几种分子标记方法相结合建立的新型分子标记方法. 生物学通报, 2007, 42(1): 26–28]
Yu Y. Development of molecular markers and their applications in selective breeding of the Pacific white shrimp,. Doctoral Dissertation of Institute of Oceanology, Chinese Academy of Sciences, 2014 [于洋. 凡纳滨对虾分子标记的开发及其在遗传育种中的应用. 中国科学院研究生院(海洋研究所)博士研究生学位论文, 2014]
Zhang H, Gao TX, Zhuang ZM,. Comparative analysis of the mitochondrial control region between the cultured and wild populations of Chinese shrimp (). Journal of Fisheries of China, 2010, 34(8): 1149–1155 [张辉, 高天翔, 庄志猛, 等. 中国对虾养殖群体与野生群体线粒体控制区序列的比较. 水产学报, 2010, 34(8): 1149–1155]
Zhang L, Li YL, Li YP,. A population genetic analysis of continuously selectedpopulations. Journal of Ocean University of China, 2018, 17(4): 913–919
Zhang RL, Wang WJ, Feng YW,. Assessment of genetic variability and microsatellite analysis of Pacific oyster () after artificial selection of the shell width.Progress in Fishery Sciences, 2016, 37(4): 90–96 [张荣良, 王卫军, 冯艳微, 等. 长牡蛎()壳宽快速生长选育群体遗传多样性及遗传结构的微卫星标记分析. 渔业科学进展, 2016, 37(4): 90–96]
Zhang TS, Wang QY, Liu P,. Genetic diversity analysis on selected populations of shrimpby microsatellites. Oceanologia et Limnologia Sinica, 2005, 36(1): 72–80 [张天时, 王清印, 刘萍, 等. 中国对虾()人工选育群体不同世代的微卫星分析. 海洋与湖沼, 2005, 36(1): 72–80]
Zhao GT, Liu XD, Wang ZY,Genetic structure and genetic diversity analysis of four consecutive breeding generations of large yellow croaker () using microsatellite markers. Journal of Fisheries of China, 2010, 43(4): 500–507 [赵广泰, 刘贤德, 王志勇, 等. 大黄鱼连续4代选育群体遗传多样性与遗传结构的微卫星分析. 水产学报, 2010, 43(4): 500–507]
Zhao Z, Boerwinkle E. Neighboring-nucleotide effects on single nucleotide polymorphisms: A study of 2.6 million polymorphisms across the human genome. Genome Research, 2002, 12(11): 1679–1686
Analysis of Genetic Diversity in Three Generations of Breeding Populations ofBased on Reduced-Representation Genome Sequencing
WANG Fengjiao1,2,3, MENG Xianhong1,2①, FU Qiang1,2, LUAN Sheng1,2, SUI Juan1,2
(1. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Key Laboratory of Sustainable Development of Marine Fisheries, Ministry of Agriculture and Rural Affairs, Qingdao 266071;2. Laboratory for Marine Fisheries Science and Food Production Processes, Pilot National Laboratory for Marine Science and Technology (Qingdao), Qingdao 266071; 3. College of Fisheries and Life Science, Shanghai Ocean University, Shanghai 201306)
Using a reduced-representation genome sequencing (2b-RAD) technique, this study performed genome-wide single-nucleotide polymorphism (SNP) screening of 649 individuals selected from three consecutive breeding generations (G9~G11) in a population ofnamed “Huanghai 2”. Genetic structure and genetic diversity of the parents’ population were further analyzed using these markers. In total, 66985 SNPs were developed and genotyped in the 649 shrimp. A genetic analysis showed that the average nucleotide diversities (P) were 0.1439, 0.1587, and 0.1674 in G9, G10, and G11, respectively. The average observed heterozygosities (H) were 0.1388, 0.1515, and 0.1609, respectively. The polymorphic information contents (PIC) were 0.1241, 0.1360, and 0.1430. Genetic diversity parameters of the G9~G11parent population showed an undisputed upward trend, although it was not significant. An-test showed that the totalstvalue of all three generations was 0.0061, and the degree of genetic differentiation between adjacent generations was weak (G9~G100.0029, G10~G110.0026). Genetic distances of adjacent generations decreased slightly, to 0.0029 and 0.0026 in G9~G10and G10~G11, respectively. Gene flow was 62.91~94.63 in all generations, which indicated that sufficient gene exchange. This study confirmed a certain impact of artificial selection on genetic diversity and genetic structure inbreeding populations. It also showed that current breeding strategies (selection pressures of 4% to 5% in each generation), do not undermine genetic diversity in parental populations. Genetic differentiation in each generation’s breeding population was small, and genetic structures tended to be stable. The study provides a basic database for genetic analysis ofat the molecular level, and theoretical guidance and data support for the formulation of abreeding program
; Breeding populations; 2b-RAD; SNP; Genetic diversity
MENG Xianhong, E-mail: mengxianhong@ysfri.ac.cn
S966.12
A
2095-9869(2020)04-0068-09
10.19663/j.issn2095-9869.20190510001
http://www.yykxjz.cn/
王凤娇, 孟宪红, 傅强, 栾生, 隋娟. 基于简化基因组测序的中国明对虾3个选育世代遗传多样性分析. 渔业科学进展, 2020, 41(4): 68–76
Wang FJ, Meng XH, Fu Q, Luan S, Sui J. Analysis of genetic diversity in three generations of breeding populations ofbased on reduced-representation genome sequencing. Progress in Fishery Sciences, 2020, 41(4): 68–76
* 重点研发计划蓝色粮仓科技创新项目课题(2018YFD0901302)、国家自然科学基金面上项目(41676148)和中国水产科学研究院黄海水产研究所级基本科研业务费项目(20603022017001)共同资助 [This work was supported by Key Plan Blue Granary Science and Technology Innovation Project (2018YFD0901302), National Natural Science Foundation of China (41676148), and Special Fund for Basic Scientific Research Business of Central Public Research Institutes, YSFRI, CAFS(20603022017001)]. 王凤娇,E-mail: 1730585520@qq.com
孟宪红,研究员,E-mail: mengxianhong@ysfri.ac.cn
2019-05-10,
2019-05-30
(编辑 马璀艳)
