奶牛CSF3基因遗传多态性筛查及其生物信息学分析
2020-06-24黄国明李耀东刘丽霞
吴 佳,陈 朗,姜 涛,黄国明,李 倬,李耀东,张 丽,刘丽霞
(西北民族大学 生命科学与工程学院,甘肃 兰州 730030)
奶牛乳房炎是奶牛养殖的重要疾病,对奶牛生产造成巨大的危害和经济损失,国内外众多学者已对其防治进行了深入研究,研发了大量的诊断方法、治疗药物(如抗生素、中草药及其制剂)、新型饲料添加剂、疫苗和基因工程产品等。从分子遗传育种的角度进行乳房炎的抗病育种已成为一种新型的疾病防治手段[1],研究表明,集落刺激因子3(CSF3)在乳腺炎易感奶牛群体中高表达,表明该基因可作为奶牛乳腺炎抗性研究候选基因群[2]。
CSF3是一种具有抗炎作用的保护性细胞因子,被认为是中性粒细胞产生的关键调节因子,对于清除细菌病原体以及调节炎症反应至关重要[3]。魏玉好[4]指出,CSF3与乳腺上皮细胞(MECs)炎症免疫密切相关,CSF3基因具有特异性受体,在成熟中性粒细胞中高度表达。细菌感染期间宿主防御炎症反应的强度与CSF3受体(CSF3R,或CD114/G-CSFR)信号有直接关系[5]。Hollmén等[6]确定了G-CSF在调节人和小鼠肿瘤中抗炎单核细胞和TAM表型中的重要作用,表现为侵袭性乳腺癌分泌大量的G-CSF,通过将TGF-α分泌增强的抗炎表型转变为肿瘤,浸润的单核细胞,以促进肿瘤生长。CSF3基因中的单核苷酸多态性(SNP)可能对CSF3蛋白功能具有有害作用,而且在dbFNP数据库中CSF3基因的所有SNP中有18.9%存在于编码区(CDS)中[7]。对CSF3基因研究有助于进一步分析SNP位点与奶牛疾病的关联性,探讨该基因对奶牛疾病的调控作用。
本研究通过采用DNA池和直接测序法对奶牛CSF3基因遗传多态性进行快速筛查,确定了CSF3基因的SNP位点,估算了各SNP的等位基因频率,并对该基因的特性和结构进行了预测分析,为奶牛乳房炎相关分子标记筛选提供理论参考。
1 材料与方法
1.1 奶牛血样采集及基因组DNA提取
奶牛的血样采自宁夏农垦贺兰山奶业有限公司,以随机抽样的方法对303头奶牛进行尾静脉采集全血10 mL,采用常规苯酚-氯仿法提取基因组DNA,所提取的DNA以0.8%琼脂糖凝胶进行电泳检测,并利用紫外分光光度计对其进行纯度测定。
1.2 CSF3基因的引物设计及合成
参照NCBI数据库中奶牛的CSF3基因序列(NCBI登录号:NM_174028.1),利用NCBI上的Primer-BLAST在线工具软件设计扩增引物。将CSF3基因上分成3段扩增,预期扩增片段长度分别为363、469、671 bp。引物由天津金唯智生物公司合成,引物信息见表1。
1.3 混合池的构建与PCR扩增
选取300个奶牛为DNA样品,将奶牛DNA样品浓度均调整为100 ng·μL-1,每50个样品各取1 μL构建一个DNA混合池,以混合池DNA为模板进行PCR扩增。扩增体系为:PCR反应总体积20 μL,2×PowerTaqPCR Master Mix 11 μL,混合DNA 0.8 μL,上下游引物(10 μmol·L-1)各0.4 μL,灭菌超纯水补至20 μL。扩增条件为:94 ℃预变性5 min;94 ℃变性30 s,退火60 ℃ 30 s,72 ℃延伸30 s,进行30个循环;最后72 ℃延伸10 min;4 ℃保存。PCR产物用1%的琼脂糖凝胶电泳检测,特异性好的扩增产物保存于-20 ℃冰箱备用。
表1CSF3基因引物信息
Table 1 Primer information ofCSF3 gene

位置Location引物序列Primer sequence (5′→3′)退火温度Annealing temperature/℃预期扩增片段长度Expected amplified fragment length/bpP1F: CTGGTCCCTGACAGAACCCR:GCTGGCCTCCCTCACTCA60363P2F:GCTCCTGGGTCCTTCCTTTCR:ACCCTCATGGCGTCTCTAGT60469P3F:TTAAGGGCAGAACTGAGCTGGR:TTGGGAGACTTCCCGATGCT60671
外显子1和外显子2为P1,外显子3和外显子4为P2,外显子5为P3。
Exon 1 and exon 2 were P1, exon 3 and exon 4 were P2, and exon 5 was P3.
1.4 序列测定与分析
挑选效果良好的样品送至苏州金唯智生物科技有限公司进行纯化后双向测序,利用Editseq和MEGA6.0软件对测序结果进行比对拼接,筛选CSF3基因SNP位点。
1.5 等位基因频率估算
利用MWSnap软件的度量尺工具对CSF3基因测序峰图中各SNP位点的峰高进行测量记录,并估算等位基因频率[8]
HAB=VA(VB)/(VA+VB)。
SNP位点中A或者B等位基因的频率由HAB表示,等位基因在SNP位点中的峰图峰高用VA或VB表示。
1.6 生物信息学分析软件
奶牛CSF3基因相关生物信息学分析软件如下[9]:mRNA二级结构预测,http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuiteRNAfold.cgi?tdsourcetag=s_pcqq_aiomsg;ORF区开放阅读框,http://www.ncbi.nlm.nih.gov/gorf/gorf.html;蛋白质理化性质分析,http://web.expasy.org/protparam/;蛋白质二级结构预测,https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html;蛋白质三级结构预测,http://swissmodel.expasy.org/和PyMOL软件;编码蛋白质信号肽跨膜区的预测,http://www.cbs.dtu.dk/services/SignalP/。
2 结果与分析
2.1 奶牛CSF3基因PCR扩增结果
将CSF3基因分3段进行扩增,产物用1%琼脂糖凝胶电泳检测,结果如图1所示,引物特异性良好,3段目的基因片段大小符合要求。
2.2 PCR扩增产物序列测定比对
奶牛CSF3基因与NCBI中的该基因序列(NM_174028.1)进行比对,结果如图2、图3所示,奶牛CSF3基因共有3个SNP位点,均位于外显子2上,分别为T66C、C67A、C118A,全部为错义突变,分别导致丝氨酸(Ser)、脯氨酸(Pro)变为组氨酸(His)[10]。

M,DL 2000 marker;1,外显子1和外显子2PCR产物;2,外显子3和外显子4PCR产物;3,外显子5 PCR产物。M, DL 2000 marker; 1, Exon 1 and exon 2 PCR products; 2, Exon 3 and exon 4 PCR products; 3, Exon 5 PCR product.图1 奶牛CSF3基因PCR扩增产物检测Fig.1 Detection of PCR amplification products of CSF3 gene in dairy cow

图2 奶牛CSF3基因扩增产物测序发现的SNP位点Fig.2 SNP locus found in sequencing of cow CSF3 gene amplification products

图3 奶牛CSF3基因突变位点示意图Fig.3 Schematic diagram of CSF3 gene mutation site in dairy cow
2.3 奶牛CSF3基因SNP位点基因频率
用MWSnap软件的测量尺对该基因各位点峰图峰高进行测量,根据估算公式计算出各等位基因频率。经计算得,C67A、C118A位点中等位基因C均为优势等位基因,在T66C中的T等位基因为优势等位基因(表2)。
2.4 奶牛CSF3基因mRNA二级结构预测
在NCBI中对奶牛CSF3基因(登录号NM_174028.1)突变前后预测对比结果如图4所示,C118A的自由能略有增加,为-591.50 kcal·mol-1,而T66C、C67A的自由能略有降低,分别为-592.70、-593.40 kcal·mol-1,使mRNA二级结构的稳定性发生了变化,在一定程度上影响蛋白质的翻译[11]。
2.5 奶牛CSF3基因序列的开放阅读框
表2 奶牛CSF3基因SNP位点基因频率
Table 2CSF3 gene SNP locus gene frequency

SNPs所处位置Location等位基因频率Allele frequencyT66C外显子2 Exon 2T(0.79)C(0.21)C67A外显子2 Exon 2C(0.78)A(0.22)C118A外显子2 Exon 2C(0.67)A(0.33)
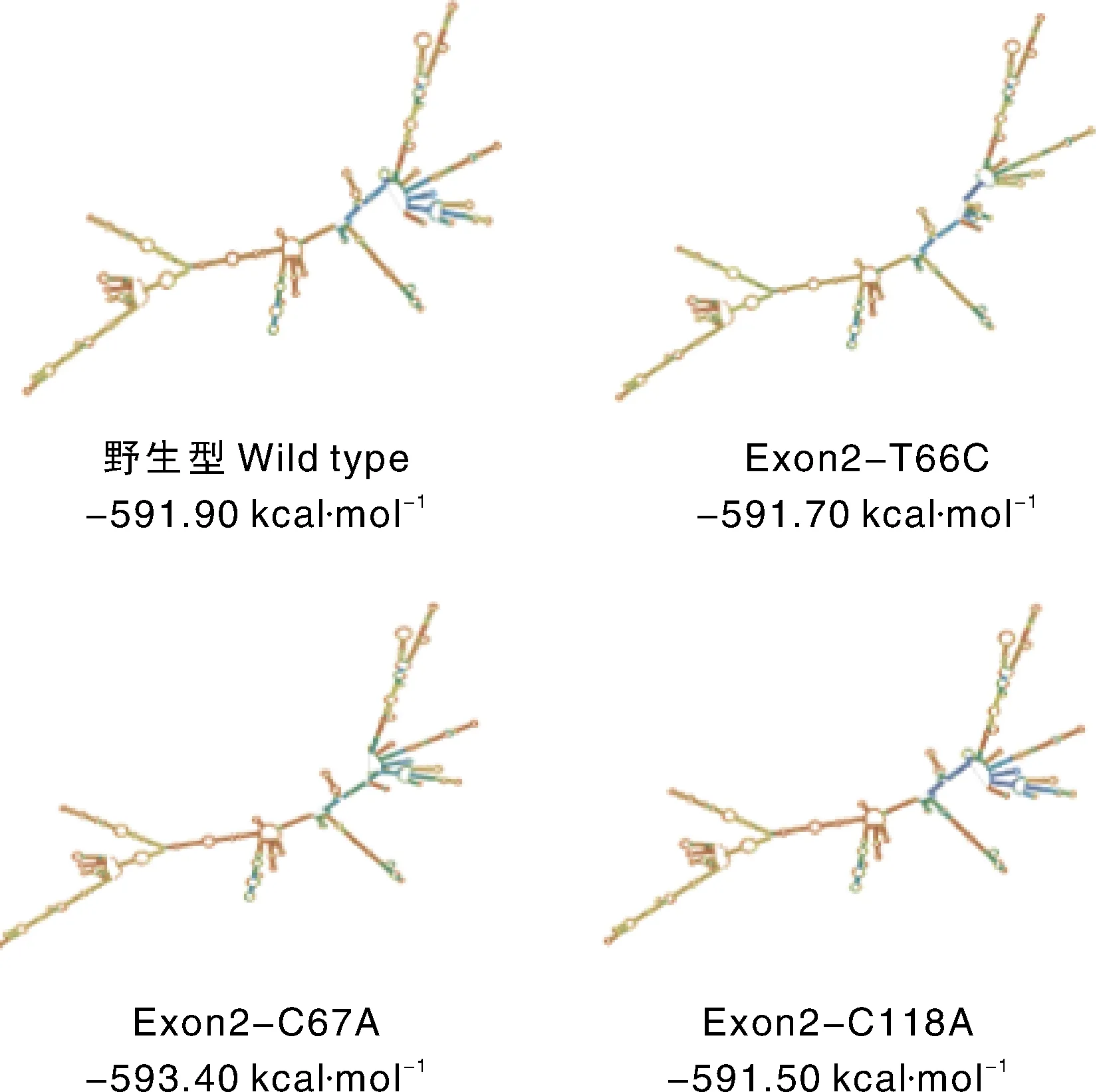
野生型为奶牛CSF3基因突变前预测结果图。Wild type is the predicted result of CSF3 gene mutation in dairy cows.图4 奶牛CSF3基因各个位点mRNA二级结构Fig.4 Secondary structure of mRNA in various sites of CSF3 gene in dairy cow
开放阅读框(open reading frame,ORF)是DNA上的一段碱基序列,有完整的起始密码子和终止密码子序列,因而编码一个蛋白[7]。分析结果显示,有588 bp的完整开放阅读框,5′端起始密码子是ATG,3′端终止密码子是TGA,编码195个氨基酸。

图5 奶牛CSF3基因ORF区开放阅读框分析Fig.5 Analysis of open reading frame of ORF region of dairy cow CSF3 gene
2.6 奶牛CSF3基因编码氨基酸理化性质分析
运用Expasy服务器上的protparam程序预测结果见图6所示,奶牛CSF3基因编码195个氨基酸,21种氨基酸中亮氨酸(Leu)数目最多,占整个氨基酸组成的20.5%,负电荷残基总数(Asp+Glu)为15,正电荷残基总数(Arg+Lys)为13,其分子式为C962H1538N262O271S10,相对分子质量为21 430.99 ku,半衰期为30 h,脂肪指数为109.18,亲水性的平均值为0.156,基因编码产物不稳定指数为62.42,大于40,显示该基因编码产物不稳定[12]。
2.7 奶牛CSF3基因编码蛋白质信号肽跨膜区的预测
奶牛CSF3基因编码产物C值为0.034 8,Y值为0.458 8,S值为0.965 2,其切割位点于20~21位的氨基酸之间,基本可以断定CSF3基因编码产物存在信号肽(图7)。
2.8 奶牛CSF3基因编码蛋白质亲/疏水性分析
利用Expasy服务器上的Protscale程序预测奶牛CSF3基因编码蛋白质的疏水性,其中第10位异亮氨酸(Ile)疏水性最强(+ 4.500);第2位的精氨酸(Arg)亲水性最强(-4.500)。在整条氨基酸肽链中,最大值为2.300,最小值为-1.322,平均亲疏水性分值为-0.99,表现为亲水性(图8)。因此,可推断CSF3基因编码的蛋白质是一种可溶性蛋白[13]。
2.9 奶牛CSF3基因编码蛋白质二级结构和三级结构的预测
奶牛CSF3基因编码蛋白质二级结构预测结果如图9所示,其中α-螺旋(h)占48.72%,β-折叠(e)占11.79%,无规则卷曲(c)占39.49%,编码蛋白质二级结构中,h和c占主导地位。

图6 奶牛CSF3基因编码蛋白质氨基酸组成Fig.6 Amino acid composition of protein encoded by CSF3 gene in dairy cow
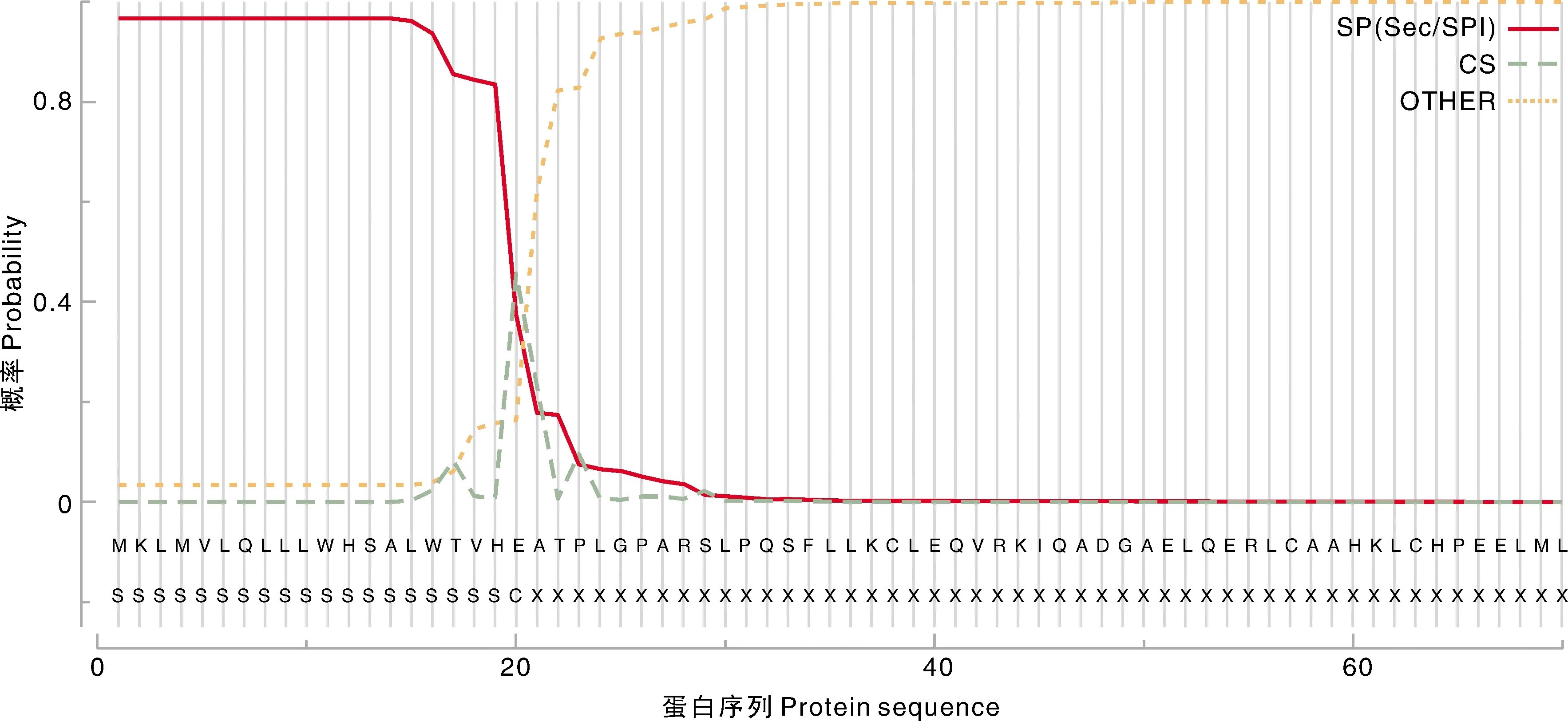
图7 奶牛CSF3基因编码蛋白质信号肽跨膜区的预测Fig.7 Prediction of transmembrane region of protein signal peptide encoded by dairy cow CSF3 gene

图8 奶牛CSF3基因编码氨基酸序列亲/疏水性预测Fig.8 Prediction of amino acid sequence affinity/hydrophobicity of dairy cow CSF3 gene
运用SWISS MODEL进行建模得以预测蛋白质三级结构(图10),其组分有3种,主要成分为h和c,与二级结构预测结果一致[9]。
3 讨论
CSF3基因在哺乳动物上具有较丰富的多态性,对该基因进行生物信息学分析可以提供物种的遗传多样性信息,对动物种质资源的保护和利用具有重要的生物学意义。目前国内外对于人、鸡、小鼠等动物CSF3基因的研究已有报道[14-16],张天等[17]发现GM-CSF3对化疗期间乳腺癌患者BCR-CDR3受体库有影响。赵淑清等[18]研究表明,CSF3是体内非常重要的细胞生长因子,能够调节粒系细胞的增殖、分化与存活,诱导T细胞免疫耐受,可以抑制急性移植物抗宿主病的发生。张灿[16]发现,小鼠GM-CSF3基因与GenBank中的序列相比存在点变异,α-螺旋在蛋白质结构中的占比在50%以上,有1~2个糖基化位点。本研究表明,CSF3基因中有3个SNP位点,全部为错义突变,突变位点使mRNA二级结构改变,进而在某种程度上影响基因编码产物[19-20]。

h,α-螺旋;e,β-折叠;c,无规则卷曲。h, α-helix; e, β-sheet; c, Random coli.图9 奶牛CSF3基因编码蛋白质二级结构预测结果Fig.9 Prediction results of secondary structure of protein encoded by CSF3 gene in dairy cow

图10 奶牛CSF3基因编码蛋白质三级结构预测结果Fig.10 Dairy cow CSF3 gene encoding protein tertiary structure prediction results
蛋白质是生物功能的主要载体,而氨基酸是蛋白质的构件分子。氨基酸的内在性质在一定程度上影响着蛋白质的功能。基因突变位点可能通过影响编码氨基酸序列,间接影响相关蛋白质的二、三级结构,进而影响蛋白质对应的相关调控功能[21]。本研究发现,在CSF3基因的CDS区筛选到3个SNP突变位点,并且全部为错义突变,导致两个氨基酸发生突变。核苷酸的改变直接影响蛋白质的结构,而结构进一步影响蛋白质的功能。奶牛CSF3蛋白质二级结构中,α-螺旋和无规则卷曲占主导地位,而且占比高达48.72%和39.49%,奶牛CSF3编码蛋白的二级结构为混合型,奶牛CSF3蛋白是以α-螺旋和无规则卷曲为主的混合型蛋白,可能会对二级结构稳定性造成一定影响[22]。奶牛CSF3蛋白由195个氨基酸折叠而成,是不稳定指数(Ⅱ)大于40的不稳定蛋白,同时该蛋白的平均亲疏水性分值为-0.99,判定奶牛CSF3蛋白质是可溶性蛋白。一般情况下,蛋白质半衰期越长则稳定性越高,奶牛CSF3编码产物的半衰期较长为30 h,却是不稳定蛋白,可能与基因编码区的突变位点存在某种关系[21]。因此,本研究利用生物信息学分析软件对奶牛CSF3基因编码区的结构及序列特征进行分析,可为今后深入研究奶牛CSF3的基因功能及乳腺炎的有效防控提供参考资料。
