龙眼DlICE1基因克隆及低温胁迫表达分析
2020-06-19霍雯李佳蜜徐小萍赖瑞联陈晓慧林玉玲陈裕坤赖钟雄
霍雯 李佳蜜 徐小萍 赖瑞联 陈晓慧 林玉玲 陈裕坤 赖钟雄


摘 要:本研究以龙眼胚性愈伤组织为材料,根据龙眼转录组数据库,采用RACE-PCR和RT-PCR技术获取龙眼DlICE1 cDNA全长序列,并对其进行生物信息学分析与低温胁迫下表达分析。结果表明,龙眼DlICE1 cDNA全长为2327 bp,其中开放式阅读框(ORF)序列长1614 bp,共编码537个氨基酸。生物信息学分析表明,龙眼DlICE1编码的蛋白质属于不稳定的弱酸性蛋白,不具有典型的信号肽,且不属于分泌蛋白;亚细胞定位表明其位于细胞核内,与甜橙的同源蛋白亲缘关系最近;实时荧光定量PCR表明,龙眼DlICE1能够有效响应低温胁迫,低温诱导其表达量提高。研究结果显示,龙眼DlICE1基因参与龙眼胚性愈伤组织抗寒过程。
关键词:龙眼;胚性愈伤组织;DlICE1基因;基因克隆;實时荧光定量PCR
中图分类号:S667.2 文献标识码:A
Cloning and Expression of Low Temperature Stress of DlICE1 in Dimocarpus longan Lour.
HUO Wen, LI Jiami*, XU Xiaoping, LAI Ruilian, CHEN Xiaohui, LIN Yuling, CHEN Yukun,
LAI Zhongxiong**
Institute of Horticultural Biotechnology, Fujian Agriculture and Forestry University, Fuzhou, Fujian 35002, China
Abstract: In this experiment, the embryogenic callus of longan were used as the materials, the cDNA sequence of DlICE1 was cloned from longan embryogenic callus by the method of RACE-PCR and RT-PCR, then it was analyzed by bioinformatical methods and expression analysis under low temperature stress. The full-length sequence of DlICE1 was 2327 bp, which contained an 1614 bp open reading frame encoded 537 amino acids. The bioinformatics analysis indicated that DlICE1 belonged to the unstable acidic protein, which didnt has a typical signal peptide and wasnt a secreted protein; subcellular localization indicated that it is located in the nucleus, which was closest to the homologous protein of sweet orange. The qRT-PCR results indicated that the longan DlICE1 could effectively respond to low temperature stress and induce increased expression at low temperature. In summary, the study suggests that the longan DlICE1 gene may be involved in the cold resistance in longan embryogenic callus.
Keywords: Dimocarpus longan Lour.; embryogenic callus; DlICE1 gene; gene cloning; real-time fluorescent quantitative PCR;
DOI: 10.3969/j.issn.1000-2561.2020.05.016
植物在生长发育过程中,不断受到各种非生物胁迫而严重影响作物的产量、品质以及生长发育。低温是一类重要的非生物因子,可以使植物受到不同程度的伤害甚至死亡。很多植物在寒冷环境下长期生长逐渐进化形成了一套感受和传导寒冷信号的系统,并能够产生一系列适应寒冷环境的变化,以增强抗寒能力[1]。低温诱导植物抗寒性提升的过程称为冷驯化[2],Weiser等[3]首次提出植物在冷驯化过程中基因表达发生改变的观点。
CBF转录因子调控信号路径是目前为止研究最广泛、最受认可的非依赖于ABA途径的转录因子之一。具体过程为ICE1(inducer of CBF expression 1)——CBF转录因子——CRT/DRE(C- repeat binding factor/dehydration-responsive element binding)基序——冷诱导基因COR(cold- regulated)表达——植物抗寒性提升。ICE1基因属于bHLH基因家族,能编码一种特定蛋白结合到CBF3基因启动子的MYC顺式元件上,进而诱导CBF3编码CBF转录因子,CBF与COR基因的顺式作用元件结合,促使COR下游基因表达,进而提高植株抗寒能力[4]。故ICE1的过量表达可以使植物抗寒能力提高[5]。在低温胁迫下,过表达水稻RsICE1可以使其下游冷调控基因OsD REBL和OsTPP1在转录水平上显著上调,说明RsICE1参与了CBF/DREB1冷调控信号网络[6]。王意程等[7]发现低温处理有利于苹果愈伤组织花青苷的累积;低温可以诱导苹果愈伤组织冷信号基因MdICE1以及花青苷合成相关转录因子基因MdMYB10和MdbHLH3的表达。
龙眼(Dimocarpus longan Lour.)是无患子科(Sapindaceae)龙眼属(Dimocarpus)植物,属于亚热带果树,喜温惧冷,年均适应生长的温度为20~22 ℃。温度是影响龙眼生长、结实的重要因素,对其产量和品质均会造成不利影响。因此,研究龙眼抗寒相关基因,有助于进一步探讨龙眼抗寒机理的相关问题,对提高龙眼产量、品质有着重要意义。本研究以龙眼‘红核子胚性愈伤组织及其转录组数据为材料和基础,对DlICE1基因进行克隆以及生物信息学分析,同时采用qRT-PCR技术检测不同温度处理下DlICE1的表达情况,以期为龙眼抗寒机制的后续研究提供科学依据。
1 材料与方法
1.1 材料
福建农林大学园艺植物生物工程研究所提供的‘红核子品种龙眼胚性愈伤组织(LC2细胞系)[8]。龙眼胚性愈伤组织的转录组数据GenBank登录号为SRA059205。
1.2 方法
1.2.1 材料处理 将生长状态良好的龙眼胚性愈伤组织置于不同温度条件下暗培养24 h,设置的温度梯度为0、4、8、12、16、20、24、28、32、36 ℃。暗培养完成后将材料放入液氮中冷冻15 min,随后放入?80 ℃冰箱保存备用。
1.2.2 总RNA提取及cDNA第一链合成 采用Tripure法提取龙眼胚性愈伤组织总RNA,经超微量分光光度计测定RNA浓度及吸光值(OD值),以A260/A280下OD值在1.8~2.0之间的测定结果为宜;采用SMART RACE试剂盒将RNA逆转录为cDNA第一链用于基因克隆;采用TaKaRa公司提供的荧光定量试剂盒逆转录成cDNA用于qRT-PCR实验。
1.2.3 引物设计 从龙眼转录组数据库中查找、提取DlICE1已有的部分转录组序列(1599 bp),将其序列在NCBI数据库中BLAST,与其他物种进行同源性对比。下载DlICE1基因同源性较高的几个序列,如甜橙(Citrus sinensis)、杨树(Populus trichocarpa)、胡杨(Populus euphratica)等,进行DNAMAN同源对比,参考PCR引物设计原理,运用DNAMAN 6.0软件设计3′RACE、5′RACE及全长验证引物,同时根据全长验证获得序列结合实时荧光定量PCR引物设计原则设计实时定量引物(表1),并将引物序列发送至北京六合华大基因有限公司进行引物合成。
1.2.4 目的基因的扩增 以逆转录的龙眼cDNA为模板,将酶、通用引物、设计的RACE引物和无菌水同时加入PCR反应体系进行PCR扩增。PCR反应体系为:94 ℃预变性4 min;94 ℃变性1 min,54 ℃退火45 s,72 ℃延伸30 s,35个循环;72 ℃延伸10 min。参照林玉玲[9]的试验方法,将获得的PCR产物目的片段用1%凝胶电泳鉴定条带是否为目的片段,切割目的片段,进行胶回收,PMD18-T载体进行目的片段的连接,随后在目的片段连接产物中加入大肠杆菌感受态细胞DH5α,进行目的基因的转化;将转化后的产物进行涂板,使其在含LB加Amp的培养板上培养12 h,随后进行挑菌,挑取多个单克隆,进行菌液PCR验证,经凝胶电泳验证,将阳性克隆子发送给上海博尚生物技术有限公司进行基因测序。
1.2.5 生物信息学分析 对克隆得到的基因序列使用DNAMAN获得对应氨基酸序列。采用ExPASy对DlICE1蛋白进行基本理化性质分析;采用SignalP 4.0 Server进行信号肽预测;采用Prot Comp进行亚细胞定位分析;采用TMHMM分析蛋白跨膜结构域;采用NCBI-Protein Tools进行蛋白质保守结构域预测;采用NetPhos 2.0 Server进行磷酸化位点与其他功能位点预测;利用COILS Server预测卷曲结构;利用Jpred3预测蛋白二级结构;利用SWISS-MODEL预测蛋白三级结构;采用MEGA 6.0进行同源性和分子系统进化树分析;使用MEGA 6.0将以上序列载入,采用采用最小邻近法,设置Bootstrap为1000进行分子同源系统进化树分析。
1.2.6 实时荧光定量PCR表达分析 按照赵双宜等[10]建立的方法分别提取不同温度处理后的龙眼胚性愈伤组织总RNA并编号,经1%琼脂糖凝胶电泳检测合格后,采用PrimeScript? RT Reagent Kit(TaKaRa)试剂盒进行cDNA逆转录。实时荧光定量反应参照Lin等[11-12]的步骤方法进行,以龙眼EIF4a、EF-la和FSD作为内参基因,采用罗氏LightCyclers? 480和SYBR? Prumix EX Taq? Ⅱ对不同温度处理下龙眼DlICE1基因进行qRT-PCR实验,重复3次。最后按照Livak[13]等的方法进行定量计算,并通过Excel软件对Cp值进行统计分析与图标制作,采用SPSS 19.0进行数据显著性差异分析。
2 结果与分析
2.1 龙眼DlICE1 cDNA全长序列获得
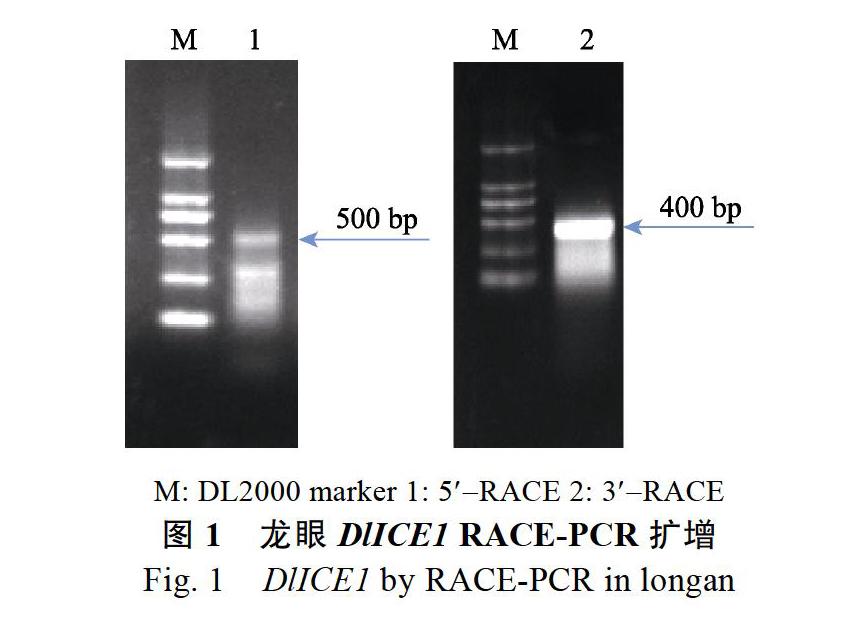
以龙眼‘红核子胚性愈伤组织为材料提取总RNA并进行反转录,采用RACE-PCR技术进行PCR扩增,PCR产物进行琼脂糖凝胶电泳,得到两条目的条带,5′-RACE和3′-RACE所得片段长度分别约为500 bp和400 bp,与目的片段大小一致,产物电泳结果如图1所示。
拼接后进行全长验证,获得龙眼DlICE1 cDNA序列全长为2327 bp,其中ORF区域长度为1614 bp,编码537个氨基酸。将所得cDNA序列在NCBI上进行在线BLAST同源性比对,结果显示该序列与其他物种的DlICE1基因同源性均较高,其中與甜橙和河岸葡萄的ICE1同源性最高,其相似率都高达82%,试验结果可确认所得序列为龙眼DlICE1基因的序列。运用DNAMAN获取ORF序列和氨基酸序列如图2所示。
随着近期龙眼基因组的公布[28] ,人类对龙眼的认识取得突破性进展,以后的科研试验可以从基因组水平上对龙眼的生长发育和进化起源等重大问题入手,有利于新基因的发现与物种的改良等。在此研究基础上,结合龙眼基因组数据的运用,研究ICE1基因与其他相关基因在龙眼抗寒过程中的相互作用以及培育龙眼抗寒新品种有了新方向。
参考文献
向殿军, 满丽莉, 张 娣, 等. LsICE1基因表达及调控转基因水稻抗冷通路研究[J]. 核农学报, 2013, 27(10): 1424- 1430.
Chinnusamy V, Zhu J, Zhu J K. Cold stress regulation of gene expression in plants[J]. Trends in Plant Science, 2007, 12(10): 444-451.
Weiser R L, Wallner S J, Waddell J W. Cell wall and extensin mRNA changes during cold acclimation of pea seedlings[J]. Plant Physiology, 1990, 93(3): 1021-1026.
刘德春. 枳PtrICE1、PtrHOS1和PtrLOS2基因克隆及PtrICE1遗传转化研究[D]. 武汉: 华中农业大学, 2010.
Chinnusamy V, Ohta M, Kanrar S, et al. ICE1: a regulator of cold-induced transcriptome and freezing tolerance in Arabidopsis[J]. Genes and Development, 2003, 17(8): 1043-1054.
Man L, Xiang D, Wang L, et al. Stress-responsive gene RsICE1, from Raphanus sativus, increases cold tolerance in rice[J]. Protoplasma, 2017, 254(2): 945-956.
王意程, 王 楠, 許海峰, 等. 红肉苹果冷信号基因MdICE1的表达及其蛋白与MdMYB的互作[J]. 园艺学报, 2018, 45(5): 817-826.
赖钟雄, 潘良镇, 陈振光. 龙眼胚性细胞系的建立与保持[J]. 福建农业大学学报, 1997(2): 33-40.
林玉玲. 龙眼体胚发生过程中SOD基因家族的克隆及表达调控研究[D]. 福州: 福建农林大学, 2011.
赵双宜, 吴耀荣, 夏光敏. 介绍一种简单高效的植物总RNA提取方法[J]. 遗传, 2002(3): 337-338.
Lin Y L, Lai Z X. Reference gene selection for qPCR analysis during somatic embryogenesis in longan tree[J]. Plant Science, 2010, 178(4): 359-365.
Lin Y L, Lai Z X. Comparative analysis reveals dynamic changes in miRNAs and their targets and expression during somatic embryogenesis in longan (Dimocarpus longan Lour.)[J]. PLoS One, 2013, 8(4): e60337.
Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2?ΔΔCT method[J]. Methods, 2001, 25(4): 402-408.
蒋 瑶, 陈文波, 周泽斯, 等. 植物抗寒转录激活因子ICE1和ICE2基因的生物信息学分析[J]. 贵州农业科学, 2015, 43(3): 24-30.
林元震, 郭 海, 刘纯鑫, 等. 赤桉抗寒转录因子ICE1基因的分子克隆与表达分析[J]. 植物生理学报, 2011, 47(5): 488-494.
林元震, 张志毅, 刘纯鑫, 等. 甜杨抗冻转录因子ICE1基因的in silico克隆及其分析[J]. 分子植物育种, 2007(3): 424-430.
赵玲玲. 苹果MdSAMDC2和MdICE1基因的功能鉴定以及抗逆性研究[D]. 泰安: 山东农业大学, 2007.
向殿军, 殷奎德, 满丽莉, 等. 大白菜低温胁迫转录因子BcICE1的克隆及表达分析[J]. 分子植物育种, 2011, 9(3): 364-369.
Wang X, Liu S, Liu X, et al. Molecular cloning and characterization of a CBF gene from Capsella bursa-pastoris[J]. DNA Sequence, 2004, 15(3): 180-187.
张腾国, 常 燕, 王 娟, 等. 油菜BnICE1的克隆及表达分析[J]. 中国农业科学, 2013, 46(1): 205-214 .
郑银英, 崔百明, 常明进, 等. 转拟南芥ICE1基因增强烟草抗寒性的研究[J]. 西北植物学报, 2009, 29(1): 75- 79.
黄 莹, 徐志胜, 王 枫, 等. 胡萝卜低温胁迫转录因子DcICE1基因克隆与非生物逆境响应分析[J]. 西北植物学报, 2015, 35(1): 30-36.
時 慧. 茶树ICE1基因的克隆及转拟南芥功能验证[D]. 青岛: 青岛农业大学, 2013.
曹慧明, 史作民, 周晓波, 等. 植物对低温环境的响应及其抗寒性研究综述[J]. 中国农业气象, 2010, 31(2): 310-314, 319.
Dong C H, Agarwal M, Zhang Y, et al. The negative regulator of plant cold responses, HOS1, is a RING E3 ligase that mediates the ubiquitination and degradation of ICE1[J]. Proceedings of the National Academy of Sciences, 2006, 103(21): 8281-8286.
Miura K, Rus A, Sharkhuu A, et al. The Arabidopsis SUMO E3 ligase SIZ1 controls phosphate deficiency responses[J]. Proceedings of the National Academy of Sciences, 2005, 102(21): 7760-7765.
肖小娥, 徐小萍, 李佳蜜, 等. 龙眼胚性愈伤组织DlHOS1基因克隆与表达分析[J]. 热带作物学报, 2018, 39(2): 293-299.
Lin Y, Min J, Lai R, et al. Genome-wide sequencing of longan (Dimocarpus longan Lour.) provides insights into molecular basis of its polyphenol-rich characteristics[J]. Gigascience, 2017, 6(5): gix023.
