分子标记和代谢模型的新方法可缩短作物育种周期(2020.5.19 Plant Biotechnology Journal)
2020-06-08
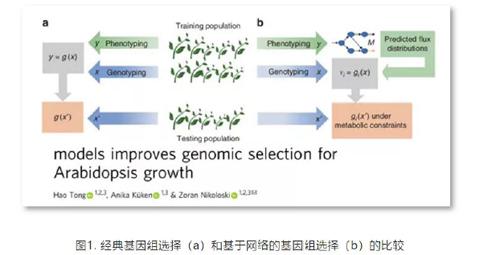
分子标记技术在准确度、精密度和产量方面的进展为改良重要农艺性状提供了新途径。基因组选择是目前最有希望加速新基因型开发和利用的育种方法。它使用机器学习对训练群体进行建模,将给定性状的表型数据与分子标记结合起来,从而识别具有改进性状(如生长)的优良基因型。进一步,性状模型被用于预测已被基因分型但表型未知的试验群体中,根据植株的基因型估计基因组估计育种值(图1a)。提高全基因组选择的精确度可以大大缩短育种周期。然而,能否提高全基因组选择的准确性,尤其是跨环境预测的准确性,仍然是个未知数。
尽管全基因组选择通过统计检验估计了分子标记的效应,但它并没有整合可植物的细胞网络信息。代谢物是遗传调控途径最下游物质,直接参与到植物的生长发育中。代谢反应将营养物质转化为生长发育所需物质,直接影响植物表型。因此代谢网络模型研究被越来越多的应用到植物研究中。代谢网络模型包括所有已知的影响生长的代谢酶功能。目前,植物代谢模型主要被用于研究:(i)通過主要途径预测反应速率,(ii)研究调控途径的影响,(iii)估计营养缺乏对生长的影响,以及(iv)比较不同类型的光合作用。
近日,来自德国波兹坦大学生物化学与生物学研究所,马克斯·普朗克分子植物生理学研究所的Zoran Nikoloski团队在Nature Communications杂志上在线发表了题为“Integrating molecular markers into metabolic models improves genomic selection for Arabidopsis growth”的研究论文。该研究提出一种结合分子标记和代谢模型的方法,该方法能够显著提高拟南芥生长特性的全基因组选择的准确性。
该研究重点关注植物生长这一农艺性状上,而植物的生长在很大程度上取决于可用营养物质转化为生物量组成部分的速率。该研究证明,代谢反应速率是一个多基因性状,可由全基因选择模型预测,并且可估计给定基因型的生长。为进一步提高预测的准确性,作者提出了一种基于网络的全基因组选择(netGS)方法,该方法使用代谢模型,并提高了经典GS对环境内和环境间生长的预测精度(图1b)。与现有67份材料的经典全基因组选择结果相比,新方法在最优氮含量和低氮处理情况下的生长预测准确率分别提高了32.6%和51.4%,从最佳氮环境转移到低碳环境下的生长预测准确率提高了50.4%。这些结果表明,将分子标记整合到代谢模型中,为预测与代谢直接相关的性状提供了一种方法,并且可以通过收集作物中的匹配数据集来检验其在育种中的实用性。
