基于杂交链式反应的适配体传感器用于卡那霉素的比色检测
2020-05-11田润陶晴卞晓军朱福琳颜娟
田润 陶晴 卞晓军 朱福琳 颜娟

摘 要 基于醛基化磁珠颗粒 (Aldehyde magnetic beads,AMBs)和DNA 杂交链式反应(Hybridization chain reaction,HCR)的信号放大策略,设计了检测卡那霉素(Kanamycin,Kana)的比色适配体生物传感器(Aptasensor)。制备了Mbs@DNA复合物,其上的卡那霉素适配体链与卡那霉素发生特异识别后,释放出互补链,互补链含有设计好的引发序列,可作为信号探针,引发HCR反应,生成的双链DNA(Double-stranded DNA,dsDNA)表现出强负电性,与溶液中带负电金纳米颗粒(Goldnanoparticles,AuNPs)相互排斥,在高盐条件下,AuNPs的稳定性被破坏而发生聚集,导致溶液的颜色从红色变为紫色,吸收光谱也发生变化。本方法检测卡那霉素的线性范围为1.6~32.0 nmol/L,检出限为0.9 nmol/L(S/N = 3);实际样品牛奶和蜂蜜样品中卡那霉素的加标回收率在96.0%~105.0%之间,相对标准偏差在3.3%~5.0%之间,说明本方法实用性良好。本方法对卡那霉素的检测具有较高的灵敏度和特异性,操作简单便捷,适用于现场检测等。同时,根据不同待测物具有与其特异性结合的适配体,本方法为其它目标物的检测提供了参考,在食品安全检测与监管中具有良好的应用前景。
关键词 卡那霉素;杂交链式反应;核酸适配体;金纳米颗粒;食品安全
1 引 言
卡那霉素(Kanamycin,Kana)是一种氨基糖苷类抗生素,因其价格低廉、抗菌活性好,常作为饲料添加剂或治疗剂,应用于农业、畜牧业及水产业中[1,2]。然而,卡那霉素的滥用会造成其在动物源性食品中残留,长期摄入含有卡那霉素残留的食品,会引起毒性、肾毒性及抗生素耐药性等副作用[3]。欧盟和日本等国际组织和国家明确规定了动物源性制品中卡那霉素的最大残留量≤200 μg/kg[4,5],牛奶中的最大残留量为200 μg/kg[6]。我国国家标准《食品安全国家标准 食品中农药最大残留限量GB 2763-2016》[7]对其在食品中的残留量也做了明确规定。
目前,对卡那霉素的检测技术有酶联免疫法[8]、高效液相色谱法[9]、毛细管电泳法[10] 等,这些方法虽然准确度高,但需要昂贵的大型设备和专业的操作人员,以及复杂的前处理过程,耗时长。因此,需要建立快速、灵敏、低成本的检测食品中卡那霉素残留的方法。
適配体是通过指数富集的配体系统进化技术(Systematic evolution of ligands by exponential enrichment,SELEX)筛选出来的单链寡核苷酸片段,可与各种靶标(如小分子,抗生素和细胞等)发生特异性识别并结合,形成稳定复合物[11]。与抗原抗体相比,适配体的生物稳定性更好,而且制备简便、特异性好、亲和性高,基于适配体的生物传感器已被广泛应用[12]。但是,仅依靠适配体捕获靶标后引起的构象变化产生的信号进行检测的策略[7],在生物检测分析中的应用较为有限。多种核酸信号放大技术,如聚合酶链式反应(Polymerase chain reaction,PCR)[13]、滚环扩增技术(Rolling circle amplification,RCA)[14]、环介导等温核酸扩增技术(Loop-mediated isothermal amplification,LAMP)[15] 以及杂交链式反应(Hybridization chain reaction,HCR)[16] 的使用,大大提高了适配体生物传感器的检测灵敏度。目前,虽有很多文献报道了采用适配体对卡那霉素进行检测的方法[17~19],然而,这些信号放大技术中,PCR技术需要严格地控制温度,同时需要设计复杂的引物;RCA则需要设计及制备环状模板;LAMP虽然不需要模板,但需要多条引物;并且这些扩增技术都需要有聚合酶参与反应,容易出现假阳性或者假阴性信号,检测成本也较高相比之下,杂交链式反应(Hybridization chain reaction,HCR)是一种自组装信号放大的技术,在常温下进行,且不需要酶参与,操作简便,只需通过碱基互补配对进行的交替杂交的信号放大技术。无酶的参与能够避免由于酶导致的假信号出现,从而提高检测的准确性。同时,HCR对外部条件要求简单,不需要额外辅助变温调控,在室温下就可以进行反应,因此,HCR作为一种简单且有效的新型信号放大策略备受关注[20~24]。HCR通过适配体之间的竞争杂交,自组装成核酸纳米结构,实现信号放大[16]。HCR的构成原件包括引发探针和两条可以互补杂交并带有粘性末端的发夹型DNA(H1和H2)。当没有引发序列时,H1和H2可以稳定存在,但当引发探针出现时,H1的二级结构被引发探针打开,H1释放出的茎端将会打开H2的二级结构,H2释放出的茎端与引发探针的序列相同,又会打开H1的二级结构,H1和H2如此循环往复地被相互打开,最后形成一条含有缺口的杂交长双链共聚物[25]。HCR与各类检测技术都有较好的兼容性,可提高核酸、蛋白、小分子以及重金属等生物分子检测的灵敏度。纳米金也具有良好的生物相容性,易于进行表面修饰,且具备独特的光学特性,因此,比色法是最早采用金纳米粒子进行分析检测的一种方法,利用金纳米粒子的聚集、溶液颜色的改变,反映溶液相中目标检测物的量[26]。本研究利用HCR反应前后,溶液中不同的DNA链与纳米金之间的相互作用导致的纳米金的变色程度,实现体系中卡那霉素的快速灵敏检测。
2 实验部分
2.1 仪器与试剂
UV-2540紫外可见分光光度计(日本岛津公司);Synergy2 SLFPTAD 多功能酶标仪(美国伯腾仪器有限公司);Mixer 4K涡旋混合器(生工生物工程(上海)股份有限公司);HCM100-Pro恒温振荡金属浴、D1008掌上离心机(大龙兴创实验仪器(北京)有限公司)。
四氯金酸水合物(HAuCl4·H2O,百灵威科技公司);柠檬酸三钠(Na3C6H5O7·2H2O,国药集团化学试剂有限公司);磷酸盐缓冲液(20 × PBS,pH 7.4~7.6);Tris-HCL缓冲液(含50 mmol/L NaCl,5 mmol/L MgCl2,5 mmol/L KCl,pH 7.0);琼脂糖M;8600-10氨基磁珠(美国 Polysciences公司);电泳缓冲液(50×TAE,pH 8.4);4S Red Plus 核酸染色剂(10000× 水溶液);牛血清白蛋白(BSA,美国Amresco公司);卡那霉素(Kanamycin)、氯霉素(Chloramphenicol)、四环素(Tetracycline)、氧氟沙星(Ofloxacin)、环丙沙星(Ciprofloxacin)均购于生工生物工程(上海)股份有限公司;实验用水为超纯水。DNA 序列(表1)由生工生物工程(上海)股份有限公司合成,其中,H1、H2使用前需先分别加热到 95℃,并维持5 min,随即冰上孵育5 min,再于室温放置2 h后,备用。
2.2 实验方法
2.2.1 AuNPs的合成 参照文献[27]的方法,在圆底烧瓶中加入99 mL水、 1 mL 1% 氯金酸,加热回流直至沸腾,加入 3.5 mL 1%现配的柠檬酸三钠溶液,反应 20 min 后,停止加热,室温下搅拌过夜,产物于4℃保存,备用。
2.2.2 Mbs@ds-DNA的制备 按照产品说明对磁珠(50 mg/mL,1.5 μmol/L)进行活化,使磁珠上的氨基转化为醛基。取80 μL 醛基化磁珠,磁分离,弃上清液。加入160 μL 1×PBS 缓冲液和16 μL 100 μmol/L 氨基修饰的Kana-Aptamer,37℃孵育过夜后,磁分离,加入200 μL 1×PBS洗涤1次,用2% BSA室温封闭1 h,磁分离后,沉淀重悬于400 μL 1×PBS 缓冲液中,备用。取8 μL 100 μmol/L Kana-Aptamer的部分互补序列Cs-I加入到92 μL Mbs@Kana-Aptamer中,37℃振荡孵育2 h,磁分离,用100 μL 1×PBS洗涤1次,重悬在100 μL Tris-HCl溶液中,4℃保存,备用。
2.2.3 卡那霉素的测定 20 μL MBs@ds-DNA和30 μL不同浓度的卡那霉素,充分混匀后,37℃振荡孵育40 min。反应结束后,磁分离,上清液中加入 H1、H2(体系终浓度为1 μmol/L)进行HCR,37℃孵育2 h。采用AuNPs比色检测,取30 μL HCR产物,加入225 μL AuNPs,在室溫下孵育30 min后,加入30 μL 0.3 mol/L NaCl溶液,静置10 min,测定体系在520 nm波长处的吸光度。
3 结果与讨论
3.1 实验原理
提出了基于杂交链式反应信号放大检测卡那霉素的策略,引入磁性生物探针Mbs@ds-DNA,通过磁分离消除复杂样本体系中基质干扰。如图1所示,连接在磁珠上的适配体序列(Kana-Aptamer)作为捕获探针,与信号探针(Cs-I)部分互补配对,形成Mbs@ds-DNA复合物。卡那霉素存在时,Mbs@ds-DNA复合物上的适配体链和卡那霉素发生特异性结合,释放出信号探针。磁分离后,上清液中游离的信号探针上的引发链部分引发HCR,生成的ds-DNA结构表现出强的负电性,与溶液中带负电AuNPs相互排斥。随着盐浓度增加,AuNPs的稳定性持续降低,并发生聚集,导致溶液的颜色从粉色变为紫色。当体系中不存在卡那霉素时,信号探针不会游离到上清液中,单独的DNA发夹结构(H1,H2)不会发生杂交链式反应,即稳定存在于溶液中,其单链粘性末端通过静电作用吸附在AuNPs表面上,保护AuNPs免受高盐浓度引起的聚集,溶液仍保持红色。
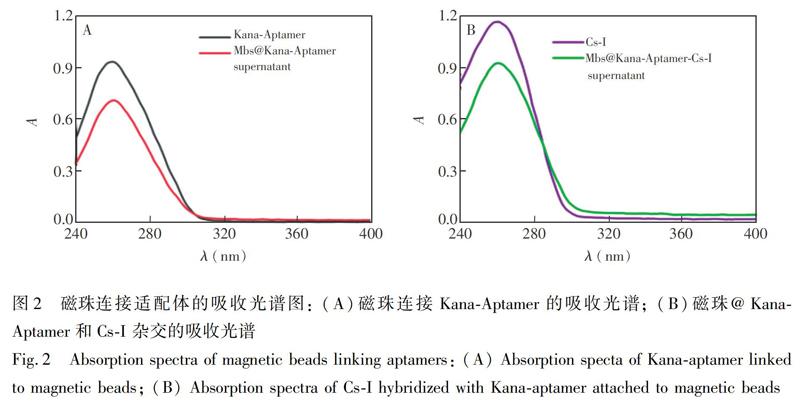
3.2 MBs@ds-DNA的紫外可见吸收光谱
采用吸收光谱对MBs@ds-DNA的组装进行表征。如图2A所示,测得Kana-Aptamer在260 nm处的紫外吸收峰值为0.933,收集磁珠和Kana-Aptamer连接后的上清液,测得其在260 nm处的紫外吸收峰值为0.707,紫外吸收值降低,说明NH2-Kana-Aptamer成功组装在醛基化磁珠表面,体系中游离Kana-Aptamer的数量减少。同理,将MBs@Kana-Aptamer与Cs-I进行孵育后,Cs-I在260 nm处的紫外吸收峰值也降低,说明MBs@Kana-Aptamer与Cs-I杂交,即MBs@ds-DNA成功制备(图2B)。
3.3 琼脂糖凝胶电泳表征
如图3A所示,琼脂糖凝胶电泳结果表明,发夹探针H1(5 μmol/L)、H2(5 μmol/L)不发生HCR(泳道1);Cs-I(2 μmol/L)可与H1杂交,形成很短的双链DNA,进行电泳测试时,出现小段拖尾,但是,大部分发夹H1未打开(泳道2);仅存在H2和Cs-I时,不发生HCR,故电泳中没有拖尾(泳道3);只有当H1、H2、Cs-I同时存在时,才会发生HCR(泳道4)。分别将无引发链存在时HCR体系与加入引发链进行HCR体系的液体滴在干净平整的云母片上,氮气吹干后,用原子力显微镜观察(图3B和3C)。在没有引发链Cs-I时,只有发夹结构H1和H2的微小斑点(图3B),加入互补链条CS-I后,有清晰可见的伸展的ds-DNA长链(图3C),说明发生了HCR。
3.4 比色生物感器的可行性验证
为了验证本研究建立的比色检测方法,将制备的MBs@ds-DNA复合物分别与0和15 μmol/L的卡那霉素标准品溶液孵育后,磁分离,将上清液进行HCR后,再进行纳米金比色检测。当存在卡那霉素时,HCR产物的ds-DNA结构表现出强的负电性,与溶液中带负电AuNPs相互排斥。随着盐浓度增加,AuNPs的稳定性持续降低,并且发生聚集,导致溶液的颜色从红色变为紫色。不存在卡那霉素时,单独的DNA发夹H1、H2的单链粘性末端可保护AuNPs免于高盐条件下的聚集,溶液仍保持红色(图4A)。透射电镜图像(图4B和4C)进一步验证了本实验的可行性,当卡那霉素浓度由0增加到15 μmol/L后,纳米金粒子由良好的分散状态(图4B)变为聚集状态(图4C)。
3.5 基于杂交链式反应的适配体传感器对卡那霉素的检测性能
采用构建的适配体传感器,检测不同浓度的卡那霉素标准品。如图5A中插图所示,随着卡那霉素浓度增加(0~800 nmol/L),溶液颜色由红色逐渐变为紫色(图5A插图),同时吸收光谱(图5A)也随之发生红移。体系在520 nm处的吸光度比值(A0-A)/A(A0和A分别为空白和样品在520 nm的吸光度)与卡那霉素浓度在1.6~32.0 nmol/L之间呈线性关系(图5B),线性方程为y=0.0047x+0.1237(R2=0.9708),检出限为0.9 nmol/L(S/N=3)。如表2所示,与其它检测方法相比,本方法具有操作简单、易于观察检测结果等优势。
采用本方法检测卡那霉素(10 nmol/L)与4种对照物(氯霉素、四环素、氧氟沙星、环丙沙星,1 μmol/L),考察本方法对卡那霉素检测的特异性。如图5C所示,当检测4种对照物时,纳米金溶液颜色无明显变化(酒红色),吸光度比值变化幅度较小;当检测目标卡那霉素时,溶液颜色由红色迅速变为紫色,吸光度比值变化尤为明显。因此,本方法对卡那霉素检测的特异性良好。
3.6 实际样品分析
采用本方法检测牛奶及蜂蜜样品。将纯牛奶在室温下以10000 r/min离心10 min,除去上层脂肪,然后加入一定浓度的卡那霉素进行检测;取1 mL蜂蜜溶于9 mL去离子水中,加入一定浓度的卡那霉素进行检测,结果见表3。牛奶样品的加标回收率在97.3%~99.4%之間,相对标准偏差(RSD)在3.3%~5.0%之间;蜂蜜的加标回收率在96.0%~105.0%之间,RSD在3.5%~4.8%之间,说明本方法灵敏度较高,特异性较好,可用于牛奶和蜂蜜等实际样品中卡那霉素残留的检测,实用性良好。
4 结 论
基于金纳米粒子体系吸光度的变化和HCR构建了适配体传感器,用于卡那霉素的比色检测。此策略无需标记,无需酶的参与,不涉及复杂的引物设计及变温调控,即可实现卡那霉素的特异检测,检出限为0.9 nmol/L,并可用于实际样品如牛奶和蜂蜜中的卡那霉素检测。本方法操作简单便捷,适于现场检测,具有良好的应用前景,对其它抗生素或目标物检测方法的建立具有参考价值。
References
1 Ramezani M,Danesh N M,Lavaee P,Abnous K,Taghdisi S M. Sens. Actuators B,2016,222: 1-7
2 Sharma A,Istamboulie G,Hayat A,Catanante G,Bhand S,Marty J. L. Sens. Actuators B, 2017,245: 507-515
3 Guo W,Sun N,Qin X,Pei M,Wang L. Biosens. Bioelectron., 2015,74: 691-697
4 Kaufmann A,Butcher P,Maden K. Anal. Chim. Acta,2012,711: 46-53
5 Bogialli S,Bruno M,Curini R,Di Corcia A,Lagana A,Mari B. J. Agric. Food Chem.,2005,53(17): 6586-6592
6 Jiang Y,Sun D W,Pu H,Wei Q. Talanta,2019,197: 151-158
7 GB 2763-2016. Maximum Residue Limits for Pesticides in Food. National Food Safety Standard
食品安全国家标准. 食品中农药最大残留限量GB 2763-2016
8 Galvidis I A,Burkin M A. Russ. J. Bioorg. Chem.,2010,36(6): 722-729
9 Chen C,Zhai W T,Lu D D,Zhang H B,Zheng W G. Mater. Res. Bull.,2011,46(4): 583-587
10 Long Y H,Hernandez A,Kaale E,Van Schepdael A,Roets E,Borrull F,Calull M,Hoogmartens J. J. Chromatogr. B,2003,784(2): 255-264
11 Tuerk C,Gold L. Science,1990,249(4968): 505-510
12 Kim Y S,Raston N H,Gu M B. Biosens. Bioelectron.,2016,76: 2-19
13 Kiss M M,Ortoleva-Donnelly L,Beer N R,Warner J,Bailey C G,Colston B W,Rothberg J M,Link D R,Leamon J H. Anal. Chem.,2008,80(23): 8975-8981
14 Mohsen M G,Kool E T. Acc. Chem. Res.,2016,49(11): 2540-2550
15 Huang X,Lin X,Urmann K,Li L,Xie X,Jiang S,Hoffmann M R. Environ. Sci. Technol.,2018,52(11): 6399-6407
16 Dirks R M,Pierce N A. Proc. Natl. Acad. Sci. USA,2004,101(43): 15275-15278
17 Lin X,Su J,Lin H,Sun X,Liu B,Kankala R K,Zhou S F. Talanta,2019,202: 452-459
18 Zeng R,Zhang L,Luo Z,Tang D. Anal. Chem.,2019,91(12): 7835-7841
19 Chen X,Hao S,Zong B,Liu C,Mao S. Biosens. Bioelectron.,2019,145: 111711
20 Zhao Y,Chen F,Li Q,Wang L,Fan C. Chem. Rev.,2015,115(22): 12491-12545
21 Zhou J,Ralston J,Sedev R,Beattie D A. J. Colloid. Interface Sci., 2009,331(2): 251-262
22 Zhen S J,Xiao X,Li C H,Huang C Z. Anal. Chem.,2017,89(17): 8766-8771
23 Zhu D,Huang J,Lu B,Zhu Y,Wei Y,Zhang Q,Guo X,Yuwen L,Su S,Chao J,Wang L. ACS Appl. Mater. Interfaces,2019,11(23): 20725-20733
24 Zhu D,Lu B,Zhu Y,Ma Z,Wei Y,Su S,Wang L,Song S,Zhu Y,Wang L,Chao J. ACS Appl. Mater. Interfaces,2019,11(12): 11220-11226
25 Chen X,Wang J,Shen H Y,Su X,Cao Y,Li T,Gan N. ACS Sens.,2019,4(8): 2131-2139
26 Ikbal J,Lim G S,Gao Z. TrAC-Trend. Anal. Chem.,2015,64: 86-99
27 Frens G. Nat. Phys. Sci., 1973,241(105): 20-22
28 Xu W,Wang Y,Liu S,Yu J,Wang H,Huang J. New J. Chem.,2014,38(10): 4931-4937
29 Bai X,Hou H,Zhang B,Tang J. Biosens. Bioelectron.,2014,56: 112-116
30 Han X,Yu Z,Li F,Shi W,Fu C,Yan H,Zhang G. Bioelectrochemistry, 2019,129: 270-277
31 Ma X,Qiao S,Sun H,Su R,Sun C,Zhang M. Front. Chem.,2019,7: 29
