肠炎沙门菌sifA基因缺失株的无痕构建及其在巨噬细胞内的增殖特性研究
2020-05-05刘桂凤杨家兴王耀楠耿士忠潘志明焦新安
刘桂凤,杨家兴,丁 炜,王耀楠,蔡 媛,张 健,王 洋,耿士忠,潘志明,焦新安
肠炎沙门菌(SalmonellaEnteritidis, SE)是一种兼性细胞内寄生的革兰阴性菌,为重要的人兽共患病原菌之一。该菌无特异性宿主,不仅能引起畜禽发病死亡造成严重的经济损失,而且被污染的畜禽产品作为肠炎沙门菌的携带者,引发人食源性疾病危害公共卫生安全[1-2]。持续感染和免疫逃逸是沙门菌一个致病特征,主要是由于其能够在细胞内进行复制和增殖。沙门菌接触到宿主细胞后能黏附于细胞表面,侵袭进入宿主细胞内部,在细胞内生存繁殖,即沙门菌的胞内增殖。为了能够在细胞内生存和繁殖,沙门菌适应性地产生一种沙门菌囊泡结构(Salmonella-containing vacuole, SCV)来逃避细胞对其的吞噬清除[3-4]。沙门菌的III型分泌系统(Type Ⅲ Secretion Systems, T3SS)是一个由膜内/外蛋白组成的多组份跨膜通道,参与沙门菌囊泡的生物发生和成熟过程。作为“分子注射器”的T3SS主要功能是分泌和转运效应蛋白,它主要通过效应蛋白来改变宿主细胞的生理活动,促进沙门菌侵入细胞并在细胞内生存[5-6]。III型分泌系统包括T3SS-1和T3SS-2,T3SS-1由沙门菌毒力岛1(SPI-1)基因组基因编码,其效应物作用于宿主细胞质膜上以使细菌入侵和SCV生物发生;由沙门菌毒力岛2(SPI-2)编码的T3SS-2效应物作用于囊泡膜,促进SCV成熟、SIF形成和囊泡内沙门菌复制[7-8]。
sifA基因是沙门菌的一个毒力相关基因,由T3SS-2分泌的SifA蛋白在SCV的形成过程中发挥重要作用,sifA基因缺失突变后无法形成SifA蛋白,对沙门菌诱导形成SCV造成严重影响,使其不能在吞噬细胞内正常复制生存[9]。SifA蛋白可能参与沙门菌入侵宿主细胞[10]及沙门菌引起机体免疫功能受损[11]。通过对巨噬细胞系J774.2与上皮细胞系HeLa等一些比较成熟的细胞模型中SCV形成的研究,沙门菌在宿主细胞内SCV的形成机制已经逐渐被阐明,但这只针对少数血清型的沙门菌,其中研究最多的模式菌就是鼠伤寒沙门菌。而对于肠炎沙门菌在细胞内增殖及SCV形成机制的研究比较匮乏,是否与鼠伤寒沙门菌一致,需要进一步深入研究予以验证。
本实验室无痕敲除靶基因技术成熟[12],利用具有lacZ基因的pGMB152自杀质粒作为中间载体,通过蓝白斑筛选的方式,直观地筛选重组菌,构建肠炎沙门菌sifA基因缺失株C50041ΔsifA及双基因缺失株C50041ΔspiCΔsifA,并研究其基本生物学特性及其对胞内肠炎沙门菌增殖的影响,为更深入地探索沙门菌胞内增殖规律奠定基础。对于肠炎沙门菌在细胞内SCV形成机制的研究,将有助于进一步探究肠炎沙门菌致病机理,为肠炎沙门菌的防控提供重要理论依据。
1 材料与方法
1.1菌株、质粒与细胞 本研究中所使用的菌株、质粒及细胞信息见表1。
表1 实验所用菌株、质粒与细胞
Tab.1 Bacterial strains, plasmids and cells used in this study
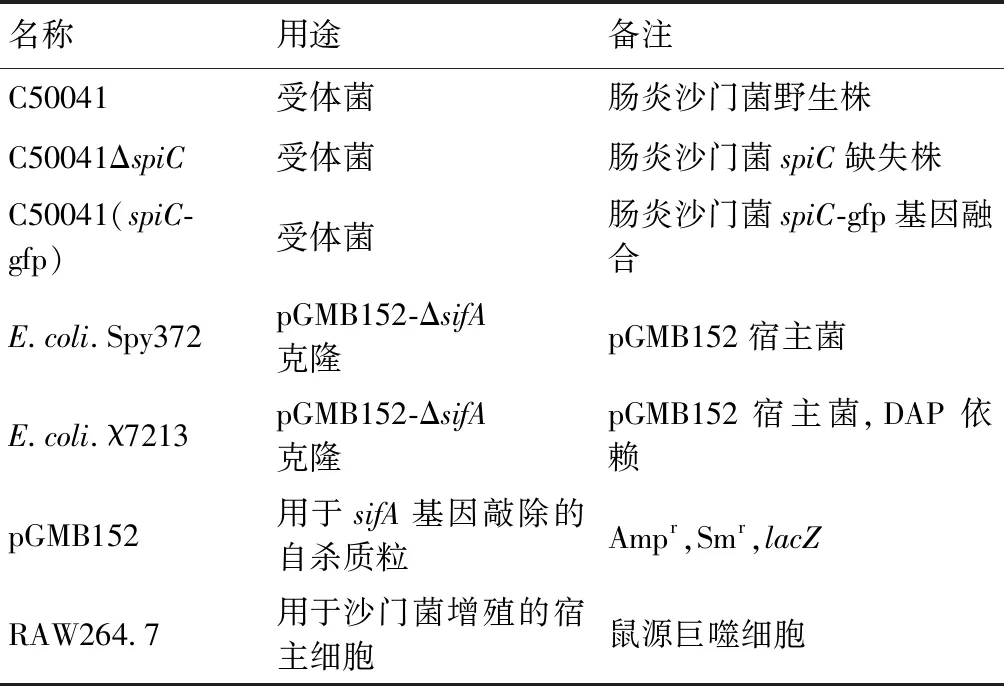
名称用途备注C50041受体菌肠炎沙门菌野生株C50041ΔspiC受体菌肠炎沙门菌spiC缺失株C50041(spiC-gfp)受体菌肠炎沙门菌spiC-gfp基因融合E.coli.Spy372pGMB152-ΔsifA克隆pGMB152宿主菌E.coli.χ7213pGMB152-ΔsifA克隆pGMB152宿主菌,DAP依赖pGMB152用于sifA基因敲除的自杀质粒Ampr,Smr,lacZRAW264.7用于沙门菌增殖的宿主细胞鼠源巨噬细胞
注:来源均为本实验室保存。
1.2主要试剂 DNA胶回收试剂盒、SalⅠ限制性内切酶、质粒纯化试剂盒、细菌基因组提取试剂盒、氨苄青霉素(Amp)和链霉素(Sm)等抗生素、DNA Marker、IPTG、PrimeScriptTMRT reagent Kit、Primestar max DNA Polymerase均购自宝生物工程(大连)有限公司;ClonExpress One Step Cloning一步法连接试剂盒以及2×Taq Master mix购自南京Vazyme生物科技有限公司;FastStart Universal SYBR Green Master(ROX)试剂盒购自罗氏公司;API 20E肠道菌鉴定卡购自法国Bio Mérieux公司;DMEM培养基、胎牛血清、0.25%胰酶、青链霉素购自美国ThermoFisher公司;快速质粒小提试剂盒、细菌及细胞总RNA提取试剂盒购自天根生化科技(北京)有限公司。高效感受态细菌制备试剂盒及其他常规试剂购自生工生物工程(上海)股份有限公司。
1.3引物的设计与合成 本研究中所使用的引物碱基序列见表2,引物由南京金斯瑞生物有限公司合成。
表2 用于PCR扩增的引物
Tab.2 Primers used for PCR in this study

PrimesSequence(5′-3′)Size/bpsifA-U-FCCCCCCCTGCAGGTCGACTAATGGGTGGCGCGAAAAAC620sifA-U-RCGCTTTGTTGTTCTGAGCGAATTCGTATTCCTTGGGGAGTTGGsifA-D-FCCAACTCCCCAAGGAATACGAATTCGCTCAGAACAACAAAGCG730sifA-D-RCTTATCGATACCGTCGACCATCACGATATCCGGCGACAsifA-U-FCCCCCCCTGCAGGTCGACTAATGGGTGGCGCGAAAAAC1349(Δ); 2173(WT)sifA-D-RCTTATCGATACCGTCGACCATCACGATATCCGGCGACApGMB152-FCGTGGAGGCCATCAAACCAC283(Δ); 1632(WT)pGMB152-RCGCGAAATAAACGACCGGGAnsifA-FGTTGTCTAATGGAACCGATAATATCGqPCRnsifA-RCTACCCCCTCCCTTCGACAT16S-FCGGGGAGGAAGGTGTTGTGqPCR16S-RGAGCCCGGGGATTTCACATC
注:黑色粗体部分为与pGMB152SalⅠ酶切位点侧翼序列互补配对片段,sifA-U-R和sifA-D-F为互补序列。
1.4 目的基因与重组自杀载体的制备
1.4.1肠炎沙门菌基因组DNA的提取 将冻存的肠炎沙门菌C50041接种于LB固体平板培养单菌落,将单菌落再接种于液体培养基中,37 ℃ 180 r/min连续震摇培养过夜,取4 mL菌液利用细菌基因组提取试剂盒提取肠炎沙门菌C50041基因组DNA。
1.4.2sifA基因上下游同源臂sifA-U和sifA-D片段的扩增 以所提C50041基因组DNA为模板,分别以sifA-U-F/R和sifA-D-F/R为引物,利用PCR扩增sifA基因上下游同源片段sifA-U、sifA-D片段。PCR扩增体系(50 μL):Primestar max DNA Polymerase 22 μL,引物F(10 μmol/L)2 μL,引物R(10 μmol/L)2 μL,模板DNA 2 μL,灭菌超纯水SW 22 μL。PCR反应条件:98 ℃ 5 min;98 ℃ 10 s,63 ℃ 15 s,72 ℃ 40 s,30个循环;72 ℃ 10 min。PCR反应结束后,利用0.7%的琼脂糖凝胶电泳鉴定PCR产物,利用DNA胶回收试剂盒回收纯化目的片段。
1.4.3pGMB152质粒载体的制备 挑取E.coli. Spy372 (pGMB152)单菌落转接于100 mL含Amp(终浓度100 μg/mL)和Sm(终浓度100 μg/mL)的LB液体培养基中,于37 ℃ 180 r/min摇床内震摇培养过夜,常规方法提取质粒pGMB152后,再用质粒纯化试剂盒纯化。
纯化后的pGMB152质粒利用SalⅠ限制性内切酶进行单酶切。酶切体系(150 μL):H Buffer 15 μL,SalⅠ 10 μL,pGMB152质粒100 μL,灭菌SW 25 μL。酶切反应条件:37 ℃,2 h。利用DNA胶回收试剂盒回收纯化酶切产物,回收产物即为本研究所用线性自杀质粒载体。
1.5sifA基因缺失株C50041ΔsifA无痕构建
1.5.1同源重组自杀质粒pGMB152-ΔsifA构建 利用ClonExpress One Step Cloning一步法连接试剂盒将sifA基因上下游同源片段sifA-U、sifA-D与线性酶切质粒PGMB152进行连接,构建同源重组自杀质粒并将其命名为pGMB152-ΔsifA。
1.5.2E.coli. Spy372与E.coli. χ7213感受态细菌的制备 分别用LB平板和含 DAP(终浓度100 μg/mL)的平板复苏E.coli. Spy372和E.coli. χ7213,于37 ℃培养箱培养单菌落。将E.coli. Spy372转接于LB液体培养基中,E.coli. χ7213转接于含DAP的LB液体培养基中,于摇床震摇培养13 h至对数生长后期,1∶50扩大培养至含100 mL LB液体培养基中,震摇培养至OD600=0.5左右,按照高效感受态细菌制备试剂盒说明书制备E.coli. Spy372与E.coli. χ7213感受态细菌。
1.5.3重组沙门菌C50041(pGMB152-ΔsifA的制备 10 μL连接产物(pGMB152-ΔsifA)加入到100 μLE.coli.Spy372感受态中,置于冰上30 min使其充分接触,于42 ℃金属浴热击90 s,将连接产物热转化进入E.coli.Spy372,将转化后的重组菌涂布在含X-gal(终浓度40 μg/mL)、IPTG(终浓度24 μg/mL)、Amp和Sm的LB固体平板上,形成蓝色重组菌E.coli. Spy372(pGMB152-ΔsifA),经自杀质粒特异性引物pGMB152-F/R进行菌落PCR验证正确后,利用快速质粒小提试剂盒提取重组菌E.coli. Spy372(pGMB152-ΔsifA)的重组质粒。利用热转化将重组质粒转入E.coli.χ7213,转化后的重组菌涂布在含DAP、Amp、Sm、X-gal和IPTG的LB平板上,形成蓝色重组菌E.coli.χ7213(pGMB152-ΔsifA),鉴定正确后,以肠炎沙门菌野生株C50041为受体菌(构建C50041ΔspiCΔsifA时受体菌为C50041ΔspiC),重组菌E.coli.χ7213(pGMB152-ΔsifA)为供体菌,按受体菌∶供体菌=1∶4的比例做固相杂交,进行接合转移[13],固相杂交后混合细菌用PBS稀释至合适梯度后涂布在含Amp、Sm、X-gal和IPTG的平板上形成蓝色重组沙门菌C50041(pGMB152-ΔsifA)。
1.5.4质粒载体的脱除 挑取沙门菌重组菌C50041(pGMB152-ΔsifA)单菌落接种于不含NaCl、含10%蔗糖的LB液体培养基中震摇培养过夜,以脱除自杀质粒,然后将菌液稀释至合适的梯度涂布在含X-gal、IPTG的LB平板上,筛选白色菌落进行PCR鉴定,将PCR鉴定正确的沙门菌命名为C50041ΔsifA。
1.6 C50041ΔsifA鉴定
1.6.1抗生素耐药性鉴定 将缺失株C50041 ΔsifA与接合转移后的重组沙门菌C50041(pGMB152-ΔsifA)在同一块含Amp、Sm的LB固体平板上划线进行抗生素耐药性验证。
1.6.2菌落蓝白色鉴定 将缺失株C50041 ΔsifA与接合转移后的重组沙门菌C50041(pGMB152-ΔsifA)在同一块含X-gal、IPTG的LB平板上划线进行蓝白斑鉴定。
1.6.3PCR鉴定 在缺失株的构建中主要通过菌落PCR进行鉴定,挑取一个单菌落于50 μL的灭菌SW中混匀,此即进行菌落PCR验证的模板。 PCR扩增体系(25 μL):2×Taq mix 12.5 μL,灭菌SW 9.5 μL,引物F 1 μL,引物R 1 μL,模板 1 μL。反应条件为:94 ℃ 5 min;94 ℃ 45 s,63 ℃ 45 s,72 ℃ 45 s,30个循环;72 ℃ 10 min。PCR结束后,通过0.7% 琼脂糖凝胶电泳鉴定PCR产物。
1.7 生物学特性鉴定
1.7.1生长特性鉴定 挑取C50041、C50041-ΔsifA及C50041ΔspiCΔsifA(构建方法与C50041ΔsifA单基因缺失株构建方法相同)单菌落接种于LB液体培养基培养过夜,次日分别转接到20 mL的LB液体培养基中并用培养基调节OD600=0.05。在37 ℃ 180 r/min的摇床中连续培养12 h,每隔1 h利用分光光度计测定各个菌液的OD600值,并绘制细菌生长曲线,以野生株C50041为对照,观察sifA基因对肠炎沙门菌生长速度的影响。
1.7.2生化特性鉴定 挑取新鲜的C50041、C50041ΔsifA及C50041ΔspiCΔsifA菌落于生理盐水中,利用比浊仪调整比浊度为0.8左右。参照使用说明书的要求向鉴定卡中加入菌液,将API 20E生化鉴定板置于37 ℃培养箱培养24 h后观察并记录结果。以野生株C50041为对照,观察sifA基因对肠炎沙门菌生化特性的影响。
1.8 C50041ΔsifA在巨噬胞内增殖
1.8.1巨噬细胞培养 用含10%的FBS、1%青链霉素的DMEM培养基于37 ℃含5% CO2的条件下培养鼠源巨噬细胞RAW264.7。在感染细胞前,用0.25%胰酶消化细胞1 min,换用含10% FBS无抗生素的DMEM培养基吹打悬浮细胞,活细胞计数后将细胞转移到24孔板中继续培养,细胞接种量为4.0×105CFU/孔,培养18 h待细胞长满细胞培养孔底时,进行沙门菌感染。
1.8.2沙门菌感染巨噬细胞 挑取C50041、C50041ΔspiC、C50041ΔsifA及C50041ΔspiCΔsifA单菌落接种于LB液体培养基中,37 ℃ 180 r/min的条件下培养过夜,用灭菌PBS洗2遍后将细菌浓度调整至OD600=1.0。过夜培养的24孔细胞板中的鼠源巨噬细胞换用新的含血清但无抗生素的培养基,每孔细胞加入100 μL的菌液进行感染,每个菌株做3个重复,加完细菌后将细胞板以1 000 r/min的转速离心10 min,使细菌与巨噬细胞充分接触,加速细菌黏附;离心后将细胞板于细胞培养箱放置30 min,此阶段为细菌黏附细胞的过程;弃掉上清培养基,用PBS洗2遍后加入含100 μg/mL庆大霉素、10% FBS的培养基,杀死未进入细胞的细菌,并将细胞板置于细胞培养箱培养1 h,此时为细菌的侵袭过程;再次弃掉上清,用PBS洗2遍后加入含10 μg/mL庆大霉素、10% FBS的培养基于细胞培养箱继续培养。加入含较低浓度的庆大霉素的培养基时为增殖时间点的零点,分别取增殖0 h、8.5 h、20 h、24 h的时间点,用0.2% Triton X-100溶液裂解细胞而释放出胞内的沙门菌,并进行细菌计数。以0 h时的细菌量为初始值。
1.9sifA基因的表达
1.9.1胞外菌sifA基因的表达 过夜震摇培养的肠炎沙门菌C50041、C50041ΔspiC、C50041ΔsifA和C50041ΔspiCΔsifA1∶50扩大培养至20 mL的LB液体培养基中,震摇培养至OD600=0.65左右,5 000 r/min离心10 min弃上清,加入1.5 mL PBS和3 mL的RNA保护剂重悬混匀后5 000 r/min离心10 min,弃上清收集细菌;再利用RNA提取试剂盒提取RNA,利用反转录试剂盒进行反转录,以反转录所得cDNA为模板进行荧光定量PCR,测定sifA基因表达量的荧光定量PCR引物为nsifA-F/R,以16S RNA引物16S-F/R为内参引物,将野生株C50041的sifA基因表达量定为1。荧光定量PCR体系(20 μL):FastStart Universal SYBR Green Master(ROX)10 μL,引物F(10 μmol/L)0.8 μL,引物R(10 μmol/L) 0.8 μL,模板(cDNA) 2 μL,RNase free Water 6.4 μL。反应条件为:95 ℃ 30 s;95 ℃ 5 s,60 ℃ 34 s,40个循环。
1.9.2胞内沙门菌sifA基因的表达 选择肠炎沙门菌C50041、C50041ΔspiC、C50041ΔsifA和C50041ΔspiCΔsifA在鼠源巨噬细胞内增殖1 h、8.5 h、19 h、20 h的时间点,收集细胞提取RNA。沙门菌在胞内增殖一定时间后弃掉细胞培养基,用PBS洗2遍后每孔加入1 mL的细胞RNA保护剂,移液枪反复吹打混匀细胞,细胞悬浮后将所有液体转移到去RNA酶的1.5 mL的离心管中,每个菌每个时间点做3个重复。然后利用RNA提取试剂盒提取细胞内的RNA,并用反转录试剂盒进行反转录得到cDNA,此即用于荧光定量PCR的模板。荧光定量PCR体系和程序与胞外sifA基因表达的荧光定量PCR的体系、程序相同。通过荧光定量PCR测定各个时间点的细胞内sifA基因的表达情况。
1.10数据分析 使用GraphPad Prism 8.0.1软件对实验数据进行统计与整理,并利用该软件制图。检验方法采用t-test检验,检验水准为ɑ=0.05。
2 结 果
2.1C50041ΔsifA基因缺失株鉴定
2.1.1重组自杀质粒的构建及鉴定 PCR扩增sifA基因上下游同源片段,利用一步连接法试剂盒将sifA基因上下游同源片段连接到SalⅠ酶切的自杀质粒pGMB152载体上,形成重组自杀质粒pGMB152-ΔsifA,转化E.coli. Spy372感受态细菌,构建重组菌E.coli. Spy37(pGMB152-ΔsifA)。获得的重组质粒经sifA-U-F/R和sifA-D-F/R引物对扩增,扩增条带分别为620 bp和730 bp,大小正确(图1)。引物PGMB152-F/R进行菌落PCR验证,扩增条带约为1 632 bp(图2),与预期结果一致。
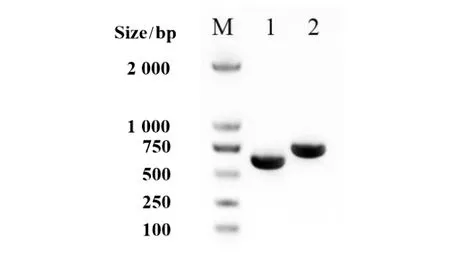
M:DL2000 Maker;1:sifA-U同源片段;2:sifA-D同源片段图1 重组质粒中sifA基因上下游同源片段的PCR验证Fig.1 PCR identification of sifA-U and sifA-D of pGMB152-ΔsifA
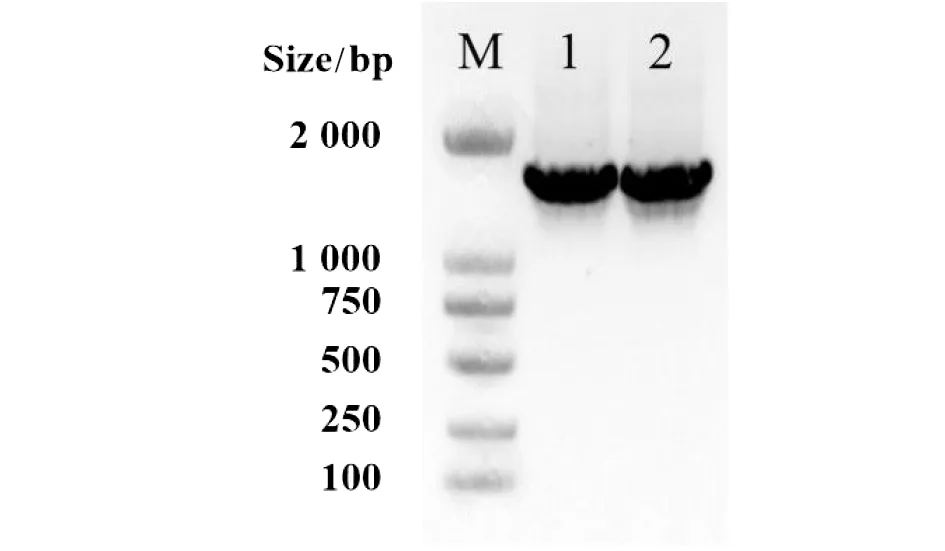
M:DL2000 Maker;1:E.coli. Spy372(pGMB152-ΔsifA);2:E.coli. χ7213(pGMB152-ΔsifA)图2 载体特异引物pGMB152-F/R PCR验证重组菌中pGMB152-ΔsifAFig.2 PCR identification of pGMB152-ΔsifA by pGMB152-F/R
2.1.2构建供体重组菌E.coli. χ7213(pGMB152-ΔsifA) 从重组菌E.coli. Spy372(pGMB152-ΔsifA)提取质粒pGMB152-ΔsifA,再转化E.coli. χ7213感受态细菌中构建重组菌E.coli. χ7213(pGMB152-ΔsifA),利用引物PGMB152-F/R进行菌落PCR验证,扩增出预期条带(图2)。该菌具有DAP生长依赖性,以作供体菌,便于与受体菌C50041进行接合转移和筛选。
2.1.3重组沙门菌C50041(ΔsifA)构建与鉴定 以重组菌E.coli. χ7213(pGMB152-ΔsifA)为供体与C50041受体进行接合转移,利用DAP生长依赖性去除供体菌E.coli. χ7213(pGMB152-ΔsifA),抗生素去除受体菌C50041,以及LacZ特性筛选蓝色重组沙门菌C50041(pGMB152-ΔsifA),重组质粒pGMB152-ΔsifA进入C50041后进行上下游同源片段交叉互换,形成蓝色重组沙门菌C50041(pGMB152-ΔsifA)。
自杀质粒pGMB152的复制需要一种特定的蛋白,而肠炎沙门菌C50041中不含该蛋白,所以pGMB152质粒在C50041中无法复制。用不含NaCl含10%蔗糖的LB液体培养基传代后在含X-gal、IPTG的LB平板上的白色菌落有两种可能:一是同源片段双交换,sifA基因缺失的菌株,另一种则可能是同源片段不交换,目的基因片段与pGMB152质粒一起丢失而回复为野生株。在传代过程中利用引物sifA-U-F/sifA-D-R进行菌落PCR验证。经过两次脱质粒,最终获得质粒脱除的sifA基因缺失株C50041ΔsifA。并分别用sifA-U-F/sifA-D-R和PGMB152-F/R的引物进行了PCR验证,证明其正确性,结果如图3、图4。利用sifA-U-F/sifA-D-R的引物扩增条带大小与预期结果一致,且PGMB152-F/R的引物未扩增出条带证明PGMB152质粒已脱除,C50041ΔsifA构建成功。

M:DL2000;1:C50041(pGMB152-ΔsifA);2:C50041ΔsifA; 3: 野生株C50041图3 引物sifA-U-F/sifA-D-R PCR重组沙门菌结果Fig.3 PCR identification of the recombinant SE strains with primer sets sifA-U-F/sifA-D-R

M:DL2000;1:C50041(pGMB152-ΔsifA);2:C50041ΔsifA;3:野生株C50041图4 引物pGMB152-F/R PCR重组沙门菌结果Fig.4 PCR identification of the recombinant C50041-ΔsifA with primer pGMB152-F/R
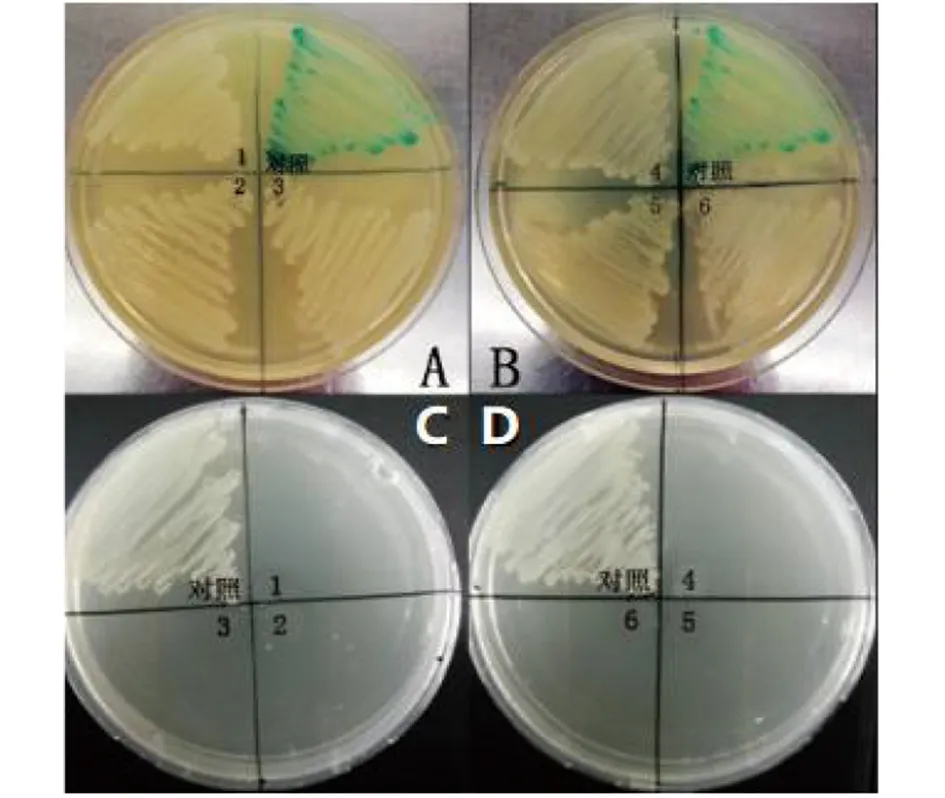
A、B:6株sifA基因缺失株的蓝白斑验证;C、D:6株sifA基因缺失株的抗生素耐药性验证对照:重组沙门菌C50041 (pGMB152-ΔsifA);1-6:6株sifA基因缺失株图5 C50041ΔsifA蓝白斑验证和抗生素耐药性分析Fig.5 Analysis of blue/white and antibiotic resistances of C50041ΔsifA strains
2.1.4C50041ΔsifA缺失株的抗生素耐药性和蓝白斑表型验证 未脱除质粒的重组菌中含有lacZ基因和氨苄青霉素、链霉素抗性基因,在IPTG的诱导下它能够利用X-gal产生蓝色物质,表现为蓝色菌落,也可以在含Amp、Sm的平板上正常生长;而缺失株 C50041ΔsifA完全脱除质粒后,失去pGMB152质粒所携带的lacZ基因和抗性基因,所以在含X-gal、IPTG的平板上表现为白色菌落,在含Amp、Sm的平板上不能正常生长。验证结果如图5,缺失株C50041 ΔsifA在含X-gal、IPTG的平板上表现为白色菌落,在含Amp、Sm的平板上不能生长,进一步说明自杀质粒已完全脱除。各菌株筛选特性见表3。
表3 沙门菌sifA基因敲除过程中各菌株筛选特性
Tab.3 Characteristics of bacterial strains during C50041ΔsifAconstruction

BacteriaCharacteristicsblue/whiteSmrAmprDAP dependenceC50041白无无无E.coli. Spy372(pGMB152-ΔsifA)蓝有有无E.coli. χ7213 (pGMB152-ΔsifA)蓝有有有C50041 (pGMB152-ΔsifA)蓝有有无C50041 ΔsifA白无无无
2.2C50041ΔsifA的生物学特性
2.2.1C50041ΔsifA的生长速度 调节C50041、C50041ΔsifA、C50041ΔspiCΔsifA初始菌液浓度均为OD600=0.05,每隔1 h测一次OD600的值,绘制生长曲线如图6。由此图可以看出缺失株的生长曲线与野生株基本保持一致,说明sifA基因缺失没有改变其生长速度。
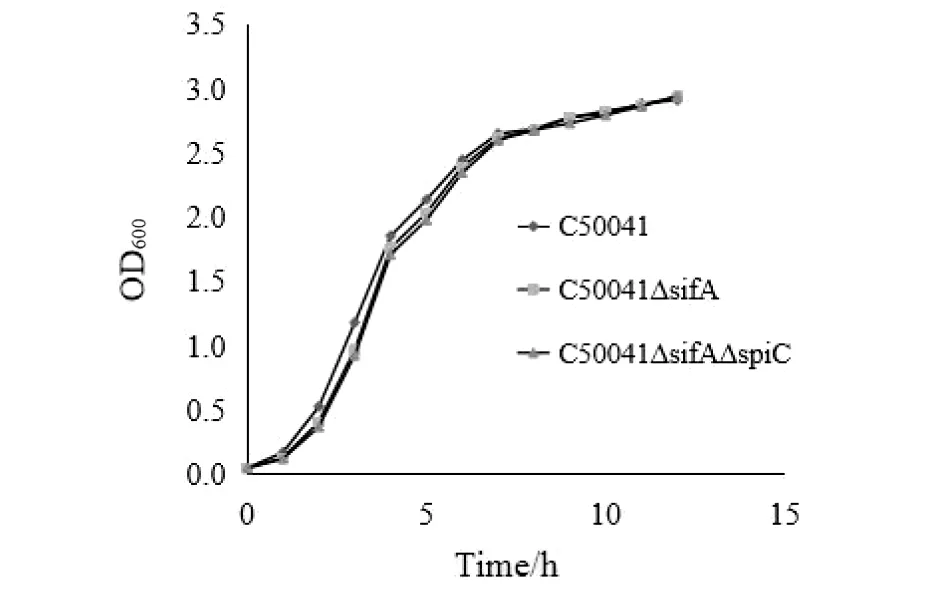
图6 C50041、C50041ΔsifA和C50041ΔsifAΔspiC的生长曲线Fig.6 Growth curves of C50041, C50041ΔsifA and C50041ΔsifAΔspiC
2.2.2C50041ΔsifA的生化特性 通过API 20E生化鉴定卡进行生化鉴定,鉴定了半乳糖苷酶(ONPE)(-),精氨酸双水解酶(ADH)(-),尿素酶(URE)(-),色氨酸脱氨酶(TDA)(-),吲哚产生(IND)(-),乙酰甲基甲醇(VP)(-),明胶酶(GEL)(-),肌醇发酵(INO)(-),蔗糖发酵(SAC)(-),苦杏仁苷发酵(AMY)(-),赖氨酸脱羧酶试验(LDC)(+),鸟氨酸脱羧酶(ODC)(+),柠檬酸利用(CIT)(+),硫化氢产生(H2S)(+),葡萄糖发酵(GLU)(+),甘露醇发酵(MAN)(+),山梨醇发酵(SOR)(+),鼠李糖发酵(RHA)(+),密二糖发酵(MEL)(+),阿拉伯糖发酵(ARA)(+)等20个生化特性,C50041、C50041ΔsifA和C50041-ΔsifAΔspiC的这些生化特性完全一致,所以sifA基因的缺失未改变肠炎沙门菌的生化特性。(“+”代表阳性,“-”代表阴性)。
2.3C50041ΔsifA在巨噬细胞内增殖情况 分别取增殖的第0 h、8.5 h和24 h的时间点,将细胞裂解后进行胞内沙门菌计数,以0 h的细菌数量为初始值,比较胞内沙门菌的增长量。结果发现spiC的缺失提高肠炎沙门菌在巨噬细胞内的增殖能力(t=28.64,P<0.001),sifA的缺失降低肠炎沙门菌在巨噬细胞内的胞内增殖能力(ΔsifAvsWT,t=3.077,P<0.05;ΔspiCvsΔspiCΔsifA(t=29.66,P<0.001)。结果如图7。

图7 肠炎沙门菌在巨噬细胞内的增殖情况Fig.7 The proliferation of intracellular SE strains
2.4肠炎沙门菌sifA基因的表达情况
2.4.1sifA基因在胞外沙门菌中表达 提取细菌mRNA并反转录进行荧光定量PCR,将野生株C50041的sifA基因的表达量设置为1,结果显示sifA基因在spiC缺失株中表达量显著上升(t=4.432,P<0.05)。结果如图8。

图8 LB培养肠炎沙门菌sifA基因相对表达量Fig.8 mRNA level of sifA gene of SE in the LB media by qPCR
2.4.2sifA基因在胞内沙门菌中表达 选取细菌在巨噬细胞内增殖1 h、8.5 h和19 h的时间点提取mRNA反转录,进行荧光定量PCR,结果同样证明spiC基因缺失能上调sifA基因的表达,尤其是在沙门菌感染巨噬细胞8.5 h和19 h显著上升(t=29.80,P<0.01)(t=6.007,P<0.01)。结果如图9。
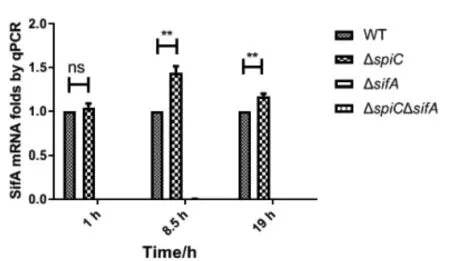
图9 感染巨噬细胞中肠炎沙门菌sifA基因表达量Fig.9 mRNA level of sifA gene of SE in macrophage by qPCR
3 讨 论
沙门菌是一种重要的人兽共患病病原菌,严重危害动物,威胁人的健康,是引起突发性食物中毒事件的重要病原菌之一,也是导致食源性疾病暴发最主要的原因[14]。2013年,27个欧盟(EU)成员国报告了82 694例沙门菌病例,每10万人有20.4例感染者。其中两种最常见的沙门菌血清型是肠炎沙门菌和鼠伤寒沙门菌,分别占所有报告与确诊人类病例相关的血清型的39.5%和20.2%,肠炎沙门菌感染病例超越了鼠伤寒沙门菌[15]。在我国肠炎沙门菌也是引起沙门菌食物中毒最常见的血清型之一[16]。但目前国内外对于肠炎沙门菌致病机制的研究较少,其致病机制主要参照模式菌—鼠伤寒沙门菌。
在鼠伤寒沙门菌致病机制研究中,沙门菌进入宿主细胞内部,能有效躲避细胞的杀菌作用,然后在细胞内生存繁殖,这是导致其持续感染和免疫逃逸的一个重要原因。
沙门菌侵入宿主细胞后,能重塑宿主细胞内吞区室而形成一个特殊的结构—沙门菌囊泡,沙门菌在SCV内生存并复制,使沙门菌可以获得宿主的膜和所需营养物质,还能保护病原菌免受细胞溶质杀菌应激反应的影响,确保其在细胞内正常生存和复制[9]。
沙门菌侵入细胞并在细胞内增殖是由多个沙门菌毒力岛(SPI)介导完成的,其中SPI1和SPI2是最重要的,分别表达III型分泌系统T3SS1和T3SS2,它们作为分子注射器能将40多种效应蛋白注入宿主细胞胞质中[5]。当沙门菌接触到细胞膜时,T3SS1分泌效应蛋白能作用于宿主细胞质膜而引起细胞骨架的重排,使细菌侵袭进入细胞[17];沙门菌进入细胞后,通过修饰吞噬小体并改变吞噬小体的成熟过程形成SCV来保护自身不被降解。囊泡中沙门菌的T3SS2被激活分泌30多种作用于囊泡膜的效应蛋白,来促进SCV成熟、SIF形成和囊泡内沙门菌复制。细菌在细胞内的复制开始于感染细胞后的2~3 h,在这段时间里,沙门菌会不断地调节SCV中的环境来使自己得以生存。
细胞中SCV的成熟过程同样遵循内体从早期至晚期的成熟途径。SCV的形成可以分为3个阶段:感染细胞后10~60 min为早期SCV形成,T3SS1效应蛋白SopA、SopB/SigD、SipA/SspA、SopD和SopE参与;感染1~4 h时形成中期SCV,此过程不仅需要T3SS1效应蛋白SipA和SopB的参与,还需要T3SS2效应蛋白SifA、SseG和SseF的参与;感染4 h以后为SCV形成后期,T3SS2效应蛋白PipB2、SifA、SifB、SopD2、SseF、SseG、SseJ和SpvB参与此过程[18]。SifA蛋白主要参与SCV形成的中晚期。
在SCV形成的早期阶段表达的T3SS2效应蛋白是SpiC,它是第一个被鉴定的T3SS2效应蛋白,它在沙门菌感染细胞后的0.5~1 h内表达,并可以抑制SCV与溶酶体的融合,它的缺失会明显降低沙门菌的毒力[19]。SCV移动至核区时即SCV形成中期,T3SS2的效应蛋白SseG和SseF相互作用,诱导细胞内晚期内体再分配维持SCV的稳定性并与SifA蛋白协同作用,改变SCV的运输路径促使SCV进入成熟期[20]。在SCV成熟期,SifA蛋白通过抑制甘露醇-6-磷酸受体的招募来阻止溶酶体对沙门菌的清除[7],sifA基因缺失可明显减弱沙门菌的毒力[21]。SifA蛋白的形成依赖于sifA基因,它通过与宿主蛋白SKIP相互作用,下调SCV对微管驱动蛋白的招募,若sifA基因缺失则对SCV的形成造成影响,使沙门菌在细胞内不能正常复制,所以sifA基因是影响沙门菌在细胞内增殖的一个重要基因。
本研究利用同源重组的原理,通过一步法连接、颜色辅助筛选等改进方法,构建了肠炎沙门菌C50041株的sifA基因缺失株C50041ΔsifA、双基因缺失株C50041ΔsifAΔspiC,该方法简化了沙门菌基因敲除过程,加快了实验进程。基因的无痕敲除则避免残留碱基可能导致的不可预期的结果。各菌株的生长生化特性保持一致,表明sifA基因的缺失对其生物学特性并无影响。肠炎沙门菌在鼠源巨噬细胞内增殖的结果表明,与野生株相比,sifA基因缺失株巨噬细胞内沙门菌的数量显著减少,并且能降低C50041ΔspiC在细胞内的增殖量,这说明sifA基因缺失能有效降低胞内沙门菌的增殖。通过荧光定量PCR结果显示无论是在细胞内还是细胞外,spiC基因缺失后sifA基因的表达量上调,可能导致产生更多的SCV丝状物,以吸收更多的营养[22],该结果部分解释了肠炎沙门菌spiC基因缺失株在巨噬细胞内超级复制增殖的现象。该研究为进一步研究肠炎沙门菌sifA基因功能及SCV的形成规律奠定基础。
利益冲突:无
