基于线粒体COI基因的DNA条形码技术对小叶蝉亚科昆虫种类的分子鉴定
2020-04-23高娅蓉熊康宁袁周伟苑晓伟陈晓晓宋月华
高娅蓉,熊康宁,袁周伟,苑晓伟,谭 超,陈晓晓,宋月华
(贵州师范大学 喀斯特研究院/国家喀斯特石漠化防治工程技术研究中心,贵州 贵阳 550001)
【研究意义】DNA条形码 (DNA Barcoding) 技术是昆虫基因组中一段被公认的、相对较短的DNA序列进行物种鉴定的一种分子生物学技术,可以快速、准确鉴定物种,对物种分类和系统发育研究有着重要意义[1]。种属的分类鉴定是小叶蝉亚科昆虫研究的重要内容,受形态学鉴定与传统分类的局限,叶蝉分类学的发展急需新技术的支撑[2]。DNA条形码的分子分类方法能够弥补传统形态学分类的缺憾[3]。【前人研究进展】基于线粒体DNA COI基因的条形码技术的物种鉴定手段在其他类昆虫鉴定中已有广泛应用[4-7]。张媛等[8]对鞘翅目昆虫COI基因的序列分析和系统进化树的构建指出,DNA条形码技术在鞘翅目昆虫分子系统发育研究中具有重要的应用价值。戴仁怀等[9]基于线粒体COI基因对中国常见的广头叶蝉亚科种类进行系统发育研究,为物种快速鉴定打下了基础。胡泽章等[10]研究证明,运用DNA条形码技术对小花蝽属昆虫进行物种快速分子鉴定是可行的。【本研究切入点】应用于DNA条形码工作中的基因很多,但线粒体细胞色素C氧化酶亚基基因(COI)拥有相对保守性的特点,种间变异较大,合适的序列长度能提供丰富的系统发育信息,是目前测序最多、结构和进化动力学研究较清楚的基因,已被广泛应用于近缘种间的系统分类研究[11]。小叶蝉亚科为叶蝉科第二大亚科,种类很多,而在小叶蝉亚科的低级阶元,近缘种普遍存在,根据传统分类学方法很难区分,采取新的特征和手段,可为近似种的鉴定提供更为可靠的证据。【拟解决的关键问题】应用DNA条形码技术对小叶蝉亚科物种进行鉴定,旨在弥补小叶蝉亚科传统分类方法的不足[12],为近似种难以从形态上鉴定区分的问题提供更为可靠的依据[13],该鉴定方法快速、方便且准确,对物种精确鉴定、生物多样性评估和系统发育关系方面的研究具有重要意义和科学价值[14]。为能快速、准确地鉴定小叶蝉亚科种类,通过PCR扩增对小叶蝉亚科5个族的线粒体COI基因片段序列进行测序和比对,然后将同源序列的碱基多样性及系统进化关系进行分析[15],探讨线粒体COI基因片段作为DNA条形码准确鉴定小叶蝉亚科昆虫种类的可行性,以期为更进一步研究小叶蝉亚科昆虫分子鉴定技术提供理论依据和实践基础。
1 材料与方法
1.1 材料
叶蝉标本于2017年8月和2018年6月在贵州梵净山国家自然保护区采集,浸泡于无水乙醇中,保存于贵州师范大学喀斯特研究院6楼实验室,-20 ℃冰箱备用。选取的2种标本类型分别编号为YXYC(眼小叶蝉族Alebrini)与 XLYC(小绿叶蝉族Empoascinisp.)。对比COI基因序列源于从基因库GenBank中下载的20种小叶蝉亚科昆虫COI基因序列(表1)。
动物组织快速提取试剂盒 (VWI)Easy Pure Genomic DNA kit,北京全式金生物技术有限公司。
1.2 方法
1.2.1 总DNA提取 取出无水乙醇保存的叶蝉标本,除去腹部和翅膀,然后将其余的动物组织使用无菌研磨棒将其磨成粉末状置于无菌的1.5 mL离心管中,然后按照动物组织快速提取试剂盒说明书提取总DNA,检测样品的浓度和纯度后马上保存于-20 ℃冰箱备用。
1.2.2 COI基因扩增及测序 ①引物合成。通过文献查找筛选适合的COI通用引物[16],引物均由上海生物工程有限公司合成。COI引物分为COIF(上游引物):GGTCAACAAATCATAAAGATATTGG;COIR(下游引物):TAAACTTCAGGGTGACCAAAAAATCA。②PCR反应体系。2×EasyTaqPCR SuperMix(+dye)25 μl,模板DNA 2 μl,上下游引物各1 μl,加ddH2O 21 μl,总共50 μl。PCR主要反应过程:94 ℃预变性3 min;94 ℃变性30 s,56 ℃退火30 s,72 ℃延伸1 min,共有34个循环;最后72 ℃延伸10 min。PCR产物通过琼脂糖凝胶电泳检测合格后由上海生物工程有限公司进行直接测序。
1.2.3 序列处理与分析 测序后得到的结果使用Chromas进行读取与确认。运用MEGA6.0进行序列的拼接与手工校正,确定分析的序列[17]。利用NCBI中的BLAST进行相似性检索,确定序列方向及目的片段;将确定的序列及Genbank中下载的20种小叶蝉的基因序列载入Clustal X 1.83[18]进行序列比对,输出格式为FASTA。比对结果导入MEGA6.0[19]计算各物种间的遗传距离[20],转换和颠换值及其比值(R值),保守位点(Conserved sites,C)及变异位点(Variable sites,V)等数值[21]。基于MEGA6.0运用邻接法(Neighbor-Joining, NJ)与Kimura 2-parameter遗传距离模型[22]分析20种小叶蝉物种同2个外群物种YXYC(眼小叶蝉族)与XLYC(小绿叶蝉族)间的遗传距离和采用Bootstrap重复抽样1000次用以检验分子系统树每个分支的置信度,构建系统发育树。
2 结果与分析
2.1 COI基因序列组成与变异
叶蝉测序得到的结果及基因库下载的相关小叶蝉序列运用MEGA6.0拼接剪切成统一长度(646 bp)的片段,没有出现缺失和插入的情况。变异位点为512个,保守位点134个,简约信息位点415个,自裔位点97个。整个序列结构中A、G、C和T碱基组成的含量分别为27.6 %、15.5 %、15.3 %和41.6 %。A+T含量为69.2 %,明显高于G+C含量,呈现出明显的A+T碱基偏好,满足昆虫线粒体基因碱基组成的基本特征[23]。

表1 小叶蝉亚科线粒体COI基因片段信息
2.2 碱基替换
从表2看出,全部位点的转换产生于T和C间,颠换主要发生在A与T间,转换/颠换比值(R)为0.63。密码子位点分析,转换与颠换主要产生在密码子第3位点,R为0.81,转换与颠换数值邻近,符合昆虫线粒体特别属性,替换主要产生在密码子第3位点。第1位点、2位点和第3位点的R分别为0.58、0.57和0.81。整体与密码子各位点的R均小于2,表明该段序列的转换与颠换没有达到饱和,一般在建立系统发育树时需要考虑转换与颠换的发生比率;说明该段基因准确度高,运用该基因得到的进化树是具有准确性的。
2.3 遗传距离
从表3看出,小绿叶蝉族叶蝉物种间的遗传距离为0.021~0.284,眼小叶蝉族间的种内改为间遗传距离删除相差最大,为2.57。不同叶蝉物种间的遗传距离在0.292~6.183,物种间的平均遗传距离为0.204,其中近缘种叉脉叶蝉族与小叶蝉亚科间的遗传距离最小,为0.017;蛇葡萄斑叶蝉和眼小叶蝉族间的遗传距离最大,为7.491。由此可知,相同叶蝉物种间不受地理位置的影响;叶蝉同一属种的种间遗传距离差异较明显,将此段序列运用于分析和鉴别物种的依据具有可行性。
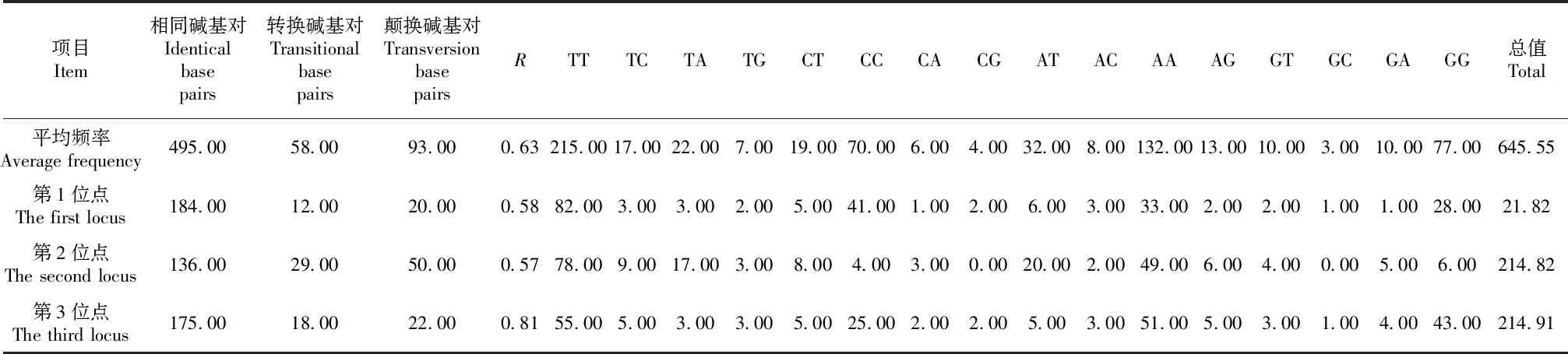
表2 小叶蝉亚科的核苷酸碱基替换值

表3 22种小叶蝉种内的种间遗传距离及标准误差
注:对角线以下为遗传距离, 对角线以上为标准误差。1,眼小叶蝉族;2,叉脉叶蝉族;3,凯小绿叶蝉;4,马铃薯小绿叶蝉;5,小叶蝉亚科;6,茶小绿叶蝉;7,小叶蝉亚科;8,叉脉叶蝉族;9,假眼小绿叶蝉;10,蛇葡萄斑叶蝉;11,小叶蝉亚科;12,小叶蝉亚科; 13,小绿叶蝉属;14,小绿叶蝉属;15,小绿叶蝉属;16,切尔塔斑叶蝉;17,小绿叶蝉属;18,蛇葡萄斑叶蝉;19,小绿叶蝉属;20,小绿叶蝉属;21,番木瓜小绿叶蝉;22,小绿叶蝉属。
Note:The values below the diagonal and above the diagonal are genetic distance and standard error respectively.. 1,Alebrini;2,Dikraneurini; 3,Empoascakaicola; 4,Empoascafabae; 5, Typhlocybinae; 6,Empoascaonukii; 7, Typhlocybinae; 8,Dikraneurasp.; 9,Empoascavitis; 10,Erythroneuratricincta; 11, Typhlocybinae; 12, Typhlocybinae ; 13,Empoascasp.; 14,Empoascasp.; 15,Empoascasp.; 16,Erythroneuracerta; 17,Empoascasp.; 18,Erythroneuratricinct; 19,Empoascasp.; 20,Empoascasp.; 21,Empoascapapaya; 22,Empoascasp.
2.4 系统发育树的构建
从图1看出,两种外群YXYC(眼小叶蝉族Alebrini) 与XLYC(小绿叶蝉族Empoascinisp.)位于树的基部,说明,叉脉叶蝉族和眼小叶蝉族种类亲缘关系较近且较原始,这与形态学分类相一致。与内群其他小叶蝉亚科种类分开。不同两个外群种均属小叶蝉亚科,聚为同一支,形成姐妹群,且分支自展值均为 100 %;在内群中,近缘种能聚集在一起,如MF931089和KR567994两个未分类的小叶蝉种类,其置信度也很高(≥97 %)。同族间的KJ867505和KJ867506亲缘关系最相近,不同种的KR564575和KR570673差异明显。总的来说,叶蝉种类间的亲缘关系与形态学的划分基本一致。
3 讨 论
对小叶蝉亚科的COI基因序列进行拼接与手工校正,并采用基因库中Blast进行序列的相似性分析结果表明,所测序列都属于小叶蝉亚科,COI基因序列且同源性均在98 %以上,说明此段序列准确性强、可信度高。同时,在小叶蝉亚科中,眼小叶蝉族和叉脉叶蝉族的亲缘关系较近,而小绿叶蝉族和斑叶蝉族的亲缘关系较远。以线粒体基因COI序列信息为基础的DNA条形码技术能有效地对形态特征变异丰富的小叶蝉亚科昆虫进行物种鉴定[24]。在此基础上,采用Kimura 2-parameter 双参数模型建立而成的NJ树,相同叶蝉物种类型聚为同一小支,且分支自展值为100 %;叶蝉物种间的近缘种聚集在一起,置信度相对较高(≥97 %);不同叶蝉物种间差异表现较明显,置信度较高,系统进化树得出的结果与叶蝉的形态学分类结果基本一致,可以满足叶蝉种类分类鉴定的需要。基于线粒体COI基因的DNA条形码技术是可以作为小叶蝉亚科物种鉴定的有利工具,对小叶蝉亚科昆虫分子鉴定具有可行性。
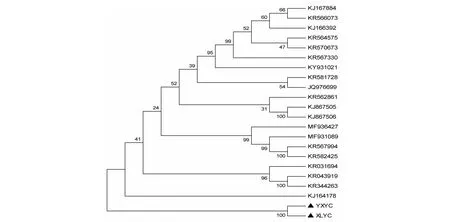
分支处上方数值表示重复1000次后的自展值The value above branches is bootstrap value (1000 replicates)
4 结 论
叶蝉测序得到的结果及基因库下载的相关小叶蝉序列运用MEGA6.0拼接剪切成统一长度(646 bp)的片段,没有出现缺失和插入的情况。变异位点为512个,保守位点134个,简约信息位点415个,自裔位点97个。整个序列结构中A、G、C和T碱基组成的含量分别为27.6 %、15.5 %、15.3 %和41.6 %。A+T含量为69.2 %,明显高于G+C含量,呈现出明显的A+T碱基偏好,符合昆虫线粒体基因碱基组成的基本特征。转换与颠换结果显示:该段序列没有饱和,可以得到准确的进化分析。同物种间与不同物种间的遗传距离分别为0.025~0.044和0.024~0.291,平均为0.358。基于COI基因序列构建的邻接法系统发育树(NJ树)显示,同一物种聚为同一小支,分支自展值均为100 %:近缘种能聚集在一起,且置信度很高(≥97 %)。结果表明,昆虫COI序列能够区分不同物种,COI基因片段的DNA条形码进行小叶蝉亚科昆虫分类鉴定具有可行性。
