兴安落叶松SSR反应体系的建立与优化
2020-04-09冷海楠张玉徐明仪王丽媛付晓玲崔福星杨立宾朱道光
冷海楠 张玉 徐明仪 王丽媛 付晓玲 崔福星 杨立宾 朱道光
摘要: 采用L16(45)正交试验设计,对影响兴安落叶松SSR-PCR 反应体系的主要因素(模板DNA、dNTPs、Mg2+、Taq聚合酶浓度及引物浓度)进行了试验分析,其结果表明:兴安落叶松SSR-PCR体积为20 μL的最佳反应体系是模板DNA用量50 ng、正反向引物浓度均为5μmol·L-1、dNTPs浓度为2.5 mmol·L-1、Mg2+的浓度为2.5 mmol·L-1、Taq聚合酶用量为1.0 U、最佳退火温度为60 ℃。试验所建立的反应体系可进一步用于兴安落叶松SSR遗传图谱构建、多样性分析、良种选育等方面的研究中。
关键词: 兴安落叶松; SSR-PCR; 正交优化试验
中图分类号: S 791. 222 文献标识码: A 文章编号:1001 - 9499(2020)01 - 0012 - 03
兴安落叶松(Larix gmelinii)是松科落叶松属植物,主要分布在我国的大、小兴安岭地区,是大兴安岭地区寒温带针叶林的主要建群种[ 1 ]。兴安落叶松具有成材快、适应能力强、耐寒耐湿等特点,因此对兴安落叶松开展研究具有很强的经济及生态意义。
SSR(Simple sequence repeats)分子标记是一种高效的分子生物学研究手段,凭借其共显性、多态性高、检测方法简单、准确率高等特点广泛应用于林木遗传育种、植物遗传图谱建立等研究中[ 2 - 6 ]。本研究利用正交设计试验对兴安落叶松SSR-PCR反应体系的各个参数进行优化,得出一套高效、可靠、稳定的反应体系,为兴安落叶松遗传多样性、良种选育等相关应用性研究奠定基础。
1 试验材料
试验材料选用大兴安岭地区呼中国家级自然保护区(122°42'14″ ~ 123°18'05″E,51°17'42″ ~
51°56'31″N)内的兴安落叶松。将采集的兴安落叶松鲜嫩针叶迅速保存于液氮罐中,实验室保存于-20 ℃冰箱中。
2 试验方法
2. 1 DNA的提取和检测
使用天根(TIANGEN)公司生产的DNA提取试剂盒(DP350)进行DNA提取,提取10组DNA作为样本;提取完成后,利用0.8%的琼脂糖凝胶电泳进行检测、核酸蛋白检测仪进行含量测定。
2. 2 SSR-PCR反应体系及程序
以提取得到的兴安落叶松DNA为模板,对模板DNA用量、正反向引物浓度、dNTPs浓度、Mg2+的浓度、Taq聚合酶用量5个因素进行正交设计试验,每个因素4个水平(表1),2个重复,共32个处理。反应程序设置为:94 ℃预变性,5 min;94 ℃变性,35 s;60 ℃退火35 s;72 ℃延伸 1 min;35个循环;72 ℃再延伸7 min;琼脂糖凝胶电泳进行检测。
2. 3 反应体系验证
以不同样本提取的DNA为模板DNA进行PCR试验,使用1.0%琼脂糖凝胶电泳对扩增产物进行检测,以检验反应体系的稳定性。
表1 兴安落叶松反应体系的正交设计试验L16(45)
3 结果与分析
3. 1 核酸检测
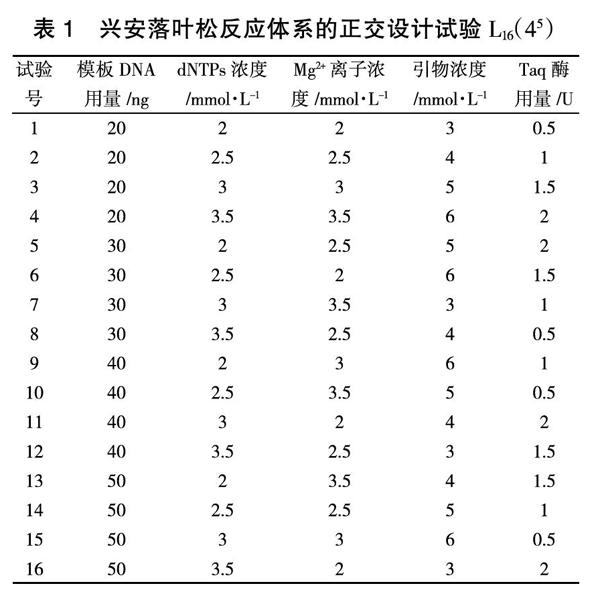
由总DNA提取电泳(图1)可知:电泳条带清晰明亮,无异物滞留在点样孔。核酸蛋白检测仪测定DNA的OD260/OD280值为2.0~2.5,浓度为150~ 240 ng/μL。由此可见,获得的DNA样本纯度高,几乎不含杂质,适合试验使用。
图1 总DNA提取电泳
3. 2 正交设计试验结果
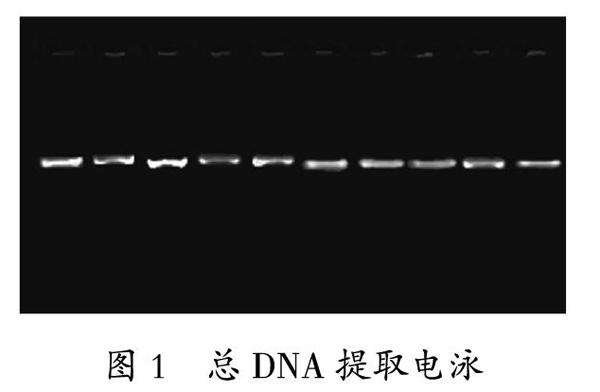
从SSR-PCR正交试验结果(图2)可以看出:不同因素、不同浓度组合所产生的扩增结果有明显差异。试验1、5、6、15的扩增条带显示不明显,其余12组都能扩增出清晰的条带,其中第14组在两次重复试验中都表现出主带明显、条带清晰的特征,可以确定为最优体系。
兴安落叶松SSR-PCR体积为20 μL的最佳反应体系是:模板DNA用量50 ng、正反向引物浓度均为5μmol·L-1、dNTPs浓度为2.5 mmol·L-1、Mg2+的浓度为2.5 mmol·L-1、Taq聚合酶用量为1.0 U。
3. 3 反应体系验证结果
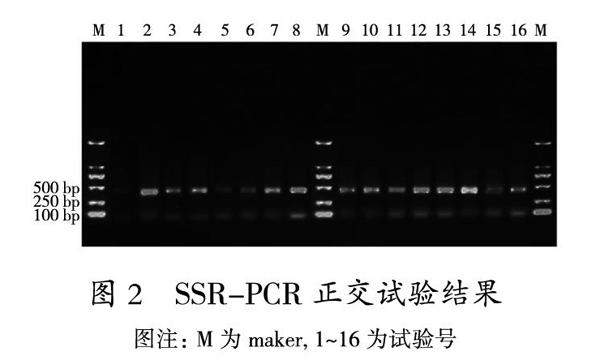
以提取10组兴安落叶松样本DNA作为模板,利用优化获得的反应体系进行SSR-PCR扩增(图3)得到了清晰的扩增条带。说明获得的反应体系稳定性高,可以用于日后的遗传多样性及分子育种研究。
图3 10个兴安落叶松样本的SSR-PCR扩增结果
4 结论与讨论
4. 1 在植物遗传多样性及林木遗传育种研究中,建立和优化出一套高效、稳定、可靠的SSR-PCR反应体系是SSR分子标记应用的基础[ 8 - 9 ]。近年来,分子生物学手段被广泛用于生态学的研究中,因此兴安落葉松SSR-PCR反应体系的建立不仅是研究兴安落叶松遗传多样性的研究基础,也将为分子生态学的研究奠定基础。
4. 2 本研究获得的最佳反应体系与其余松科植物相比,在DNA模板用量、dNTPs、Mg2+的浓度上虽然有差异,但差异并不大,而在引物浓度、Taq酶用量上却有比较大的差异[ 10 - 15 ]。这说明在整个SSR-PCR反应体系中,影响最终扩增结果的是多因素共同作用的结果。同科属树种由于是多个因子影响最佳反应体系,因此同科属树种的最佳反应体系多是不同的。
4. 3 本研究利用正交试验获得了兴安落叶松的最佳反应体系,为兴安落叶松遗传结构的分析、林木遗传育种研究以及分子生态学研究奠定了基础。
参考文献
[1] 刘世荣, 柴一新, 蔡体久, 等. 兴安落叶松人工群落生量物与净初级生产力的研究[J]. 东北林业大学学报, 1990, 18(2) :40 - 46.
[2] Li Y,Wang D W,Li Z Q,et al. A molecular genetic linkage map of Eucommia ulmoides and quantitative trait loci(QTL) analysis for growth traits[J]. International Journal of Molecular Sciences, 2014, 15(2) : 2 053 - 2 074.
[3] Sanchez M, Ingrouille M J,Cowan R S, et al. Spatial structure and genetic diversity of natural populations of the Caribbean pine, Pinus caribaea var.bahamensis(Pinaceae), in the Bahaman archipelago[J]. Botanical Journal of the Linnean Society,2014,17(3) : 359-383.
[4] 謝文刚, 张新全, 彭燕, 等. 鸭茅SSR-PCR反应体系优化及引物筛选[J].分子植物育种, 2008,6(2) : 381 - 386.
[5] 王志勇, 郭海林, 刘建秀. 正交设计优化狗牙根SSR-PCR反应体系[J]. 分子植物育种, 2007, 5(S1) : 201 - 206.
[6] Thao D V,Yamashita M,Watanabe A,et al.Development of tetranucleotide microsatellite markers in Pinus kesiya Royle ex Gordon[J].Conservation Genetics Resources,2012, 5(2) : 405 - 407.
[7] 张振. 兴安落叶松基本群体遗传多样性分析[J]. 东北林业大学, 2012
[8] 周静, 陈书霞, 程智慧, 等. 大蒜SSR体系的建立与优化[J]. 西北农业学报,2011, 20(11) : 117 - 122.
[9] 郭鑫, 倪州献, 罗建中, 等.桉树SSR-PCR体系优化及引物筛选应用[J]. 分子植物育种, 2017,15(2) : 626 - 632.
[10] 蔡娟娟, 季孔庶. 马尾松大配子体的SSR多样性及其遗传作图策略研究[J]. 分子植物育种, 2009, 7(5): 934 - 940.
[11] 杨娅. 利用微卫星(SSR)分子标记进行油松无性系种子园及其子代群体结构的研究[D]. 河南农业大学, 2006.
[12] 张冬梅, 杨娅, 沈熙环, 等. 油松SSR-PCR 引物筛选及反应体系的建立[J]. 北京林业大学学报, 2007, 29(2): 13 - 17.
[13] 张瑞丽, 许玉兰, 王大玮, 等. 云南松SSR-PCR反应体系的建立与优化[J]. 生物技术通报, 2012(4) : 93 - 97.
[14] 郭鑫, 倪州献, 罗建中, 等. 桉树SSR-PCR 体系优化及引物筛选应用[J]. 分子植物育种, 2017, 15(2) : 626 - 632.
[15] 方攀, 张运成. 樟子松SSR反应体系优化[J]. 基因组学与应用生物学. 2013, 32(3): 413 - 419.
第1作者简介: 冷海楠(1983-), 男, 助理研究员, 在读博士, 从事分子生态学研究。
收稿日期: 2019 - 11 - 01
(责任编辑: 李 丹)
Optimization of SSR-PCR Reaction System for Larix gmelinii
LENG Hainan
( Institute of Natural Resources and Ecology, HAS. National and Provincial Joint Engineering Laboratory of Wetlands and Ecological Conservation, Heilongjiang Harbin 150040)
Abstract The important basis of the researches on SSR marker assisting selective breeding of Larix gmelinii is to establish an SSR-PCR reaction system and get some polymorphism primers. In order to lay a foundation for the researches on applying SSR marker technology to establishment of genetic diversity and molecular breeding, some main impact factors in the Larix gmelinii SSR-PCR reaction system were analyzed by using combination method of L16(45) orthogonal test and single factor tests, including DNA templates,primer, dNTPs, Mg2+, Taq polymerase, and annealing temperature. The results showed that the optimized PCR reaction mixture was 20 μL total volume including 50ng template DNA, 5 μmol/L forward and reverse primers, 2.5 mmol/L dNTPs, 2.5 mmol/L Mg2+ and 1.0 U Taq DNA polymerase, and the optimum annealing temperature was 60℃. This result indicated that the optimized SSR-PCR reaction system could provide a vital theoretic and technical support for genetic maps establishment and molecular marker assisted breeding in Larix gmelinii.
Key words Larix gmelinii; SSR-PCR; Orthogonal optimization test
