悬钩子属DNA条形码通用序列的初步筛选
2020-04-08巫伟峰沈玺龙杨鼎元李永霞张群英
巫伟峰 沈玺龙 陈 哲 杨鼎元 李永霞 王 瑶 张群英*
(1.贵州省植物园,贵阳 550000; 2.中国科学院庐山植物园,九江 332900)
悬钩子属(Rubus)是蔷薇科(Rosaceae)的一个大属,又称树莓属,共1 000余种[1],主要分布在北半球温带地区,少数生长在热带和南半球。我国是悬钩子属植物野生资源非常丰富的国家,有194种,88变种,其中特有种138种[2],种类之多仅次于北美起源中心。在悬钩子属植物的遗传与繁殖过程中存在无融合生殖、多倍化、频繁杂交等现象,这导致其遗传背景复杂,物种的分类与鉴定困难较大[3]。目前,国际上悬钩子属的植物学分类以Focke[4~6]的12亚属划分最受认可。而在中国,Yu等将中国悬钩子属植物分为8组[7](多数与Focke系统的属相对应),并被广泛应用于我国悬钩子属植物的物种鉴定[3]。这两种分类系统都基于形态学差异,在实际应用中易受专业性强、植株生长环境及发育阶段影响等问题的限制,较大影响了悬钩子属植物物种鉴定效率。
近年新兴的DNA条形码技术,利用一个或几个标准DNA序列实现对物种的分类与鉴别[8]。因其简便、通用性高、稳定性好、不受生长环境和生长发育阶段的限制等特点被广泛用于分子系统学和物种鉴定[9~12]、生物多样性[13]和生态学[14]等研究,是传统鉴定方法的有效补充[15],这为悬钩子属植物的快速鉴定提供了可能。目前,在DNA条形码研究报道中应用较多的标准序列有ITS、ITS2、matK、psbA-trnH、rbcL、rpoC1等[16]。其中matK和rbcL在2009年被国际条形码协会植物工作组提出作为陆地植物通用的条形码序列,并建议将叶绿体基因片段psbA-trnH和核糖体DNA ITS作为补充条形码[17]。2011年,中国植物条形码研究组通过对1 757种植物的psbA-trnH、ITS/ITS2及rbcL+matK序列或序列组合进行鉴别能力评价,提出将ITS/ITS2序列作为种子植物的核心条形码,psbA-trnH作为辅助条形码序列[18]。在悬钩子属植物研究中,陈小露等利用ITS2序列对茅莓根及其混伪品进行了鉴定,成功区分了茅莓根与其混伪品[19]。Wang等将3个叶绿体基因组片段rbcL、rpl20-rps12和trnG-trnS,3个核基因组片段nrITS、GBSSI-2和PEPC共6个DNA片段组合重构了中国悬钩子属系统发育树[3]。吕群丹等基于ITS2序列对畲药搁公扭根及其同属易混种进行了分子鉴定,成功鉴别了搁公扭根基源植物及其9种同属易混种[20]。但迄今未见悬钩子属植物DNA条形码筛选的相关报道。
NCBI的GenBank数据库是国际上权威的序列数据库,其收集的数据具有来源广泛、认可度高、标准化等特点,是基因克隆与表达分析、分子标记挖掘、基因功能预测等生物学研究最常用的数据共享平台。其中也收集了大量物种的条形码序列,这为不同物种DNA条形码通用序列的初步筛选与评估提供了可靠的数据源,有利于缩减DNA条形码筛选周期、节约成本。本研究通过barcoding gap、遗传变异、建树等分析方法,基于GenBank数据初步评估了ITS、ITS2、matK、rbcL、trnH-psbA、trnL-trnF6个DNA条形码候选序列对悬钩子属植物的鉴别能力,旨在筛选获得适用于悬钩子属植物的DNA条形码通用序列,为悬钩子属植物的DNA条形码分子鉴定研究提供参考依据。
1 材料与方法
1.1 材料
从GenBank数据库中下载悬钩子属ITS、ITS2、matK、rbcL、trnH-psbA、rtnL-trnF相关序列。其中18种的ITS、ITS2序列各50条,15种的叶绿体matK序列45条,20种的叶绿体rbcL序列60条,11种的叶绿体trnH-psbA序列30条,14种的叶绿体rtnL-trnF序列42条。
1.2 数据分析
根据GenBank注释去除trnH-psbA和rtnL-trnF序列两端的编码区,及ITS序列两端的18S和26S区段。并基于隐马尔可夫模型HMMer真核生物注释方法,去除序列两端5.8S和26S区段获得ITS2间隔区序列[21]。matK、rbcL为部分编码序列。用ClustalX v2.1软件[22]对序列进行多序列比对,BioEdit软件[23]对比对结果校正切齐。用SpeciesIdentifier软件进行gap barcoding检验[24]。用MEGA X软件[25]基于K2P模型分别计算种内及种间遗传距离、序列变异信息,并使用邻接法(Neighbor-Joining,NJ)构建NJ一致树(空隙采用完全缺失处理,bootstrap进行1000次重复抽样检验获得分支支持率)。
2 结果与分析
2.1 序列特征
对6条候选序列分别进行序列分析(表1)。ITS、matK、rbcL3个候选条形码序列长度大于400 bp,ITS2、trnH-psbA、rtnL-trnF的序列长度小于400 bp。ITS、ITS2、rbcL的G+C含量介于40%~60%,matK、trnH-psbA、rtnL-trnF的GC含量小于40%,尤其是trnH-psbA的G+C含量介于22.1%~30.6%。变异位点占比最高的3个候选条形码依次为trnH-psbA、ITS2、ITS。
2.2 种间与种内遗传变异评估
由表2所示,6个候选条形码中trnH-psbA的总变异最高,远高于其他候选条形码,总变异高低次序为trnH-psbA>ITS2>ITS>matK>rtnL-trnF。6个候选条形码中种内变异与种间变异差异最为明显的是trnH-psbA,其次为matK、rbcL、rtnL-trnF。为了量化种间变异与种内变异的差异程度,本研究引入“变异分辨率”的概念,百分比越高说明种内变异与种间变异的差异越大,区分能力也越强。α表示变异分辨率(variation resolution),α=(种间变异-种内变异)/(种间变异+种内变异)。α的大小次序为trnH-psbA>matK>rbcL>rtnL-trnF>ITS>ITS2。6个候选条形码的种内最大变异均大于种间最小变异。
表1 候选序列的序列结构及变异信息
Table 1 Sequence structure and variation information of candidate sequences

序列名称Sequence name序列长度Sequence length(bp)G+C含量G+C content(%)变异位点Variable sites(%)ITS569~57254.1~58.223.0ITS2193~19653.1~61.341.0matK65933.0~34.37.6rbcL50244.4~45.26.2trnH-psbA129~14922.1~30.648.4rtnL-trnF364~37829.1~30.610.5
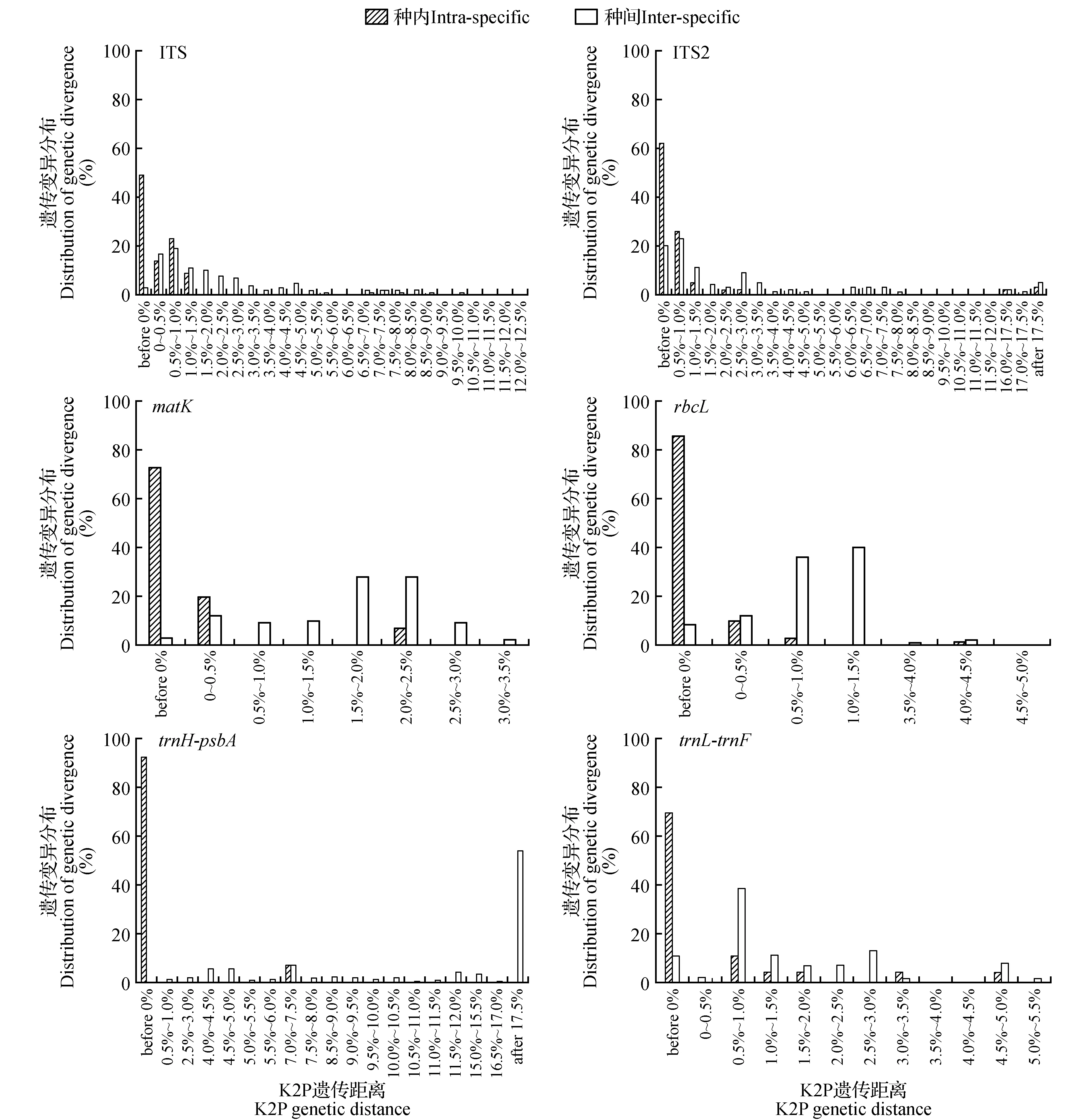
图1 种内变异与种间变异的barcoding gap图Fig.1 Barcoding gap diagram of intraspecific variation and interspecies variation
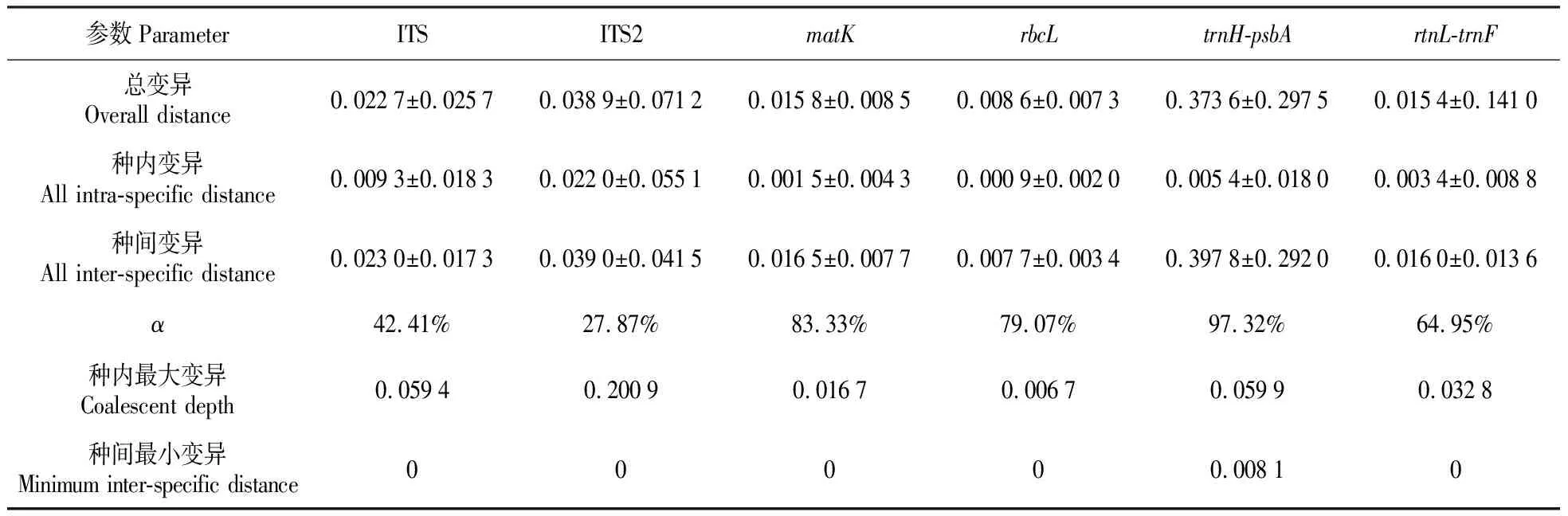
表2 候选条形码的遗传变异
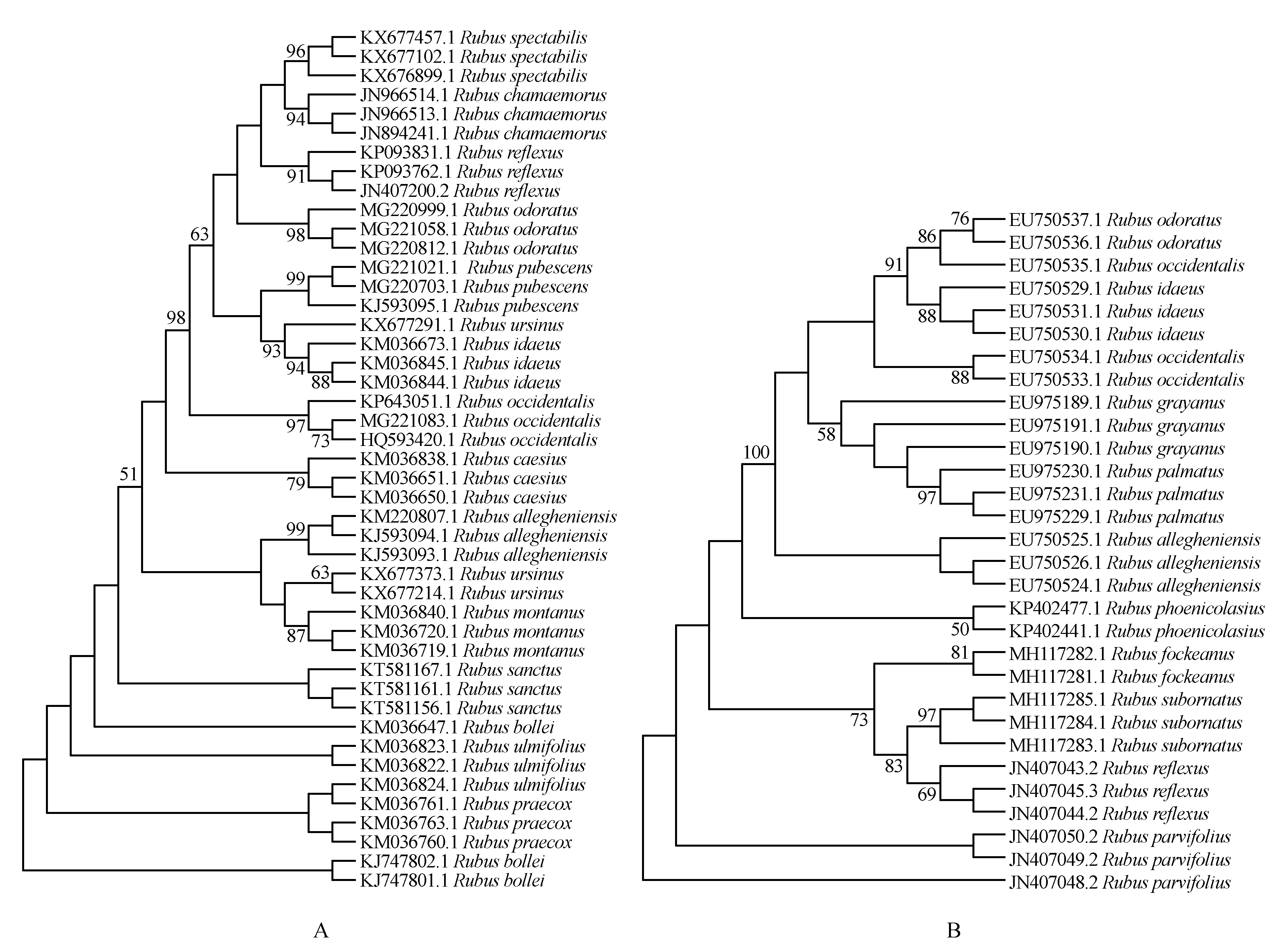
图2 基于matK、trnH-psbA构建的NJ一致树 A.基于matK构建的NJ一致树;B.基于trnH-psbA构建的NJ一致树 分支仅显示50%以上的bootstrap value。Fig.2 NJ consistent tree based on matK,trnH-psbA The branch only shows more than 50% of the bootstrap value,A is an NJ consistent tree based on matK,and B is an NJ consistent tree based on trnH-psbA.
2.3 Barcoding gap评估
理想的DNA条形码鉴定序列种内遗传变异应明显小于种间遗传变异,两者之间具有明显的间隙,即barcoding gap[26]。如图1所示,ITS、ITS2的种间与种内变异频率分布重叠较大,matK、rbcL、trnH-psbA、rtnL-trnF4个条形码的种间与种内变异分布频率具有较明显的间隙(gap),其中matK和trnH-psbA的gap最明显。
2.4 NJ一致树分析
在物种的DNA条形码分子鉴定中,NJ一致树也是一种常用的条形码评估方法,良好的条形码序列能将不同物种材料进行有效鉴别,形成单系性分支,并获得高支持率。本研究分别基于6个候选条形码构建NJ一致树,计算获得各候选条形码有效单系性(支持率>50%)比例分别为ITS:33%,ITS2:28%;matK:67%,trnH-psbA:64%,rbcL:30%,rtnL-trnF:43%。其中,matK和trnH-psbA的有效单系性比例最高,能有效区分60%以上的参试物种,相比ITS、ITS2、rbcL、rtnL-trnF对悬钩子属植物具有较高的鉴别能力,如图2为matK、trnH-psbA构建的NJ一致树。
3 讨论
评价不同序列的barcoding gap是判断不同条形码优劣的标准之一[13,27]。理想的DNA条形码序列检测到的种间遗传变异应明显大于种内遗传变异,在遗传变异分布频率图中(即barcoding gap检测图)种内变异与种间变异之间具有明显的gap[26]。本研究的遗传变异分析显示,6个候选条形码中种内变异与种间变异差异较大的有trnH-psbA、matK、rbcL和rtnL-trnF,变异分辨率分别为97.32%、83.33%、79.07%、64.95%,matK、trnH-psbA、rbcL和rtnL-trnF的barcoding gap也较为明显。可见,从遗传变异的角度来看matK、trnH-psbA、rbcL和rtnL-trnF4个候选条形码对悬钩子属植物具有较强的鉴别潜能。除利用遗传变异分析种内变异与种间变异的差异程度、检测barcoding gap外,构建系统进行树进行物种单系性分析也是筛选DNA条形码的一条重要途径[13]。本研究通过构建NJ一致树,结果显示matK的单系性比例最高(67%),其次为trnH-psbA(64%),而rtnL-trnF的单性系比例为43%,rbcL仅30%。因此,从建树分析角度来看,以matK、trnH-psbA作为悬钩子属物种鉴定的条形码是较优选择。综合考虑,笔者认为matK、trnH-psbA2个条形码序列在悬钩子属的物种鉴定中具有较大的鉴定潜力。陈小露等曾利用ITS2序列对茅莓根及其混伪品(粗叶悬钩子、山莓、锈毛莓、空心泡、百花悬钩子、插田泡等)进行鉴别,NJ建树分析显示茅莓及其混伪品能够表现良好的单系性,ITS2对茅莓、粗叶悬钩子、山莓、锈毛莓、空心泡、百花悬钩子、插田泡具有较好的鉴别能力[19]。而本研究中ITS2在NJ一致树中的单系性比例仅28%,其中山莓(R.corchorifolius)(79%)、山楂叶悬钩子(R.crataegifolius)(89%)、R.odoratus(100%)、北悬钩子(R.arcticus)(58%)获得较好的区分,但茅莓(R.parvifolius)、锈毛莓(R.reflexus)、插田泡(R.coreanus)没有获得有效鉴别,这与取样的范围相关,GenBank数据来源范围的扩大可能会降低条形码的鉴别力。
越来越多的研究表明靠单一片段不太可能实现对所有植物物种进行准确鉴定[28~30]。因此筛选条形码不能仅关注单个片段,必要时可以考虑增加片段数量(即片段组合策略)。Kress等2005年在其研究中最早提出片段组合观点,并建议将ITS+trnH-psbA作为被子植物鉴定的条形码组合[31];2007年Kress和Erickson又提出使用rbcL+trnH-psbA组合对陆生植物进行识别与鉴定[32];2009年,国际生命条形码联盟植物工作组建议将rbcL+matK组合作为陆地植物的核心条形码[17];近年我国学者也在DNA条形码研究领域做了大量突出的研究工作,2011年中国植物条形码研究组通过对1 757种植物的psbA-trnH、ITS/ITS2及rbcL+matK序列或序列组合进行鉴别能力评价,提出将ITS/ITS2序列作为种子植物的核心条形码,psbA-trnH作为辅助条形码序列[18]。从本研究的遗传变异、NJ一致树等分析结果来看,单个片段无法实现对悬钩子属植物100%的鉴别。6个候选条形码中matK和trnH-psbA对悬钩子属植物的鉴别潜力最大,而ITS、ITS2、rbcL、trnL-trnF对悬钩子植物的鉴别潜力相对较差,但也具有一定的鉴别力。因此,笔者认为在对悬钩子属植物进行DNA条形码鉴定时可以适当增加片段数量,将具有较高鉴别潜力的matK和trnH-psbA作为悬钩子属植物鉴定的核心条形码,而鉴别潜力相对较弱的ITS、ITS2、rbcL、rtnL-trnF作为辅助条形码。
