甜玉米种子营养品质主要性状全基因组关联分析
2020-04-07王长进徐运林程昕昕余海兵
王长进,徐运林,程昕昕,周 毅,余海兵
(安徽科技学院 农学院,安徽 凤阳 233100)
持续推进优化玉米种植结构,增加鲜食糯玉米、甜玉米有效供给是农产品供给侧结构性改革的重点。鲜食甜玉米(ZeamayssaccharataSturt.)是玉米属的一种重要类型,受su1、su2、sh1、sh2、sh4、du、ae、bt1、bt2、se等隐性突变基因的影响,籽粒乳熟期碳水化合物转化为淀粉的效率低,籽粒皱缩干秕,发芽率差。因此,籽粒营养品质性状是控制甜玉米产量的重要指标,也是直接影响种子出苗和产量的关键因素。
种子营养品质性状是一种受多基因控制的复杂数量性状。种子中贮存的营养物质是影响种子萌发效率的主要因子,主要包括淀粉、蛋白质和脂肪等多种成分。在不同种质资源中存在的不同等位基因可能是造成营养品质表型差异的重要原因。连锁定位和关联分析是鉴定表型性状与分子标记或候选基因间关系的重要分析方法。连锁定位需要构建作图群体,然后依据标记的重组率确定数量性状基因座的位置,其定位准确度受到群体和标记密度的影响。关联分析(genome-wide association study,GWAS)最早应用于人类是以连锁不平衡为基础的,通过分析关联群体表型与分子标记之间的相关程度,筛选出与目标性状显著关联的标记,并依据基因组的连锁不平衡程度确定候选基因区间[1]。与连锁定位相比,关联分析具有耗时短、定位精度高等优点。随着第三代分子标记技术的单核苷酸多态性(single nucleotde polymorphism,SNP)芯片技术的快速发展,基于SNP标记技术的关联分析已在水稻[2]、小麦[3]、玉米[4-5]、拟南芥[6]、大豆[7]等模式植物中广泛应用。
玉米是最早应用关联分析的植物。在种质资源分类方面,吴金凤等[8]利用NJ聚类法将51份玉米自交系划分为7个杂种优势群;史亚兴等[9]利用1 031个SNP标记将39份甜玉米自交系划分为5个类群。在籽粒品质方面,Beló等[10]利用553份玉米自交系和8 590个SNP标记定位到控制玉米籽粒脂肪酸含量的位点,并确定了候选基因fad2; Yang等[11]利用527份玉米自交系定位到控制玉米籽粒油分的74个显著关联位点。在玉米农艺性状方面,Mao等[12]利用GWAS鉴定到控制玉米苗期干旱性状、编码NAC转录因子的基因ZZmNAC111;Rashid等[13]对368个玉米自交系进行高粱霜霉病的全基因组关联分析,检测到26个SNP与高粱霜霉病显著相关。但有关鲜食甜玉米籽粒营养品质的GWAS分析鲜有报道。本研究利用玉米育种安徽省工程技术中心提供的不同地理来源的100份甜玉米种质资源对种子的3个营养品质性状的表型进行鉴定,利用均匀覆盖全基因组的37 297个高质量SNP标记进行全基因组关联分析,旨在检测控制甜玉米籽粒品质性状的主效QTL,挖掘出表现稳定的QTL优异等位基因,为甜玉米分子标记辅助选择和品质育种提供理论基础。
1 材料与方法
1.1 实验材料
本研究利用玉米育种安徽省工程技术中心提供的包含100份甜玉米自交系的育种资源群体进行实验。
1.2 实验方法
1.2.1 表型性状鉴定
在甜玉米自交系果穗吐丝前进行套袋,抽雄散粉后人工辅助授粉,在授粉后42 d分别收获3个自交果穗,取其中部籽粒混匀,进行干燥,待测。
淀粉含量采用旋光法测定[14]。蛋白质含量采用凯氏定氮蒸馏法测定[14]。脂肪含量采用索氏抽提法提取测定[15]。
1.2.2 DNA提取
利用SDS法提取DNA[16]。
1.2.3 SNP质量控制
利用北京中玉金标记生物技术股份有限公司根据玉米基因组开发的56K SNP芯片进行实验。为了减少标记之间强LD对关联结果的影响,利用Plink软件计算成对SNP间的r2并对连锁不平衡强度较高(r2>0.3)的标记进行过滤,最终共有37 297个SNP标记用于后续分析。
1.2.4 关联分析
采用SPAGeDi进行亲缘关系分析,并计算亲缘关系值的矩阵(K矩阵)[17]。Structure 2.2软件进行群体结构分析,通过ΔK确定群体亚群数目,最适K值重复运算计算Q矩阵。在TASSEL 2.1[18]软件中,采用一般线性模型,将Q矩阵作为协变量(Q,GLM),对甜玉米的淀粉、蛋白、脂肪含量进行全基因组关联分析(P≤0.05表示标记与性状间存在显著关联)[19]。
1.3 数据分析
利用SAS软件和PASW18.0软件对关联群体籽粒营养品质性状进行方差分析。
2 结果与分析
2.1 甜玉米群体结构分析
利用Plink软件对56 000个SNP标记中的连锁不平衡强度较高(r2>0.3)的标记进行过滤,得到37 297个SNP标记,均匀覆盖甜玉米每条染色体,且每条染色体上的SNP密度为7.65~21.08 Mb-1,多态信息含量为0.194~0.195,不同染色体的基因多样性变幅是0.361~0.377,平均值为0.371。
选取过滤后的标记进行群体结构分析(图1):当K为2时,对应的ΔK值最大,然后ΔK迅速下降并趋于平缓。因此,可以将这100份甜玉米划分为2个亚群,其中,亚群1包括92份种质,亚群2包括8份种质。
表1 甜玉米10条染色体PIC值和基因多态性
Table1PIC values and gene diversity in 10 chromosome of sweet corn
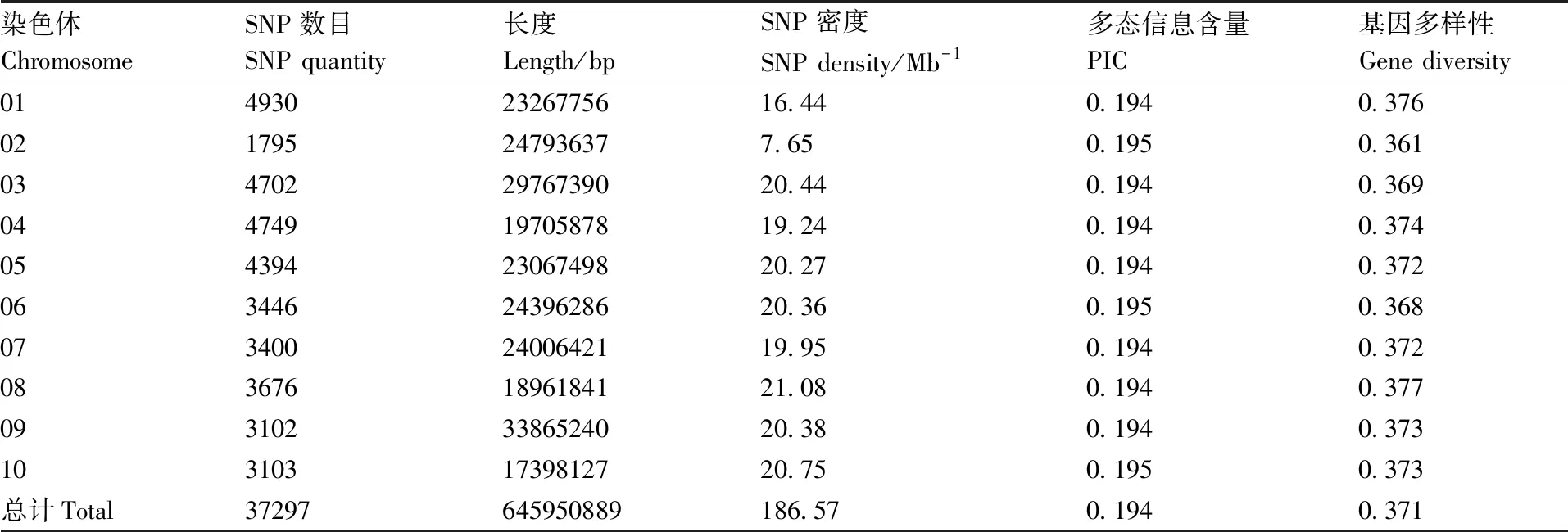
染色体ChromosomeSNP数目SNP quantity长度Length/bpSNP密度SNP density/Mb-1多态信息含量 PIC 基因多样性Gene diversity0149302326775616.44 0.1940.376021795247936377.65 0.1950.3610347022976739020.44 0.1940.3690447491970587819.24 0.1940.3740543942306749820.27 0.1940.3720634462439628620.36 0.1950.3680734002400642119.95 0.1940.3720836761896184121.08 0.1940.3770931023386524020.38 0.1940.3731031031739812720.75 0.1950.373总计Total37297645950889186.57 0.1940.371
2.2 甜玉米自然群体种子品质表型数据分析
淀粉、蛋白质和脂肪含量是甜玉米种子的主要营养成分。从表2可以看出,甜玉米种子中3种营养成分含量变幅较大,其中淀粉含量变幅为53.91%~73.70%,蛋白质含量变幅为8.96%~18.01%,脂肪含量变幅为5.53%~18.50%,进一步表明甜玉米自交系种子主要营养品质性状差异较大。偏度系数表明,淀粉含量、蛋白质含量呈正态分布。
2.3 关联分析
利用GLM-Q检测到与甜玉米种子淀粉含量显著关联的SNP位点14个(表3),分别分布在1、2、3、4、5、6、7、8号染色体上,能够解释表型变异的13.34%~20.90%。其中,在3、4、5号染色体上分别检测到2个SNP位点,在8号染色体上检测到4个SNP位点。在-log10P>3.50水平下,检测到显著关联位点3个,3号染色体上qSTA-3-1位于Affx-115329496处,贡献率最大,约为20.90%。
共检测到控制甜玉米种子蛋白质含量的SNP位点15个,主要分布在1、2、4、8、9号染色体上,贡献率为3.45%~51.69%。在-log10P>18水平下,检测到显著关联位点5个,其中:1号染色体上qPRO-1-1位于Affx-91181539,贡献率达到51.69%;9号染色体上qPRO-9-2位于Affx-115333989处,贡献率高达45.25%(表4)。
甜玉米种子脂肪含量关联分析共定位到20个SNP位点,分别位于1、3、4、5、6、7、8、9、10号染色体上,能够解释表型变异的13.69%~17.89%,在-log10P>3.50水平下,检测到显著关联位点1个,位于5号染色体上的Affx-91137282处,qFAT-5-1对表型变异的解释率为17.06%(表5)。
表2 甜玉米自然群体种子主要品质性状表型数据统计分析
Table2Phenotypes statistical analysis of seed quality in sweet corn natural population
表3 甜玉米种子淀粉含量显著关联的位点
Table3Significant markers associated with starch content of sweet corn
表4 甜玉米种子蛋白质含量显著关联的位点
Table4Significant markers associated with protein content of sweet corn
表5 甜玉米种子脂肪含量显著关联的位点
Table5Significant markers associated with fat content of sweet corn

序号No.QTL序号QTL IDSNP染色体Chromosome位置Position-log10P贡献率Contribution rate/%1qFAT-5-1Affx-9113728251879614161.26×10-417.06 2qFAT-5-2Affx-908299095927988453.13×10-415.47 3qFAT-9-1Affx-11533223691163385473.44×10-415.60 4qFAT-5-3Affx-9128306251325999713.91×10-415.08 5qFAT-10-1Affx-9007067010462557424.52×10-414.68 6qFAT-10-2Affx-9037425510545984764.52×10-414.68 7qFAT-10-3Affx-9109614710548678144.52×10-414.68 8qFAT-1-1Affx-9096290912956589234.72×10-414.61 9qFAT-10-4Affx-9109788610459631454.80×10-414.72 10qFAT-10-5Affx-9016085710548677134.87×10-414.69 11qFAT-1-2Affx-1153315981652435155.58×10-417.89 12qFAT-10-6Affx-9129081010664084286.08×10-414.30 13qFAT-6-1Affx-9067240961480439876.21×10-414.12 14qFAT-3-1Affx-9109074331910080676.37×10-414.64 15qFAT-1-3Affx-9009999012553703856.65×10-414.00 16qFAT-7-1Affx-9118114971672303537.03×10-415.05 17qFAT-6-2Affx-905323646404341817.30×10-414.24 18qFAT-8-1Affx-9099956381104459367.56×10-414.47 19qFAT-10-7Affx-9117929310964770087.58×10-414.04 20qFAT-4-1Affx-904824134700196037.93×10-413.69
3 结论与讨论
种子的营养品质是受多基因控制的复杂数量性状,主要包括淀粉、蛋白质和脂肪等。本研究发现,甜玉米种子中3种营养成分含量变幅较大,其中淀粉含量变幅为53.91%~73.70%,蛋白质含量变幅为8.96%~18.01%,脂肪含量变幅为5.53%~18.50%,这种表型变异可能是不同种质资源中存在的等位基因差异造成的。
群体的表型变异是全基因组关联分析的基础。群体内不同材料之间表型变异越丰富,群体的重组率越高,连锁不平衡就越低,越适于进行全基因组关联分析。本研究发现,100份甜玉米自交系可划分为两个亚群,其中亚群1包括92份种质,亚群2包括8份种质。利用56K SNP基因芯片获得了均匀覆盖甜玉米基因组的37 297个高质量SNPs,PIC值集中在0.19,基因多样性变幅集中在0.36~0.38。
全基因组关联分析是通过扫描覆盖整个基因组的大量标记对群体的目标性状进行分析,以期关联到控制目标性状的位点。玉米是较早进行关联分析的植物,前人已经对玉米产量、株高、抗病性等多个性状进行了全基因组关联分析[20-24]。其中,2001年Thornsberry等[21]关联定位到控制玉米自交系开花期的dwarf8基因;2011年Kump等[22]关联检测到控制影响叶枯病的32个QTL;2012年Farfan等[23]利用346个玉米自交系定位到控制产量、株高、开花期的10个QTL。Hu等[24]对282个玉米自交系进行发芽试验,利用2 271 584个SNP对玉米萌发过程的抗寒性进行全基因关联分析,共检测到17个位点与抗寒性有关。Liu等[25]利用263个玉米自交系56 110个均匀分布的SNP对淀粉含量进行关联定位,发现4个SNP与淀粉含量显著相关,预测了淀粉合成相关的候选基因77个,其中APS1基因与淀粉合成紧密相关。本研究利用GLM-Q模型关联定位到控制甜玉米籽粒淀粉含量、蛋白质含量、脂肪含量的SNP位点14、15和20个,能够解释表型变异的3.45%~51.69%。其中,在-log10P>3.50水平下,位于3号染色体上Affx-115329496处的qSTA-3-1对淀粉含量贡献率最大,约为20.90%;与Liu等[25]关联定位的3号染色体上关联位点较一致。
种子全基因组蛋白质功能研究是当前研究的一个热点,蛋白质功能预测主要受生化反应(通路、细胞、组织等)、环境条件、多功能性等多面的影响。本研究发现,在-log10P>3.50水平下,位于甜玉米1号染色体Affx-91181539处的qPRO-1-1位点和9号染色体上Affx-115333989处的qPRO-9-2与籽粒蛋白质含量显著关联。
