花生幼苗响应低温胁迫的转录组分析
2020-04-02史晓龙任婧瑶董佳乐赵新华蒋春姬于海秋
张 鹤,史晓龙,任婧瑶,董佳乐,赵新华,蒋春姬,于海秋
(沈阳农业大学 农学院/花生研究所,沈阳 110161)
花生(Arachis hypogaea L.)是世界范围内重要的经济作物和油料作物,广泛种植于热带和亚热带地区[1]。近年来,随着对花生需求量的日益增加,其种植面积不断地向高纬度地区扩张。东北地区花生因黄曲霉污染率低、品质优良、出口量大而成为我国花生主产区之一,目前的种植面积已经达到46.7万~53.3万hm2[2]。然而,在全球气候变化的背景下,低温、干旱等极端气候事件在东北地区的发生频率和发生强度逐年增加,严重地限制了花生的生长发育、产量和品质形成[3]。因此,深入解析花生的耐寒机理,创制花生耐寒新种质,对提高东北地区的花生生产和品质具有重要意义。
植物从感受到低温信号到做出生理生化响应来适应低温胁迫,期间需要经历复杂的信号转导过程,受多个基因和代谢过程调控[4]。明确植物耐寒的分子机制能更好地阐释植物的耐寒性,也是快速培育耐寒种质的基础。RNA-Seq作为挖掘相关性状基因的一种方法,可在全基因组水平上进行相关基因差异表达的研究,已经被广泛地应用于不同物种、不同生育时期的耐冷性研究中,包括玉米[5]、水稻[6]、大豆[7]、小麦[8]和苜蓿[9]等,差异表达基因广泛涉及糖、脂类、蛋白和核酸等生物大分子代谢,次生产物代谢及能量代谢等过程,并且通过代谢途径的富集发现植物激素信号转导、光合作用途径、核糖体翻译机制和苯丙烷类生物合成等代谢途径在低温胁迫下变化明显。随着野生种和栽培种花生基因组测序工作的陆续完成,转录组测序技术在花生研究中也开始被大范围应用,ZHAO等[10]以花生栽培种J11为试验材料,对干旱胁迫下的花生根部进行转录组测序分析,共筛选出4648个差异表达基因,显著富集在碳代谢、光合作用和柠檬酸循环等代谢途径。SUI等[11]通过分析盐胁迫处理下花生幼苗叶片的RNA-Seq数据,分别在脂类代谢通路和脂肪酸代谢通路上鉴定出26和11个盐胁迫响应基因。但是,目前关于花生对低温胁迫的响应机理却少有报道。
为了从全基因组水平上系统地揭示花生对低温胁迫的响应机制,本研究以辽宁省大面积栽培的耐寒型花生品种农花5号为试验材料,利用RNA-Seq转录组测序技术分析低温胁迫下花生幼苗叶片的基因表达水平变化,并对差异表达基因进行功能注释和代谢通路分析,以期明确花生在低温胁迫下的转录机制,挖掘出响应低温胁迫的关键功能基因,为利用基因工程技术改良花生的耐寒能力提供理论基础。
1 材料与方法
1.1 材料
以辽宁省大面积栽培的耐寒型花生品种农花5号(NH5)为试验材料,所有花生种子均由沈阳农业大学花生研究所提供。
1.2 方法
选取籽粒饱满,大小一致的花生种子,浸泡12h后播种至装有沙土的塑料花盆中,于温度为28℃,光照/黑暗为16h/8h的人工气候室内培养,每天定量补充1/2Hoagland营养液。待幼苗长至3叶1心期,于温度为6℃,光照/黑暗为16h/8h的条件下进行低温处理,分别在处理0(T0)、12(T1)和24h(T2)后采集叶片,进行转录组测序,各处理组3次重复。
1.2.1 总RNA提取、cDNA文库构建和测序 采用TRIzol法提取总RNA,并对RNA浓度、纯度、完整性进行检测。样品检测合格后,用带有Oligo(dT)的磁珠富集真核生物mRNA。加入Fragmentation Buffer将mRNA进行随机打断,以mRNA为模板,用六碱基随机引物合成第一条cDNA链,然后加入缓冲液、dNTPs、RNAase H和DNA polymeraseⅠ合成第二条cDNA链,利用AMPure XPbeads纯化cDNA,纯化的双链cDNA再进行末端修复、加A尾并连接测序接头,然后用AMPure XPbeads进行片段大小选择,最后通过 PCR扩增,纯化PCR产物,得到最终文库。文库质量检测合格后,不同文库按照目标下机数据量进行pooling,用IlluminaHiSeq平台进行测序。
1.2.2 测序数据 分析将下机数据进行过滤得到Clean Data,与花生栽培种基因组 (Tifrunner.gnm1.ann1.CCJH)进行序列比对,对得到的Mapped Data进行插入片段长度检验、随机性检验等文库质量评估。以FPKM(Fragments Per Kilobase of transcript per Million fragments mapped)≥1为标准筛选有效表达基因。利用基于负二项分布的 DEGseq软件,以差异表达倍数(Fold Change,FC)≥2和错误发现率(False Discovery Rate,FDR)<0.01为标准筛选差异表达基因(Differentially expressed genes,DEGs)。
1.2.3 差异表达基因GO分析和KEGG分析 采用基于Wallenius non-central hyper-geometric distribution的GOseq软件方法进行GO富集分析。差异基因的功能注释和分类依托KEGG公共数据库,Pathway富集分析采用 KOBAS(2.0)进行。
1.2.4 实时荧光定量PCR验证 从转录组数据库中随机选取6个差异表达基因进行qRT-PCR分析验证,以6℃低温处理0,12,24h的花生幼苗叶片RNA为材料,使用日本TAKARA公司试剂盒(PrimeScriptTMRT Master Mix)进行反转录合成cDNA,以花生Actin作为内参基因,利用Primer Premier 5.0软件设计引物 (表 1),进行qRT-PCR分析。每个处理进行3次生物学重复,用2-ΔΔCT法计算基因的相对表达量。

表1 荧光定量PCR的引物序Table 1 Primers used for real time PCR analysis
2 结果与分析
2.1 转录组数据质控分析
对T0、T1和T2的花生幼苗叶片进行RNA-Seq测序,测序结果显示 (表2),9个样品共获得56.91 Gb Clean Data,各样本均产出超过 5.82Gb的高质量数据,GC比在 44.13%~46.49%之间,Q30碱基百分比均在93.17%及以上。

表2 低温胁迫下花生叶片样本转录组数据统计Table 2 Statistics of transcriptome sequencing of peanut leaf samples under cold stress
分别将各样品的Clean Reads与花生栽培种基因组(Tifrunner.gnm1.ann1.CCJH)进行序列比对,比对效率可以看出转录组数据的利用率,结果如表3,9个样本的成功比对率在89.57%~95.36%之间,比对效率较高。质量控制和序列比对数据说明,低温胁迫下9个花生样本的转录组测序数据质量较好,可以用于后续生物学分析。
2.2 基因表达量分析
对低温胁迫下9个花生样本的转录组测序数据进行表达量统计,共获得35344个有效表达基因(FPKM≥1)。通过相关性热图,对样本间基因表达量的生物学重复相关性进行评估,将皮尔逊相关系数r作为评估指标,r2越接近1,说明两个重复样品相关性越强。由图1A可知,每个处理间3次重复的相关性较强,而各处理间相关性较小,其中T0处理的3个重复间相关性最高。同时,对9个样本的基因表达量进行主成分分析,找出离群样本,判别相似性高的样品簇。结果表明(1B),相同处理的样本之间聚集在一起,相似度较高,而不同处理之间基因表达差异显著,分别形成不同的集群,这与热图结果一致。
2.3 基因表达的差异性分析
依据FPKM值对样品间基因表达的差异性进行分析,差异基因火山图可以简明地看出低温处理前后的差异基因分布,其中显著上调的基因以红色表示,显著下调的基因以绿色表示,无显著性差异的基因以黑色表示(图2)。结果显示,与0h相比,低温处理12 h后有8943个基因差异表达,其中4639个基因上调表达,4304个基因下调表达;低温处理24 h后有6835个基因差异表达,其中4390个基因上调表达,2445个基因下调表达,上调的基因数明显高于下调基因数。

表3 转录组数据与参考基因组的比对情况Table 3 Statistics of transcriptome sequencing data and reference genome alignment
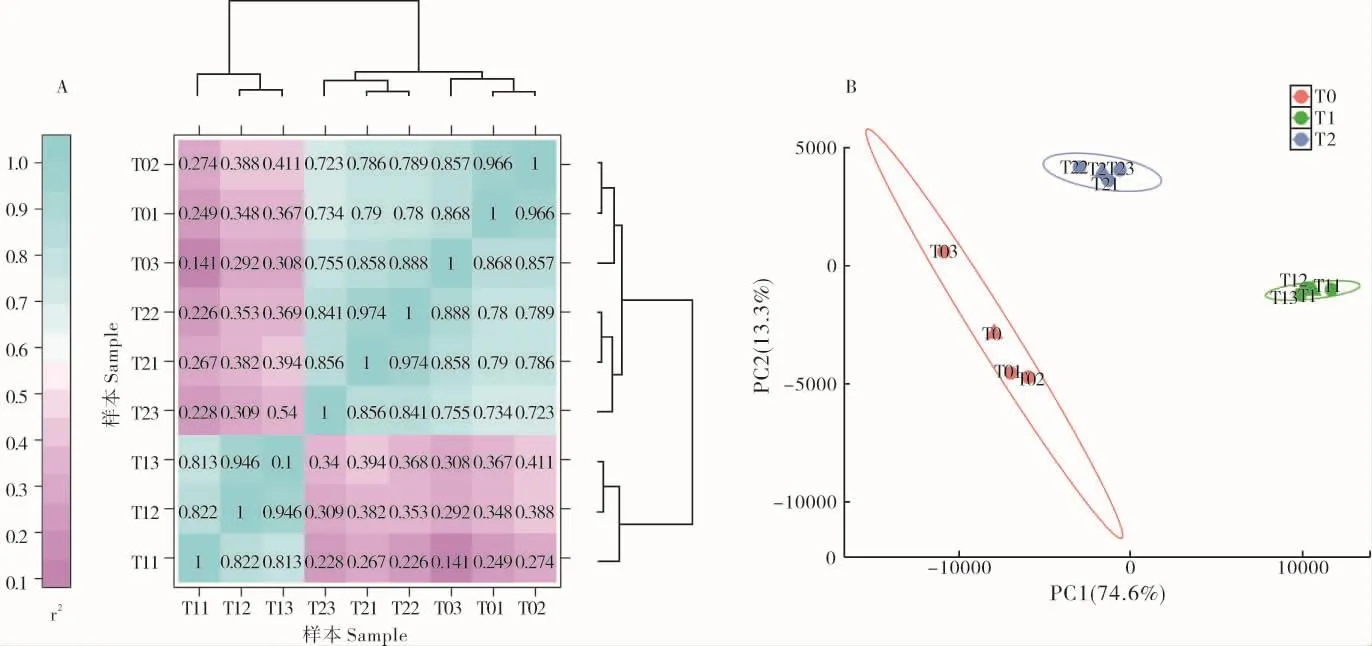
图1 低温胁迫下9个花生样本间的表达量分析(A.相关性分析;B.主成分分析)Figure 1 Expression level analysis of nine peanut samples under cold stress(A.The correlation analysis;B.The PCA analysis)
通过韦恩图(图3)进一步对对差异表达基因进行分析可知,低温胁迫下,在上调表达的DEGs中,有2428个基因为持续差异表达基因,即在T1和T2中均上调表达;在下调表达的DEGs中,有1482个持续差异表达基因,上调持续表达基因显著多于下调持续表达基因。由此可见,低温胁迫24h内有大量的冷响应基因被激活,部分基因受到抑制。
2.4 持续差异表达基因的GO富集分析
根据差异基因检测结果,对持续差异表达基因进行GO(Gene ontology)功能分类,结果如图4A所示,2044个持续差异表达基因被注释到43个功能组中,主要分为生物学过程(20组)、细胞组分(12组)和分子功能(11组)三大类别。其中,生物学过程中的代谢过程(1145个)、细胞过程(904)和单器官过程(713),以及分子功能中的催化活性(1214)和结合(1017)等功能组富集到的持续差异表达基因较多,而细胞组分中相对较少。

图2 差异表达基因火山图Figure 2 Volcanic diagram of different expressed genes
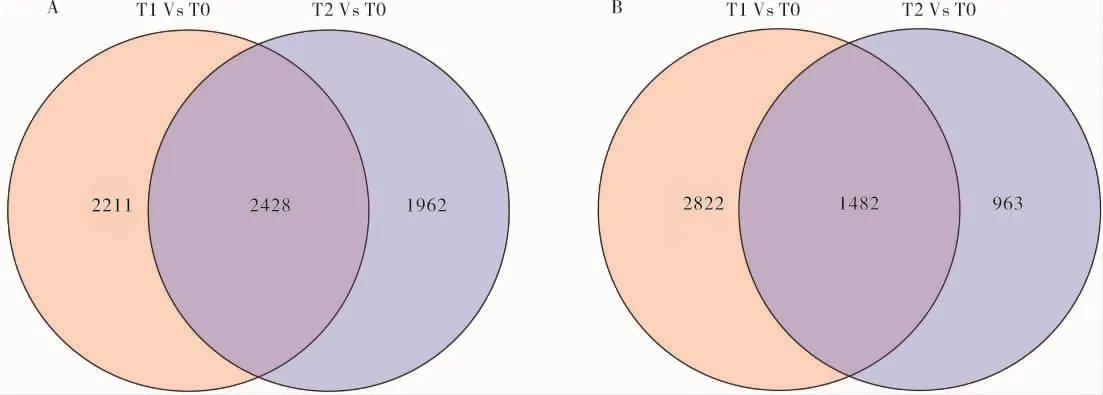
图3 差异表达基因韦恩图(A.上调差异表达基因;B.下调差异表达基因)Figure 3 Venn diagram of different expressed genes(A.Up-regulated DEGs;B.Down-regulated DEGs)
在持续差异表达基因GO功能分类的基础上,进一步对持续上调和持续下调表达的基因进行GO注释(4B),结果发现,花生幼苗在持续低温胁迫下大多数功能组分中的上调差异表达基因显著多于下调表达基因数目,这表明低温胁迫对这些功能组基因的促进作用大于抑制作用。只有运动、大分子复合物、电子载体活性和结构分子活性四个功能组中的下调基因数量大于上调,低温胁迫对这些功能组基因具有一定的抑制作用。
2.5 持续差异表达基因的代谢途径分析
生物个体在行使其生物学功能时,往往需要不同层面的基因共同协调完成,通过对代谢通路的分析,可以更好的揭示DEGs的生物学功能。对3910个持续差异表达基因进行KEGG (Kyoto encyclopedia of genes and genomes)分析,共富集到116个代谢通路中,分为有机系统、遗传信息处理、细胞过程、环境信息处理和代谢过程五大类别(图5A),其中有机系统中的环境适应,环境信息处理中的信号转导,以及代谢过咸亨中的碳水化合物代谢和脂类代谢等通路富集到的持续差异表达基因数目较多。进一步对各组分的代谢通路进行显著性富集分析,结果表明(5B),持续低温胁迫下,花生叶片中的DEGs显著富集在植物激素信号转导(66个)、淀粉和蔗糖代谢(53个)和植物-病原菌互作(52个)等代谢通路上。
由表4可知,植物激素信号转导、淀粉和蔗糖代谢、植物-病原菌互作、氨基酸的生物合成和甘油磷脂代谢等代谢途径中上调基因显著多于下调基因,并且,植物昼夜节律和α-亚麻酸代谢途径中的持续差异表达基因均为上调表达,说明这些代谢途径在花生响应低温胁迫时被高度激活,具有重要作用。而核糖体途径中的持续差异表达基因全部下调表达,说明低温胁迫下花生中的核糖体途径受到严重抑制。
2.6 qRT-PCR验证

图4 持续差异表达基因的GO功能分类Figure 4 GO classification of continuous differentially expressed genes under cold stress

图5 持续差异表达基因的KEGG功能分类Figure 5 KEGG enrichment of continuous differentially expressed genes under cold stress
为了验证RNA-Seq结果的准确性,从转录组数据库中随机选取6个DEGs进行qRT-PCR分析。由图6可知,6个DEGs的表达水平在RNA-Seq和qRT-PCR存在差异,但表达趋势基本一致,证明了转录组数据的分析结果是可靠的。
3 讨论
植物对逆境信号的响应是一个极其发杂的过程,低温胁迫下植物表型和生理生化水平上发生的改变往往是其内在复杂的分子网络调控的结果[12]。为了从全基因组水平上明确低温胁迫下花生的基因表达情况,本研究以耐寒型花生品种NH5为试验材料,采用RNA-Seq技术对6℃低温胁迫0,12,24h的花生幼苗叶片进行转录组测序分析,筛选出3910个持续差异表达基因。DEGs的GO功能注释发现,生物学过程中的代谢过程富集到的持续差异表达基因数目最多,且多数功能组中上调基因数量大于下调,说明持续低温胁迫能促进某些功能基因上调表达,并对代谢过程的影响最大。通过KEGG注释对代谢过程进一步分析发现,持续上调表达基因大部分富集在植物激素信号转导、淀粉和蔗糖代谢、植物-病原菌互作、植物昼夜节律和α-亚麻酸代谢途径上,说明这些途径可能在花生苗期冷胁迫响应中起积极作用。
张高华等[13]对耐寒性不同的高油酸花生品种在低温胁迫下的转录组进行了比较分析,结果表明植物-病原相互作用和植物激素信号转导通路在花生耐寒中起着重要作用,与本研究的结果具有一定的相似性,也充分证明了花生对低温胁迫的响应与这两个途径密切相关。目前,植物激素信号转导途径中研究较多的是ABA依赖途径,ABA信号途径能够促进多数逆境应答基因表达[14]。低温胁迫下,水稻内源ABA升高,其受体PYP/PYL/
RCAR蛋白与ABA结合并与蛋白激酶PP2C互作,抑制蛋白激酶SnRK2与PP2C结合,SnRK2便能磷酸化转录因子,磷酸化后的转录因子激活ABA响应基因,进而提高对低温的耐受性[15]。除此之外,JA在花生响应低温胁迫中可能也具有重要作用,α-亚麻酸是JA合成的前体[16],本研究中α-亚麻酸代谢途径被显著富集,且所有DEGs均上调表达,可能对JA的合成及信号转导途径具有促进作用,值得进一步探究。

表4 持续上调和持续下调表达基因的KEGG富集分析Table 4 KEGG enrichment analysis of up-regulated and down-regulated continuous differentially expressed genes under cold stress

图6 差异表达基因的qRT-PCR验证Figure 6 The qRT-PCR validation of differentially expressed genes
植物对低温响应的分子调控机制是一个多基因参与、多种代谢过程相互影响的复杂过程[17],目前研究已经表明涉及到蛋白激酶、钙调蛋白、光合作用、激素生物合成与代谢、生物节律等系列过程[18]。本研究从转录水平上系统分析了花生对低温胁迫的响应机制,为解析花生耐寒的调控途径,挖掘花生耐寒的关键功能基因奠定了基础,对加速花生耐寒性的遗传改良具有重要意义。
