烟草DGAT基因家族全基因组鉴定与分析
2020-03-30牛永志王国平郑昀晔马文广
牛永志 王国平 郑昀晔 马文广
摘 要:二酰甘油酰基转移酶(DGAT)是三酰甘油合成的关键酶和唯一限速酶。在烟草中鉴定DGAT 基因对于研究油脂代谢途径和创造具有附加值的新型油料作物具有重要意义。本研究从烟草基因组鉴定DGAT 家族基因,并对序列特性、基因结构、亞细胞定位、进化模式、表达模式等进行分析。结果表明,共获得25 个烟草DGAT 家族蛋白,其平均氨基酸长度为485,大部分为碱性蛋白和亲水性蛋白,有13 个DGAT 蛋白定位到8 条连锁群上;有16 个蛋白定位到内质网,6 个定位叶绿体,3 个定位到胞外;烟草DGAT 蛋白包含典型的WS_DGAT 结构域和LPLAT 结构域;系统发育树将DGAT 家族分为DGAT1、DGAT2、DGAT3 和WSD 等4 个类群,但烟草DGAT 仅划分到DGAT2 和WSD 两个类群;不同烟草DGAT基因的时空表达特性有所差异,并表现出一定的组织特异性,主要集中在衰老叶片和花器官中大量表达。
关键词:基因家族;DGAT;烟草;生物信息
植物油脂不仅可作为细胞膜组成的脂肪酸供体,还可以以三酰甘油(Triacylglycerol,TAG)的形式存贮,作为真核生物主要的能量来源,对植物生长发育具有重要作用[1]。同时,植物油含有丰富的不饱和脂肪酸,是人类健康饮食的重要物质。植物油脂经甲基化处理后也可作为一种可再生生物柴油,在替代石油燃料方面具有潜在价值[2]。因此,了解油脂的生物合成也将有助于创造具有附加值的新型油料作物。
三酰甘油是油脂贮藏的一种主要形式。虽然在不同的器官和组织中,三酰甘油的生物合成存在多种途径[3],但最经典的还是三磷酸甘油途径(Kennedy途径)[4]。在该途径中,二酰甘油酰基转移酶(Diacylglycerol acyltransferase, DGAT)催化最后一步反应,将二酰甘油转化为三酰甘油,是影响三酰甘油含量的关键酶,也是唯一的限速酶[5]。目前,DGAT 基因家族共发现4 种亚型,分别为DGAT1、DGAT2、DGAT3 和WS/DGAT。其中DGAT1、DGAT2 是主要亚型,在大多数真核生物中广泛存在。DGAT1 已经在旱金莲[6]、蓖麻[7]、玉米[8]、芝麻[9]、油菜[10]等作物中被鉴定,DGAT2 已经在蓖麻[11]、油桐[12]、油菜[10]、橄榄[13]等作物中被鉴定。研究表明DGAT1 和DGAT2 在真核生物进化过程中是独立进化的,并且其功能趋于同质化[14]。DGAT3 和WS/DGAT 是近几年发现的亚型,只在很少的作物中被报道。其中DGAT3 是一种可溶性酶,在花生[15]和拟南芥[16]等作物中被鉴定。WS/DGAT 是一种双功能酶,其主要功能是催化蜡酯的合成,同时也参与少量TAG 的合成[17-18]。与DGAT1 和DGAT2 一样,DGAT3 和WS/DGAT 似乎也是独立进化的[19]。
烟草是一种叶用经济作物,但其种子油含量高,总体上占到种子重量的33.6%~39.4%,有的甚至可以达到48%[20]。而且烟籽油营养价值高,不饱和脂肪酸含量高于橄榄油、茶籽油、大豆油、玉米油、芝麻油、亚麻油等,仅次于核桃油[21-22],可以作为一种潜在的食用油。此外,烟草种子油经过酯交换反应后也可用于可再生生物燃油[23]。目前,烟草中的一个DGAT 基因已经被鉴定[24],但还有很多基因功能尚待解析。本研究利用生物信息学方法对烟草DGAT 基因家族进行系统分析,为深入研究油脂代谢途径以及培育高油含量烟草新品种奠定基础。
1 材料与方法
1.1 DGAT 基因家族序列鉴定与特征分析
本研究从茄科数据库(https://solgenomics.net/)下载普通烟草品种K326 的基因组数据(2017 年发布,包括GFF 文件、蛋白序列文件和核酸序列文件)。通过两种方式获得烟草DGAT 家族基因,一种是从Pfam 数据库(http://pfam.xfam.org/)下载DGAT 基因家族的HMM模型文件(PF03982),利用HMMER3.0 软件检索烟草基因组数据,E 值设为1e-5;另一种是以拟南芥的DGAT1(At2g19450)、DGAT2(At3g51520)、DGAT3(At1g48300)和WSD1(At5g37300)为参考序列,通过HMMER 3.0 软件构建HMM 模型,然后利用该模型检索烟草基因组数据,E 值设为1e-5。将两种方式获得的基因去除重复后,即为初始的烟草DGAT 家族基因。然后将初始基因上传至Pfam 网站进行结构域预测,E 值设为1e-5,剔除不含DGAT 家族典型蛋白结构域的序列,剩余的即为烟草DGAT 基因家族成员。
利用ExPaSy 网站(https://www.expasy.org/)的ProtParam 工具分析蛋白的氨基酸含量、分子量、等电点、脂肪族氨基酸指数和疏水性指数等理化性质。从GFF 文件中提取基因的染色体位置。
1.2 亚细胞定位及跨膜区预测
利用在线网站Softberry(http://linux1.softberry.com/)的ProtComp 工具进行亚细胞定位预测。利用在线工具TMHMM 2.0(www.cbs.dtu.dk/services/TMHMM-2.0/)进行跨膜区预测。
1.3 进化树构建
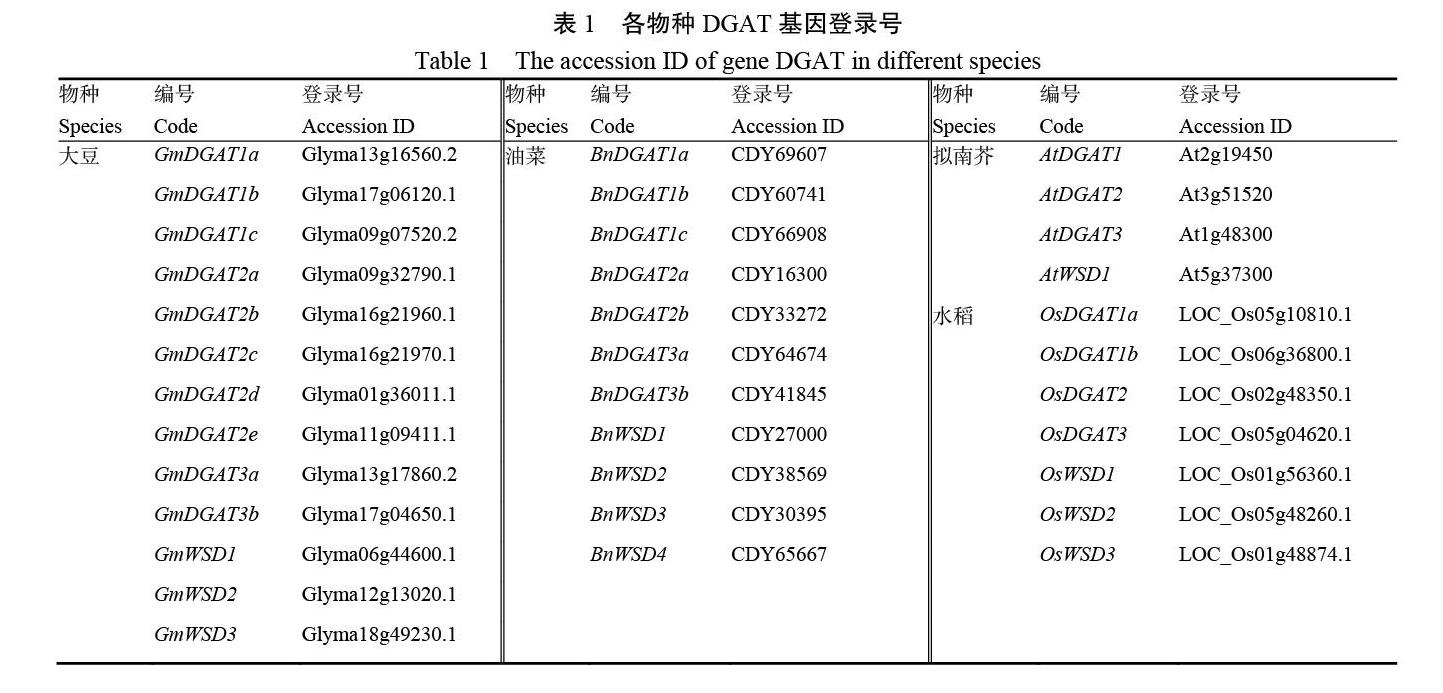
从Uniprot 数据库(https://www.uniprot.org)分别下载拟南芥、大豆、油菜、水稻的部分DGAT 基因(表1)。利用MEGA 软件进行多序列比对(采用ClustalW 比对方式,默认参数),并构建NJ 树(检验方法为Bootstrap 法,检验次数设为1000 次,gap处理选择Pairwise Deletion)。利用在线网站iTOL(https://itol.embl.de/)对进化树进行可视化。
1.4 蛋白结构域及基因结构分析
利用在线网站MEME(http://meme-suite.org/)进行保守结构域的预测,Motif 长度范围为6~100,检索的最大Motif 数为10,其他选择默认参数。利用在线网站EvolView(https://www.evolgenius.info/evolview/#login)对蛋白结构域进行可视化。利用NCBI 的CDD 工具(https://www.ncbi.nlm.nih.gov/cdd/?term=)鉴定结构域的类型。利用TBtools 软件对基因结构进行可视化。
1.5 表达模式分析
烟草基因表达图谱(TobEA)由EDWARDS 等于2010 年发布,该图谱包含了早期衰老叶、中早期衰老叶、中后期衰老叶、后期衰老叶、成熟叶、茎生叶、幼叶、上部茎、下部茎、成熟根、幼根、种子、未开花蕾、开放花蕾、花朵、幼芽、茎顶端、子叶和花顶端19 个样品组织,贯穿从种子萌发到植株衰老的整个生命周期。从EMBL-EBI 网站下载TobEA数据,登录号为E-MTAB-176(http://www.ebi.ac.uk/arrayexpress/experiments/E-MTAB-176/),通过blastp 比对检索DGAT 家族成员匹配的样品编号,通过样品编号获取对应的基因表达谱数据。将数据整理后利用Cluster 3.0 软件和Java TreeView 工具绘制热图。
2 结 果
2.1 不同物种DGAT 家族基因统计
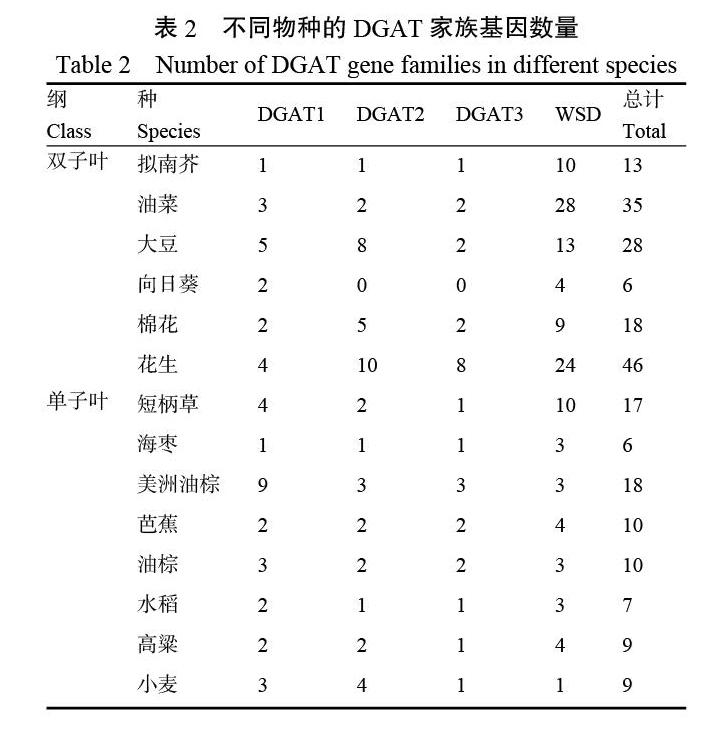
根据以往研究报道[25-26],DGAT 基因家族已在14 种被子植物中得到鉴定(表2)。除了向日葵中没有鉴定到DGAT2 和DGAT3 基因,其他物种中的DGAT 家族均包含4 种类型,其中数量较多的是花生(46 个)、油菜(35 个)和大豆(28 个)。整体上看,双子叶植物比单子叶植物的DGAT数量更多,WSD 亚型的基因数量比其他亚型更多。本研究中共鉴定得到烟草DGAT 家族基因25 个,数量仅次于大豆,其中包含17 个WSD 基因和8 个DGAT基因。
2.2 烟草DGAT 基因特征分析
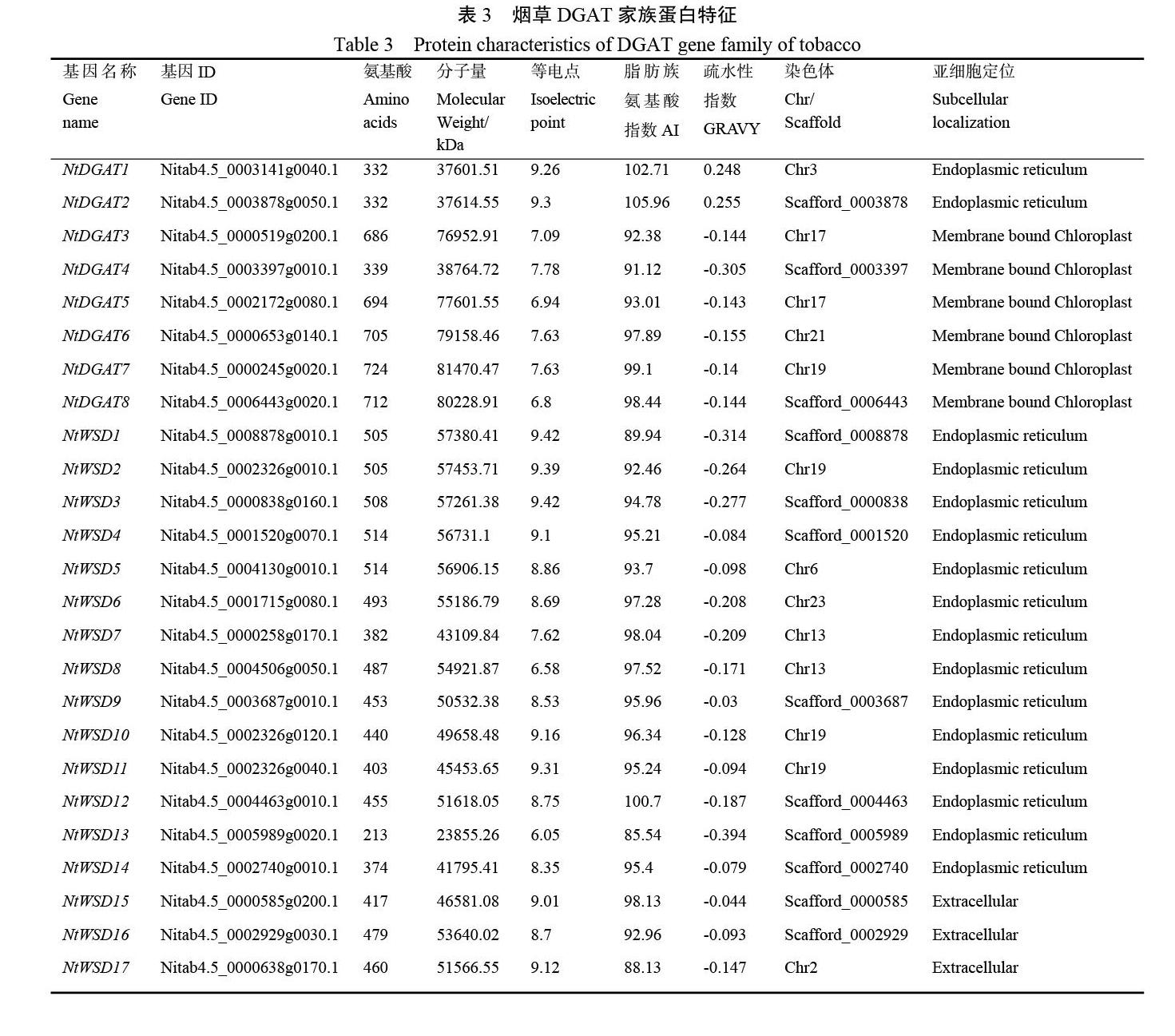
分析DGAT 家族基因的序列特征发现(表3),平均氨基酸长度为485,范围在213~724 之间;平均分子量为54521.81 kDa , 范围在23855.26~81470.47 kDa 之间;平均等电点为8.34,范围在6.05~9.42 之间,其中酸性蛋白(等电点小于7)有4 个,碱性蛋白(等电点大于7)有21 个,大部分的蛋白质呈碱性;平均脂肪族氨基酸指数为95.52,范围在85.54~105.96 之间;平均疏水性指数为-0.13,范围在-0.39~0.26 之间,除了NtDGAT1 和NtDGAT2 表现为疏水性,其余全部表现为亲水性,NtDGAT1 和NtDGAT2 可能参与了细胞膜的组成。
染色体定位分析显示(表3),有13 个烟草DGAT 基因定位到8 条连锁群上,其中19 号连锁群含有最多的4 个基因,13 和17 号连锁群分别含有2 个基因,2、3、6、21 和23 号连锁群上分别含有1 个基因。其余12 个DGAT 基因分别定位到不同的Scaffold 上。
2.3 亚细胞定位分析与跨膜区预测
亚细胞定位分析显示(表3),有16 个基因(NtDGAT1、NtDGAT2 以及NtWSD1~NtWSD14)定位到内质网,有6 个(NtDGAT3~NtDGAT8)定位到叶绿体,有3 个(NtWSD15~NtWSD17)定位到胞外。以往研究表明DGAT1 和DGAT2 亚型蛋白是一种膜结合蛋白,而且主要在内质网上,WSD亚型蛋白表现出多样性,一些是可溶性蛋白,其余是膜结合蛋白[27]。水溶性蛋白可能定位于胞外,而膜结合蛋白可能定位于内质网或叶绿体,这与本研究结果一致。跨膜区预测结果显示,NtDGAT1 和NtDGAT2 分别包含2 个跨膜区,NtWSD5、NtWSD7、NtWSD11 和NtWSD12 分别包含1 个跨膜区,其余蛋白没有检测到跨膜区。结合疏水性指数、亚细胞定位和跨膜区预测结果,说明NtDGAT1 和NtDGAT2是一种膜蛋白。
2.4 进化分析
选取水稻(7 个)、拟南芥(4 个)、油菜(11个)和大豆(13 个)的DGAT 家族基因(表1),与本研究鉴定的25 个烟草DGAT 家族基因一同构建进化树。结果显示(图1),整个进化树分为DGAT1、DGAT2、DGAT3 和WSD 4 个类群,但是烟草DGAT基因仅划分到DGAT2 和WSD 两个类群,其中NtDGAT1~NtDGAT8 划分到DGAT2 类群中,NtWSD1~NtWSD17 划分到WSD 类群中,烟草中未发现DGAT1 和DGAT3 亚型基因。大部分的烟草基因是聚在一起的,其中NtDGAT3~NtDGAT8 基因聚在一簇,NtWSD1~NtWSD13 基因聚在一簇,共形成了10 个姊妹系,这说明烟草中存在大量冗余基因,可能是在进化过程中发生了基因复制。不同的是,烟草NtDGAT1 和NtDGAT2 基因与大豆的GmDGAT2a 、GmDGAT2b 、GmDGAT2d 和GmDGAT2e 单独聚在一起, 烟草NtWSD15~NtWSD17 與大豆GmWSD1~GmWSD3 单独聚在一起,它们的功能可能更相似。而且亚细胞定位结果也显示,这些基因与其他基因的定位结果不同,这也进一步说明了这些基因的特殊性。从进化时间上看,各类群中均分布有水稻、拟南芥、油菜和大豆基因,这说明DGAT 家族基因的分化时间可能早于物种的分化时间。从亲缘性上看,油菜、拟南芥、大豆和烟草之间的亲缘关系更近,水稻与他们的亲缘关系较远,因为水稻属于单子叶植物,而其他属于双子叶植物。此外,DGAT1 和DGAT2亚型聚在一簇,其亲缘关系较近,而DGAT3 和WSD亚型分别聚成一簇,与DGAT1 和DGAT2 亚型亲缘关系较远,这与以往研究结果一致。
2.5 蛋白结构域与基因结构分析
由于WSD 亚型的主要功能是参与蜡酯的合成,其次参与少量TAG 的合成,与DGAT1、DGAT2 和DGAT3 亚型在功能和结构上差异较大,所以,本研究单独分析了WSD 亚型的蛋白结构域。结果显示(图2a),WSD 亚型蛋白检测到2 个WS_DGAT 结构域(motif1 和motif2),除了NtWSD13 缺失了motif2 和NtWSD14 缺失了motif1 外,其余的WSD基因都包含了2 个WS_DGAT 结构域(motif1 和motif2)。另外,也检测到4 个功能未知的结构域DUF1298(motif4、motif5、motif9 和motif10)。DGAT亚型蛋白检测到3 个LPLAT(LysophospholipidAcyltransferases)结构域(motif11、motif16、motif20)和一个Hydrolase_4 结构域(motif14)。其中,LPLAT是一个溶血磷脂酰基转移酶超基因家族,催化甘油磷脂的合成。LPLAT 包含了GPAT、AGPAT、MGAT、LPEAT、LPCAT、DHAPAT、TAZ 等众多成员。Hydrolase_4 是一个水解酶家族。NtDGAT1 和NtDGAT2 检测到所有的3 个LPLAT 结构域,其余的仅检测到2 个LPLAT 结构域(motif11 和motif16)。除了NtDGAT1、NtDGAT2 和NtDGAT4 外,其余均檢测到Hydrolase_4 结构域。
基因结构分析结果显示(图2b),聚成一簇的基因结构相似,WSD 亚型基因序列平均外显子数目为6.24 个,范围为2~8 个,DGAT 亚型基因序列平均外显子数目为11.25 个,范围为5~14 个。WSD亚型基因外显子数目相对较少,基因序列相对较短。这在一定程度上也反映了基因的进化关系。
2.6 基因表达分析
基因表达分析结果显示(图3),不同基因的表达模式有所差异,并表现出一定的组织特异性。基因NtDGAT1~NtDGAT8 的表达模式类似,在未开放花蕾、已开放花蕾、花顶端和花等花器官以及茎生叶中的表达量均较低,在早期、早中期、中后期、后期衰老叶、成熟叶等叶片以及幼芽中的表达量均较高,在种子、幼叶、子叶、茎尖等幼嫩组织以及上部茎中的表达量中等。但其中基因NtDGAT1、NtDGAT2、NtDGAT3 和NtDGAT5 在下部茎、成熟根、幼根中的表达量较高,而基因NtDGAT4、NtDGAT6、NtDGAT7 和NtDGAT8 在这些组织中的表达量中等。基因NtWSD13、NtWSD14 和NtWSD15除了在种子、幼叶和茎尖的表达量较低外,在其余组织中的表达情况与NtDGAT1 、NtDGAT2 、NtDGAT3 和NtDGAT5 的表达模式基本一致。基因NtWSD16 和NtWSD17 在幼芽、子叶、幼叶和茎端的表达量较高,在花和幼根中的表达量较低,在其余组织中的表达量趋于中等。基因NtWSD1~NtWSD12 的表达模式相似,并且与基因NtDGAT1~NtDGAT8 的表达模式相反,在未开放花蕾、已开放花蕾、花顶端和花等花器官以及茎生叶中的表达量均较高,在早期、早中期、中后期、后期衰老叶、成熟叶以及幼芽中的表达量均较低,在种子、幼叶、子叶、茎尖等幼嫩组织以及上部茎中的表达量中等。总体来看,DGAT 家族基因主要集中在衰老叶片和花器官中大量表达。
3 讨 论
植物DGAT基因对控制油脂的合成具有重要作用。目前,虽然在一些植物中已经发现了不同的DGAT 基因,但尚未得到全面的解释,因此,关于植物中TAG 合成的基因还需要更深入的研究。利用生物信息学方法鉴定DGAT 基因家族,并对其序列特性、基因结构、亚细胞定位、进化模式和表达模式等进行系统分析,对于更好地理解它们在TAG生物合成中的作用至关重要。
在物种进化过程中,发生过多次的基因复制和全基因组复制事件,这也助推了基因和物种的进化和多样化[28-29]。本研究共鉴定到烟草DGAT 家族基因25 个,数量仅次于花生(46 个)、油菜(35 个)和大豆(28 个)。油菜、花生和烟草都是异源四倍体,大豆是由古四倍体演变而来的二倍体,这些作物含有较多的基因数,很可能是由于在进化过程中发生全基因组复制所致。
DGAT 家族的亚细胞定位存在差异,亚型DGAT1 和亚型DGAT2 大多定位在内质网上,但是其位置并不重叠,桐树的DGAT1 和DGAT2 定位在内质网的不同点状区域,功能也没有发生冗佘[12]。大豆DGAT2a 主要定位在内质网膜和油体膜上,菠菜叶中DGAT1 主要定位在叶绿体中[30],而亚型DGAT3 是一种可溶性蛋白,存在于细胞质中[15]。亚型WSD 蛋白表现出多样性,一些是可溶性蛋白,其余是膜结合蛋白[27]。本研究中,NtDGAT1 和NtDGAT2 定位到内质网上,NtDGAT3~NtDGAT8 定位到叶绿体上,它们可能属于膜蛋白,只是大部分定位到叶绿体而非内质网,这与以往结果有所差异。此外,大部分WSD 蛋白(NtWSD1~NtWSD14)定位到内质网上,可能属于膜结合蛋白,少量WSD蛋白(NtWSD15~NtWSD17)定位到胞外,可能属于可溶性蛋白,这与以往报道一致。结合疏水性指数、亚细胞定位和跨膜区预测3 个结果,可以确定NtDGAT1 和NtDGAT2 是一种内质网膜结合蛋白。
进化分析结果显示,DGAT1、DGAT2、DGAT3和WSD 亚型分别单独聚成一簇,这说明DGAT 家族基因的分化早于物种分化时间,或者类似于DGAT1 和DGAT2,有独立的起源[14],这与以往研究结果一致[19]。研究报道,DGAT1 和DGAT2 与DGAT3 和WS/DGAT 相似性较低[14,19]。而本研究中也表现出相似的结果,DGAT1 和DGAT2 亚型聚在一簇,其亲缘关系较近,而DGAT3 和WSD 亚型分别聚成一簇,与DGAT1 和DGAT2 亚型亲缘关系较远。
DGAT 基因主要参与了油脂的积累,还参与植物种子萌发、幼苗发育和叶片衰老等生物过程中的脂类的代谢,因此,DGAT 基因应该在种子、花朵、叶片等众多组织中都有表达,但表达水平具有组织特异性。研究表明,旱金莲DGAT1 只在发育的种子中表达[6]。莲座DGAT1 基因的表达量随着叶片的衰老不断提高[31]。桐树的DGAT1 在各器官中的表达水平几乎没有差异,但DGAT2 在发育的种子中表达量极高[12]。拟南芥DGAT3 在胚根、花粉、衰老叶、叶原基、木质部的表达量较高,WSD 在花序、花、雌蕊、柱头、子房和花梗的表达量较高。烟草DGAT 家族基因表现出时空特异性表达。NtDGAT1~NtDGAT8 以及NtWSD13~NtWSD15 在不同时期的衰老叶片中表达量较高,在不同的花器官中表达量较低,这些基因可能主要参与了叶片脂类的合成。而NtWSD1~NtWSD12 的表达情况与此相反,他们可能主要参与了种子油脂的合成。
4 结 论
本研究从烟草基因组鉴定得到25 个DGAT 家族基因;其平均氨基酸长度为485,大部分为碱性蛋白和亲水性蛋白,有13 个DGAT 蛋白定位到8条连锁群上;有16 个蛋白定位到内质网,6 个定位到叶绿体,3 个定位到胞外;烟草DGAT 蛋白包含典型的WS_DGAT 结构域和LPLAT 结构域;系统发育树将DGAT 家族分为DGAT1、DGAT2、DGAT3和WSD 4 個类群,但烟草DGAT 仅划分到DGAT2和WSD 两个类群;不同烟草DGAT 基因的时空表达特性有所差异,并表现出一定的组织特异性,主要集中在衰老叶片和花器官中大量表达。
参考文献
[1] FARESE R V, WALTHER T C. Lipid droplets finally get a littleR-E-S-P-E-CT[J]. Cell, 2009, 139: 855–860.
[2] DYER J M, STYMNE S, GREEN A G, et al. High-value oils fromplants [J]. Plant Journal, 2008, 54: 640–655.
[3] LIU Q, SILOTO R M P, LEHNER R, et al. Acyl-CoA:diacylglycerol acyltransferase: molecular biology, biochemistry andbiotechnology[J]. Progress in Lipid Research, 2012, 51: 350-377.
[4] KENNEDY E P, WEISS S B. Function of cytidine coenzymes in thebiosynthesis of phospholipides[J]. Journal of Biological Chemistry,1956, 222: 193-213.
[5] ICHIHARA K, TAKAHASHI T, FUJII S. Diacylglycerolacyltransferase in maturing safflower seeds–its influences on thefatty acid composition of triacylglycerol and on the rate oftriacylglycerol synthesis[J]. Biochimica Et Biophysica Acta, 1988,958:125-129.
[6] XU J Y, FRANCIS T, MIETKIEWSKA E, et al. Cloning andcharacterization of an acylCoA-dependent diacylglycerolacyltransferase 1 (DGAT1) gene from Tropaeolum majus, and astudy of the functional motifs of the DGAT protein usingsite-directed mutagenesis to modify enzyme activity and oil content[J]. Plant Biotechnology Journal, 2008, 6(8): 799-818.
[7] HE X H, GRACE Q C, LIN J T, et al. Regulation ofdiacylglycerolacyl transferase in developing seeds of castor[J].Lipids, 2004, 39(9): 865-871.
[8] LI Q , HU L , GUO J , et al. Molecular characterization of two type Iacyl-CoA: diacylglycerol acyltransferase genes in maize[J].Biotechnology & Biotechnological Equipment, 2016, 30(3):453-461.
[9] WANG Z, HUANG W, CHANG J, et al. Overexpression ofSiDGAT1, a gene encoding acyl-CoA:diacylglycerol acyltransferasefrom Sesamum indicum L. increases oil content in transgenicarabidopsis and soybean[J]. Plant Cell, Tissue and Organ Culture(PCTOC), 2014, 119(2): 399-410.
[10] NYKIFORUK C L, FURUKAWASTOFFER T L, HUFF P W, et al.Characterization of cDNAs encoding diacylglycerol acyltransferasefrom cultures of Brassica napus and sucrose-mediated induction ofenzyme biosynthesis[J]. Biochimica Et Biophysica Acta, 2002,1580(2): 95-109.
[11] KROON J T M, WEI W, SIMON W J, et al. Identification andfunctional expression of a type 2 acyl-CoA: diacylglycerolacyltransferase (DGAT2) in developing castor bean seeds which hashigh homology to the major triglyceride biosynthetic enzyme offungi and animals[J]. Phytochemistry (Amsterdam), 2006, 67(23):2541-2549.
[12] SHOCKEY J M. Tung tree DGAT1 and DGAT2 have nonredundantfunctions in triacylglycerol biosynthesis and are localized to differentsubdomains of the endoplasmic reticulum[J]. The Plant Cell Online,2006, 18(9): 2294-2313.
[13] BANILAS G, KARAMPELIAS M, MAKARITI I, et al. The oliveDGAT2 gene is developmentally regulated and shares overlappingbut distinct expression patterns with DGAT1[J]. Journal ofExperimental Botany, 2011, 62(2): 521-532.
[14] TURCHETTO-ZOLET A C, MARASCHIN F S, DE MORAIS G L,et al. Evolutionary view of acyl-CoA diacylglycerolacyltransferase(DGAT), a key enzyme in neutral lipid biosynthesis[J]. BMC Evolutionary Biology, 2011, 11: e263.
[15] SAHA S, ENUGUTTI B, RAJASEKHARAN R R. Cytosolictriacylglycerol biosynthetic pathway in oilseeds. molecular cloningand expression of peanut cytosolic diacylglycerol acyltransferase[J].Plant Physiology, 2006, 141(4): 1533-1543.
[16] M LUISA HERN?NDEZ, WHITEHEAD L, HE Z, et al. A cytosolicacyltransferase contributes to triacylglycerol synthesis insucrose-rescued arabidopsis seed oil catabolism mutants[J]. PlantPhysiology, 2012, 160(1): 215-225.
[17] KALSCHEUER R, ALEXANDER STEINB?CHEL. A novelbifunctional wax ester synthase/acyl-CoA: diacylglycerolacyltransferase mediates wax ester and triacylglycerol biosynthesis inAcinetobacter calcoaceticus, ADP1[J]. Journal of BiologicalChemistry, 2003, 278(10): 8075-8082.
[18] LI F, WU X, LAM P, et al. Identification of the wax estersynthase/acyl-coenzyme A: diacylglycerol acyltransferase WSD1required for stem wax ester biosynthesis in arabidopsis[J]. PlantPhysiology, 2008, 148(1): 97-107.
[19] TURCHETTO-ZOLET A C, CHRISTOFF A P, KULCHESKI F R,et al. Diversity and evolution of plant diacylglycerol acyltransferase(DGATs) unveiled by phylogenetic, gene structure and expressionanalyses[J]. Genetics and Molecular Biology, 2016, 39(4): 524-538.
[20] FREGA N, BOCCI F, CONTE L S, et al. Chemical composition oftobacco seeds (Nicotiana tabacum L.)[J]. Journal of the AmericanOil Chemists' Society, 1991, 68(1): 29-33.
[21] 侯小東,张怀宝,张忠锋,等. 烟籽油成分分析及体内抗氧化作用研究[J]. 中国烟草科学,2016,37(4):80-84.HOU X D, ZHANG H B, ZHANG Z F, et al. Analysis of tobaccoseed oil and its antioxidant activities in vivo[J]. Chinese TobaccoScience, 2016, 37(4): 80-84.
[22] 杨琼,侯小东,刘艳华,等. 不同类型烟草种子蛋白质、脂肪及其主要活性成分分析[J]. 中国烟草科学,2019,4(6):95-102.YANG Q, HOU X D, LIU Y H, et al. Analyses of protein, fFat andmain active components in the seeds of tobacco (Nicotiana spp.)types[J]. Chinese Tobacco Science, 2019, 40(6): 95-102.
[23] PARLAK A, KARABAS H, OZSERT I, et al. Effects of tobaccoseed oil methyl ester on performance and exhaust emissions of dieselengine[J]. Journal-Energy Institute, 2016, 86(3): 147-152.
[24] ZHANG F Y, YANG M F, XU Y N. Silencing of DGAT1 in tobaccocauses a reduction in seed oil content[J]. Plant Science (Oxford),2005, 169(4): 689-694.
[25] ROZANA R, PEK-LAN C, KUANG-LIM C, et al. In Silico,characterization and expression profiling of the diacylglycerolacyltransferase family (DGAT1, DGAT2, DGAT3 and WS/DGAT)from oil palm, Elaeis guineensis[J]. Plant Science, 2018, 275: 84-96.
[26] 郑玲,单雷,李新国,等. 花生DGAT 基因家族的生物信息学分析[J]. 山东农业科学,2018,50(6):10-18.ZHENG L, SHAN L, LI X G, et al. Bioinformatics analysis of peanutDGAT gene family[J]. Shandong Agricultural Sciences, 2018, 50(6):10-18.
[27] STOVEKEN T, KALSCHEUER R, MALKUS U, et al. The waxester synthase/acyl coenzyme A: diacylglycerol acyltransferase fromAcinetobacter sp. strain ADP1: characterization of a novel type ofacyltransferase[J]. Journal of Bacteriology, 2005, 187(4): 1369-1376.
[28] NYSTEDT B, STREET N R, WETTERBOM A. The Norway sprucegenome sequence and conifer genome evolution[J]. Nature, 2013,497: 579-584.
[29] MICHAEL T P, VANBUREN R. Progress, challenges and the futureof crop genomes[J]. Current Opinion in Plant Biology, 2015, 24:71-81.
[30] MARTIN B, WILSON R F. Properties of diacylglycerolacyltransferase from spinach leaves[J]. Lipids, 1983, 18(1): 1-6.
[31] KAUP M T, FROESE C D, THOMPSON J E. A role fordiacylglycerol acyltransferase during leaf senescence[J]. PlantPhysiology, 2002, 129(4): 1616-1626.
