染色质三维构象在胃癌发生发展中的研究
2020-03-15邱劲军朱鹏曾启昂李凤艳
邱劲军,朱鹏,曾启昂,李凤艳
(深圳市坪山区人民医院,南方医科大学附属坪山总医院,广东 深圳)
0 引言
胃癌(gastric cancer, GC)是消化道常见的恶性肿瘤之一,是世界第四大常见恶性肿瘤,每年大约有70万人死于胃癌。我国是胃癌高发国家,每年新增大约40万胃癌患者(约占全世界胃癌发病人数的1/3),且呈年轻化趋势[1-2]。胃癌早期症状多缺乏特异性,I期仅有10%的检出率[3]。多数胃癌患者被确诊时已经发展至中晚期或伴有淋巴、血液转移,其五年生存率低至1/5[4]。胃镜检查是诊断胃癌的“金标准”,但因患者不适反应多,依从性差,限制了其作为胃癌筛查的应用。上消化道钡餐透视、CT和磁共振等影像学检查已广泛应用于胃癌诊断,但早期胃癌和癌前病变的影像学表现常缺乏特异性,故影像学检查在发现早期胃癌及微小病灶方面有一定的局限性。而传统的抗原类肿瘤标记物多在胃癌晚期才明显升高,且其阳性率低于40%[5]。这些传统的技术不利于肿瘤的早期诊断,临床上仍然缺乏有效的胃癌早期预警诊断方法。
胃癌的发生发展涉及多个因素,病程复杂,其发病机制尚未完全清楚。较多研究表明胃癌的发生和发展涉及基因功能的异常改变,而基因功能的改变可能与基因碱基突变和表观遗传学改变有关。这些改变会引起抑癌基因和原癌基因活性与功能的改变,从而导致胃癌的发生和发展。多数胃癌患者因发现晚而已经失去最佳治疗时机。如果临床上能及时发现早期胃癌患者,并判断其危险程度,凭借目前的医疗技术可以有效地提高患者的生存率。因此,进一步了解胃癌的遗传学分子机制,对提高胃癌的早期筛查和诊断有着十分积极的意义。目前,胃癌发病的分子机制中研究得较多的是DNA甲基化、错配修复基因等[6],这些研究基本都是针对单个基因的检测。近年来,基因组的三维空间构象和功能逐渐成为基因组学研究的新热点[3]。传统的DNA测序技术通常会将基因的表达形式描述成线性,实际上,基因除了成线性排列外,基因的两侧可能会互相成线性,非常接近三维立体的形式。染色体构象捕获(Chromosome Conformation Capture,3C),是一种研究细胞核的实验方法,通过高通量测序技术和生物信息学方法,深入分析整个染色质DNA在空间位置上的关系。本文从细胞染色质三维构象角度,探讨其与胃癌发生发展之间的关联,同时展望了细胞染色质三维构象等表观遗传图谱在胃癌临床诊治中的应用前景。
1 胃癌分子机制的研究现状
多年来,人们通过系统研究肿瘤基因组发现,癌症是由细胞不受控制的生长和扩散而引起的一大类恶性疾病,其特征是基因组的各种突变,其中包括点突变、小插入/缺失、染色体重排、拷贝数变异。癌症基因组图谱(TCGA)描述了许多类型癌症的点突变和结构变异[6]。常见的突变基因包括TP53、KRAS和PIK3CA。
这些基因与主要的细胞增殖途径相关,包括MAPK信号、PI(3)K信号和转录调控。后续也有研究表明,癌症会发生表观遗传学的改变[7]。目前,胃癌的发病机制中研究得较多的是DNA甲基化、错配修复基因等分子机制,但仍旧未完全阐释清楚胃癌的发病机制。越来越多研究报道,染色质三维结构在基因表达调控,细胞分化,以及疾病发生等过程中起着至关重要的作用。
(1)DNA甲基化
DNA甲基化是一种重要的表观遗传,也是基因活性抑制的一个重要的机制,以s-腺苷甲硫氨酸为甲基供体,将甲基转移到特定的碱基上的一种反应。DNA甲基化会抑制肿瘤基因被或沉默转录,在肿瘤的发生的早期阶段就发挥着重要的作用[8]。随着整体测序的完成,人们发现在很多肿瘤抑制基因的启动子区存在着大量CpG岛,提示DNA甲基化可能在肿瘤抑制基因失活方面发挥着重要作用。已有多个研究表明表观遗传学修饰在胃癌的发生过程中扮演着重要的角色。目前已有多个研究报道,DNA甲基化直接参与了胃癌肿瘤细胞分化、细胞周期、肿瘤转移等过程 ,提示DNA甲基化有可能参与了胃癌发生的整个过程[9,10]。谷胱甘肽过氧化物酶GPX3是GPX的一种重要的细胞亚型,GPX3基因启动子CpG岛的高甲基化可使GPX3的蛋白表达下调,导致体内过氧化物含量增多,参与胃癌的发病[11]。现已确认EBV感染与胃癌发生密切相关,初步研究证实EBV感染可诱导相关抑癌基因启动子区呈高甲基化状态,这是EBV引起肿瘤抑制基因功能失活的重要机制之一[12],但其具体机制有待进一步的研究。有研究分析胃癌组织发现,细胞中3号染色体短臂(3p)抑癌基因启动子区CpG岛呈高度甲基化。这些发现可能为胃癌的早期诊断和预后监测等方面提供重要帮助。
(2)错配修复基因系统
错配修复(mismatch repair,MR)系统属于一种DNA复制后的修复系统,由一系列保守基因组成,具有修复错配DNA碱基、增强DNA复制准确性、维持基因组稳定性、降低自发突变的功能。它的缺陷导致基因组不稳定而对肿瘤易感。[13-15]。DNA错配修复(MMR)系统是保证基因组相对稳定的重要组成部分,由一系列特异性修复DNA碱基错配的MMR基因调控的酶分子组成,在保持遗传物质的完整性方面起重要作用。细胞一旦出现MMR功能缺陷,就会引起基因突变的累积,最终导致肿瘤发生。错配修复基因hMLHl能识别多种基因的突变并加以修复,当细胞内突变基因积累时,hMLHl蛋白表达上调,这可能是由于基因突变在癌旁黏膜上皮细胞有所积累的结果[14]。胃癌旁黏膜上皮出现hMLHl蛋白核表达增高,提示具有该蛋白高表达的胃黏膜上皮细胞可能是易发生癌变细胞。胃黏膜上皮细胞核hMLHl蛋白的表达显著高,可能是胃癌发生的前兆,该蛋白在细胞核和细胞浆同时出现可能是胃癌发生的标志[15]。检测胃黏膜细胞核hMLHl蛋白的表达可能有助于临床预警胃癌的发生及判定胃癌的高危人群。
(3)遗传与表观遗传失调对染色质结构产生影响
在染色质组成完整的情况下,非编码基因的遗传或表观遗传失调可以对染色质三维结构产生深远的影响。尤其是,转录因子结合位点的序列或表观遗传环境的变化可能改变染色质的交互作用,这能深远影响到基因的表达和细胞身份的表达。全基因组多态性关联分析研究发现,相隔甚远的调控元件之间也能发生一种很强的相互作用[16]。后续有研究关联了这些疾病相关的非基因区域的多态性,发现该区域存在的调控因子参与染色质的形成和成环[17,18]。最近有研究发现,结直肠癌(colorectal cancer,CRC)的某一特定基因亚型存在CCCTC结合因子(CCCTC-binding factor,CTCF)结合位点突变优势[19]。而且,这些CTCF结合位点突变与一种已被证实的癌症突变信号AT>GC突变高度相关,并且这种信号富集在CTCF保守序列的特定位置上[20]。在大样本中CTCF结合位点突变呈单峰分布,类似地,小样本中的大多数突变发生在CTCF结合位点上[21]。在一个全基因组研究中,CTCF结合位点被耦合到延迟复制定时域,并且已有研究报道这些CTCF/粘连蛋白结合位点不是被主导DNA多聚酶复制而是有另外的非典型多聚酶复制的[22]。综上所述,这些结果提示部分CRC患者在修复CTCF结合位点突变方面可能存在全局性缺陷。而且,这种突变特征并不是CRC独有的,在其他多种癌症类型中也观察到了。
CTCF结合模式也会受到其结合位点表观遗传修饰的影响。CTCF保守结合序列的DNA甲基化已被证明可以控制细胞类型特异CTCF结合,说明CTCF结合可以通过可逆的表观遗传改变来调节[23]。因为DNA甲基化的破坏是多种癌症的标志,DNA甲基化的改变可能直接或间接影响CTCF的结合。一项关于IDH突变型胶质瘤的研究发现,CTCF结合位点的高甲基化与重要结构域边界的溶解和强致癌基因的异常表达有关[24]。以上研究表明,DNA甲基化的改变是癌症中染色质结构被破坏的一种潜在机制。所以,表观遗传学、染色质结构以及基因突变对细胞基本生命活动都具有重要影响,也可能是诱导癌症等疾病的关键因素。
2 染色质DNA构象的研究
DNA测序技术是分子生物学研究中最常用的技术,它的出现极大地推动了生物学的发展。自从1953年Watson和Crick发现DNA双螺旋结构后,人类就开始了对DNA序列的探索,在世界各地掀起了DNA测序技术的热潮[25]。1977年Maxam和Gilbert[26]报道了通过化学降解测定DNA序列的方法。同一时期, Sanger[27]发明了双脱氧链终止法。20世纪90年代初,荧光自动测序技术的出现将DNA测序带入自动化测序的时代。这些技术统称为第一代DNA测序技术。最近几年发展起来的第二代DNA测序技术则使得DNA测序进入了高通量、低成本的时代。目前,基于单分子读取技术的第三代测序技术已经出现,该技术测定DNA序列更快,并有望进一步降低测序成本,推进相关领域生物学研究。
然而,即使解码了几乎所有的遗传信息,人们认识到线性的DNA序列尚不能去解释整个基因组是如何指导复杂生命过程完成的。实际上,编码在DNA序列上的遗传信息的表达很大程度上依赖于其所处的染色质环境——染色体空间结构。科学家们再次将研究方向转到最初的染色体结构研究,试图结合宏观水平(基因组空间结构)去理解微观水平上基因功能。大量证据表明基因组三维空间结构及其变换具有一定的规律性,并且与细胞核一些基础功能(如基因转录、基因复制、基因易位等)密切相关[28-30],深入理解基因组三维空间结构信息对于认识基因表达调控具有重要的意义。基因组的空间结构是由染色体内和染色体间错综复杂的交互形成并由细胞核内部元件介导,参与这一过程核内部调控元件包括转录因子、转录和复制工厂、多家族蛋白体以及核纤层的交联等。
在过去的几十年里,一系列分子生物学和基因组学技术的发展使得人们能够更加深入地去认识基因组的3D结构,这些技术包括荧光原位杂交(FISH)和染色质构象捕获(3C)及其衍生技术。传统测序是基于核苷酸的一维排列不同,而3C及其衍生技术测序是基于染色质的三维构象,如图1所示。近些年来,结合3C技术和高通量测序技术,再加上新的数据分析方法,能够在全基因组范围内捕获不同基因座之间的空间交互,揭示基因组空间结构的规律性及其与基因调控之间的关系。然后,基于这些高通量交互数据,进一步采用数学建模方法,极大拓展了我们对基因组空间结构的认识。Duan等人[31]首次预测了酿酒酵母基因组的空间三维结构,揭示酵母基因组也具有层次化结构。Bau等人[32]通过对人类第16条染色体中包含alpha-globin的基因域进行研究,发现该基因域也具有球形结构:活化基因在空间会聚集在球形中央,而非活化基因则零散地分布各个基因环(gene loop)上。Erceg等[33]利用Hi-C技术通过测量人类正常淋巴细胞染色体中基因座空间交互信息,证实了染色体地域(chromosome territories,CTs)的存在。此外,他们还发现该细胞基因组在空间结构上呈现出开合和闭合两种相互隔离的构象区域。进一步,他们还通过生物物理模型预测这个基因组在空间上具有一种分形的三维结构。

图1 左边为传统DNA测试,右图为3C技术测序
通过探究基因组的空间三维结构,发现染色体空间结构和基因共表达之间具有十分重要的联系。这些实验及计算结果对揭示基因组空间层次结构起到了重要作用,但是它们无法确定究竟是何种基因相互结合在一起,空间结构又是如何对基因实施具体调控的。Bellush等人[34]使用新技术对人类基因组中的特定调控因子,雌激素a受体的空间交互信息进行了研究,发现绝大多数的结合位点都通过远程的染色体空间交互而聚集在一起。进一步的实验表明,这些聚集在一起的基因比其它基因具有更高的表达水平,证明了基因组空间三维结构与基因调控之间具有直接的关联。
基于高通量染色质交互数据,通过层层剖析,从分子水平层面初步揭开染色体复杂空间结构隐藏的特性:短程基因座交互数量庞大,形成初级环化过程;远程基因座交互,如增强子和启动子、启动子和启动子之间的交互,将不同的短程环连接一起,形成高级环化过程(A类和B类结构域);最后,少量的超远程基因座交互将不同层次的空间结构进一步结合在一起,形成更高层次的复杂空间结构,如图2所示。这些研究为更加深入地理解基因组三维空间特征,寻找易感基因[35-37]。
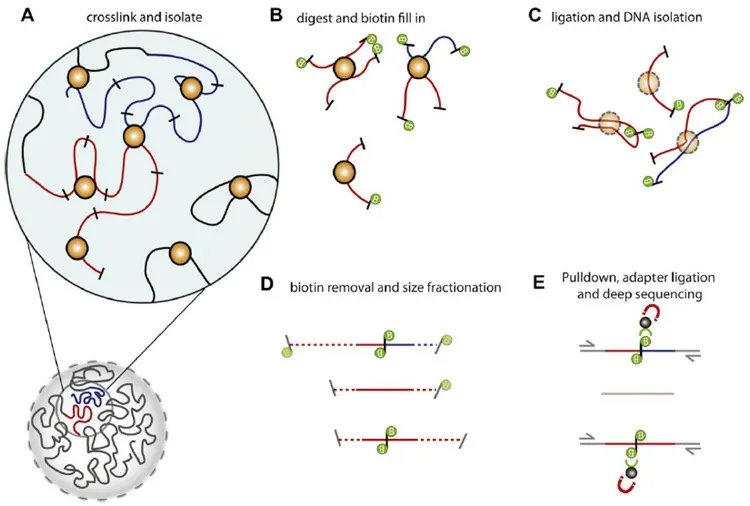
图2 细胞核空间结构Hi-C测序原理示意图
3 染色质三维结构异常导致癌症
虽然近几十年来,我们已经了解了很多关于癌症中的基因组序列如何发生突变,但我们对基因组三维结构在癌症发展中的作用知之甚少。 近年来,多项研究对乳腺癌、前列腺癌、胶质瘤、结直肠癌、多发性骨髓瘤以及多种血液癌症的基因组三维结构进行了分析[38-45]。研究发现,癌症中基因组在A/B结构域切换、TADs和染色质相互作用等三维结构上发生了改变[46]。(1)染色质中发生A/B结构域转换失调。在乳腺癌、多发性骨髓瘤、B细胞淋巴瘤和T细胞急性淋巴细胞白血病中,多达20%的基因组区域存在结构域转换(由A到B,反之亦然)[38,41-45]。正常乳腺上皮细胞(MCF-10A)和乳腺癌(MCF-7)细胞系的Hi-C分析显示,癌细胞中基因组的三维结构与正常细胞中存在明显差异[38]。与正常细胞相比,乳腺癌中大约12%的基因组区域有A/B结构域切换。 此外,从A结构域到B结构域的转换与基因表达下调有关,而结构域的切换改变方向则会引起基因表达上调。考虑到染色质结构域的映射算法,这种结构域切换更可能反映了癌细胞中基因表达的变化,而不是在癌症发展中起着致病作用[39]。(2)TADs数量和边界发生改变。在TADs水平看,虽然不同的癌症类型的变化特征各不相同,但健康细胞和癌细胞之间的差别特别明显。据研究报道,乳腺癌、前列腺癌、多发性骨髓瘤等类型的癌症的TAD边界发生了变化,通常伴有相应的TAD数量的增加和大小的减小。对于健康细胞和癌细胞,肿瘤细胞中TADs数量的增加程度可能有很大差异[38,40,42]。与正常的B细胞相比,多发性骨髓瘤中TADs的数量增加了25%,而前列腺癌细胞系中TADs的数量是正常前列腺上皮细胞的2-3倍。Taberlay等[42]人发现在前列腺癌中,基因组三维结构是处于无组织状态,同时发现前列腺癌细胞比正常细胞有更多的TAD边界和更小的平均TAD长度,而且许多癌症特异性区域的边界出现在拷贝数变异的区域。此外,长程染色质相互作用的变化与表观遗传修饰和基因表达的变化一致。胶质瘤、急性淋巴细胞白血病TAD界限的减弱和/或消失更为典型[41]。Wu等[44]人通过整合Hi-C、全基因组测序(whole genome sequencing, WGS)和RNA-seq数据来比较多发性骨髓瘤细胞和正常B细胞也有类似的发现。对于胶质瘤、急性淋巴细胞白血病等类型的癌症,TAD界限的减弱和/或消失更为典型。B细胞淋巴瘤中TADs的数量和结构无明显差异[41,45]。(3)染色质的相互作用异常。在乳腺癌细胞中,16号染色体与22号染色体等小而富含基因的染色体之间的相互作用频率低于正常细胞。染色体内的相互作用也不同:正常细胞中端粒和亚端粒区域的相互作用比乳腺癌细胞中更频繁[44,46]。
4 应用前景
目前,胃癌已经成为我国城市居民死亡的重要原因。据统计,我国该病发病率在20年间升高近50%,而近80%的胃癌在发现时已经是中晚期,且生存率极低[4]。如何早期发现胃癌,已经成为医学界的关注重点。与国际水平相比,国内一些大型医院的设备和诊治水平已经接近或达到发达国家水平,但对胃癌防控的能力严重不足,缺乏胃癌的早期诊断技术。最新研究报道40%胃癌患者是可以通过预防而不得癌症的,40%患者可以通过早发现、早诊断、早治疗而治愈,20%患者可以带癌生存,这充分说明了癌症早发现早诊断的必要性和重要性[47]。但目前的检测手段存在有创性,不利于胃癌早期诊断,难以大规模人群普查,可见,实现胃癌早期无创检测是亟需解决的重大科学问题。新一代DNA测序技术是高通量特性进行测序分析,能够发现血浆中微小的游离DNA变化,同时结合生物信息分析技术,能够实现对胃癌早期诊断、个性化治疗和治疗预后的监测。因此,利用高通量测序方法,将胃癌基因组三维结构图绘制出来,比较正常组织与胃癌患者组织基因组的三维结构,定位胃癌组织中基因组结构的变化,并进行系统分析,找到所有致癌和抑癌基因的微小变异,了解胃癌癌细胞发生、发展的机制,为胃癌的诊断、治疗奠定基础。建立该研究技术平台,开发出实体胃癌及血液系统胃癌的早期诊断和治疗监测试剂盒,构建自动化生物信息学分析平台,将有助于实现对胃癌早期诊断、个性化治疗、治疗预后的监测。
