我国75份小麦品种SNP和SSR指纹图谱构建与比较分析
2020-03-15刘丽华刘阳娜张明明李宏博庞斌双赵昌平
刘丽华, 刘阳娜, 张明明, 李宏博, 庞斌双, 赵昌平
(北京市农林科学院北京杂交小麦工程技术研究中心, 杂交小麦分子遗传北京市重点实验室, 北京 100097)
小麦是我国的第二大口粮作物,优良新品种的选育和推广对国民经济发展与人民生活水平提高具有重要意义。截至目前,在中国种业网的大数据平台上(http://202.127.42.145/)可查询到的小麦审定品种3 766个。随着近年我国《主要农作物品种审定办法》的改革,品种审定渠道拓宽,每年参加区试统一试验或联合体的小麦品种已达2 000余份。不同生态区过度集中利用少数骨干优良亲本导致育成品种的同质化严重,遗传背景趋于狭窄。传统的田间种植鉴定周期长,易受环境因素的影响,耗时费力。因此,面对品种数量多、同质化严重以及鉴定方法的局限性等问题,准确有效地鉴定小麦品种成为小麦品种管理、市场监管、执法和司法仲裁等首要难题。以SSR和SNP分子标记为代表的DNA 指纹鉴定技术具有准确可靠、简单快速、易于自动化的优点,是国内外公认的农作物DNA指纹鉴定技术,通过构建农作物标准样品的DNA 指纹及DNA指纹数据库管理系统能够实现对农作物品种进行精准快速的鉴定。
SSR标记为第二代分子标记,具有数量丰富、多态性高、操作简单、成本低等优点,被国际植物新品种保护联盟(International Union for The Protection of New Varieties of Plants, UPOV)推荐为作物品种鉴定的优选标记[1],SSR标记技术历经二十余年的发展,在品种鉴定中的研究与应用已基本趋向成熟。截至目前,我国已建立了小麦[2]、玉米[3]、水稻[4]等15种作物SSR标记技术的品种鉴定标准,并广泛应用于作物标准样品指纹图谱构建,为我国农作物品种管理和质量监管提供了有力技术支撑。SNP标记为第三代分子标记,通常为双等位基因,较SSR标记具有数据统计简单、数据兼容性强、通量高等优点。国内外学者已经开始研究基于SNP标记的指纹图谱构建和品种鉴定技术,并取得了一定效果。赵仁欣等[5]利用油菜60K Illumina SNP芯片成功构建了我国224份冬油菜参试品种DNA 指纹图谱,筛选出5 374个SNP 标记可用于甘蓝型油菜品种特异性和一致性的鉴定分析;朱国忠等[6]利用棉花 KonSNP80K芯片对 326 份不同来源的陆地棉种质进行 SNP分型,筛选出适于陆地棉品种指纹图谱绘的核心位点4 857个,99%以上的陆地棉材料均能够被准确鉴定。北京市农林科学院玉米研究中心利用Maize SNP384 芯片完成4 632份审定玉米品种的SNP 指纹库构建[7];魏中艳等[8]选用分布于13 个基因的23个SNP 标记对599 份大豆表型精准鉴定种质进行基因型分析,筛选出14个SNP用于品种鉴定,并构建了100份种质可扫描身份证。Shirasawa等[9]研究发现, 利用8个SNP标记可以区分43个水稻品种。然而,对于异源六倍体小麦来说,其基因组复杂,基因组测序完成和SNP芯片开发较晚,在小麦品种鉴定中的应用相对滞后,主要停留在评估芯片检测平台、遗传多样性和群体结构等方面的研究[10-13],目前,还没有同时利用SNP和SSR构建小麦DNA指纹图谱及两者技术特征比较的相关报道。本研究以96份代表样品为材料,利用 Illumina 公司的小麦90K SNP芯片进行基因分型,筛选适于小麦指纹图谱构建的SNP标记;以筛选出的SNP标记和标准[2]中的SSR标记构建代表品种的指纹图谱,并进行比较分析,以期为今后小麦品种DNA指纹检测技术的选择、推广和应用奠定基础。
1 材料与方法
1.1 试验材料
小麦样品共96份(表1),由北京市农林科学院北京杂交小麦工程技术研究中心的低温种质库提供。其中育成品种75份(编号1~75,为不同生态区骨干亲本、生产上大面积推广应用品种等)、地方品种3份(编号76~78)、育种中间材料10份(编号79~88)、杂交小麦及其双亲3份(编号89~91)、DH系样品1份(编号92),为区分纯合SNP位点的内参以及4个品种的第二次重复(重复内参,编号93~96)。

表1 96份代表样品信息Table 1 Information of 96 representative samples
1.2 基因组DNA提取
每份材料取50粒种子,置于培养皿浸种至露白(36 h左右),剥取胚芽30个,放入2.0 mL离心管中,利用植物基因组DNA提取试剂盒(天根生化科技有限公司)提取其基因组DNA,用0.5×TE溶解保存。DNA质量和浓度用紫外分光光度计和琼脂糖凝胶电泳进行测定,要求DNA样本浓度在50 ng·μL-1以上,OD260/OD280应在1.7~2.1之间,DNA总量应大于4 μg。
1.3 SNP基因分型
利用 Illumina公司的Wheat 90K iSelect Bead Chips全基因组芯片(SNP位点数目为81 587个)对96份样本进行分析。在获得原始数据后将其直接导入GenomeStudio软件进行基因分型,获得每个样本每个位点的SNP基因分型数据。
1.4 SSR基因分型
基于毛细管电泳平台,采用农业行业标准《主要农作物品种真实性SSR分子标记检测 普通小麦》(NY/T2859—2015)[2]中的42对SSR引物构建75份育成品种的指纹。采用仪器自带的Date Collection Ver.1.0软件收集原始数据并形成FSA文件。采用北京市农林科学院玉米研究中心开发的SSR指纹分析器(软件登记号:2015SR161217)对FSA 文件进行分析,采集指纹数据。
1.5 统计分析
以75份育成品种为材料,利用Powermaker V3.25软件[14]分析SNP和SSR位点的遗传多样性指数,包括基因多样性、多态性信息量(polymorphism information content,PIC)和最小等位变异频率(minimum allelic variation frequency,MAF)等。计算品种间的Nei’s(1983)遗传距离,并进行UPOVE聚类分析。利用Excel计算SNP和SSR位点的鉴别力(discrimination power,DP)。采用杜邦先锋公司提供的Uniqueness软件和Minimal marker软件[15]进行统计分析(基于组合最优化算法),获得小麦品种鉴定最少的SNP和SSR位点组合。
2 结果分析
2.1 适于指纹图谱构建的SNP位点组合筛选
2.1.1SNP位点的筛选 从81 587个SNP标记中筛选出得分值在0.60~1.00 之间的标记共49 539个(占比60.7%);剔除不具备重复性、不符合遗传规律、不可靠、无多态性的位点,剩余14 940个SNP标记;剔除仅有AA或BB基因型、三种基因型所占面积比例不正常、缺失率较高、整体信号较弱、杂合率较高、连锁的标记,最后筛选出高质量、单拷贝、无连锁的理想位点384个(图1),用于指纹图谱构建。
2.1.2SNP位点的分布 384个SNP位点的染色体分布如图2所示,总体上来看,D染色体组以及同源群3和4上的标记比较少,可能与90K芯片中D染色体组以及第3和4同源群上的标记比较少有关。
2.1.3SNP位点的多态性分析 SNP位点的PIC值变化范围为0.07~0.38,平均0.33。MAF值(图3)变化范围为0.04~0.50,平均0.33,83%的位点MAF值大于0.2,表明筛选出的大部分SNP位点多样性较高。
2.2 SSR和SNP标记指纹图谱分析
利用筛选出的384个SNP标记组合和42个SSR标记组合对75份育成品种进行基因分型并获得指纹图谱。对于SNP位点,指纹图谱格式采用SNP变异实际的A/T/C/G碱基字母直接编码,如某品种在某个位点上的特征编码为“AA”。对于SSR位点,指纹图谱格式采用等位变异名称表示,如某品种在某个位点上特征编码为“223/223”。固定SNP或SSR标记顺序,串联每个品种固定标记的特征编号,即为每个品种的特征指纹图谱。表2列出了不同麦区4份代表性样品12个位点的指纹,指纹图谱的构建为品种识别及下一步分析鉴定了基础。
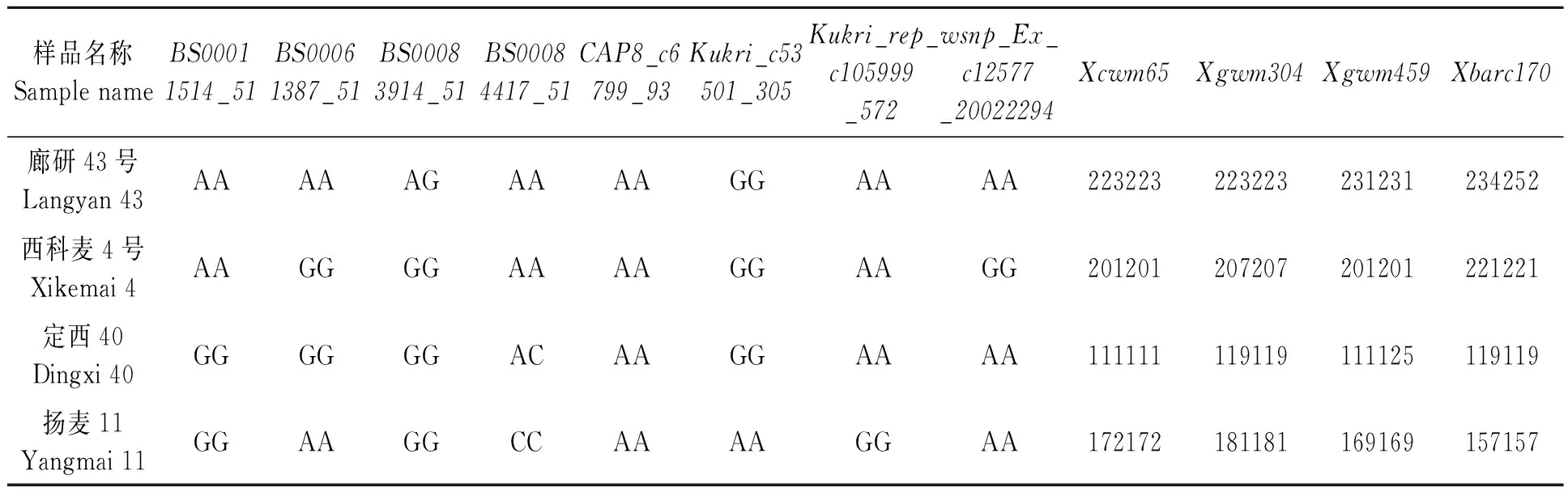
表2 4份样品的指纹信息Table 2 Fingerprint information of four samples
2.3 基于SNP和SSR标记的遗传多样性比较
基于SNP和SSR指纹图谱,分别计算多样性各参数。SNP位点的平均等位变异数为2,基因多样性和PIC值变化范围分别为0.07~0.50和0.07~0.38,平均值分别为0.41和0.33。SSR位点的平均等位变异数为9.5,基因多样性和PIC值变化范围分别为0.56~0.87和0.49~0.81,平均0.75和0.72。SNP标记揭示的遗传多样性各参数明显比SSR标记低,但也能较好地反映品种间的遗传多样性。
2.4 基于SNP和SSR标记的的遗传相似系数比较
2.4.1遗传相似系数比较 基于SNP和SSR指纹图谱,计算各育成品种之间的遗传相似系数(genetic similarity,GS),结果如图4所示。基于SNP标记,各品种之间的GS变化范围为0.45~1.00,平均0.62。廊研43号与扬麦158之间的遗传相似系数最小为0.45,农大211和农大212的遗传相似系数最大为1.00。基于42个SSR标记,各品种之间的GS变化范围为0.02~0.99,平均0.25。烟农19号和舜麦1718之间的遗传相似系数最大为0.99,宁麦8号与农大3432的遗传相似系数最小为0.02。SNP揭示的遗传相似系数高于SSR标记。
2.4.2遗传相似系数相关性分析 利用两种标记对75份育成品种进行遗传相似系数相关性分析,结果(图5)显示,利用384个SNP位点组合与42个SSR位点组合的鉴定结果呈极显著线性相关(P<0.01),说明两者之间一致性较好。
2.4.3聚类分析 基于品种间的遗传相似系数绘制SNP和SSR聚类图(图6),结果显示,编号为14和70、48和68、34和71、56和51以及1、63和69的品种均在聚类图中优先聚在一起,说明两种标记之间一致性较好。
2.5 两种标记的鉴别能力比较
基于SNP和SSR指纹图谱,计算两种标记的鉴别力(DP)。SNP位点的DP值变化范围为0.08~0.64,平均0.49,明显低于SSR位点(DP值变化范围为0.59~0.89,平均0.77)。基于遗传相似系数分析结果,42个SSR位点组合能将75份育成品种区分开,而384个SNP位点组合仅能区分74份育成品种,不能区分的品种为农大212(农大211高度相似姊妹系),说明筛选出的384个SNP位点组合鉴别近等基因系的能力低于42个SSR位点组合。去除农大212后,基于最优化算法,发现仅需8个SNP位点组合或4个SSR位点组合(表3)即可区分剩余的74份育成品种,说明8个SNP位点组合与4个SSR位点组合对本批实验材料的鉴定效率一致。
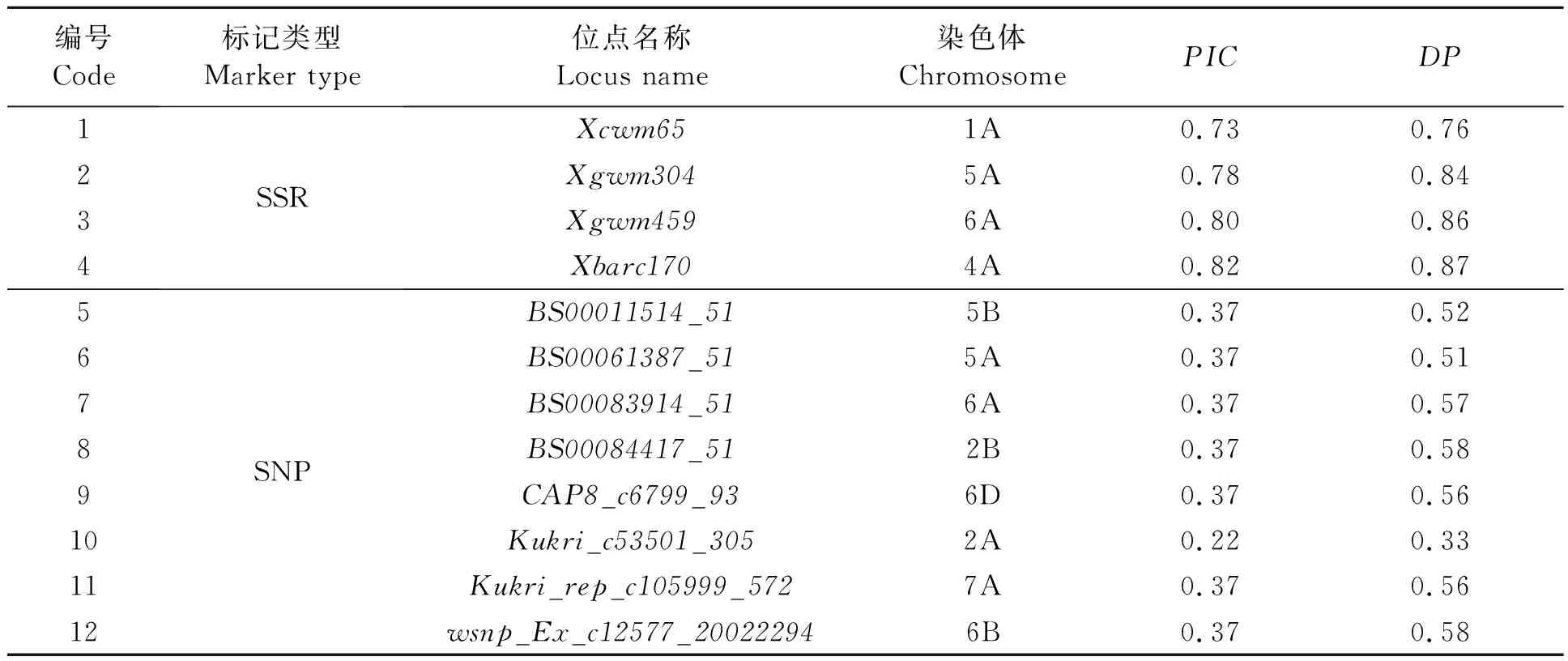
表3 8个SNP和4个SSR位点信息Table 3 Information of 8 SNP and 4 SSR loci
3 讨论
筛选重复性、稳定性和多态性好的高质量SNP标记是构建农作物高质量指纹和准确高效鉴定品种的前提。由于品种鉴定是为执法、司法仲裁等提供证明性作用的结果报告,因此其对SNP标记质量的要求远比遗传多样性分析和遗传定位等研究要高。对于异源六倍体小麦而言,所需的SNP标记除了满足“符合孟德尔遗传规律、无连锁、重复性好、稳定性好、分布较均匀、分辨率高、PIC较高、易实现高通量”等二倍体作物普遍要求外,还应为单拷贝位点。由于多倍体特性,绝大部分标记是多拷贝的,不利于基因型的准确分型、记录、统计、自动化分析等,直接影响检测的准确性和通量。本研究所用的Illumina 90K SNP芯片中的绝大部分SNP位点在基因组上具有多个拷贝,而分型软件主要是针对于二倍体开发的,无法实现准确分型。后期虽然开发了多倍体插件,但分型不能实现自动化,而且不利于基因型的记录和统计。本研究通过筛选单拷贝SNP位点,将六倍体作物二倍体化,实现了SNP标记的准确和自动化分型,为构建准确可靠的指纹图谱奠定了坚实的基础,为其他多倍体作物提供了借鉴。
SSR和SNP均具备共显性、高多态性、在基因组上均匀分布、高准确性、较好的重复性、较高通量、易实现自动化、成本低等优点,但各有优劣。相比之下,SSR 拥有更高的多态性,分辨力高;SNP标记在基因组中更为丰富,易实现数据共享、自动化、通量更高的优点。基于指纹图谱,通过比较发现SNP标记揭示的遗传多样性各参数低,但均能较好的反映小麦遗传多样性;SNP标记揭示的遗传相似系数高,但两者呈显著线性相关,聚类图验证了相关结果,说明两者之间一致性较好,与Wurschum 等[10]的研究结果不一致。他们认为基于SNPs和SSRs揭示的遗传相似性无显著的相关性,可能与研究材料和标记数量不一致有关。在品种鉴定效率方面,384个SNP位点鉴定近等基因系(农大211和农大212)的能力不如42个SSR位点。分析原因,农大211和农大212来自于同一个亲本组合,农艺性状方面仅有粒色差异,而90K芯片中可供筛选的SNP标记数量有限,筛选出的384个SNP位点又与粒色无关,因此为满足后续大规模检测与指纹数据库构建的需要,还需进行大量SNP标记的开发与筛选工作,加入更高密度的标记或者功能标记,作为扩展位点,以达到理想的品种鉴定效果。研究发现去掉农大212后,仅需8个SNP标记和4个SSR标记即可区分剩余的74份品种,说明8个SNP位点组合与4个SSR位点组合鉴定效率一致。虽然SNP和SSR位点平均等位变异数分别为2和9.5,相差将近5倍,但利用位点组合法鉴别同一批品种所需的位点数仅相差2倍,说明位点组合的优势非常明显,鉴定效率更高。另外,研究结果也说明利用SSR和SNP两种技术进行品种身份鉴定时,兼顾成本、准确性和时效性,可先采用少量位点进行初检,对于一些极近似品种,再增加高密度位点检测。
