基于非洲菊转录组测序的SSR位点信息分析
2020-03-08林发壮李锦烨安慧珍黄琦姚凤琴钟琳珊陈昌铭郭芸玮
林发壮 李锦烨 安慧珍 黄琦 姚凤琴 钟琳珊 陈昌铭 郭芸玮


摘 要:为深入开发非洲菊SSR分子标记,进而推动非洲菊遗传多样性研究,以云南红非洲菊的转录组序列为基础,对SSR位点进行挖掘。结果表明:通过MISA软件从60563条unigenes 检测出14928个SSR位点,分布于11932条unigenes中,出现频率为24.65%,平均分布距离为3.43 kb。优势重复基序为单核苷酸、二核苷酸和三核苷酸,分别占总SSR位点的63.44%、21.54%和14.06%。14928个SSR 位点由73种重复基序构成,(A/T)、(AG/CT、AC/GT)、(AAT/ATT、AAG/CTT)分别是单核苷酸、二核苷酸和三核苷酸优势重复基元,分别占总SSR重复类型的62.31%、18.13%和6.93%。利用 Primer 3.0对SSR序列设计引物9535对,成功率为63.87%。非洲菊转录组SSR位点丰富,分布密度大,具有较高的多态性潜能,可作为开发SSR标记的有效来源,为非洲菊遗传多样性研究,分子标记辅助育种、遗传图谱构建以及功能基因挖掘等提供科学依据。
关键词:非洲菊;转录组;SSR引物;位点信息
中图分类号:S682.1 文献标志码:A 文章编号:0253-2301(2020)11-0001-06
DOI: 10.13651/j.cnki.fjnykj.2020.11.001
Abstract: In order to further develop the SSR molecular markers and promote the researches on the genetic diversity of Gerbera jamesonii L., the SSR loci were mined on the basis of the transcriptome sequencing of Yunnan Red Gerbera jamesonii L..The results showed that 14928 SSR sites were detected from 60 563 unigenes by the MISA software, which were distributed among 11932 unigenes, with a frequency of 24.65% and an average distribution distance of 3.43 kb. The predominant repeat motifs were mononucleotide, dinucleotide, and trinucleotide, which accounted for 63.44%, 21.54%, and 14.06% of the total SSR loci, respectively. A total of 14928 SSR loci consisted of 73 kinds of repeat motifs. Among them, A/T, AG/CT, AC/GT, AAT/ATT, and AAG/CTT were the predominant repeat motifs of mononucleotide, dinucleotide, and trinucleotide, accounting for 62.31%, 18.13%, and 6.93% of the total SSR repeats, respectively. Primer 3.0 was used to design 9535 pairs of SSR sequencing primers, and the success rate was 63.87%. Therefore, Gerbera jamesonii L.. had abundant SSR loci with transcription sites, large distribution density, and excellent polymorphism potential. It could be used as an effective source for the development of SSR markers, and would provide scientific basis for the study of genetic diversity, molecular markerassisted breeding, genetic map construction, and functional gene mining, etc.
Key words: Gerbera jamesonii L.; Transcriptome; SSR primer; Information of loci
非洲菊Gerbera jamesonii L.为菊科大丁草属植物,其花色、瓣型等都有丰富的类型。非洲菊用途非常广泛,而且用量极大,世界五大鲜切花之一[1];不仅可以用于瓶插装饰,还可以做宿根花卉,用于庭院丛植、花海等园林绿化均有极好的效果。栽培非洲菊品种多为二倍体,种间高度杂合,基因组大小预估在2.56~3.84 G[2],至今尚未有非洲菊基因組测序相关报道,而且非洲菊ESTsSSR位点的相关研究几乎空白。本研究基于前期工作中对非洲菊转录组测序获得60563条unigenes 序列,使用SSR软件MiscroSAtellite(MISA)鉴别所有转录组的SSR位点信息,分析其分布及组成特征,以期利用SSR分子标记对非洲菊种质资源进行遗传多样性分析、遗传图谱构建和分子辅助育种等研究奠定一定的基础。
1 材料与方法
1.1 转录组数据来源
非洲菊转录组数据来源于三明市农业科学院花卉所2020年3月对云南红非洲菊品种的植株进行Illumina高通量深度测序。测序取云南红非洲菊种苗地上部材料,材料处理如下:(1)种苗;(2)与印度梨形孢真菌共生的种苗;(3)45℃热处理6 h种苗;(4)45℃热处理6 h与印度梨形孢真菌共生的种苗。委托杭州联川生物技术股份有限公司进行RNAseq转录组测序,并通过de novo方法组装得到unigenes,作为背景数据。
1.2 数据分析及SSR引物设计
采用MISA程序查找SSR位点(https://webblast.ipkgatersleben.de/misa/),参数设置标准为:单核苷酸Mononucleotide、二核苷酸Dinucleotide、三核苷酸Trinucleotide、四核苷酸Tetranucleotide、五核苷酸Pentanucleotide和六核苷酸Hexanucleotide最少重复次数分别为10、6、5、5、5和5次。采用Primer 3.0(2.3.5版,默认参数)设计引物,标准为:引物长度18~24 bp,GC含量40%~60%,理论退火温度(Tm)55.0~65.0℃,预计产物长度在150~300 bp。
2 结果与分析
2.1 非洲菊转录组中SSR位点的数量与分布
通过对非洲菊转录组60563条unigenes序列(总长为51138993 bp)进行搜索,在其中11932条unigenes 序列中检测出14928个SSR位点,SSR发生频率为19.70%,出现频率为 24.65%。其中9521條unigenes 序列仅包含1个SSR位点,2380条含有两个及两个以上SSR位点,另外有1197条unigenes序列含有复合型SSR位点,平均3.43 kb出现1个SSR位点(表1)。由于SSR长度的变异来源于重复单元次数的不同,非洲菊的转录组SSR各种重复类型均有,重复次数分布在5~46次,而且出现频率存在较大差异(表2),其中10次重复的SSR最多,占26.02%;其次是6次重复的SSR,占9.15%;5次重复的SSR占8.97%。其中5~11次重复的SSR有10195个,占总数的68.29%;12~22次重复的SSR有4412个,占比为29.56%;23次以上重复的SSR有321个,只占2.15%。其中单核苷酸、二核苷酸和三核苷酸重复类型SSR出现频率最高,四核苷酸、五核苷酸和六核苷酸重复类型 SSR数量少;单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重复单元的数量分别有9470、3216、2099、77、21和45个,分别占总SSR数的63.44%,21.54%、14.06%、0.52%、0.14%和0.30%。
2.2 转录组SSR基序重复类型和频率特征
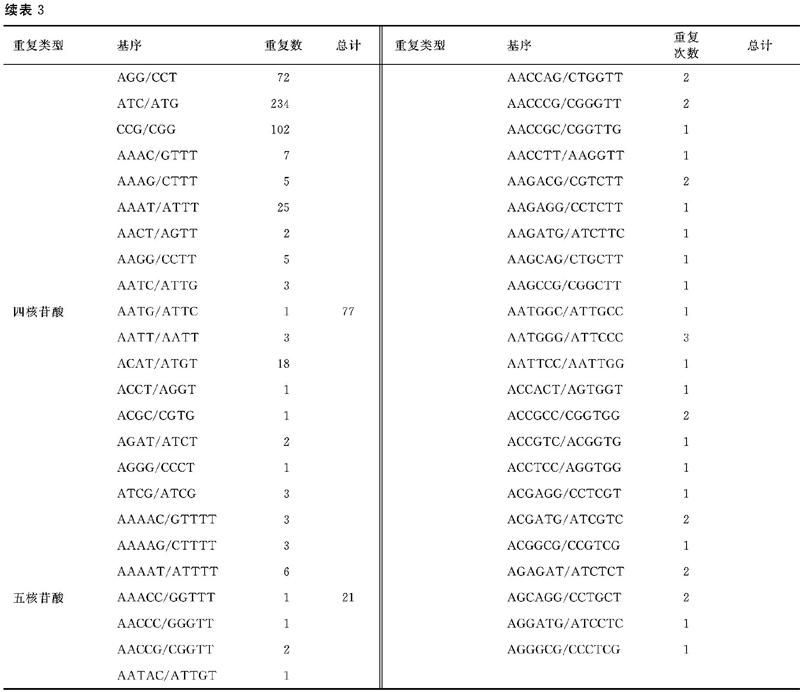
由表3可知,从非洲菊转录组SSR基序类型看,14928个位点共包含73种重复基序类型,单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重复各有2、4、10、14、11和32种;从SSR重复频率分析,其中单核苷酸重复单元有2种类型,其中A/T类型的重复基序出现频率最多有9301个,占该类型的98.22%,C/G基序类型仅有169个,占1.77%(表3)。二核苷酸重复单元有4种类型,其中AG/CT和AC/GT基序出现频率最高,分别为44.47%和39.71%,其次为AT/AT,最少为CG/CG基序类型,只有40个。三核苷酸重复单元有10种类型,其中AAT/ATT和AAG/CTT基序类型出现频率最高,分别占24.92%和24.39%。四核苷酸重复类型有14种,AAAT/ATTT和ACAT/ATGT基序类型占主导地位,分别占32.47%和23.38%。五核苷酸重复类型有11种,其中AAACC/GGTTT基序类型频率最高,占28.57%,其次是AAAAC/GTTTT和AAAAG/CTTTT基序类型,分别占14.29%和14.29%。六核苷酸重复单元类型最多有32种,其中AAAAAC/GTTTTT基序类型个数最多,有4个占8.89%,其次AATTCC/AATTGG基序型有3个,其他类型不超过2个。
2.3 非洲菊转录组中SSR基序长度及引物设计
非洲菊转录组中SSR基序长度分布如图1。从图1可知,大部分基序长度集中在12~20 bp,占47.57%(6532条);其次是小于12 bp,占35.02%(4809条);再次基序长度是21~30 bp,占8.15%(1119条);30 bp以上的基序很少,共占9.26%(1 271条),这类SSR具有较高的多态性。
利用Primer 3.0对14928个含有SSR位点的unigenes 进行引物设计,并针对预测到的每一个SSR位点分别设计了3组引物,取其中一组作为后续分析,成功设计出9535对SSR序列引物,成功率为63.87%。为进一步提高及验证非洲菊SSR引物的可用性和有效性,去除包含单核苷酸SSR位点序列,筛选得到SSR长度12 bp及以上包含复合及二核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸重复单元的SSR位点序列,成功设计出5228对引物,从中筛选出SSR长度20 bp以上序列设计出2220对引物。
3 讨论与结论
本研究从云南红非洲菊品种转录组测序获得60563条unigenes 序列,从中搜索发现其中11932条unigene序列中含有14928个SSR 位点,其中 2380条含有两个及两个以上SSR位点。SSR发生频率为19.70%,SSR的出现频率为24.65%,平均3.43 kb出现1个SSR位点,其出现频率与朱顶红(25.13%)[3]、锦绣杜鹃(24.46%)[4]、牡丹(25.52%)[5]等观赏植物相似,高于湿地松(4.80%)[6]、桂花(13.62%)[7]、三色堇(14.20%)[8]、紫斑牡丹(14.58%)[9]、山茶(19.52%)[10]、兰属植物(20.84%)[11]、紫薇(21.53%)[12]等植物;低于三角梅(44.91%)[13]、菊花(45.71%)[14]、桐花树(68.55%)[15]等植物,这表明非洲菊转录组SSR出现频率处于中等水平,这种频率差异可能与物种基因组大小差异、转录组数据来源特定时期和特定器官、SSR开发方式及筛选标准有关[16-17]。
非洲菊转录组中SSR重复基元较为丰富,6种核苷酸重复基元均有出现,优势重复基元为单核苷酸重复类型,占SSR总数的63.44%,其次为二核苷酸重复类型(21.54%)和三核苷酸重复类型(14.06%),三者共占SSR总数的99.04%;四核苷酸、五核苷酸和六核苷酸重复类型数量最少,分别占0.52%、0.14%和0.30%;这与柿[18]、黄麻[19]、三角梅[13]和黑枸杞[20]等植物单核苷酸占主导地位的研究结果一致,而已有研究报道表明大多数植物种SSR的主要类型是二、三核苷酸重复类型[21-24],也有少数植物是六核苷酸重复类型[22,25-26]。非洲菊这种以单核苷酸、二核苷酸和三核苷酸SSR基序为主体,而四核苷酸、五核苷酸和六核苷酸SSR基序占少数的分布可能是因为自身物种特异性差异及自然选择的结果。另外,作为双子叶植物的非洲菊SSR中单核苷酸出现频率最多的基序是A/T,二核苷酸出现频率最多的基序是AG/CT和AC/GT,三核苷酸出现频率最多的基序是AAT/ATT和AAG/CTT,这与其他双子叶植物上的研究结果基本一致[13,19,22]。
研究发现SSRs重复单元频率的不同将导致SSR位点的长度变化,因此研究SSRs基序长度变化是分析物种多态性高低的重要因素[27-29]。当SSRs基序长度集中在12~20 bp多态性中等,基序长度大于20 bp的SSR多态性较高[28-31]。分析非洲菊转录组SSRs重复12次以上的比例为31.71%,这表明非洲菊转录组SSR重复数较高,具有良好的多态性潜能;本研究发现非洲菊转录组中有47.57%的SSRs集中在12~20 bp,有17.41%的SSRs长度大于20 bp,这表明非洲菊的SSRs具有良好多态性。针对14928个含有SSR位点的unigenes进行引物设计,成功设计9535对引物,成功率为63.87%,得到潜在中高多态性引物7448对。本研究结果可为非洲菊遗传多样性分析和分子标记辅助育种等研究提供技术支持。
参考文献:
[1]PARTHASARATHY V A,NAGARAJU V Invitro propagation in Gerbera jamesonii bolus[J].Indian Journal of Horticulture,1999,56:82-85.
[2]REYNOIRD J P,CHRIQUI D,NOIN M,et al.Plant regeneration fromin vitro leaf culture of several Gerbera species[J].Plant Cell Tiss Organ Cult,1993,33:203-210.
[3]黄宝华,蔡家珍,刘生财,等.朱顶红转录组SSR标记的开发与遗传多样性研究[J].闽南师范大学学报(自然科学版),2019,32(4):81-87.
[4]王书珍,张羽佳,黄诗颖,等.基于锦绣杜鹃花蕾转录组的SSR标记开发及应用[J].林业科学研究,2019,32(3):97-104.
[5]贺丹,吴芳芳,张佼蕊,等.牡丹转录组SSR信息分析及其分子标记开发[J].江苏农业学报,2019,35(6):1428-1433.
[6]易敏,张露,雷蕾,等.湿地松转录组SSR分析及ESTSSR标记开发[J].南京林业大学学报(自然科学版),2020,44(2):75-83.
[7]孙陶泽,母洪娜,等.桂花(Osmanthus fragrans)转录组SSR特征分析[J].分子植物育种,2019,17(7):2258-2263.
[8]杜晓华,杨雅萍,朱小佩,等.三色堇轉录组SSR分析及分子标记开发[J].园艺学报,2019,46(4):797-806.
[9]韩平,阮成江,丁健,等.RNAseq技术开发紫斑牡丹目的基因SSR标记[J].分子植物育种,2019,17(11):3665-3673.
[10]潘丽芹,李纪元,李绍翠,等.基于山茶转录组的SSR标记开发及亲缘关系分析[J].北京林业大学学报,2019,41(7):111-120.
[11]孙叶,刘红,马辉,等.兰属植物ESTSSR标记开发及应用[J].江苏农业学报,2020,36(3):681-688.
[12]秦波,李素珍,胡玲,等.基于转录组的紫薇SSR分子标记开发[C]//中国观赏园艺研究进展,2018:526-532.
[13]赵彤,常圣鑫,冷青云,等.基于三角梅转录组测序的SSR分子标记的开发[J].分子植物育种,2019,17(13):4331-4341.
[14]丁红旭.基于全长转录组序列的菊花SSR标记开发与应用[D].开封:河南大学,2020.
[15]赵宇航,张守信,冉闽媛,等.桐花树叶片的转录组分析[J].分子植物育种,2019,17(7):2133-2143.
[16]BISWAS M K,CHAI L J,MAYER C,et al.Exploiting BACend sequences for the mining, characterization and utility of new short sequences repeat(SSR)markers in Citrus[J].Molecular biology reports,2012,39(5).
[17]张华丽,丛日晨,王茂良,等.基于万寿菊转录组测序的SSR标记开发[J].园艺学报,2018,45(1):159-167.
[18]杜改改,孙鹏,索玉静,等.基于柿雌雄花芽转录组测序的SSR和SNP多态性分析[J/OL].中国农业大学学报,2017,22(10):45-55.
[19]陶爱芬,陈婉婷,祁建民,等.基于转录组测序的黄麻SSR分子标记开发与初步验证[J].中国农业大学学报,2018,23(6):24-32.
[20]黄兴发,尹跃,赵建华,等.黑果枸杞基因组SSR标记开发及遗传多样性分析[J].西北农林科技大学学报(自然科学版),2021(1):1-10.
[21]黄海燕,杜红岩,乌云塔娜,等.基于SSR分子标记的杜仲遗传多样性体系建立[J/OL].林业科学研究,2013,26(6):795-799.
[22]李炎林,杨星星,张家银,等.南方红豆杉转录组SSR挖掘及分子标记的研究[J].园艺学报,2014,41(4):735-745.
[23]林发壮,姚凤琴,安慧珍,等.红掌佛焰苞转录组测序数据组装及基因功能注释[J].分子植物育种,2020,18(19):6256-6265.
[24]郭俊,朱婕,谢尚潜,等.油梨转录组SSR分子标记开发与种质资源亲缘关系分析[J].园艺学报,2020,47(8):1552-1564.
[25]MORGANTE M,HANAFEY M,POWELL W.Microsatellites are preferentially associated with nonrepetitive DNA in plant genomes [J].Nature Genetics,2002,30 (2):194-200.
[26]吴琼,段小群,陈旭,等.基于高通量测序的红豆杉ESTSSRs标记研究[J].中国中药杂志,2012,37(24):3728-3733.
[27]THAO M,YAMASHITA A,WATANABE,S,et al.Development of tetranucleotide microsatellite markers in Pinus kesiya Royle ex Gordon[J].Conservation Genetics Resources,2013,5(2):405-407.
[28]賈展慧,宣继萍,张计育,等.薄壳山核桃转录组中的SSR位点信息分析[J].分子植物育种,2019,17(10):3305-3311.
[29]付燕,杨芩.枇杷花转录组SSR位点信息分析[J/OL].分子植物育种,2020:https://kns.cnki.net/kcms/detail/46.1068.S.20201012.1040.002.html.
[30]TEMNYKH S,DECLERCK G,LUKASHOVA A,et al.Computational and Experimental Analysis of Microsatellites in Rice(Oryza sativa L.):Frequency,Length Variation,Transposon Associations,and Genetic Marker Potential[J].Genome Res,2001,11(8):1441-52.
[31]李珊珊,孙春玉,蒋世翠,等.SSR分子标记及其在植物遗传育种中的应用[J].吉林蔬菜,2014(5):33-38.
(责任编辑:柯文辉)
