基于90K芯片的小麦穗长和旗叶长QTL分析
2020-03-05姚俭昕张传量宋晓朋许小宛邢永锋吕栋云宋鹏博杨孟于孙道杰
姚俭昕,张传量,宋晓朋,许小宛,邢永锋,吕栋云,宋鹏博,杨孟于,孙道杰
(1.西北农林科技大学农学院,陕西杨凌 712100;2.周口市农业科学院,河南周口 466000;3.驻马店市农业科学院,河南驻马店 463000)
小麦(TriticumaestivumL.)是全球重要的粮食作物,提供人类所需的热量和蛋白质[1-2]。小麦育种的基础目标是增加单位面积产量,而影响小麦产量的大部分农艺相关性状由微效多基因控制[3]。其中,穗长与小麦产量三要素(单位面积穗数、穗粒数和粒重)均密切相关[4],小麦旗叶长则与穗粒数和单株籽粒产量呈显著正相关[5-6]。因此,小麦的穗长和旗叶长是小麦育种过程中重要的目标性状,对其进行QTL定位研究,可为提高小麦产量提供参考依据。
迄今为止,已有许多国内外学者在不同环境下对小麦穗长和旗叶长的遗传机制进行了大量研究,并取得了一定的进展。然而,调控穗长的主要基因还未被确认,但是定位了大约400个控制穗长的QTL,定位信息覆盖了整个染色体组[7-16]。Wu等[17]研究发现,与穗长相关的一些效应较大的或在多个环境中检测到的QTL,分布在2D、3A、4A、4B、5A、6A、6B、7A、7B和7D染色体上,且QSpl.nau-2D(HL1)被定位到2D染色体的短臂上。Zhai等[12]利用RIL群体经多环境的联合分析,发现QSl.cau-2D与矮杆基因Rht8定位区间一致,为相同的基因,在不改变小穗数的情况下,可降低穗长长度。Ma等[8]基于RIL群体和F2群体,在7D染色体上定位到效应较大的数量位点QSpl.nau-7D;Yao等[18]利用近等基因系对该位点进行分析,发现该位点的优良等位基因能够增加穗长,提高小穗数和穗粒数。随着定位技术水平的提高,挖掘到的QTL数量逐渐丰富,但仍需继续挖掘新的控制穗长的QTL,以深入了解穗长的遗传机制。
近些年,关于小麦旗叶长的QTL定位研究逐渐增多,小麦的旗叶大小极易受环境影响,属于典型的数量性状[19],控制旗叶大小的QTL在小麦21条染色体上均能被检测到。例如,Isidro等[20]在硬粒小麦中,发现控制旗叶长的QTL分布在2A、3B、5B、7A和2B染色体上。Hussain等[21]基于基因型测序(GBS)技术,以Harry(耐旱)与Wesley(干旱易感)两个品种构建的RIL群体为材料,对旗叶相关性状进行QTL定位,共检测到7个控制旗叶长的QTL,分布在2D、5A、6A、7A和7B染色体上。Liu等[22]利用半野生小麦藏1817和普通小麦宁冬3331构建的含有213个株系的RIL群体进行QTL定位,共检测到7个控制旗叶长的QTL,分布在2B、3A、4B和5A染色体上。Fan等[23]利用科农9024与京411两个亲本构建的含有188个株系的重组自交系为材料,进行旗叶长的QTL定位,共检测到11个控制旗叶长的QTL,分布在1B、2B、4A、4B、5B和5D染色体上,其中,有2个QTL能够在8个环境中检测到,为稳定QTL。大量的研究也表明,旗叶相关性状与产量相关性状之间存在相关性,适当的旗叶大小将有利于产量的提高。例如,Liu等[22]研究发现,部分QTL同时控制旗叶和产量相关性状。Ma等[27]通过条件QTL分析发现,旗叶大小和角度之间的适当平衡可以提高产量。Zhao等[19]利用3个不同亲本组合的RIL群体为材料,发现10个同时控制旗叶和产量相关性状的QTL簇。
虽然目前针对小麦穗长和旗叶长已经开展了大量研究工作,但结论却各不相同,这可能与选择的遗传材料不同有关。随着生物技术的不断发展,分子标记技术在作物育种研究中得到广泛应用,其中,SNP标记由于具有数量丰富、多态性高和稳定性好等优点,近年来被广泛应用。因此,本研究以小偃81和西农1376为亲本构建的含120个株系的RIL群体为研究对象,利用90K SNP芯片对穗长和旗叶长进行QTL定位,挖掘在多环境下能够稳定表达的主效QTL,以期为相关基因克隆和分子育种提供参考。
1 材料与方法
1.1 试验材料
以优良品种(系)小偃81(XY81)和西农1376(XN1376)构建的包含120个株系的F9∶10代RIL群体为材料,于2016年10月至2017年6月和2017年10月至2018年6月,分别在陕西杨凌和河南南阳(分别用2017YL、2017NY、2018YL和2018NY表示)进行试验,各试验点均采用随机区组设计,每个株系种植2行,行长2 m,每行70株,行距 0.3 m。重复5次。其他田间管理措施同当地生产管理。
1.2 表型鉴定
于小麦蜡熟期,每个株系选取长势良好且均匀一致的5个单株(排除边际效应),对其主茎穗长和旗叶长进行测量。利用R/leme4计算各个环境的表型数据,并进行W-test检验,对亲本性状的数值进行多重比较及遗传力计算,广义遗传力的计算公式为:
H2=VG/(VG+VGY/y+VGE/e+VE/nr)× 100%
其中,y代表年数,e代表环境数,n代表重复数。根据多环境的表型数据进行随机效应的线性分解,对BLUP育种值(best linear unbiased prediction)进行预测,利用各个环境的平均值和BLUP育种值进行QTL作图[24]。
1.3 遗传图谱的构建
以小麦幼嫩叶片为材料,采用CTAB法提取小麦基因组DNA。利用Illumina 基因分型技术测试平台(北京博奥生物有限公司)的微珠芯片技术(Bead Array)对小麦90K SNP芯片标记进行检测,利用Genomestudio v1.0软件进行多态性分析。构建图谱的大致流程为:芯片标记分型成功之后,导入EXCEL,筛选出两亲本间存在多态性的标记,剔除偏分离标记(P<0.001)和缺失率>20%的标记,然后用CARTHAGENE[25]进行遗传图谱的构建,LOD> 5,distance<0.3。
1.4 QTL的检测
利用完备复合区间模型(ICIM)[26]对4种环境下的表型值及育种值(BLUP)分别进行QTL定位。在a=0.05显著水平下进行1 000次的排列检验,确定LOD值的大小,采用正反逐步回归法控制背景,步长设为1 cM。定位结果与中国春的物理位置(IWGSC RefSeq v1.0)进行比对,以确定具体的物理位置。
2 结果与分析
2.1 表型数据分析
由表1可以看出,不同环境的穗长和旗叶长在亲本XY81与XN1376间均存在显著差异。穗长和旗叶长的遗传力分别为0.82和0.74,说明均不易受环境影响;穗长的遗传力大于旗叶长,两性状均出现不同程度的超亲分离。W-test检验结果发现,两性状在各环境下的P值皆大于 0.01,符合正态分布,属于典型的数量性状。
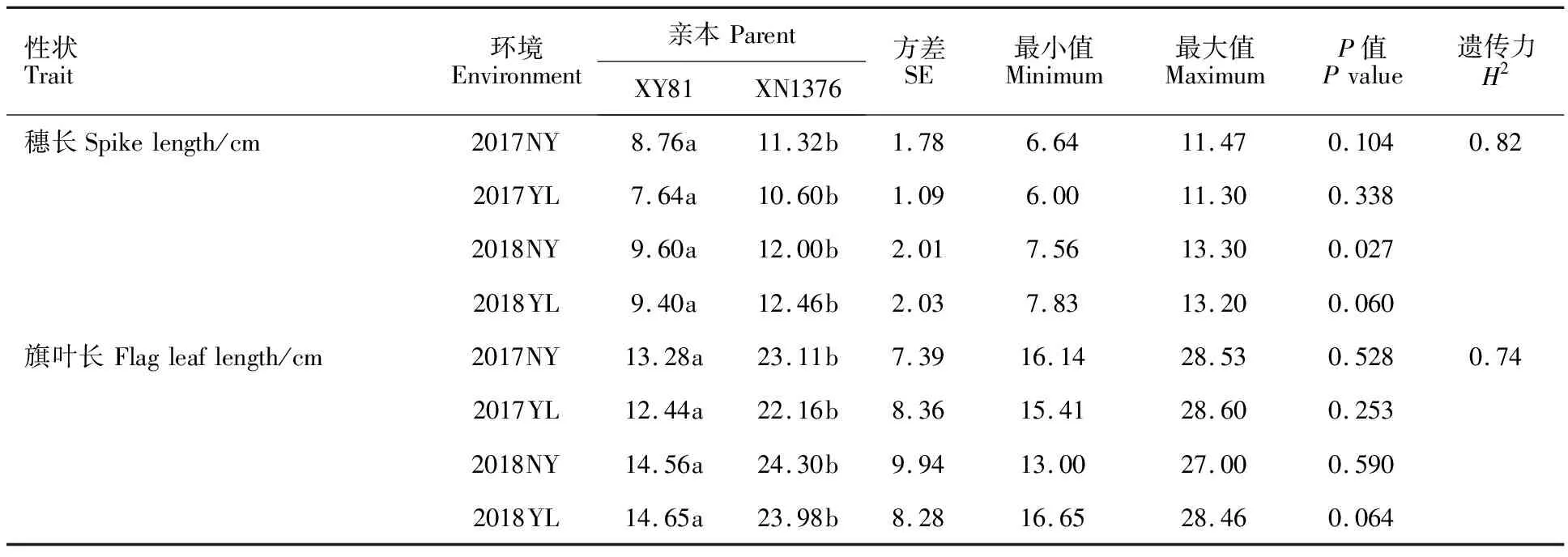
表1 亲本及RIL家系的穗长和旗叶长的统计分析
2.2 遗传图谱的构建
利用小麦90K SNP芯片81 587个标记对XY81和XN1376及其组配的RIL群体进行多态性标记筛选,共筛选出9 728个多态性标记。对多态性标记进行筛选,设定偏分离P的阈值为 0.001,缺失率的阈值为20%,最终5 531个标记被挂载到连锁群上,连锁群全长为3 172.49 cM,平均图距0.57 cM,覆盖小麦21条染色体,平均每个株系单条染色体的交换率为1.48次。
遗传图谱的初步分析结果(表2)表明,90K SNP芯片的连锁标记数在小麦基因组A、B和D间分布不均衡,表现为A亚组的标记数>B亚组的标记数>D亚组的标记数;A亚组的连锁群长度>B亚组的连锁群长度>D亚组的连锁群长度。

表2 遗传图谱中 SNP 标记在小麦染色体组中的分布
2.3 穗长和旗叶长的QTL分析
2.3.1 单环境QTL定位
从表3、图1可以看出,共检测到3个控制穗长的QTL,分别分布在2B、2D和5D染色体上,表型变异的贡献率为6.78%~22.51%,其中,Qsl.nwsuaf-2D和Qsl.nwsuaf-5D在多个环境中均能被检测到,其平均表型变异解释率分别为20.74%和12.33%,为主效QTL;Qsl.nwsuaf-2B仅在1个环境中被检测到,其表型变异解释率为6.78%,为微效QTL,这3个QTL位点的增效等位基因均来源于XY81。检测到2个控制旗叶长的QTL,均分布在5A染色体上,表型变异贡献率为8.30%~16.36%。其中,Qfll.nwsuaf-5A.1在多个环境下均能被检测到,且其平均表型变异贡献率大于10%,为主效QTL。Qfll.nwsuaf-5A.1的增效等位基因来源于XY81,而Qfll.nwsuaf-5A.2的增效等位基因则来源于XN1376。
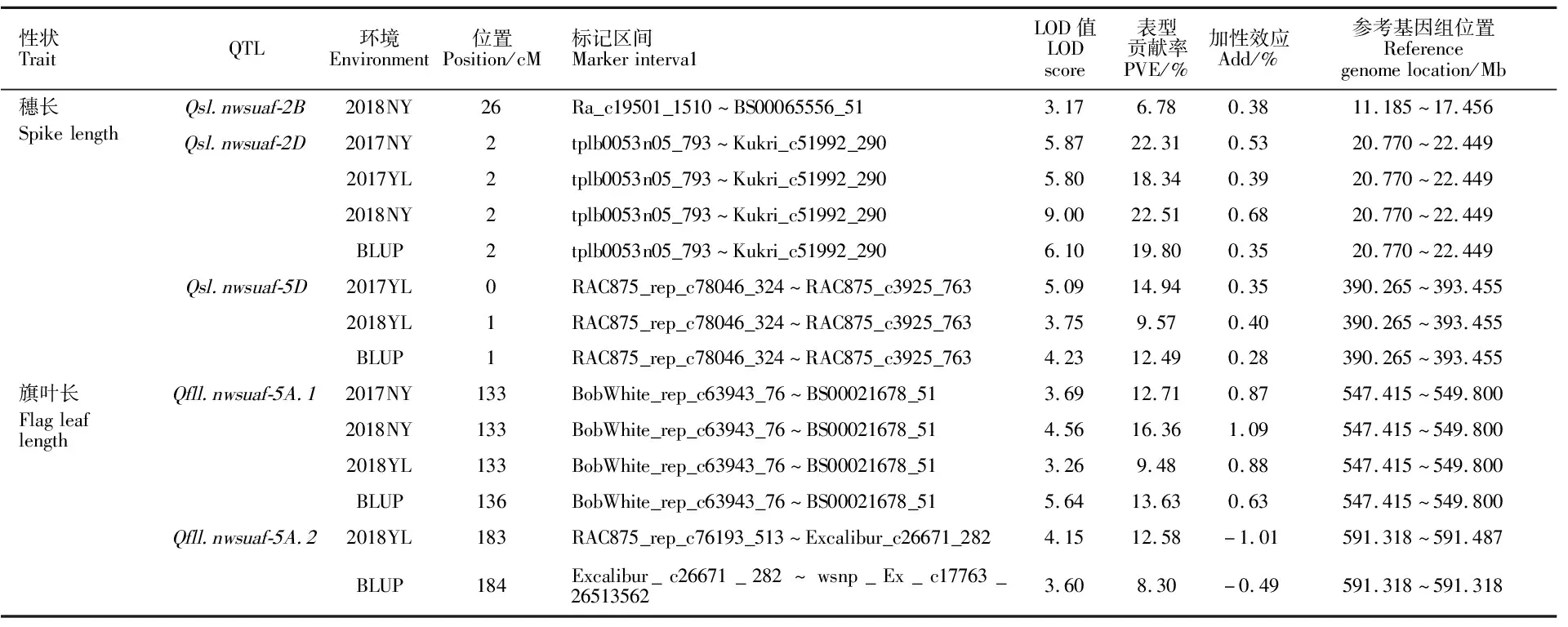
表3 穗长和旗叶长的定位信息

图1 穗长和旗叶长QTL在染色体上的分布
2.3.2 环境互作QTL定位
从表4、图1可以看出,共检测到3个控制穗长的QTL,分别为Qsl.nwsuaf-2D、Qsl.nwsuaf-4D和Qsl.nwsuaf-5D,分布在2D、4D和5D染色体上,表型变异解释率分别为15.91%、4.89%和10.81%,环境互作解释的表型变异PVE(AbyE)分别为0.53%,0.33%和0.54%。其中,Qsl.nwsuaf-2D和Qsl.nwsuaf-5D正向调控穗长,Qsl.nwsuaf-4D则负向调控穗长。共检测到4个控制旗叶长的QTL,分别为Qfll.nwsuaf-4B、Qfll.nwsuaf-5A.1、Qfll.nwsuaf-5A.2和Qfll.nwsuaf-6A,分布在4B、5A和6A染色体上,表型变异解释率分别为4.26%、14.03%、7.53%和7.07%,PVE(AbyE)分别为1.44%、1.69%、3.11%和0.23%。其中,Qfll.nwsuaf-4B和Qfll.nwsuaf-5A.1正向调控旗叶长,Qfll.nwsuaf-6A和Qfll.nwsuaf-5A.2负向调控旗叶 长度。
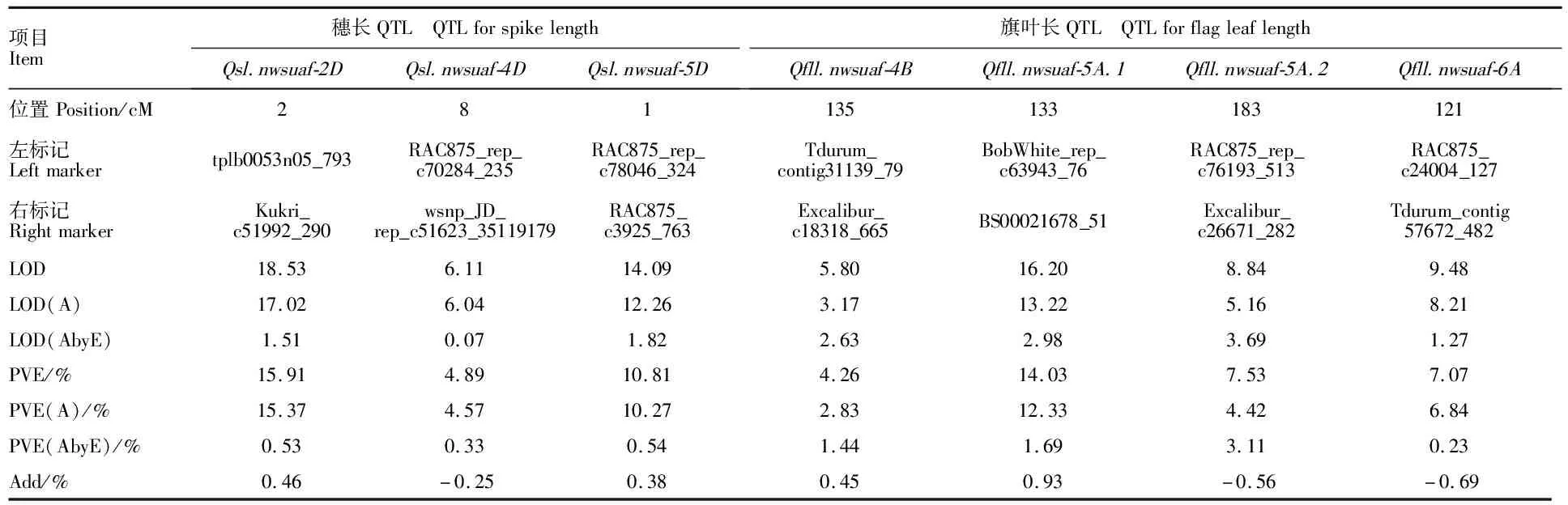
表4 多环境下穗长和旗叶长的QTL定位信息
2.3.3 定位结果综合分析
通过对单环境和环境互作QTL定位结果进行比对,发现Qsl.nwsuaf-4D、Qfll.nwsuaf-4B和Qfll.nwsuaf-6A这3个QTL均未在单环境中检测到,表明这些位点受环境影响较大;而Qsl.nwsuaf-2D、Qsl.nwsuaf-5D、Qfll.nwsuaf-5A.1和Qfll.nwsuaf-5A.2用两种定位方法均能检测到,其中,Qfll.nwsuaf-5A.2在单环境QTL定位结果中,仅在一个环境中检测到,但在多环境QTL定位结果中,PVE(AbyE)占总表型变异解释率的41.29%,说明该位点易受环境影响;Qsl.nwsuaf-5D仅在两个环境中检测到。综合结果表明,Qsl.nwsuaf-2D和Qfll.nwsuaf-5A.1为环境钝感QTL。
3 讨 论
构建高密度且分布范围广的遗传图谱是提高QTL定位准确性的前提。由于SNP标记具有分布均匀且数目众多的优异特性,因此,可用于构建高密度且覆盖所有染色体的连锁图谱,有利于将QTL定位到较小的区间内,极大的降低候选基因预测和基因克隆的难度,提高遗传分析的精度和效率。小麦为异源六倍体,基因组构成复杂,与其他作物相比,小麦SNP标记的开发与利用起步较晚,基于SNP标记的小麦群体遗传分析相对较少。本研究利用小麦Illumina 90K基因芯片,共筛选出5 531个标记,连锁群全长为3 172.49 cM,平均图距0.57 cM,构建了覆盖小麦21条染色体的高密度遗传图谱。但从染色体组的覆盖度来看,D亚组的分子标记少,连锁群长度短,说明90K芯片对普通小麦D染色体组的覆盖程度具有局限性[12]。从定位结果来看,定位的遗传距离为1.0~6.5 cM,物理距离为2.915 Kb~65 Mb,相较于传统分子标记遗传研究,定位区间进一步缩小。
本试验利用西农1376和小偃81构建的RIL群体,基于完备复合区间模型(ICIM)对4种环境下的表型值采用单环境和多环境分别进行QTL定位,结果表明,在单环境QTL分析中,共定位到3个控制穗长的QTL,其中包括一个多环境下稳定表达的主效位点Qsl.nwsuaf-2D,该位点在基因组上的物理距离为20.770~22.449 Mb,此结果与Zhai等[17]定位结果一致,推测此位点和Rht8基因为同一个基因,而Chai等[14]则利用近等基因系材料证明此位点为控制株高和穗长的耦合位点。位于5D染色体上的主效位点Qsl.nwsuaf-5D,其变异解释率为9.57%~14.94%,前人在5D染色体上尚未发现有控制穗长的QTL,推测可能为新的位点。位于2B染色体上的位点Qsl.nwsuaf-2B,参考基因组上位置为11.185~17.457 Mb,与Zhai等[12]定位到的Qsl.cau-2B.2相差11 Mb,推测可能为新的位点,但是不排除是同一QTL的可能;共定位到2个控制旗叶长的QTL,其中5A染色体上控制旗叶长的主效位点Qfll.nwsuaf-5A.1参考基因组位置为547.415~549.801 Mb,与Hussain等[21]在干旱条件下定位到的位点在参考基因组上出现部分重叠,可能为同一位点,此位点在干旱条件下能够稳定表达;而在多环境QTL定位中共定位到7个QTL,其中,Qfll.nwsuaf-4B和Qfll.nwsuaf-5A.2的PVE(AbyE)占总表型变异解释率的33.69%和 41.29%,受环境影响较大。
综上所述,结合单环境和多环境两种定位方法,共定位到3个相同的QTL,其中Qsl.nwsuaf-2D和Qfll.nwsuaf-5A.1在两种方法中的贡献率均大于10%,属于环境钝感QTL,说明两位点为多环境下稳定表达的主效位点,不易受环境影响。这两个QTL为进一步挖掘小麦穗长和旗叶长的遗传机制奠定了基础,并为后续精细定位和标记开发提供参考。
