基于转录组测序的大熊猫多态性微卫星标记筛选
2020-02-27涂洪梅周闯王冠楠成美玲岳碧松孟杨
涂洪梅, 周闯, 王冠楠, 成美玲, 岳碧松, 孟杨
(四川大学生命科学学院,生物资源与生态环境教育部重点实验室,成都610065)
大熊猫Ailuropodamelanoleuca是全球生物多样性保护的旗舰物种(Weietal.,2012)。由于受到人类人口扩张和栖息地丧失、破碎化等的影响,大熊猫的生存面临严重威胁(Huangetal.,2015)。近几十年来,我国为保护这一珍稀物种采取了一系列的保护措施,使得全国大熊猫种群得以逐渐复壮。全国第四次大熊猫数量调查显示,截止2014年底,野生大熊猫数量为1 864只,与第三次调查相比增长了16.8%(唐小平等,2015)。如今大熊猫栖息地存在不同程度的破碎化,野生大熊猫种群被进一步分割,部分小种群还面临着灭绝的风险,大熊猫的生存状况不容乐观,因此对大熊猫的保护及遗传多样性研究仍是重点(青菁,2016)。
微卫星标记称为简单重复序列(simple sequence repeats,SSRs)或短串联重复序列,由核心序列和侧翼序列组成,其中核心序列由1~6 bp核苷酸序列组成,在真核生物基因组中分布广泛。微卫星标记多态性含量丰富、易检测、小片段或者降解的DNA都能因含有足够的微卫星而进行PCR扩增,同时微卫星的检测在种间、种外都能进行(张正义等,2017)。作为分子生物学中最常用的一种遗传标记(Maetal.,2004;Selkoe & Toonen,2006),微卫星分子标记被广泛应用于物种鉴定、群体遗传学和遗传图谱的研究(Huangetal.,2015;修云芳等,2015;Wangetal.,2016)。二代测序(next-generation sequencing,NGS)技术又被称为高通量测序,近年来应用十分广泛,使用二代测序技术开发微卫星标记的研究日趋增多。传统的微卫星分子标记开发方法,如DNA文库筛选和利用亲缘物种跨种扩增等往往耗时长、数量少、工作量大。与传统方法相比,使用NGS筛选微卫星分子标记成本更低、效益更高(Zhengetal.,2013;Liuetal.,2017)。因此,高通量测序由于其高效性迅速成为开发DNA分子标记的首选(岳华梅等,2016)。
从GenBank下载大熊猫完整基因组,使用本实验室自主开发的Krait(Duetal.,2017)对基因组中微卫星序列进行搜索和统计。结合本实验室已测得的6只大熊猫转录组数据,使用CandiSSR(Xiaetal.,2016)对基因组和转录组中多态性微卫星进行对比与筛选。CandiSSR是一种能快速有效地进行多态性微卫星筛选的软件,能够基于多个组装序列鉴定出具有多态性的微卫星。本研究根据CandiSSR鉴定出的大熊猫基因组中的多态性微卫星,并挑选其中评分较为理想的20个,提取25只大熊猫血液DNA为模板进行PCR扩增,将扩增产物送至成都擎科梓熙生物科技有限公司分型并分析结果,验证所选位点的多态性及其他遗传参数。本研究结果开发了更多优良微卫星位点,为大熊猫种群遗传学的进一步研究提供了资源。
1 研究方法
1.1 基因组中的微卫星统计分析
GenBank下载大熊猫全基因组(AilMel_1.0)。微卫星的检索标准分别限定为单碱基重复12次以上,二碱基重复7次以上,三碱基重复5次以上,四、五和六碱基重复4次以上,这与之前使用Krait进行研究的方法一致(Qietal.,2015;Xuetal.,2016)。在本研究中,重复单位模式为循环排列和/或反向互补的被归类为一种SSR类型。例如,AGC模式由AGC、GCA、CAG、GCT、TGC和CTG组成(Jurka & Pethiyagoda,1995)。
1.2 RNA-seq数据分析
采集6只大熊猫个体的RNA-seq数据。使用FastQC(https://www.bioinformatics.babraham.ac.uk/projects/fastqc)对reads进行质控,使用Cutadapt(Martin,2015)去除接头序列和较低质量reads(Phred score<20)。使用Trinity(Haasetal.,2013)在默认参数下对clean reads进行组装,保留长度超过200 bp的转录本进行分析。采用CD-HIT-EST(Li & Godzik,2006)删除带有默认参数的冗余序列,并使用UniGenes发现微卫星标记。
1.3 多态性微卫星检测及引物设计
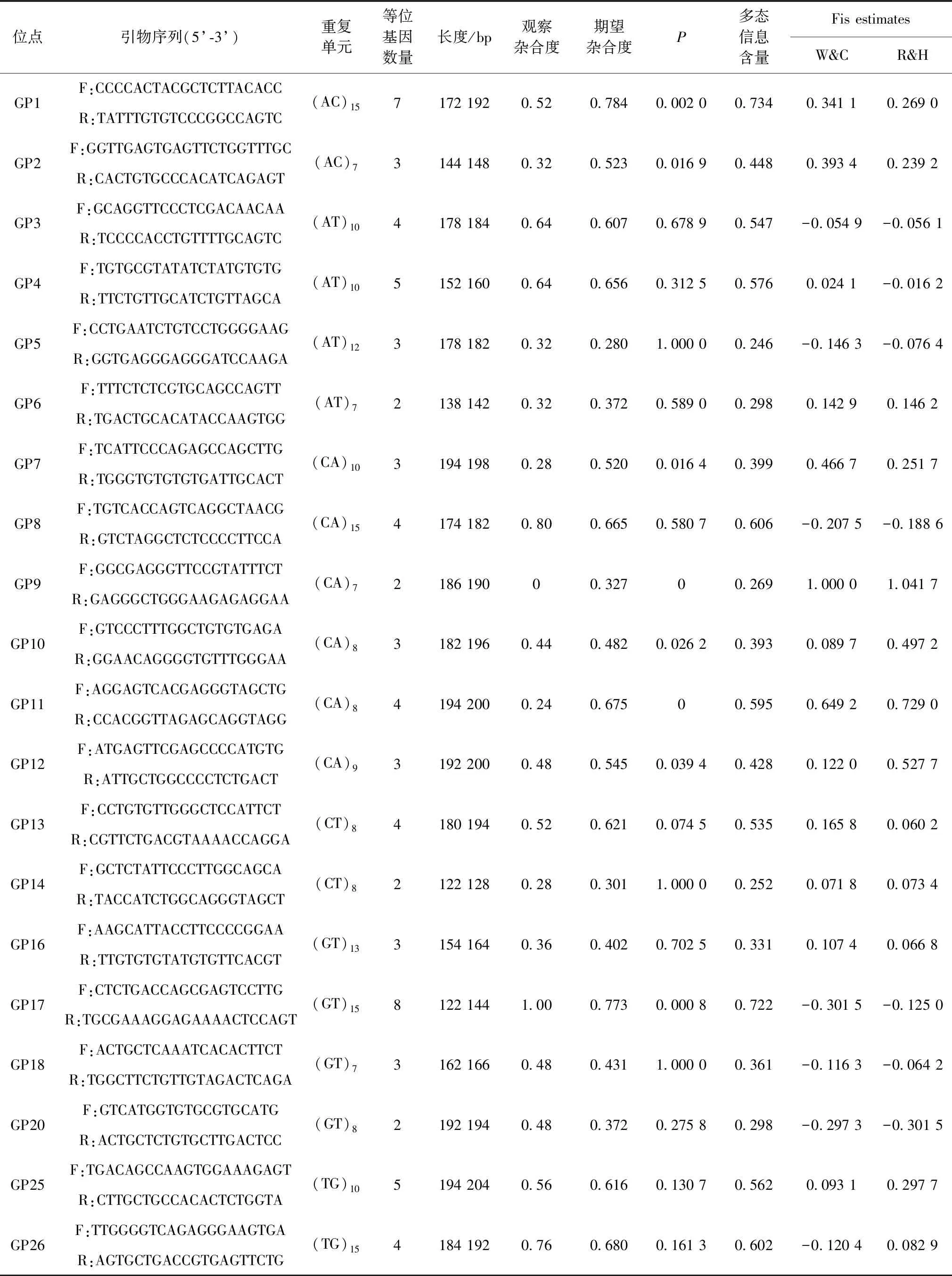
基于本实验室已组装好的6只大熊猫的转录组序列文件和从GenBank下载的大熊猫全基因组序列,使用CandiSSR(Xiaetal.,2016)检索多态性微卫星并由此设计引物。从筛选结果中选择20个多态性二碱基微卫星位点,用于25个大熊猫血液DNA样品的多态性验证。每个引物对(表4)的正向引物在5’-末端用FAM荧光染料标记进行基因分型分析。
1.4 样品采集和DNA提取
25只大熊猫个体的血液样本来自成都大熊猫繁育研究基地。使用DNA Extraction Kit(Tsingke,北京)试剂盒,并按照说明书从血液样品中提取基因组总DNA。以提取的基因组DNA为模板进行PCR扩增。
1.5 PCR条件和扩增
PCR反应体系为20 μL:2.5 μL 10×Taq缓冲液,1.0~3.0 μL dNTP(各2.5 mmol·L-1),各引物1 μL(10 mmol·L-1),0.2 μL ExTaq聚合酶(5 U·mL-1;TaKaRa,日本),基因组总DNA约200 ng作为模板。PCR反应程序为:95 ℃ 5 min;94 ℃ 30 s,55~63 ℃ 30 s,72 ℃ 12 s,35个循环;72 ℃ 10 min,10 ℃保存。
1.6 基因分型
所有样品的基因分型检测在成都擎科梓熙生物科技有限公司进行。基因分型时将每个荧光引物扩增的产物单独放在一条泳道里电泳分型。所有样品的基因分型在ABI 3730 DNA Analyzer上进行,使用GeneMapper 4.0检测各样本等位基因数,等位基因大小相对于分子内标GS500LIZ决定。
1.7 微卫星数据分析
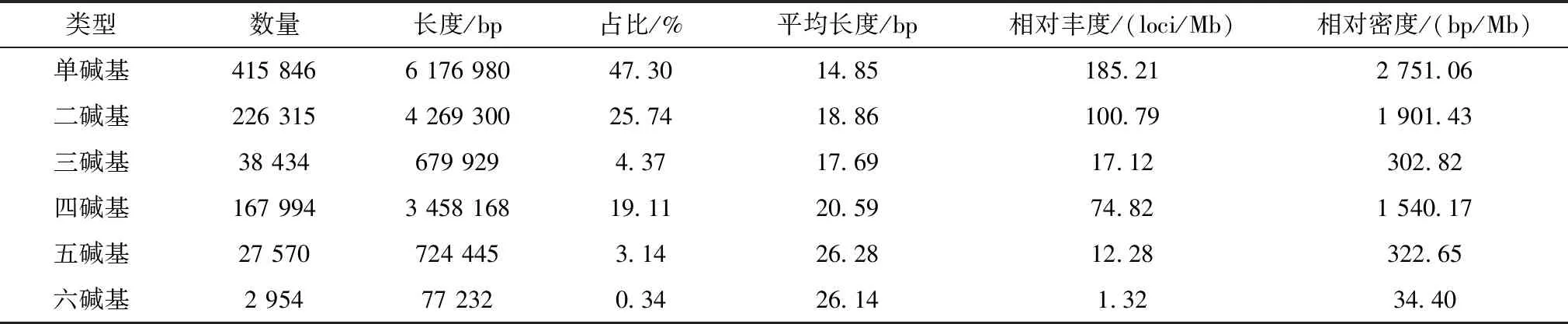
使用CERVUS(Marshalletal.,1998)计算等位基因数、观察杂合度、期望杂合度和多态信息含量(polymorphic information content,PIC)。使用GENEPOP(Raymond & Rousset,1995)计算哈迪-温伯格平衡和连锁不平衡的偏差。作为衡量多态信息含量的指标,PIC>0.50为高度多态性位点,0.25 Krait搜索和统计结果显示,在大熊猫的基因组中,不完美的微卫星是最常见的类型,共有3 177 509个,相对丰度为1 415.17 loci/Mb,相对密度为40 727.66 bp/Mb。其次是完美微卫星,数量最少的是复合型微卫星,只有53 787个,相对丰度为23.96 loci/Mb,相对密度为980.90 bp/Mb(表1)。完美微卫星的数量、长度、相对丰度和相对密度如表2所示。6种完美微卫星总数为879 113个,总长度为15 386 054 bp。其中最常见的类型是单碱基微卫星,有415 846个,且长度最长,达到了6 176 980 bp,最高丰度为185.21 loci/Mb,最高密度为2 751.06 bp/Mb,占基因组微卫星总数的47.30%。其次是二碱基微卫星,占微卫星总数的25.74%。而三碱基、四碱基和五碱基微卫星较少,六碱基微卫星则最少,仅占微卫星总数的0.34%。在大熊猫基因组中前10个最常见的重复单元是(A)n、(AC)n、(AG)n、(AAAT)n、(AAAG)n、(AT)n、(C)n、(AAAC)n、(AAGG)n和(AAT)n。 表1 大熊猫基因组3种类型微卫星统计Table 1 The number and frequency of 3 categories of microsatellites in the giant panda genome 表2 大熊猫基因组中完美型微卫星分布概况Table 2 The summary of perfect microsatellite types in the giant panda genome CandiSSR分析结果显示,从大熊猫转录组与基因组中共筛选出326个多态性微卫星,其中二碱基最多,占69.94%,共228个。三、四、五、六碱基所占比例分别为9.51%、14.11%、5.21%、1.22%。本研究根据标准差、缺失率以及序列长短等因素,选出20个二碱基微卫星用于后续的多态性评估验证(表3)。 选出的20个微卫星位点能够在25只大熊猫个体的血液DNA中成功扩增。每个位点的等位基因数为2~8。观察杂合度为0~1.00,平均值为0.472,期望杂合度为0.280~0.784,平均值为0.532。使用Bonferroni校正后,有4个微卫星位点显著偏离哈迪-温伯格平衡(P<0.01),20个微卫星位点未观察到显著的连锁不平衡(P>0.01)。20个位点多态信息含量为0.246~0.734,其中具有高度多态性的位点9个(PIC>0.50),11个位点呈中度多态性(0.25 转录组平台的构建在很大程度上推动了DNA分子标记的开发。近年来利用转录组数据获得含有微卫星的序列,并对其进行遗传多样性的研究在国际上已有很多成功报道(Tianetal.,2014;Xiaoetal.,2015),而利用微卫星标记进行野生大熊猫遗传多样性分析从2001年就已开始(Luetal.,2001)。在二代测序技术广泛应用之前,开发微卫星标记的方法较多,比较常用的方法包括构建DNA文库筛选法、小片段DNA克隆法、富集法、利用亲缘物种筛选等。Shen等(2005)利用Dynal磁珠法筛选了37对大熊猫微卫星引物,Wu等(2008)使用富集法筛选出33个微卫星位点,Li等(2010)通过构建DNA文库法筛选出7个稳定的四碱基微卫星位点。这些位点的筛选与标记开发在一定程度上可以用于大熊猫种群遗传多样性评估、遗传结构分析等相关研究。由于部分微卫星位点扩增不稳定,在使用粪便或者毛发等样本提取的DNA扩增效果不好时就限制了这些位点的应用。随着测序成本的降低及测序平台通量的不断扩大,利用二代测序技术对基因组进行随机测序即可获得海量的微卫星序列。与传统微卫星标记开发相比,二代测序技术对样品的处理和使用简单、处理速度快、通量高,能在较短时间内开发出数量多且类型丰富的微卫星。目前利用二代测序技术对微卫星位点进行筛选与开发也越来越多(Ritchieetal.,2016;李薇等,2017;宋琪等,2019)。 表3 20个多态性微卫星参考信息Table 3 Reference indicators of 20 polymorphic microsatellite loci 表4 20个微卫星位点遗传特征Table 4 Characterization of 20 microsatellite loci isolated 本研究对大熊猫基因组中的微卫星序列进行搜索和统计,使用的是本实验室开发的Krait:一种用于全基因组微卫星调查和引物设计的超快工具(Duetal.,2017)。该软件是在2013年本实验室开发的微卫星搜索及统计软件MSDB(Duetal.,2013)基础之上进行开发的,有着更强大的功能。Krait可以很容易地用于筛选微卫星标记,用户可以高效地设计用于微卫星放大的引物。李午佼等(2014)曾使用MSDB分析了大熊猫基因组微卫星的分布特征。研究表明,Krait比MSDB搜索检测速度更快、更高效,能够快速研究DNA序列中的微卫星(Duetal.,2017)。对比2个软件的分析结果,发现Krait不仅能快速筛选微卫星标记,还能有效地检测到更多微卫星重复序列,在种类及丰度等分布特征的识别上更为准确。因此本研究使用Krait对大熊猫基因组中微卫星序列进行检测的结果是可信、可靠的。 到目前为止,也有较多的微卫星标记成功用于野生大熊猫的种群遗传多样性分析(Heetal.,2008;Huetal.,2010)。据统计,目前公布的大熊猫的微卫星标记共109种,其中超过60%是二碱基微卫星(乔麦菊等,2019)。根据目前研究进展,用于野生大熊猫遗传多样性调查研究的微卫星标记多,且存在差异,故无法进行统一的比较分析,未来关于大熊猫微卫星标记的开发在稳定实用的基础上,应尽量统一标准。本研究中使用6只大熊猫的转录组数据与大熊猫基因组相结合,利用软件筛选多态性微卫星,共筛选出了326个多态性微卫星,处理过程耗时短,减少了很多工作量。而从中挑选的20个微卫星位点通过实验验证,显示部分位点多态性较高,证明该方法是可行的,可以对后续位点筛选提供资源。 群体的遗传多样性主要表现在等位基因数、杂合度和多态信息含量3个方面(Senananetal.,2004)。本研究选取的20个位点中,高度多态性位点有9个,中度多态性位点有11个,其中,有4个显著偏离哈迪-温伯格平衡(P<0.01)。后续实验可将这20个位点中多态性较高的位点选用粪便DNA进行扩增和其他参数的评估,筛选出能够应用于野生大熊猫遗传多样性研究的微卫星标记,为野生大熊猫种群的群体结构及遗传研究提供一定的理论指导。 根据分析结果,本研究认为NGS是一种有效开发大熊猫微卫星标记的方法。利用大熊猫的NGS序列数据可以开发大量的微卫星标记。微卫星分子标记的开发将有助于评估大熊猫的遗传多样性和种群结构,并制定有效的保护和管理策略。2 结果
2.1 微卫星类型和丰度

2.2 多态性微卫星位点筛选
2.3 多态性微卫星位点验证
3 讨论