基于生长和抗逆功能基因SNP分子标记的凡纳滨对虾野生及选育群体遗传多样性分析
2020-02-22申淑慧戴习林
申淑慧 戴习林
摘要:【目的】分析凡納滨对虾野生群体与选育群体的遗传多样性及遗传差异,为进一步改良优化凡纳滨对虾种质提供参考依据。【方法】通过凡纳滨对虾转录组测序数据挖掘候选SNP位点,设计扩增引物,采用10尾野生凡纳滨对虾进行直接测序验证,筛选获得11对有效引物及24个位于生长和抗逆功能基因上的SNPs位点,并用于分析2个野生群体(Y1和Y2)和2个选育群体(F1和F2)的遗传多样性。【结果】凡纳滨对虾野生群体的观测杂合度(Ho)、期望杂合度(He)及多态信息含量(PIC)等遗传参数平均值明显高于选育群体。在2个野生群体中,Y2的各项遗传参数平均值均高于Y1;在2个选育群体中,F2的各项遗传参数平均值均高于F1,且在CATL、ANT、Tryptase、Crustin-like和Penaeidin4c等功能基因的某些SNP位点上,2个选育群体的各项遗传参数平均值为0。4个凡纳滨对虾群体中均存在SNP位点不同程度偏离Hardy-Weinberg平衡现象。野生群体Y1与选育群体F1间的近交系数(Fis)最高(0.0875),且基因流(Nm)也较高(4.2728);野生群体Y2与选育群体F1间的遗传分化指数(Fst)最高(0.0947),但Nm最低(2.3886),表明二者间的遗传背景较远。4个凡纳滨对虾群体的遗传变异主要来源于群体内(85.15%),仅有14.85%变异来源于群体间。4个凡纳滨对虾群体的遗传相似性系数为0.9351~0.9747,遗传距离为0.0256~0.0671;基于凡纳滨对虾群体遗传距离的UPGMA聚类分析结果显示,Y1、F1和F2聚为一支,而Y2单独聚为一支。【结论】凡纳滨对虾野生群体在抗逆功能基因SNP分子标记中的遗传多样性显著高于选育群体,且野生群体与选育群体间的遗传距离较远,具有较高的遗传改良潜力。在实际生产中,可通过杂交措施将野生群体的优良抗逆基因引入选育群体,提高凡纳滨对虾选育群体的免疫及抗病能力。
关键词: 凡纳滨对虾;野生群体;选育群体;SNP分子标记;生长功能基因;抗逆功能基因;遗传多样性
中图分类号: S945.49 文献标志码: A 文章编号:2095-1191(2020)11-2836-10
Genetic diversity analysis in wild and selected populations of Penaeus vannamei based on SNP markers in growth
and stress resistance genes
SHEN Shu-hui1,2,3, DAI Xi-lin1,2,3*
(1National Demonstration Center for Experimental Fisheries Science Education(Shanghai Ocean University), Shanghai 201306, China; 2Key Laboratory of Freshwater Aquatic Genetic Resources, Ministry of Agriculture and Rural Affairs (Shanghai Ocean University), Shanghai 201306, China; 3Shanghai Collaborative Innovation for Aquatic Animal
Genetics and Breeding(Shanghai Ocean University), Shanghai 201306, China)
Abstract:【Objective】The genetic diversity and genetic differences between wild populations and selected populations of Litopenaeus vannamei were analyzed to provide reference for improvement and optimization of germplasm resources. 【Method】Single nucleotide polymorphisms(SNPs) found from amplification primers, which designed with high-throughput sequencing of L. vannamei, were verified using ten wild L. vannamei by sequencing. Finally, 11 primers and 24 identified SNPs in growth and stress resistance genes, were selected to analyze genetic diversity of Y1, Y2, which were wild populations, and F1, F2, which were selected populations. 【Result】The results showed that the genetic parameters such as observed heterozygosity(Ho), expected heterozygosity(He) and polymorphism information content(PIC) in Y1 and Y2 were higher than those in F1 and F2. Generally, all the genetic parameters in Y2 were higher than Y1. Similarly, in two selected families, genetic parameters of F2 were higher than F1. In addition, the genetic parameters in the two selected populations were 0 at some SNP loci of functional genes such as CATL, ANT, Tryptase, Crustin-like and Penaeidin4c. Deviation from Hardy-Weinberg equilibrium existed in all four populations differently. The coefficient of inbreeding (Fis) was the highest(0.0875) and the gene flow(Nm) was also high (4.2728) between wild population Y1 and selected population F1. The genetic differentiation index(Fst) between Y2 and F1 was the highest(0.0947), but Nm was the lowest (2.3886), indicating a distant genetic relationship between the two populations. AMOVA analysis found that 85.15% genetic variation of four populations was mainly within the population, and only 14.85% was found between populations.The genetic similarity coefficients of the four populations ranged from 0.9351 to 0.9747, and the genetic distance varied from 0.0256 to 0.0671. UPGMA clustering analysis showed that Y1, F1 and F2 clustered into a cluster, while Y2 clustered into a single cluster. 【Conclusion】Generally, in wild populations, genetic diversity of SNP markers in stress resistance function gene is significantly higher than that of selected populations. And wild populations are far from the selected populations, which shows a high potential for breeding. Therefore, it is necessary that the excellent stress resistance genes from wild populations should be introduced into the selected population through hybridization in production process, to improve the immunity and disease resistance of the selected population.
Key words: Litopenaeus vannamei; wild population; selected populations; SNP markers; growth functional genes; stress resistance genes;genetic diversity
Foundation item: Special Project of Shanghai Shrimp Industry Technology System Construction (HNKGZ〔2014〕7-1-11)
0 引言
【研究意义】凡纳滨对虾(Litopenaeus vannamei)又称南美白对虾,原产于太平洋沿岸,因具有含肉率高、抗逆性强、生长快、饵料需求低、便于活虾运输及耐高密度和低鹽度养殖等优点,已发展成为世界上最主要的虾类养殖品种,约占虾类总产量的85%(杨铭等,2017;李强勇等,2020)。随着对虾养殖业的不断发展,因病害频发和种质退化造成的对虾抗病力下降、生产周期延长及产量下降等问题日益凸显,给对虾养殖带来巨大经济损失,严重制约了凡纳滨对虾养殖业的持续健康发展(戴习林等,2018;蒋葛等,2019)。因此,培育抗逆性强且生长快的优良对虾养殖品种,是对虾养殖业今后发展的重要任务(杨铭等,2017;叶宁等,2017)。其中,引进原种或改良现有养殖品种是恢复和优化凡纳滨对虾种质的有效途径,但在遗传改良过程中必须掌握已有群体的种质资源现状及遗传背景。【前人研究进展】对虾的遗传育种工作起步相对较晚,但随着PCR和分子标记技术的不断成熟及推广应用,近年来对虾遗传育种研究取得了长足进展。于洋(2014)、郝敏(2016)、李东宇等(2016)、王全超(2017)、Yu等(2019)先后从凡纳滨对虾遗传图谱构建、数量性状定位、遗传多样性分析、亲权鉴定及分子辅助育种等方面开展研究,研究结果为凡纳滨对虾的良种选育提供了参考依据。目前,针对凡纳滨对虾的遗传学研究主要是利用等位酶、单拷贝核DNA、线粒体DNA、限制性片段长度多态性(RFLP)、扩增片段长度多态性(AFLP)及微卫星(SSR)等分子标记进行群体遗传背景分析(张灵侠等,2008;叶宁等,2017;陈锦豪等,2019),其中又以SSR分子标记在凡纳滨对虾遗传多样性研究中的应用最广泛。Rezaee等(2016)、刘洪涛等(2018)、方振朋等(2019)、黄小帅等(2019)分别研发出SSR分子标记并应用于凡纳滨对虾群体遗传多样性研究,但SSR分子标记也存在一些缺陷,包括可能存在无效等位基因、无法存在于功能基因中及费时费力等。单核苷酸多态性标记(Single nucleotide polymorphism,SNP)作为第三代分子标记,属于共显性标记,虽然每个标记的基础信息量较少,但能稳定、均匀、广泛地分布于整个基因组中,且可能出现在功能基因内部,更有利于开展生长、抗逆及繁殖等相关功能基因的研究,是分析遗传变异的有效分子工具(张晓萌等,2013;王全超,2017;陈静等,2018)。有关SNP分子标记在水产动物群体遗传学方面的应用研究已有较多报道,Agarwal等(2016)通过高通量测序获得大量罗氏沼虾SNP分子标记,并用于分析印度不同区域罗氏沼虾群体的遗传背景;于磊等(2018)基于酶切测序技术开发获得38个多态性较高的SNP分子标记,并以此分析辽宁丹东和福建三沙2个日本鳗鲡群体的遗传结构及其本地适应性;Thongda等(2018)研究表明,开发获得的58个东方牡蛎SNP分子标记均可用于遗传分化、群体结构分析及亲缘关系鉴定等相关研究。【本研究切入点】SNP分子标记是一种研究物种遗传变异的理想分子标记,但至今鲜见应用于凡纳滨对虾遗传多样性分析的研究报道。【拟解决的关键问题】基于凡纳滨对虾转录组数据,挖掘与生长和抗逆相关候选基因中的SNP位点,并以此分析凡纳滨对虾野生群体与选育群体的遗传多样性及遗传差异,为进一步改良优化凡纳滨对虾种质提供参考依据。
1 材料与方法
1. 1 样品采集及DNA提取
凡纳滨对虾样本为2个厄瓜多尔野生群体和2个选育群体。其中,厄瓜多尔野生群体为2016和2019年从厄瓜多尔引进的野生凡纳滨对虾,捕捞自瓜亚基乐湾水域,分别命名为Y1和Y2;选育群体是以美国关岛大学引进家系选育获得的子二代,分别命名为F1和F2。从各群体中随机取30尾样本,无水酒精固定后以天根海洋动物组织基因组DNA提取试剂盒提取其DNA,-20 ℃冰箱中保存备用。
1. 2 SNP位点发掘与引物筛选
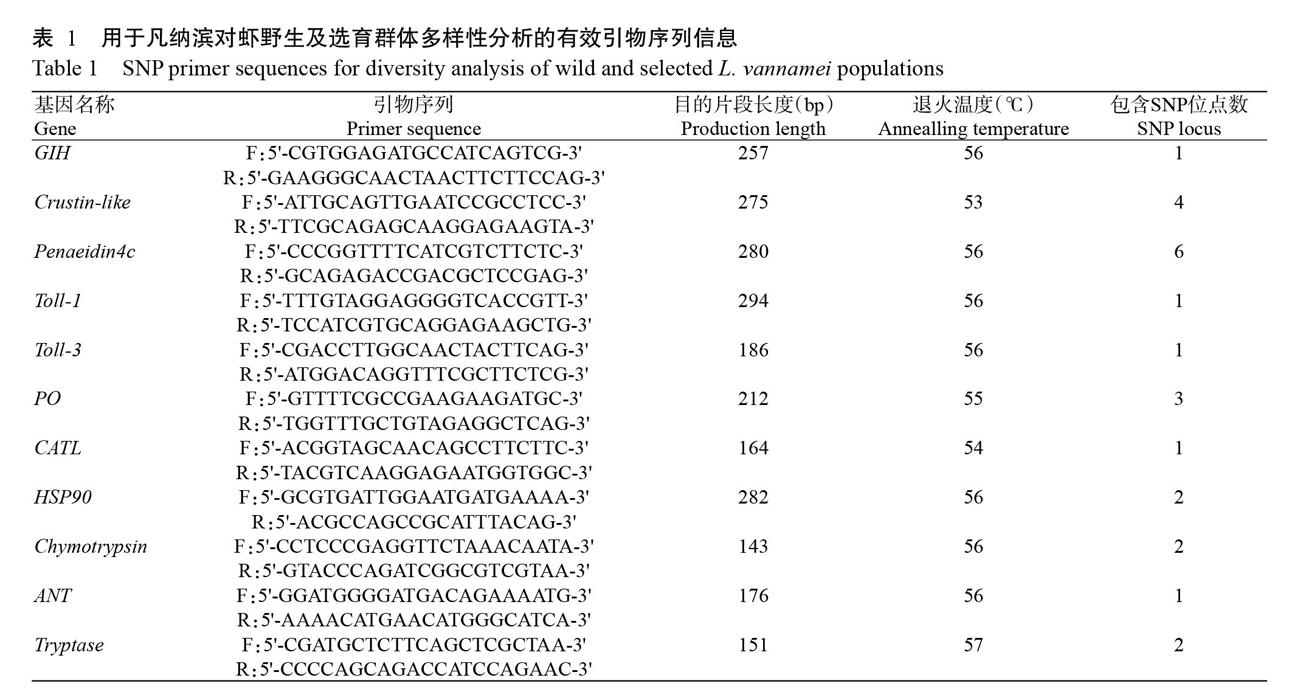
根据农业农村部淡水水产种质资源重点实验室已有转录组数据中的基因注释,挖掘与凡纳滨对虾生长和抗逆相关候选基因的预测SNP位点,包括胰蛋白酶(Tryptase)、糜蛋白酶(Chymotrypsin)、保幼激素(JH)(杜育哲等,2001)、腺苷酸转移酶(ANT)、性腺抑制激素(GIH)(邱庆庆,2019)、组织蛋白酶L(CATL)、有丝分裂原活化蛋白激酶(MAPK)、Alpha淀粉酶(AMY)(刘立鹤等,2006)、热休克蛋白(HSP90)(夏西超等,2012)、ATP转运酶、对虾素(Penaeidin)(杜志强,2012,2013)、酚氧化酶(Phenoloxidase,PO)(樊廷俊,2002)、β-葡聚糖结合蛋白(BGBP) (胡世康等,2018)、Toll受体(黄旭雄等,2012)和Crustin-like(张艳艳等,2012)等功能基因。然后选用10尾野生凡纳滨对虾样本进行验证,在挑选基因序列中的预测SNP位点两端设计引物,PCR扩增结果送至上海迈浦生物科技有限公司测序。测序结果使用BioEdit进行分析,读取转录本预测位置的基因型,在10尾野生凡纳滨对虾中其杂合基因型概率达20%的SNP位点所对应的引物即为有效引物,可用于后续的凡纳滨对虾群体基因型检测。
1. 3 统计分析
获得4个凡纳滨对虾群体的基因分型数据后,使用PopGen32计算群体的观测杂合度(Ho)、期望杂合度(He)、Nei基因多样性指数(H)、Hardy-Weinberg平衡偏离指数(D)、近交系数(Fis)、遗传分化指数(Fst)、遗传相似性系数及遗传距离等(戴习林等,2017);利用Cervus 3.0计算SNP位点的多态信息含量(PIC),剔除多态性过低的SNP位点;采用Arlequin V3.5分析Fst和基因流(Nm),并以MEGA 5.0进行UPGMA聚类分析。
2 结果与分析
2. 1 SNP位点发掘及引物筛选结果
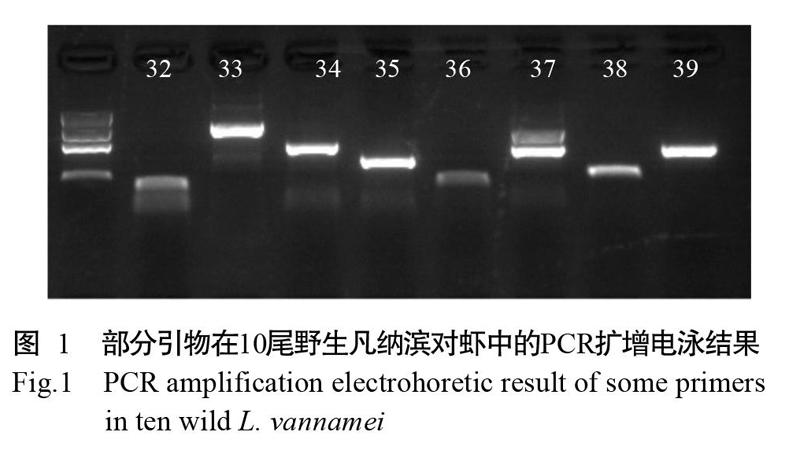
从转录组中挖掘获得70个SNPs位点,设计对应的引物并在10尾野生虾中进行PCR扩增,结果显示有46对引物能成功扩增出目的片段。图1为部分扩增结果的凝胶电泳图,将扩增获得且片段大小在预期范围内的单一明亮条带(编号33、34、35和39)送至上海迈浦生物科技有限公司测序。根据测序结果读取峰图,并统计基因型,发现SNP杂合基因型概率达20%的有效引物共计15对,包含33个SNPs位点。采用15对筛选出的有效引物对4个凡纳滨对虾群体进行PCR扩增,分别统计33个SNPs位点的基因型,并剔除在4个凡纳滨对虾群体中突变率均低于15%的SNP位点,最终获得11对有效引物(表1),包含24个SNPs位点。其中,HSP90、GIH、CATL、ANT、Tryptase和Chymotrypsin为生长功能基因,含有9个SNPs位点;Toll、Crustin-like、Penaeidin4c和PO为抗逆功能基因,含有15个SNPs位点。
2. 2 遗传多态性分析结果
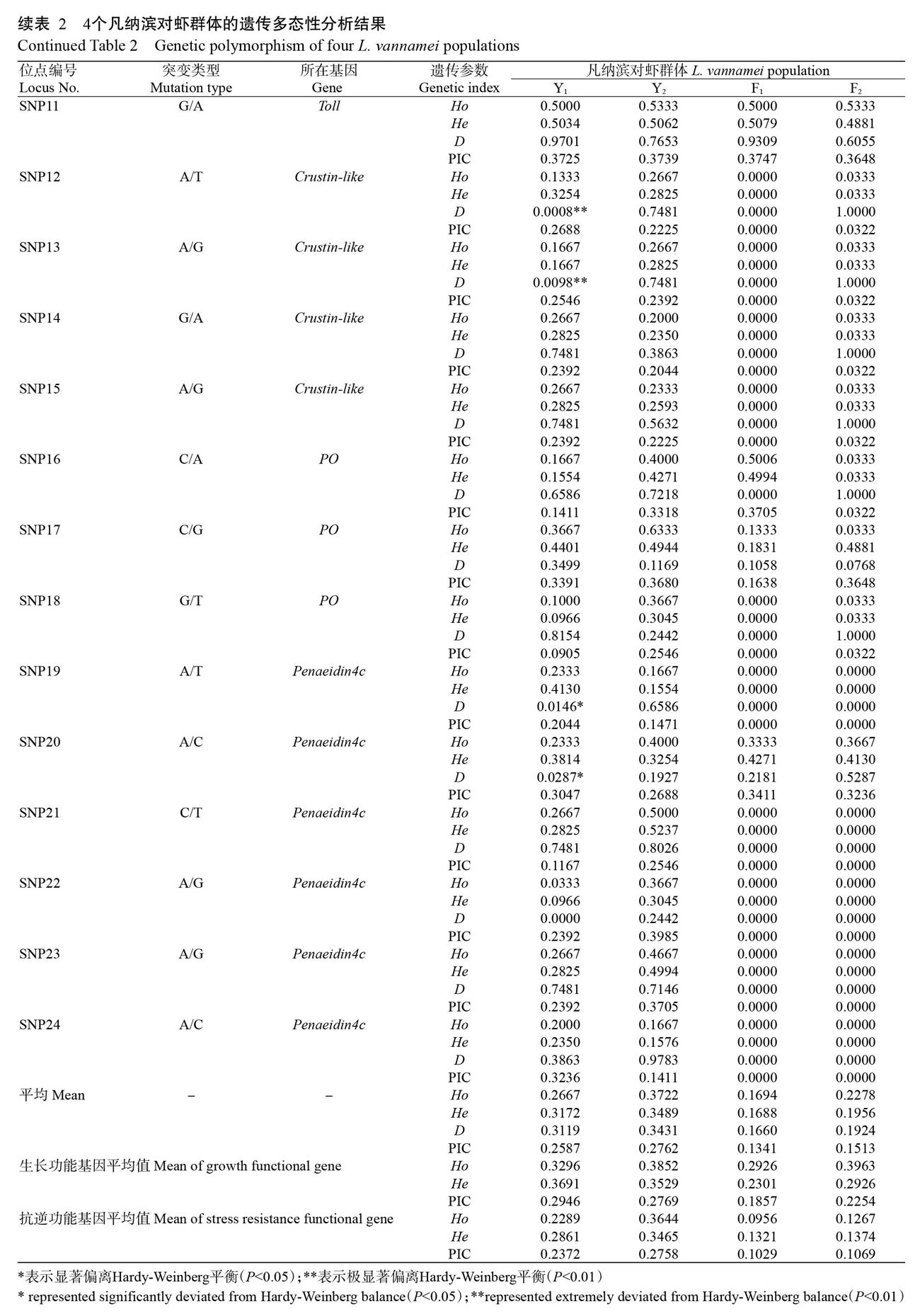
基于9个生长功能基因SNPs位点和15个抗逆功能基因SNPs位点,研究4个凡纳滨对虾群体(2个野生群体和2个选育群体)的遗传多样性。结果(表2)显示,24个SNP分子标记在4个凡纳滨对虾群体中的PIC平均值分别为Y1:0.2587、Y2:0.2762、F1:0.1341和F2:0.1513,2个野生群体明显高于2个选育群体。其中,Y1和Y2属于中度多态水平(0.25 总体上,2个野生群体(Y1和Y2)的各项遗传参数平均值明显高于2个选育群体(F1和F2)。在2個野生群体中,Y2的各项遗传参数平均值均高于Y1,在生长功能基因中Y1的PIC和He平均值高于Y2,但在抗逆功能基因中Y2高于Y1。在2个选育群体中,F2的各项遗传参数平均值均高于F1,在抗逆功能基因中F1和F2基本相同,但在生长功能基因中二者存在明显差异,表现为F2高于F1。在CATL、ANT、Tryptase、Crustin-like和Penaeidin4c等功能基因的某些SNP位点上,2个选育群体(F1和F2)的各项遗传参数平均值为0,可能是杂合子缺失所引起。4个凡纳滨对虾群体中均存在SNP位点不同程度偏离Hardy-Weinberg平衡现象(P<0.05为显著偏离,P<0.01为极显著偏离)。其中,Y1群体的SNP19和SNP20位点显著偏离Hardy-Weinberg平衡,处于Penaeidin4c基因上;SNP12和SNP13位点极显著偏离Hardy-Weinberg平衡,位于Crustin-like基因上;Y2群体的SNP3和SNP7位点显著偏离Hardy-Weinberg平衡,分别位于GIH和Chymotrypsin基因上;F1群体中除突变缺失位点外,仅SNP3位点显著偏离Hardy-Weinberg平衡,位于GIH基因上;F2群体中除突变缺失位点外,仅SNP6和SNP7位点显著偏离Hardy-Weinberg平衡,均位于Chymotrypsin基因上。 2. 3 遗传分化分析结果 4个凡纳滨对虾群体在24个SNPs位点上的Fis平均值为-0.0224,且负值较多(表3),表明不同群体间的近交程度较低。不同群体间各SNP位点的Fst变化范围为0.0050~0.2754,说明4个凡纳滨对虾群体在24个SNPs位点上表现出明显的遗传分化。4个凡纳滨对虾群体相互间的Fis、Fst和Nm详见表4。其中,Y1与F1间的Fis最高(0.0875),且Nm也较高(4.2728);Y2与F1间的Fst最高(0.0947),但其Nm最低(2.3886),表明Y2与F1间的遗传背景较远。基于SNP分子标记的凡纳滨对虾群体遗传变异分子生物学方差分析结果(表5)显示,4个凡纳滨对虾群体的遗传变异大部分来源于群体内(85.15%),仅有14.85%变异来源于群体间。 2. 4 遗传距离及聚类分析结果 由表6可知,4个凡纳滨对虾群体的遗传相似性系数为0.9351~0.9747,遗传距离为0.0256~0.0671。其中,F1与F2的遗传相似性系数最大(0.9747),Y2与F1的遗传相似性系数最小(0.9351);F1与F2的遗传距离最小(0.0256),Y2与F1的遗传距离最大(0.0671)。基于凡纳滨对虾群体间的遗传距离,采用MEGA 5.0进行UPGMA聚类分析,结果(图2)显示,Y1、F1和F2聚为一支,而Y2单独聚为一支。 3 讨论 SNP可通过改变碱基引起所处基因的表达变化,导致其编码蛋白发生结构功能变异,进而影响表型性状。目前,开展水产动物遗传多样性分析时有关分子标记的选择尚存在随机性,且主要针对相关功能基因的分子标记进行研究(崔蕾等,2012;孙成飞等,2015;武菲等,2016),利用SNP分子标记进行遗传多样性的研究相对较少。本研究基于凡纳滨对虾的生长和抗逆相关功能基因,并通过转录组注释筛选其中的候选SNP位点,最终筛选出24个SNPs位点(9个SNPs位点位于生长功能基因上,15个SNPs位点位于抗逆功能基因上)用于凡纳滨对虾野生和选育群体遗传多样性分析。生长和抗逆相关功能基因中的SNP位点差异会影响其所处功能基因表达,相对于其他中性位点更容易造成机体生长性状及抗病能力的差异,若将其应用到群体遗传背景分析中,则有望从DNA水平了解群体生长性状及抗病能力的遗传差异,更有利于后续将分子标记与性状联系起来,辅助良种选育工作的开展。 本研究通过转录组数据结合直接测序挖掘出24个功能基因相关SNPs位点(9个生长功能基因SNPs位点,15个抗逆功能基因SNPs位点),并应用于凡纳滨对虾野生和选育群体遗传多样性分析,结果表明,野生群体Y1和Y2的遗传多样性水平显著高于选育群体F1和F2,与董在杰等(2018)在鲤鱼、王军等(2018)在中国明对虾、王沈同等(2018)在草鱼中的研究结果相似,可能是由于选育过程中封闭群体的亲本数量限制、近亲交配、遗传漂变及对养殖环境适应等因素所引起。2个野生群体具有更丰富的遗传多样性,说明其进化潜力更高,即具有较高的遗传育种改良潜力和适应生存能力。此外,在2个野生群体中,Y2的各项遗传参数平均值均高于Y1,在生长功能基因中Y1的PIC和He平均值高于Y2,在抗逆功能基因中则Y2高于Y1,究其原因可能是不同野生种群在自然环境中迁移或变异所造成。在2个选育群体中,F2的各项遗传参数平均值均高于F1,在抗逆功能基因中F1和F2基本相同,但在生长功能基因中二者存在明显差异,表现为F2高于F1,说明在选择育种中F2较F1更具优势。在CATL、ANT、Tryptase、Crustin-like和Penaeidin4c等功能基因的某些SNP位点上,2个选育群体(F1和F2)的各项遗传参数平均值为0,推测F1和F2存在杂合子缺失现象,与颉晓勇等(2008)、叶宁等(2017)的研究结果相似,是由于人工选育过程中的近亲交配现象或对某一特定基因型个体的筛选,造成某些基因从该基因库中流失。凡纳滨对虾遗传多样性降低,极易造成种质资源退化、生长滞缓,进而严重影响其养殖业的健康发展(李强勇等,2020)。因此,在凡纳滨对虾选育过程中,需及时了解其遗传多样性水平,并在此基础上进行科学管理,才能在遗传改良的同时维持选育群体具有较高的遗传多样性水平,或从野生群体引种,进一步丰富凡纳滨对虾选育群体的遗传多样性。 从本研究的遗传分化及UPGMA聚类分析可看出,凡纳滨对虾野生群体Y1与2个选育群体(F1和F2)聚为一支,且Nm和Fis均较高,与三者间遗传距离较近的结果相对应;而凡纳滨对虾野生群体Y2与2个选育群体(F1和F2)間的Fst较高,且Nm较低,与三者间遗传距离最远的结果相对应,表明野生群体Y2在家系选择育种中更具引种优势和选育潜力。分子生物学方差分析结果也显示,4个凡纳滨对虾群体的遗传变异主要来源于群体内(85.15%),仅有14.85%变异来源于群体间,与童馨等(2009)、Rezaee等(2016)的研究结果类似,说明不同凡纳滨对虾群体间相似性较小且基因交流较少,仍具有较高的选育潜力。本研究还发现,在生长功能基因SNP位点遗传多样性分析中,F2的遗传多样性水平明显高于F1,且在相同养殖条件下F2的生长性能也优于F1,说明F2无论是在遗传上还是生长性状上均优于F1,在后续执行选育计划时应优先考虑。但从抗逆性状的遗传背景来看,2个选育群体(F1和F2)均不具备抗逆优势,在传染病频发的养殖现状中极易被淘汰。因此,在实际生产中非常有必要通过选育群体与野生群体杂交以提高凡纳滨对虾后代的抗逆能力,可采取抗逆性状高的野生群体Y2与选育群体相杂交的措施,将Y2的优良抗逆基因引入选育群体,优化其种质资源,提高凡纳滨对虾选育群体的免疫及抗病能力。 4 结论 凡纳滨对虾野生群体在抗逆功能基因SNP分子标记中的遗传多样性显著高于选育群体,且野生群体与选育群体间的遗传距离较远,具有较高的遗传改良潜力。在实际生产中,可通过杂交措施将野生群体的优良抗逆基因引入家系选育群体,优化其种质资源,以提高凡纳滨对虾选育群体的免疫及抗病能力。 参考文献: 陈锦豪,郑锦滨,王攀攀,李天骄,毛勇,苏永全,王军. 2019. 凡纳滨对虾养殖亲本群体遗传多样性分析[J]. 渔业研究,41(1):1-10. [Chen J H,Zheng J B,Wang P P,Li T J,Mao Y,Su Y Q,Wang J. 2019. Analysis for genetic diversity of broodstock populations of breeding Litopenaeus vannamei[J]. Journal of Fisheries Research,41(1):1-10.] 陈静,何吉祥,樊佳佳,黄龙,吴本丽,宋光同,汪翔,武松. 2018. 草鱼MyoD基因SNP和Indel标记的筛选及其与生长性状的关联分析[J]. 江苏农业学报,34(3):612-616. [Chen J,He J X,Fan J J,Huang L,Wu B L,Song G T,Wang X,Wu S. 2018. Screening of SNP and Indel mar-ker of MyoD gene and its association with growth traits in Ctenopharyngodon idella[J]. Jiangsu Journal of Agricultural Sciences,34(3):612-616.] 崔蕾,谢从新,李艳和,于永耀,刘肖莲,张杰,周小云. 2012. 斑点叉尾鮰4个群体遗传多样性的微卫星分析[J]. 华中农业大学学报,31(6):744-751. [Cui L,Xie C X,Li Y H,Yu Y Y,Liu X L,Zhang J,Zhou X Y. 2012. Analysis of genetic diversity among four different channel catfish populations by using microsatellite markers[J]. Journal of Huazhong Agricultural University,31(6):744-751.] 戴习林,刘洁,李晶晶,江宗冰,明磊,蒋飞. 2017. 罗氏沼虾种群SSR分析中样本量及标记量对遗传多样性指标的影响[J]. 水产学报,41(7):1083-1095. [Dai X L,Liu J,Li J J,Jiang Z B,Ming L,Jiang F. 2017. Effects of sample size and loci number on genetic diversity index in Macrobrachium rosenbergii with microsatellite markers[J]. Journal of Fisheries of China,41(7):1083-1095.] 戴习林,杨展昆,朱其建. 2018. 凡纳滨对虾室内养殖密度和简易水质调控措施对水质及养殖效果的影响[J]. 上海海洋大学学报,27(6):894-906. [Dai X L,Yang Z K,Zhu Q J. 2018. Effects of indoor aquaculture density and simple water quality control measures on water quality and culture efficiency of Litopenaeus vannamei[J]. Journal of Shanghai Ocean University,27(6):894-906.] 董在杰,劉念,傅建军,朱文彬,王兰梅,苏胜彦. 2018. 6个野生与选育鲤群体的微卫星遗传分析[J]. 南方水产科学,14(4):48-57. [Dong Z J,Liu N,Fu J J,Zhu W B,Wang L M,Su S Y. 2018. Genetic analysis for six wild and selection populations of common carp(Cyprinus carpio) using microsatellites[J]. South China Fisheries Science,14(4):48-57.] 杜育哲,郭世宜,刘丽. 2001. 甲壳动物保幼激素的研究[J]. 天津农学院学报,8(3):34-36. [Du Y Z,Guo S Y,Liu L. 2001. Juvenile hormone in crustacean[J]. Journal of Tianjin Agricultural College,8(3):34-36.] 杜志强. 2012. 先天免疫系统中抗菌肽的研究进展[J]. 广东农业科学,39(10):231-233. [Du Z Q. 2012. Research progress in antimicrobial peptides of innate immune system[J]. Guangdong Agricultural Sciences,39(10):231-233.] 杜志强. 2013. 凡纳滨对虾对虾素3a基因的克隆与序列分析[J]. 云南农业大学学报(自然科学),28(1):79-82. [Du Z Q. 2013. Gene cloning and sequence analysis of Penaeidin3a in Litopenaeus vannamei[J]. Journal of Yunnan Agricultural University(Natural Science),28(1):79-82.] 樊廷俊. 2002. 对虾非特异性免疫与对虾疾病监控的研究进展[J]. 海洋科学,26(4):26-31. [Fan T J. 2002. Resear-ching Progresses in shrimp non-specific immunity and di-sease control[J]. Marine Sciences,26(4):26-31.] 方振朋,孟宪红,李旭鹏,栾生,曹家旺,陈宝龙,孔杰,闫茂仓,胡利华. 2019. 基于微卫星分子标记的凡纳滨对虾商业苗种遗传多样性分析[J].渔业科学进展,41(5):101-109. [Fang Z P,Meng X H,Li X P,Luan S,Cao J W,Chen B L,Kong J,Yan M C,Hu L H. 2019. Genetic diversity analysis of domestic commercial brands seedlings of Litopenaeus vannamei based on microsatellite molecular markers[J]. Progress in Fishery Sciences,41(5):101-109.] 郝敏. 2016. 凡纳滨对与生长繁殖相关的遗传标记的筛选[D]. 上海:上海海洋大学. [Hao M. 2016. The genetic marker screening for growth and reproduction properties in Litopenaeus vannamei[D]. Shanghai:Shanghai Ocean University.] 胡世康,王博,秦海鹏,廖栩峥,孙成波,刘永胜,曲朋. 2018. 抗菌肽对凡纳滨对虾抗病性和免疫指标的影响[J]. 热带生物学报,9(3):281-286. [Hu S K,Wang B,Qin H P,Liao X Z,Sun C B,Liu Y S,Qu P. 2018. Effect of microbial antimicrobial peptides on disease resistance and immune indices of Litopenaeus vannamei[J]. Journal of Tropical Biology,9(3):281-286.] 黄小帅,徐煜,胡晓娟,徐武杰,苏浩昌,文国樑,杨铿,曹煜成. 2019. 利用微卫星标记分析7个凡纳滨对虾引进群体子一代的遗传多样性[J]. 南方水产科学,15(1):54-62. [Huang X S,Xu Y,Hu X J,Xu W J,Su H C,Wen G L,Yang K,Cao Y C. 2019. Genetic diversity analysis of first filial generation of seven introduced Litopenaeus vannamei populations using microsatellite DNA markers[J]. South China Fisheries Science,15(1):54-62.] 黄旭雄,罗词兴,郭腾飞,温文,华雪铭,周洪琪. 2012. 凡纳滨对虾Toll受体和溶菌酶mRNA组织表达的差异研究[J]. 上海海洋大学学报,21(1):28-32. [Huang X X,Luo C X,Guo T F,Wen W,Hua X M,Zhou H Q. 2012. Expre-ssions of Toll receptor and lysozyme mRNA among ti-ssues of white shrimp,Litopenaeus vannamei[J]. Journal of Shanghai Ocean University,21(1):28-32.] 蒋葛,沈辉,万夕和,乔毅,黎慧,王李宝,史文军. 2019. 凡纳滨对虾急性肝胰腺坏死综合症病虾与健康虾肠道优势菌群比较分析[J]. 江苏农业学报,35(1):142-148. [Jiang G,Shen H,Wan X H,Qiao Y,Li H,Wang L B,Shi W J. 2019. Difference analysis of intestinal flora between healthy Litopenaeus vannamei and acute hepatopancreatic necrosis syndrome L. vannamei[J]. Jiangsu Journal of Agricultural Sciences,35(1):142-148.] 李东宇,孔杰,孟宪红,栾生,罗坤,卢霞,曹宝祥. 2016. 凡纳滨对虾(Litopenaeus vannamei)微卫星多重PCR体系的建立及其在家系亲权鉴定中的应用[J]. 渔业科学进展,37(3):58-67. [Li D Y,Kong J,Meng X H,Luan S,Luo K,Lu X,Cao B X. 2016. Development of multiplex PCR systems of microsatellite markers for Pacific white shrimp(Litopenaeus vannamei) and its application for parentage identification[J]. Progress in Fishery Sciences,37(3):58-67.] 李强勇,李旻,曾地刚,朱威霖,彭敏,杨春玲,刘青云,赵永贞,陈秀荔,陈晓汉. 2020. 凡纳滨对虾微卫星分子标记的开发及不同养殖家系遗传多态性分析[J]. 南方农业学报,51(2):429-436. [Li Q Y,Li M,Zeng D G,Zhu W L,Peng M,Yang C L,Liu Q Y,Zhao Y Z,Chen X L,Chen X H. 2020. Development of microsatellite markers of Litopenaeus vannamei and genetic polymorphism ana-lysis of different cultured families[J]. Journal of Southern Agriculture,51(2):429-436. 劉洪涛,杨明秋,何玉贵,唐贤明. 2018. 凡纳滨对虾八个地理群体遗传多样性的微卫星分析[J]. 海南大学学报(自然科学版),36(2):146-152. [Liu H T,Yang M Q,He Y G,Tang X M. 2018. Microsatellite analysis of genetic diversity in eight geographical populations of Litopenaeus vannamei[J]. Natural Science Journal of Hainan University,36(2):146-152.] 刘立鹤,陈立侨,周永奎,江洪波. 2006. 甲壳动物消化酶的研究[J]. 饲料工业,27(18):56-62. [Liu L H,Chen L Q,Zhou Y K,Jiang H B. 2006. Studies on digestive enzymes of crustaceans[J]. Feed Industry,27(18):56-62.] 邱庆庆. 2019. 罗氏沼虾生长性状候选基因的克隆及mRNA表达规律研究[D]. 南宁:广西大学. [Qiu Q Q. 2019. Cloning and mRNA expression of candidate genes for growth performance of Macrobrachium rosenbergii[D]. Nanning:Guangxi University.] 孙成飞,叶星,董浚键,田园园,梁健辉. 2015. 罗氏沼虾6个养殖群体遗传多样性的微卫星分析[J]. 南方水产科学,11(2):20-26. [Sun C F,Ye X,Dong J J,Tian Y Y,Liang J H. 2015. Genetic diversity analysis of six cultured populations of Macrobrachium rosenbergii using microsate-llite markers[J]. South China Fisherise Science,11(2):20-26.] 童馨,龚世圆,喻达辉,黄桂菊,杜博,李色东. 2009. 凡纳滨对虾(Litopenaeus vannamei)不同世代养殖群体的遗传多样性分析[J]. 海洋与湖沼,40(2):214-220. [Tong X,Gong S Y,Yu D H,Huang G J,Du B,Li S D. 2009. Genetic diversity of cultured Pacific white shrimp(Litopenaeus vannamei) stocks of different generations in China[J]. Oceanologia et Limnologia Sinica,40(2):214-220] 王军,王清印,孔杰,孟宪红,曹家旺,王明珠,冯亚萍,吕丁. 2018. 中国明对虾人工选育群体与野生群体遗传多样性的SSR分析[J]. 渔业科学进展,39(2):104-111. [Wang J,Wang Q Y,Kong J,Meng X H,Cao J W,Wang M Z,Feng Y P,Lü D. 2018. SSR analysis on genetic diversity in breeding and wild populations of Fenneropenaeus chinensis[J]. Progress in Fishery Sciences,39(2):104-111.] 王全超. 2017. 凡纳滨对虾生长和抗病性状的全基因组关联分析与基因组选择育种研究[D]. 北京:中国科学院大学. [Wang Q C. 2017. Genome-wide association study and genomic selection of growth and disease resistance traits in Litopenaeus vannamei[D]. Beijing:University of Chinese Academy of Sciences.] 王沈同,沈玉帮,孟新展,王荣泉,李家乐. 2018. 草鱼野生与选育群体遗传变异微卫星分析[J]. 水产学报,42(8):1273-1284. [Wang S T,Shen Y B,Meng X Z,Wang R Q,Li J L. 2018. Genetic variability in wild and selected populations of Ctenopharyngodon idella using microsatellite markers[J]. Journal of Fisheries of China,42(8):1273-1284.] 王帅豪. 2012. 淅川乌骨鸡群体遗传多样性及绿壳蛋性状相关基因SNP分析[D]. 郑州:河南农业大学. [Wang S H. 2012. SNP analysis about green egg shell correlation gene and population genetic diversity of Xichuan silky[D]. Zhengzhou:Henan Agricultural University.] 武菲,胡文革,王翠華,郭飏,莫超. 2016. 河鲈养殖与野生群体遗传多样性比较分析[J]. 水生生物学报,40(1):181-188. [Wu F,Hu W G,Wang C H,Guo Y,Mo C. 2016. Genetic diversity of the cultivated and natural Perca fluviatilis[J]. Acta Hydrobiologica Sinica,40(1):181-188.] 夏西超,杨洪,王文锋,李昕,宁黔冀. 2012. 凡纳滨对虾热休克蛋白90基因cDNA全长克隆及表达分析[J]. 水产科学,31(11):673-678. [Xia X C,Yang H,Wang W F,Li X,Ning Q J. 2012. Molecular cloning and expression of heat shock protein 90(HSP90) gene in Pacific white leg shrimp Litopenaeus vannamei[J]. Fisheries Science,31(11):673-678.] 颉晓勇,苏天凤,陈文,张志,颜远义,江世贵. 2008. 凡纳滨对虾6个养殖群体遗传多样性的比较分析[J]. 南方水产,4(6):42-49. [Xie X Y,Su T F,Chen W,Zhang Z,Yan Y Y,Jiang S G. 2008. Analysis on genetic diversity of six cultured stocks of Litopenaeus vannamei[J]. South China Fisheries Science,4(6):42-49.] 杨铭,于洋,张晓军,王全超,刘敬文,李富花,相建海. 2017. 基于转录组数据的凡纳滨对虾微卫星标记开发[J]. 海洋科学,41(2):96-102. [Yang M,Yu Y,Zhang X J,Wang Q C,Liu J W,Li F H,Xiang J H. 2017. Development of microsatellite markers from the transcriptome sequences of Pacific white shrimp(Litopenaeus vannamei)[J]. Marine Sciences,41(2):96-102.] 葉宁,包秀凤,刘建勇. 2017. 凡纳滨对虾引进群体与养殖群体遗传多样性的AFLP分析[J]. 水产学报,41(3):339-346. [Ye N,Bao X F,Liu J Y. 2017. AFLP analysis of genetic diversity of imported Litopenaeus vannamei popu-lations and cultured stocks[J]. Journal of Fisheries of China,41(3):339-346.] 于磊,刘炳舰,刘进贤. 2018. 日本鳗鲡离散SNP标记筛选及群体遗传多样性分析[J]. 中国海洋大学学报(自然科学版),48(4):37-47. [Yu L,Liu B J,Liu J X. 2018. Outlier SNPs development and preliminary genetic diversity ana-lysis of Japanese eel(Anguilla japonica)[J]. Periodical of Ocean University of China,48(4):37-47.] 于洋. 2014. 凡纳滨对虾分子标记的开发及其在遗传育种中的应用[D]. 北京:中国科学院大学. [Yu Y. 2014. Deve-lopment of molecular markers and their applications in selective breeding of the Pacific white shrimp,Litopenaeus vannamei[D]. Beijing:University of Chinese Academy of Sciences.] 张灵侠,沈琪,胡超群. 2008. 凡纳滨对虾的遗传育种研究现状[J]. 海洋科学,32(2):91-95. [Zhang L X,Shen Q,Hu C Q. 2008. A review of genetics and breeding of Litopenaeus vannamei[J]. Marine Sciences,32(2):91-95.] 张晓萌,马普,王洪迪,王秀利. 2013. SNPs在水产动物中的研究进展[J]. 生物技术通报,(8):7-11. [Zhang X M,Ma P,Wang H D,Wang X L. 2013. Progresses of SNPs studies in aquaculture animals[J]. Biotechnology Bulletin,(8):7-11.] 张艳艳,刘小林,黄海洪,钱昭英,王宪宗,相建海,李义军. 2012. 凡纳滨对虾Crustin-like基因的克隆及在副溶血弧菌感染条件下的表达分析[J]. 西北农林科技大学学报(自然科学版),40(12):41-48. [Zhang Y Y,Liu X L,Huang H H,Qian Z Y,Wang X Z,Xiang J H,Li Y J. 2012. Molecular cloning of Crustin-like gene in the white shrimp(Litopenaeus vannamei) and its mRNA expression with Vibrio parahaemolyticus challenge[J]. Journal of Nor-thwest A & F University(Natural Science Edition),40(12):41-48.] Agarwal D,Aich N,Pavan-Kumar A,Kumar S,Sabnis S,Joshi C G,Koringa P,Pandya D,Patel D,Karnik T,Bhingarde R,Gireesh-Babu P,Chaudhari A. 2016. SNP mi-ning in transcripts and concomitant estimation of genetic variation in Macrobrachium rosenbergii stocks[J]. Conservation Genetics Resources,8(2):159-168. Rezaee S,Farahmand H,Nematollahi M A. 2016. Genetic diversity status of Pacific white shrimp(Litopenaeus vannamei) using SSR markers in Iran[J]. Aquaculture International,24(2):479-489. Thongda W,Zhao H G,Zhang D D,Jescovitch L N,Liu M,Guo X M,Schrandt M,Powers S P,Peatman E. 2018. Development of SNP panels as a new tool to assess the genetic diversity,population structure,and parentage ana-lysis of the eastern oyster(Crassostrea virginica)[J]. Marine Biotechnology(New York),20(3):385-395. Yu Y,Wang Q C,Zhang Q,Luo Z,Wang Y,Zhang X J,Huang H,Xiang J H,Li F H. 2019. Genome scan for genomic regions and genes associated with growth trait in Pacific white shrimp Litopenaeus vannamei[J]. Marine Biotechnology(New York),21(3):374-383. (责任编辑 兰宗宝) 收稿日期:2020-02-15 基金项目:上海市虾类产业技术体系建设专项(沪农科攻字〔2014〕第7-1-11号) 作者简介:*为通讯作者,戴习林(1969-),教授,主要从事水产养殖与海洋生物学研究工作,E-mail:xldai@shou.edu.cn。申淑慧(1995-),研究方向为罗氏沼虾遗传育种,E-mail:18994535472@163.com
