FtsZ小分子抑制剂的筛选及抗S.aureus活性评价
2020-02-14姚舒生汪安勇王中新
姚舒生,刘 晨,汪安勇,王中新
细菌耐药已经成为一个全球性的问题,新型抗菌药物的研发需求已变得十分迫切[1-2]。传统抗菌药物获取的主要方式是对现有抗菌药物的结构改造,但这一策略的开展现在变得越来越困难,故寻找新的抗菌药物作用靶点、开发作用于新型靶点的抗菌药物已成为一条越来越重要的途径[3]。细丝温度敏感蛋白Z(filamentous temperature-sensitive protein Z, FtsZ)属于细菌分裂蛋白家族,广泛分布在原核细胞中,高度保守,具有GTP酶活性,作为细菌分裂的起始调控蛋白,参与细菌细胞分裂的整个过程[4-5]。研究发现,抑制FtsZ的功能能够阻止细菌细胞分裂体的形成,进而导致细菌细胞死亡[6-8]。
计算机辅助药物设计(computer-aided drug design, CADD)是近年来流行的一种重要的合理药物设计方法,其基本原理通过模拟小分子药物和大分子相互作用而设计新型药物先导化合物[9]。CADD技术的发展极大减少了新药研发的盲目性和偶然性,大大缩短了新药研发的周期,节约了大量的人力、财力,成为新药研发重要的一环[10]。该研究利用CADD技术,选择了来自于金黄色葡萄球菌(S.aureus)的FtsZ晶体结构(PDB号:4DXD)[11]为靶点,开展了虚拟筛选,对筛选的化合物进行体外抗S.aureus活性评价,并利用分子动力学模拟对部分筛选结果进行了分析,希望能够获取FtsZ抑制剂,为研发新的抗菌药物提供一定参考。
1 材料与方法
1.1 主要设备与试剂计算工作使用美国Accelrys公司的Discovery Studio 2017软件完成;小分子库来源于美国Chemidiv公司,筛选出的小分子化合物均购自于该公司;化合物PC190723购自北京百诺威生物科技有限公司;对甲氧西林敏感的金黄色葡萄球菌(MSSA,ATCC29213)、耐甲氧西林金黄色葡萄球菌(MRSA,ATCC43300)由安徽医科大学第一附属医院检验科提供;营养肉汤购自于杭州百思生物技术有限公司。
1.2 方法
1.2.1靶标的准备 FtsZ 蛋白晶体解析结构在Protein Data Bank数据库下载,PDB编号为4DXD。在筛选前首先进行预处理,包括删除水分子、原配体分子PC190723、非相关的蛋白质构象;其次定义活性中心,选择原配体位置为中心,把直径18 Å的范围内的氨基酸残基定义为活性氨基酸残基,保存备用。
1.2.2软件适用性评价 评价Discovery Studio 2017软件中将用于对接的模块,包括Libdock、Ligandfit、CDOCKER和Flexible Docking。选择定义的活性中心为对接位置,分别利用4种对接模块(对接参数设置见1.2.3项虚拟筛选)将4DXD晶体复合物中的原配体PC190723对接回原来位置,用对接结果中结合能力最好构象与原配体构象之间的均方根偏差值(root-mean-square difference,RMSD)评价各模块的适用性,RMSD≤1 Å,表示对接软件及参数设置具有高度适用性。
1.2.3虚拟筛选
1.2.3.1Libdock模块筛选 从Discovery Studio 2017操作界面调出Libdock 模块,设置参数如下:Ligands栏设置为Chemidiv 公司小分子数据库,Receptor栏设置为活性中心定义后的蛋白4DXD,Docking Preferences选择为High Quality,每个小分子定义为20个构象,其余参数为默认值。筛选结束后根据Libdock Score打分保留每个小分子分值最高的构象,以原配体PC190723的最高分值构象为阀值,保留高于阀值的小分子,命名为Group 1。
1.2.3.2Ligandfit模块筛选 从工作界面调出Ligandfit模块,设置参数如下:Ligands栏设置为Group 1,Receptor栏设置为活性中心定义后的蛋白4DXD,Scoring Functions中选择Dock-Score、每个小分子定义为20个构象,其余参数为默认值。筛选结束后保留每一个小分子Dock-Score打分最高构象,选择原配体PC190723做参照,筛选出Dock-Score打分高于PC190723最高打分的小分子,命名为Group 2。
1.2.3.3CDOCKER模块筛选 运行CDOCKER模块,把预处理后的4DXD设置为指定受体,小分子配体选择Group 2,对接位置设置为活性位点,每个小分子定义为20个构象,其余参数为默认值。完成对接后,选择-CDOCKER_TNTERACTION_ENERGY参数打分,保留每个小分子最高打分构象,按降序排列,筛选出打分高于PC190723的小分子,命名为Group 3。
1.2.3.4Flexible Docking模块筛选 首先定义活性位点中关键氨基酸残基,包括Gly205、Val207、Leu209、Asn263、Ser204,选定之后设置为Class 1。运行Flexible Docking模块,对接受体设置为4DXD,对接配体设置为Group 3,Selected Residues设置为Class 1,Conformation Method设置为Best,对接次数每个小分子设置为20个构象,其余参数为默认值。对接完成后,选择-CDOCKER_TNTERACTION_ENERGY参数打分,保留每个小分子最高打分构象,按降序排列,筛选出打分高于PC190723的小分子,命名为Group 4。
1.2.3.5类药性筛选 打开Small molecules工具栏,选择Filter Ligands工具面板中的命令工具Filter by Lipinski and Veber Rule,小分子定义为Group 4,根据Veber Rule规则和Lipinski’类药性原则筛选出Group 4适合成药的小分子,命名为Group 5。
1.2.4最小抑菌浓度(Minimum inhibitory concentration, MIC)检测 抗菌实验方法参照美国临床实验室标准委员会的方法进行,采用肉汤稀释法检测将Group 5中的化合物终浓度分别为256、128、64、32、16、8、4、2、1、0.5 μg/ml时的抗菌作用。具体操作步骤:取无菌96孔板,在每孔中加入100 μl的营养肉汤培养基,第一列每孔中再补加95 μl的营养肉汤培养基,随后分别取浓度为10.24 mg/ml的化合物5 μl加入到第一列各孔中,混匀,倍半稀释,最后从第10孔中吸取100 μl液体,最后在每孔中加入预配制的菌液5 μl,设置空白对照和阴性对照,做好标记,在37 ℃恒温箱中孵育24 h,观察结果,计算MIC值。
1.2.5分子动力学模拟 将对接后的4DXD蛋白-配体复合物首先进行溶剂化处理,处理后加 CHARMm 力场进行能量优化;其次在NVT 系统下进行模拟升温,温度参数设置为0~300 K,升温时间设置为0.25 ns,压力设置为1 atm,到达预定参数值后进行0.25 ns的平衡模拟,最后对体系进行5 ns的模拟,每0.002 ns保存一次轨迹文件。使用Discovery Studio 2017的Simulation模块完成。
2 结果
2.1 对接软件适用性评价使用4种对接模块Libdock、Ligandfit、CDOCKER和Flexble Docking对原配体和FtsZ蛋白晶体对接,见图1。结果显示4种对接模块对接后配体PC190723与对接前FtsZ蛋白晶体复合物中的同一配体PC190723相比,RMSD值均小于1 Å,说明这4种对接程序及参数设置均适用于分子对接的研究,对接结果是可信的。
2.2 虚拟筛选结果对ChemDiv公司小分子库中39 005个小分子化合物经多步筛选,使用PC190723作为对照。结果显示使用Libdock和LigandFit模块快速筛选后得到5 632个小分子化合物;使用CDOCKER对筛选得到的5 632个小分子化合物各个构象在受体活性位点进行优化,精确对接后得到473个小分子化合物;使用Flexible Docking模块针对关键氨基酸残基进行全柔性对接后剩余72个小分子化合物;最后通过类药性评价,获得15个具有成药潜力的小分子化合物。见图2、3和表1。
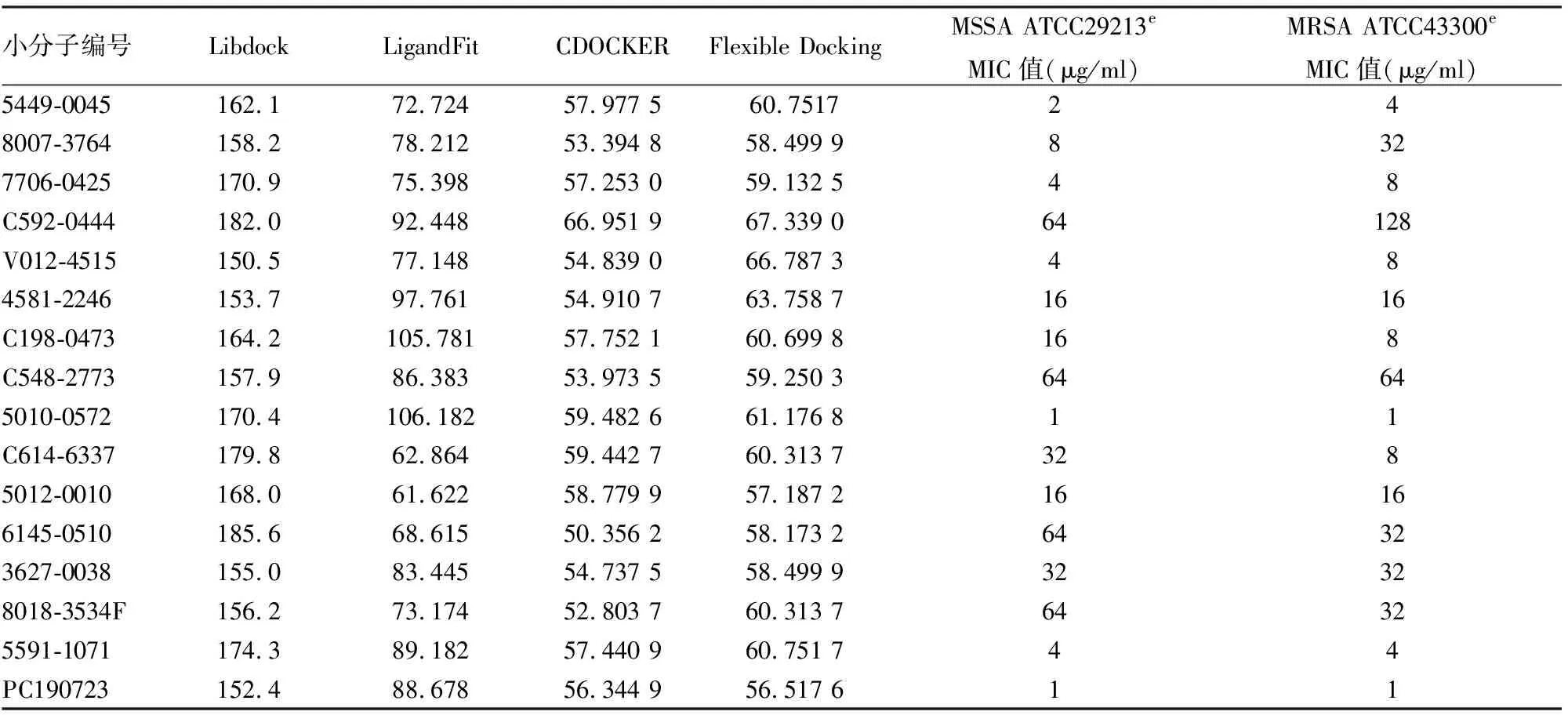
表1 Discovery Studio 2017软件4个模块筛选结果分值及体外抗S.aureus活性(MIC)评价

图1 对接软件一致性评价
A:Libdock模块对接;B:Ligandfit模块对接;C:CDOCKER模块对接;D:Flexble Docking模块对接;黄色结构为PC190723在4DXD晶体复合物构象,绿色为PC190723对接后的构象
2.3 化合物抗菌活性评价结果如表1 所示,所筛选出的化合物对MSSA和MRSA均表现出一定的抗菌活性,MIC值范围分别为1~64 μg/ml和1~128 μg/ml。其中,化合物5010-0572表现出最强的活性,MIC值均为1 μg/ml,与FtsZ抑制剂PC190723相当;化合物5591-1071和5449-0045也表现出良好的抗MSSA和MRSA的作用,MIC值分别为1~4μg/ml之间。C592-0444抗MSSA和MRSA增殖的作用最差,MIC值分别为64 μg/ml 和128 μg/ml。
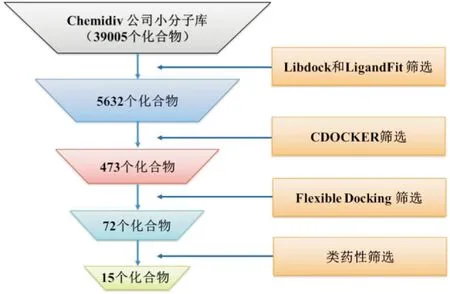
图2 虚拟筛选流程及结果
2.4 配体-受体复合物体系稳定性评价基于抗菌活性评价结果,采用分子动力学模拟评估了化合物5010-0572、5591-1071、5449-0045、C592-0444和PC190723在与FtsZ蛋白对接形成的配体-受体复合物体系稳定性。结果如图4所示,晶体复合物中的原配体PC190723在2.5 ns的时候与受体的结合便得到平衡。筛选出的4个化合物中,化合物5010-0572的RMSD变化幅度和PC190723相比最为接近,化合物5591-1071、5449-0045 RMSD依次变大,化合物C592-0444直到3.5 ns后才达到平衡,且波动幅度很大,意味着其形成的复合物体系的稳定性差。
3 讨论
FtsZ作为细菌分裂起始蛋白广泛存在于多种细菌中,其通过招募多种细菌分裂蛋白参与细菌分裂的过程,同时能够催化GTP生成GDP,为细菌分裂提供能量,已广泛被认定是一个具有研发前景的抗菌作用靶点[4-8],尤其对于抗耐药菌药物的研发[12]。FtsZ具有高度的保守性和特异性,具备小分子药物靶向识别的条件。
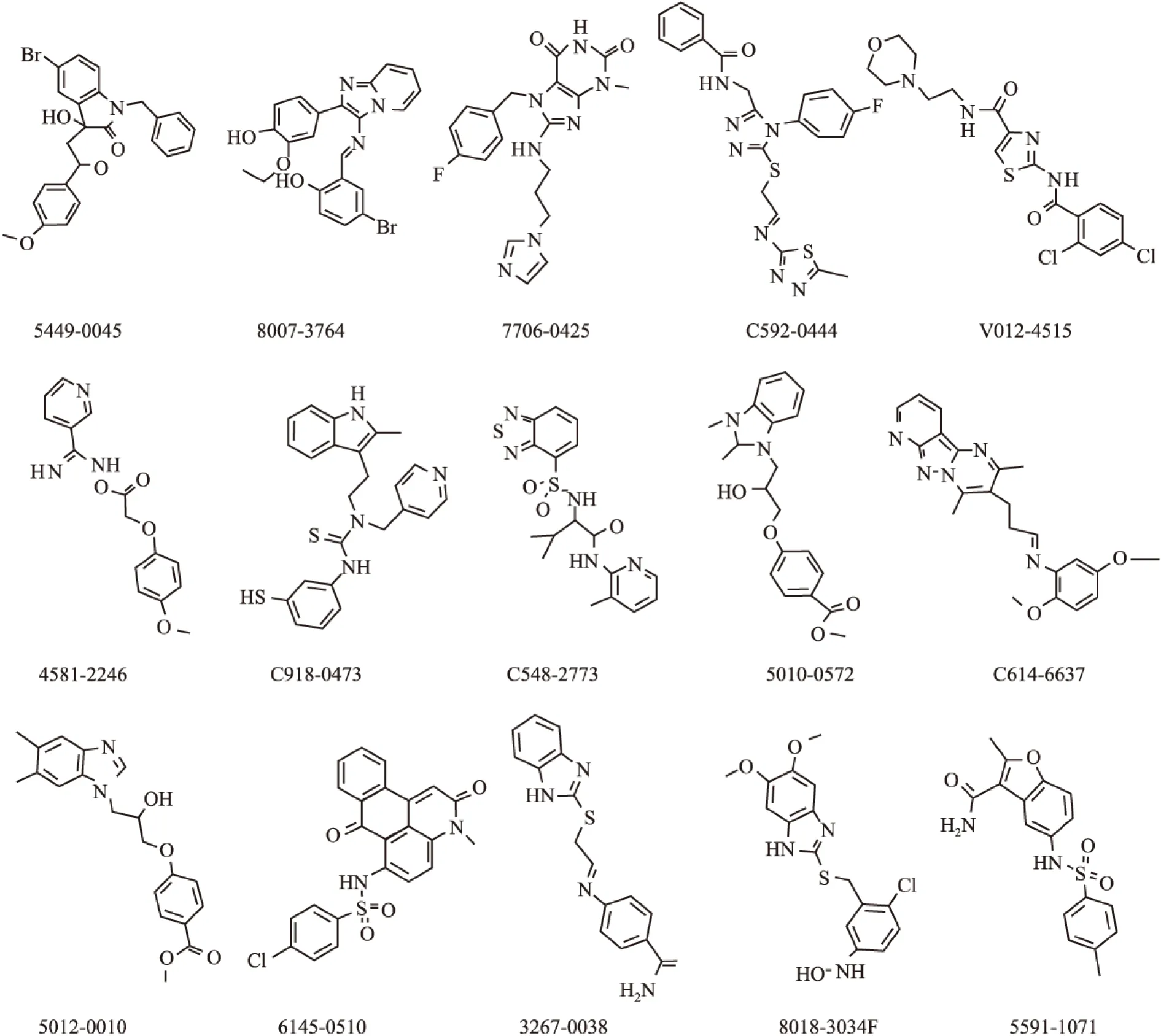
图3 虚拟筛选获得的小分子配体的结构
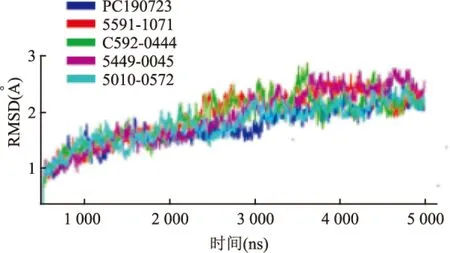
图4 配体-受体复合物体系蛋白主链碳原子的RMSD
在本研究中,利用计算机辅助药物设计Discovery Studio 2017软件技术开展了基于S.aureusFtsZ蛋白为靶标的药物筛选。在筛选过程中,首先开展了对接程序适用性的评价。对接结果的准确性关系到筛选结果的可信度,这也是评价CADD软件优劣的重要标准。结果显示,4种对接模块Libdock、Ligandfit、CDOCKER和Flexble Docking使用原配体对接后,均能够很好重现原配体与受体的相互作用模式,RMAD值在1 Å以下,说明软件的对接结果是十分可信的。随后使用4种对接模块设计了由粗犷到精细渐进式的筛选方案。其中Libdock根据热区匹配的原理将小分子构象刚性对接到活性位点口袋中,Ligandfit则是基于配体和受体的形状匹配,采用Monte Carlo method来检索分子的柔性空间构象,两种方法适用于快速的虚拟筛选;CDOCKER基于CHARMm力场,采用模拟退火的方法将各个构象在受体活性位点进行优化,从而找到全局能量最低构象,CDOCKER对接结果更加准确,但速度较慢,耗时约是Libdock的5倍;Flexible Docking 模拟生理条件受体和配体的状态,以柔性的方式进行对接,精准的研究受体-配体相互作用过程,适用于进一步更精确的筛选[14]。对小分子库中39 005个小分子经上述4个模块依次筛选后得到了72个化合物,进一步通过类药性评价最终获得15个化合物。抗菌活性评价显示,这些化合物对MSSA和MRSA均有一定的抗菌作用,其中化合物5010-0572的活性最强(MIC=1 μg/ml),与已报道FtsZ抑制剂PC190723相当[11]。
分子动力学模拟能够模拟受体-配体相互作用随时间的变化,判断受体-配体结合的稳定性,准确性高,可以用来解释复合物相互作用机理。对经虚拟筛选和抗菌活性筛选后的部分化合物研究发现,抗菌能力强的化合物5010-0572与FtsZ结合过程与蛋白晶体中的原配体PC190723最为接近,而抗菌活性最差的化合物C592-0444的结合过程与原配体相比,偏差最大,这一结果有力的印证了化合物抗菌活性评价的结果,也是对虚拟筛选结果的有力补充。
综上,本研究利用CADD技术,通过多重筛选策略筛选得到了15个靶向FtsZ蛋白的抗菌小分子化合物,化合物5010-0572表现出最强的抗菌活性,具有成为抗菌药物先导化合物的条件。
