饲料中粗脂肪和粗纤维含量的近红外光谱快速分析
2020-01-08吴文辉商庆园
郝 勇,吴文辉,商庆园
华东交通大学机电与车辆工程学院,江西 南昌 330013
引 言
饲料营养的精准配制不仅可以提高饲料的质量,还可以节约成本。粗脂肪和粗纤维是饲料主要成分,其含量检测是饲料精准配制的重要环节,传统的饲料成分方法为湿化学检测方法其灵敏度和精确度高,但操作复杂、检测时间长、成本高。近红外技术作为一种快速无污染的检测方法广泛应用于农业、林业、化工、制药等行业。如Sagrario Modrono等运用便携式光谱仪实现了饲料中蛋白质、粗纤维和淀粉等组分的实时测定[1]; Patrica A.Harris等运用近红外光谱分析方法准确分析了马蹄草的营养成分[2]; Lusia Mandrile等运用近红外光谱进行饲料中虫类蛋白含量的检测等[3]。
本文利用近红外光谱实现对饲料的粗脂肪和粗纤维的含量检测,在利用近红外光谱建立含量分析回归模型前,采用光谱预处理方法和变量选择方法来简化模型,以提高模型分析精度。
1 实验部分
1.1 样本
收集了不同厂家不同时期生产的饲料样本325个,包括乳猪饲料、中猪饲料和大猪饲料。在采集近红外光谱前先将样本利用高速万能粉碎机(FW135)粉碎,过40目筛,制成粉末状样本。饲料的粗脂肪和粗纤维的实际含量按照国家标准GB/T 6433—2006和GB/T 6434—2006确定。
1.2 近红外光谱的采集
使用瑞士步琦公司NIR-Flex N-500傅里叶近红外光谱仪,其波数范围为10 000~4 000 cm-1。为了保证光谱仪的正常运行和所获光谱的一致性,饲料样本的厚度统一为4 mm,实验室温度为10~30 ℃,湿度为30%RH~70%RH。本文共获取352条饲料光谱,每条光谱由1 501个变量组成。
1.3 近红外光谱预处理方法
建立饲料粗脂肪和粗纤维定量识别模型时,352个样本光谱根据x-y联合距离划分样本集算法(sample set partitioning based on joint x-y distance,SPXY)按照2∶1比例划分为校正集和预测集,校正集有235个样本,预测集含有117个样本[4]。
为消除背景噪声的影响需要对原始光谱进行预处理。常用的光谱预处理方法主要分为两大类: 光谱导数法和散射校正法,其中Norris-Williams导数法(Norris-Williams derivation,NW)和多元散射校正法(multiplicative scatter correction,MSC)有较好的光谱预处理效果[5-6]。
1.4 近红外光谱变量选择方法
每条饲料的近红外光谱有1 501个变量,其包含了饲料的全部信息。变量选择方法通过选择有用变量去除冗余信息来简化模型,提高模型精度。变量选择方法有很多,本文采用蒙特卡罗无信息变量消除法[7-9](Monte carlo based uninformative variable elimination,MCUVE)、变量组合集群分析法[10-12](variables combination population analysis,VCPA)和区间变量迭代空间收缩法[13-14](interval variable iterative space shrinkage approach,iVISSA)进行变量选择并比较结果,从而确定最优的变量选择方法。
1.5 模型的评价
光谱经过预处理和变量选择后,利用校正集样品建立饲料粗脂肪和粗纤维含量模型,再利用预测集样品来对模型进行验证,确定模型能否达到粗脂肪和粗纤维的含量分析要求。采用校正集相关系数Rc、预测集相关系数Rp、交叉验证均方根误差RMSECV、预测集均方根误差RMSEP等指标来评价模型的预测精度和稳健性[15-17]。
2 结果与讨论
2.1 光谱预处理方法的结果分析
饲料中粗脂肪和粗纤维的实际含量统计信息如表1所示。
表1 饲料中粗脂肪和粗纤维含量统计表(单位: %)
Table1Statisticaltableofcrudefatandcorasefibercontentinfeed(unitofcontent: %)

类别最大值最小值均值标准差粗脂肪4.182.623.460.42粗纤维4.661.913.190.68
采用NW导数和MSC对光谱进行预处理。其中NW导数包括平滑法(NW0th)、一阶导数法(NW1st)和二阶导数法(NW2nd)三种方法,同时还利用NW1st-MSC方法来进行预处理。如表2所示为不同预处理方法的饲料粗脂肪和粗纤维含量模型结果。
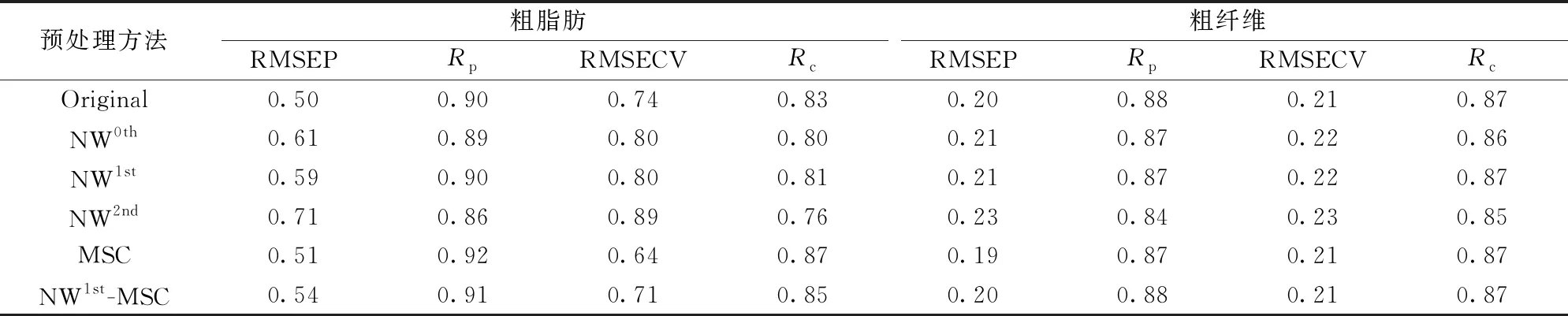
表2 不同预处理方法的饲料粗脂肪和粗纤维含量模型结果
通过表2可以看出,相比与原始光谱的PLS模型,经NW0th,NW1st和NW2nd三种方法处理后的模型都没起到明显的效果。而MSC与NW1st-MSC方法对饲料噪声去除有明显效果,对粗脂肪模型精度有明显的提升。在粗脂肪模型中,相比于NW1st-MSC方法,单独使用MSC预处理方法能够更好的实现光谱的信息提取,校正集的RMSECV由0.74下降到0.64,Rc由0.84提高到0.87。在粗纤维模型中,MSC的RMSEP相比于未经处理的有着0.01的提升,而RMSECV与Rc基本相同; NW1st-MSC处理后的效果与原始光谱模型一致。因此,针对饲料的粗脂肪和粗纤维含量分析的光谱预处理采用MSC方法。图1为饲料的原始近红外光谱和经过MSC预处理后的光谱。MSC修正了光谱间的相对基线平移和偏移,使光谱更为紧密。
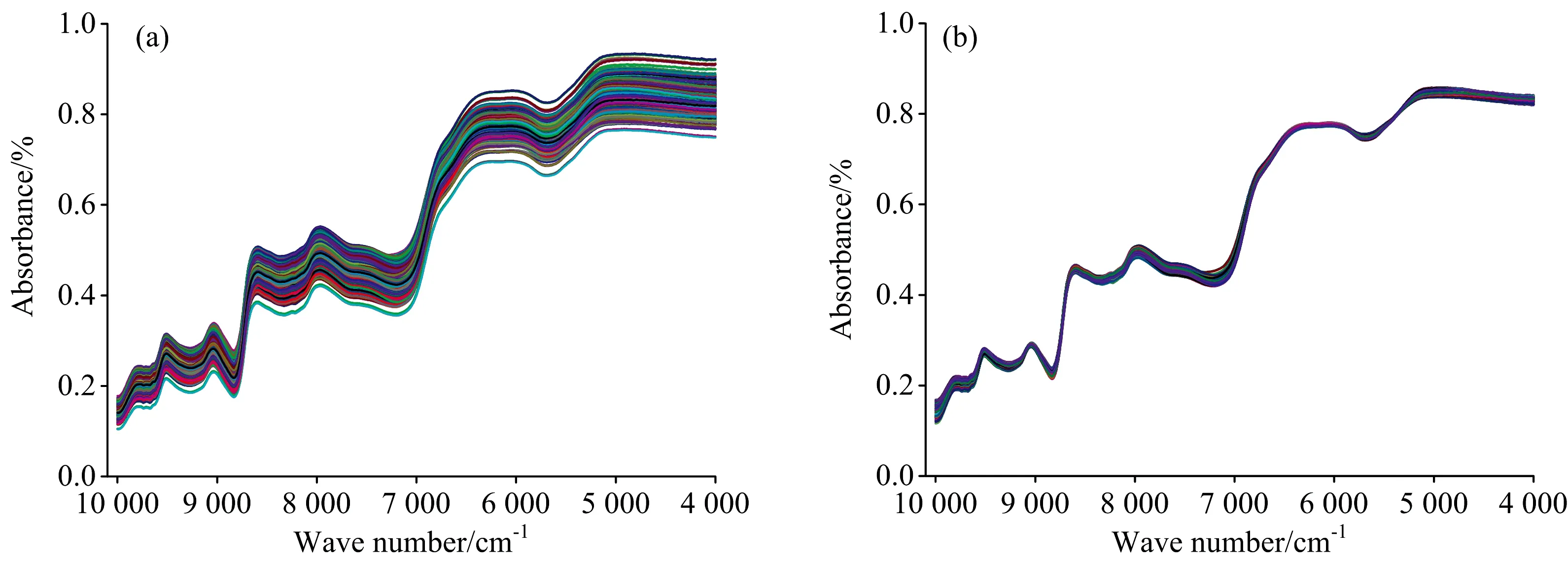
图1 饲料的近红外光谱与MSC预处理后的光谱
2.2 饲料粗脂肪模型分析
饲料光谱经过MSC预处理后,分别利用MCUVE,VCPA和iVISSA三种变量选择方法结合PLS建立饲料粗脂肪含量分析模型。图2为不同变量选择方法的脂肪含量分析PLS模型校正和测试相关图。
图2(a)为利用经过MSC预处理的光谱建立的PLS模型,其校正集和测试集的回归直线基本重合,相关系数Rc和Rp分别为0.87和0.88。模型的RMSECV和RMSEP分别为0.21和0.20。图2(b)为MSC-MCUVE-PLS模型,图中可以看出校正集与测试集相比与MSC-PLS模型分布更为紧密,Rc为0.92,RMSECV为0.17。相比于全谱的模型其校正模型更为稳定,精度更高。同时,Rp相较于MSC-PLS模型提高了0.01,且RMSEP降低了0.01,变量数Vn为400,大幅度降低。图2(c)为MSC-VCPA-PLS模型,在校正模型中Rc为0.87,RMSECV为0.21与全谱的PLS模型相同,而Rp为0.81,RMSEP为0.25,与MSC-PLS模型相比起预测结果相差较多。Vn为12个,小于全谱变量数和MCUVE筛选后的变量数。图2(d)为MSC-iVISSA-PLS模型,在此模型中Rc为0.86,RMSECV为0.21与全谱的校正集模型相同,同时预测集的Rp为0.87,RMSEP为0.20且与全谱的预测值相同,Vn为20与VCPA选择的变量数近似。
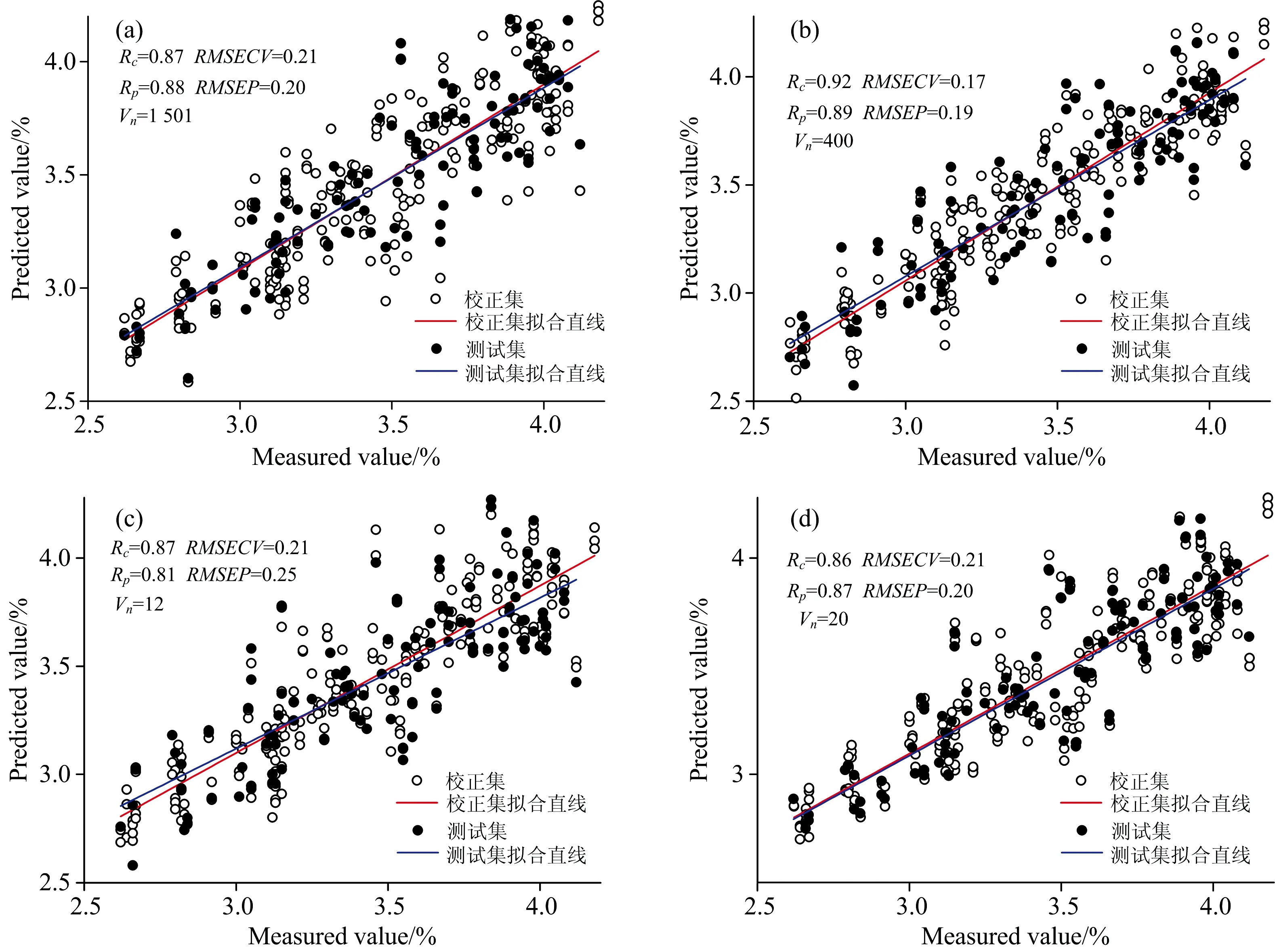
图2 不同变量选择方法的饲料粗脂肪含量分析PLS模型校正和测试相关图
通过图2可以看出三种变量选择方法中,经MCUVE方法变量选择后,其模型的各个参数都优于全谱模型。经VCPA和iVISSA方法选择后都以极少的变量建立检测模型且与全谱模型几乎相同。若精度要求不高时,可以选择VCPA和iVISSA方法进行变量选择,建立更简洁的模型; 若要提升精度,MCUVE则是最好的选择。
2.3 饲料粗纤维模型分析
如图3为经不同变量选择方法的粗纤维定量分析模型相关图。图3(a)为经MSC预处理后由全谱1501个变量建立的PLS模型,其校正集相关系数Rc为0.91,RMSECV为0.28,在测试集中Rp为0.95,RMSEP为0.25。图3(b)为经过MCUVE变量选择后的模型,选择后剩余740个变量用于模型分析,其Rc和Rp分别为0.95和0.94,较全谱模型的校正集Rc提高了0.04。同时RMSECV为0.23,较全谱模型降低了0.05,RMSEP降低了0.02,Vn为740,由此可知MCUVE变量选择法有效地选择了有用变量,提高了模型的精度。
图3(c)为经过VCPA变量选择后建立的PLS模型,由图可知,经过VCPA选择了11个变量来建立模型,相比与全谱和MCUVE方法,VCPA用极少的变量实现了几乎相同的结果,其校正集Rc为0.91,RMSECV为0.27略好于全谱校正模型,预测集Rp和RMSEP都差于全谱。图3(d)为iVISSA变量选择后的饲料粗纤维检测模型,模型的建立基于20个变量,其Rc为0.90,RMSECV为0.29,Rp为0.93,RMSEP为0.27相比与全谱和MCUVE模型较差,但较于VCPA模型更好。
对比三种变量选择方法,可以看出MCUVE变量选择方法校正集模型参数优于全谱模型,而预测集的参数与全谱预测集参数几乎一致,因其变量数少于全谱,则模型更为简洁。利用VCPA和iVISSA进行变量选择后,都剩余极少变量,部分有用信息变量被剔除,因而其模型参数较全谱都稍差一些,其优点是模型简单。
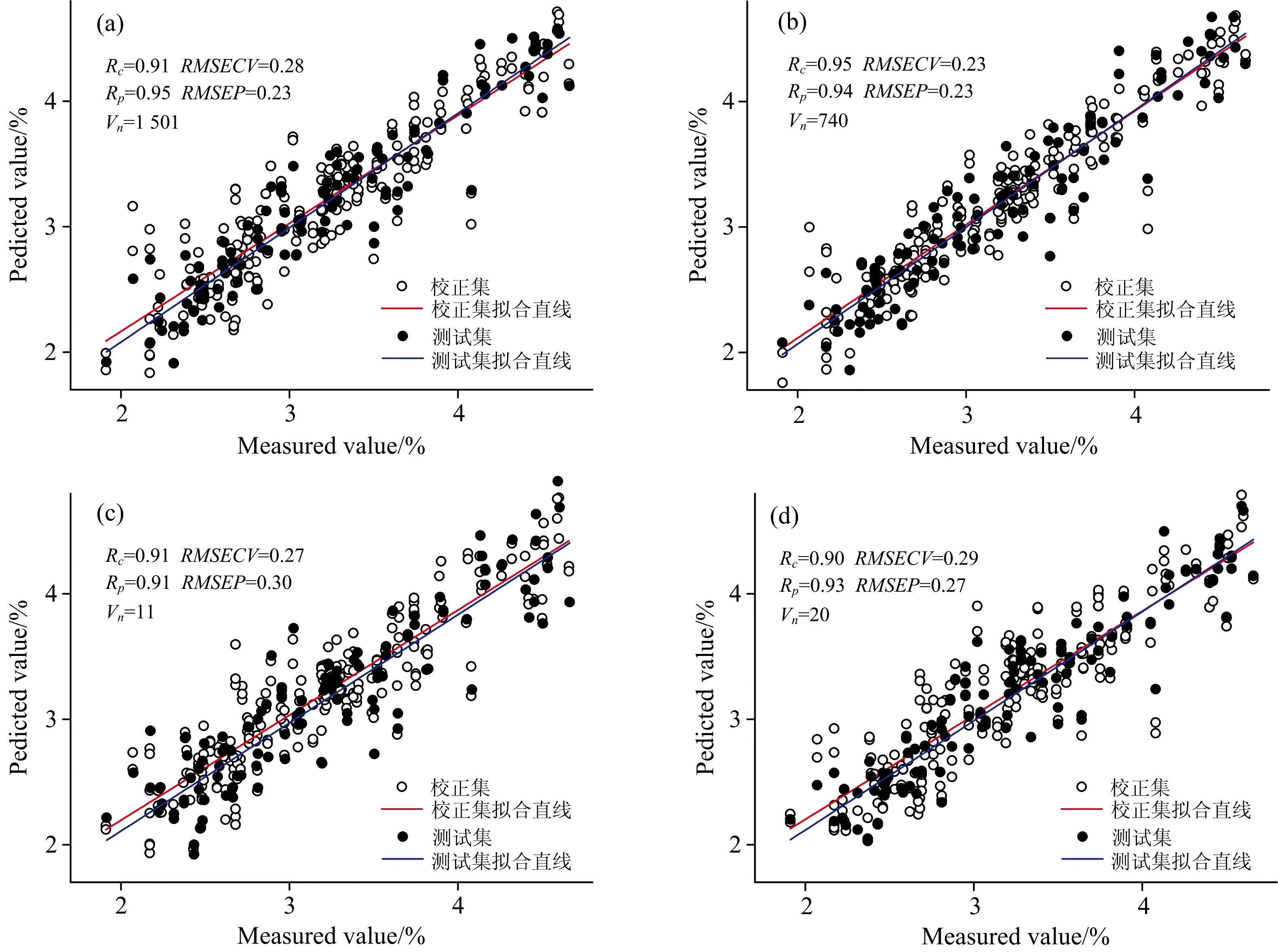
图3 不同变量选择方法的饲料粗纤维含量分析PLS模型校正和测试相关图
3 结 论
近红外光谱分析方法用于饲料中粗脂肪和粗纤维的定量分析,采用不同的光谱预处理方法和光谱变量选择方法对光谱模型进行优化。结果表明,在饲料粗脂肪定量分析中,MSC预处理方法结合MCUVE变量选择方法可以建立最优粗脂肪分析模型,模型变量数由1 501降低到400,RMSEP和RMSECV分别降低为0.19和0.17,Rp和Rc提高到0.89和0.92; 在饲料粗纤维定量分析中,利用MSC预处理方法和MCUVE变量选择方法可以实现最优的粗纤维定量分析模型,模型变量数为740,模型的RMSEP和RMSECV分别为0.23和0.23,Rp和Rc分别为0.94和0.95。因此,基于近红外光谱建立的MSC-MCUVE-PLS模型可以有效的进行饲料的粗脂肪和粗纤维的定量分析,其精度高,稳定性好且更简洁。
