斑节对虾carcininPm3的表达及其无标签多肽的获得
2020-01-06李国强李安国王潮岗
李国强,周 亮,李安国,王潮岗
深圳大学生命与海洋科学学院,深圳市海洋生物资源与生态环境重点实验室,广东省海洋藻类开发与应用工程重点实验室,广东深圳 518060
抗菌肽是一种由生物防御系统产生的对抗外源性病原体的小分子肽类物质,是体液免疫中的一种重要因子,具有广谱的抗菌活性[1],被认为是一种天然抗生素.与传统抗生素相比,抗菌肽的长期使用不会产生耐药性[2],可激活生物体内的免疫系统,参与细胞信号的传导,具有抗肿瘤、抗病毒和抗寄生虫[3]等功能,是最有希望成为替代抗生素的新型抗菌药物.据报导,目前已发现超过2 960种抗菌肽,其中,2 184种来自动物[4],而在对虾中的抗菌肽主要有:对虾素(penaeidins)、甲壳肽(crustins)和抗脂多糖因子(anti-lipopolysaccharide factors, ALFs)等[5-7].对虾素家族有富含脯氨酸的N端,且在C端含有6个半胱氨酸残基,已从凡纳滨对虾(Litopenaeusvannanmei)与白滨对虾(Litopenaeussetiferus)等多种对虾中分离出来.ALFs在中国鲎(Tachypleustridentatus)血细胞中首次被分离出来,这是一种含两个半胱氨酸形成二硫键的碱性蛋白,其氨基末端为焦谷氨酸,含有一个脂多糖结构域(lipoplysacide binding domain, LBD)[8],对革兰氏阳性菌和革兰氏阴性菌均有抑菌活性.crustin是无脊椎动物中最大抗菌肽家族之一,富含半胱氨酸.甲壳类中共有4种蛋白类型[9].I型crustin主要存在于蟹和龙虾等生物,由一个信号肽、富含半胱氨酸的结构域和C末端的单个乳清酸性蛋白(whey acidic protein, WAP)结构域构成.II型crustin主要存在于对虾中,除富含半胱氨酸外,在N末端含有一个富含甘氨酸的区域.III型crustin也称为含有单一WAP结构域蛋白质,在WAP结构域前有富含脯氨酸和精氨酸的区域[10-11].IV型crustin是含有2个WAP结构域的蛋白[11].以上4种类型的crustin在斑节对虾(Penaeusmonodon)均有发现:属于I型crustin的蛋白有2种;属于II型crustin的蛋白至少有10多种[12],其中,crustinPm1和crustinPm5仅对革兰氏阳性菌表现出抗菌活性,而crustinPm7革兰氏阳性菌和革兰氏阴性菌具有活性[13-15];属于III型crustin的蛋白有3种,对革兰氏阳性菌有抑制功能,但对革兰氏阴性菌无效,且是枯草杆菌蛋白酶A的竞争性抑制剂[16];属于IV型crustin的蛋白有1种,但未表现出明显的抑菌活性[17].
抗菌肽是无脊椎动物免疫系统的重要成分,但其在生物体内的含量比较低.可利用基因工程技术在大肠杆菌(Escherichiacoli,E.coli)中进行大量表达,研究其抗菌功能.由于抗菌肽对细菌和真菌等具有抑杀作用,很多抗菌肽的异源表达并不成功,即使获得了表达,也易形成包涵体.此外,原核表达系统为了便于纯化,往往带有标签,这会影响蛋白的正确折叠和抗体的制备等.因此,如何使抗菌肽实现高表达、高活性且不带外源标签,是目前抗菌肽表达及功能研究的难点.本研究通过比对转录组测序及抗菌肽数据库,获得carcininPm3的基因序列,与类泛素蛋白修饰分子(small ubiquitin-like modifier, SUMO)在大肠杆菌中融合表达,获得无外源标签的carcininPm3抗菌肽,为后续的功能分析及应用提供基础.
1 材料与方法
1.1 材 料
1.1.1 质粒和菌株
E.coliBL21(DE3)购自上海唯地生物技术有限公司;pSmartI-SUMO载体购于通用生物系统(安徽)有限公司.
1.1.2 主要试剂及酶
主要试剂和酶有:Amp、Kana、异丙基-β-D-硫代半乳糖苷(isopropyl β-D-thiogalactopyranoside,IPTG)、考马斯亮蓝、Tris、NaCl和咪唑(imidazole).质粒提取试剂盒购自Takara(北京)公司;Direct-loadTMColor Prestain Protein Marker(10~180 kDa, 1 Da =1 u)购自广州市康润生物科技有限公司;5× 蛋白上样缓冲液、Broadford试剂盒购自生工生物工程(上海)股份有限公司.
1.2 carcininPm3基因的合成和载体的构建
将carcininPm3的成熟肽与具有His标签的SUMO蛋白C末端融合,并在E.coliBL21(DE3)中进行原核表达.carcininPm3的核苷酸序列在两端具有SacI和XhoI酶切位点,可根据大肠杆菌密码子偏好性优化其密码子,然后交由通用生物系统(安徽)有限公司通过化学合成方法合成基因,最后通过SacI和XhoI酶切位点将合成的DNA与pSmartI-SUMO载体连接,得到重组质粒,命名为pSmartI-SUMO-carcininPm3.
1.3 重组蛋白的诱导表达及可溶性分析
利用质粒提取试剂盒提取重组表达载体pSmartI-SUMO-carcininPm3,采用热激法转化至E.coliBL21(DE3)中,获得单克隆菌落.按照以下的步骤进行诱导表达:① 挑取阳性单克隆菌落接种于含卡那霉素的LB培养基中,于37 ℃以200 r/min振荡菌液,培养过夜;② 次日将过夜培养的菌液按体积比1∶100加到新鲜的LB培养基中,于37 ℃以200 r/min振荡菌液,培养菌液至光密度D(600)≈0.6,加入IPTG使其终浓度为1.0 mmol/L,在加入IPTG诱导前取1 mL菌液作为对照;③ 在37 ℃以200 r/min振荡菌液,继续诱导6 h;④ 诱导完毕后,取1 mL诱导后的菌液,剩余的菌液以8 000 r/min离心10 min收集菌体,加入平衡缓冲液(50 mmol/L Tris,200 mmol/L NaCl,pH=8.0)重悬菌体,超声破碎菌体40 min(功率为195 W,超声2 s,停顿4 s),于4 ℃以10 000 r/min离心30 min后分离出上清与沉淀;⑤ 取诱导前菌液、诱导6 h的菌液、破碎菌液后的上清和破碎菌液后的沉淀,分别加入5×蛋白上样缓冲液,100 ℃煮沸10 min,十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis, SDS-PAGE)电泳检测蛋白表达及其可溶性的情况.
1.4 原核表达条件的优化
进行原核表达时,影响蛋白表达量的条件主要是诱导时间和IPTG浓度,为获得大量蛋白,需对蛋白表达的诱导时间和IPTG浓度进行优化.时间梯度设为0、1.5、3.0、4.5、6.0和7.5 h;IPTG终浓度的梯度为0.2、0.4、0.6、0.8和1.0 mmol/L.按照1.3节方法进行诱导,通过SDS-PAGE检测蛋白表达情况,最终确定表达的最佳条件.
1.5 carcininPm3蛋白的分离纯化
按照1.3节方法收集菌体,取100 mL培养物的菌体沉淀,加入4 mL 平衡缓冲液(50 mmol/L Tris、200 mmol/L NaCl,pH=8.0)重悬菌体,超声破碎后收集上清,并用孔径为0.45 μm的滤膜过滤.上清液中的His-SUMO-carcininPm3融合蛋白通过镍柱亲和层析进行分离纯化.先用平衡缓冲液(50 mmol/L Tris, 200 mmol/L NaCl,pH=8.0)平衡柱子,并调0,待吸收峰稳定后上蛋白样品,待流出峰经过,吸收峰下降并稳定后用洗脱液(50 mmol/L Tris,200 mmol/L NaCl,60 mmol/L imidazole, pH=8.0)洗脱未结合蛋白;然后,换收集液 1(50 mmol/L Tris,200 mmol/L NaCl,200 mmol/L imidazole,pH=8.0)洗脱并收集样品;最后,用收集液 2(50 mmol/L Tris,200 mmol/L NaCl,500 mmol/L imidazole,pH=8.0)洗脱并收集样品.收集好的样品经SDS-PAGE电泳检测蛋白纯化结果.
1.6 蛋白浓度的测定
将过柱纯化后获得的融合蛋白溶液装入透析袋,浸泡在平衡缓冲液(50 mmol/L Tris,200 mmol/L NaCl,pH=8.0)中,于4 ℃透析16 h,以去除蛋白溶液中的咪唑.根据Bradford试剂盒说明书制作蛋白标准曲线,利用多功能酶标仪测定融合蛋白光密度D(595),再根据D(595)的值与标准曲线,计算出融合蛋白浓度,最后将蛋白溶液分装并进行真空干燥,获得蛋白粉末存于-80 ℃.
1.7 SUMO融合标签的分离
由于SUMO标签相对分子质量大,可能影响carcininPm3蛋白功能,所以利用SUMO酶(购自通用生物系统(安徽)有限公司)将SUMO融合标签与carcininPm3蛋白切割分离,获得不带标签的目的蛋白carcininPm3.为能完全去除SUMO标签,取相同质量的融合蛋白carcininPm3,分别加入不同量的SUMO酶,于4 ℃酶切16 h,SDS-PAGE检测酶切结果,最终确定SUMO酶与融合蛋白的最佳切割质量比例.
1.8 利用LC-MS鉴定carcininPm3
将SDS-PAGE凝胶电泳得到的carcininPm3蛋白条带进行切胶,并放置于1.5 mL EP离心管中,加入体积分数为50%的乙腈,于37 ℃以200 r/min振荡脱色过夜;用纯乙腈使胶体变白变硬,弃上清,加入胰酶酶解,再加入覆盖液(碳酸氢铵)于37 ℃水浴16 h;经胰酶酶解后转至新的1.5 mL EP离心管,加入萃取液(水和无水乙腈按照体积比1∶4混合后,加入总体积0.5%的甲酸),超声10 min,离心,再将萃取液真空干燥4 h得到蛋白粉末;用上样液(水和无水乙腈按照体积比1∶49混合后,再加入总体积0.5%的甲酸)溶解蛋白粉末,涡旋振荡后13 000g室温下离心15 min,最后利用液相色谱-质谱联用仪(liquid chromatograph mass spectrometer, LC-MS)鉴定carcininPm3蛋白.
2 结果与分析
2.1 获得carcininPm3原核表达载体
carcininPm3成熟肽的氨基酸序列为:APQSSV LLPTPVPSCSRWCHHDVDGYLYCCQEEQGVGAHSGK CPDTPIQPHEQNILDTKGGGALHCERDGDCAKNQKC CYTKFRQQRICRNV,它是一类新型的对虾抗菌肽,仅与南美白对虾(Penaeusvannamei)有较高同源性(88%),与日本囊对虾(Penaeusjaponicus)等物种的同源性都低于45%.carcininPm3蛋白的分子质量为10.2 kDa,等电点(isoelectric point, pI)值为7.09.根据大肠杆菌密码子合成carcininPm3成熟肽的基因序列,最终连接到pSmartI-SUMO载体的SacI和XhoI上,与载体上的SUMO的蛋白C末端融合,获得pSmartI-SUMO-carcininPm3表达载体(图1).
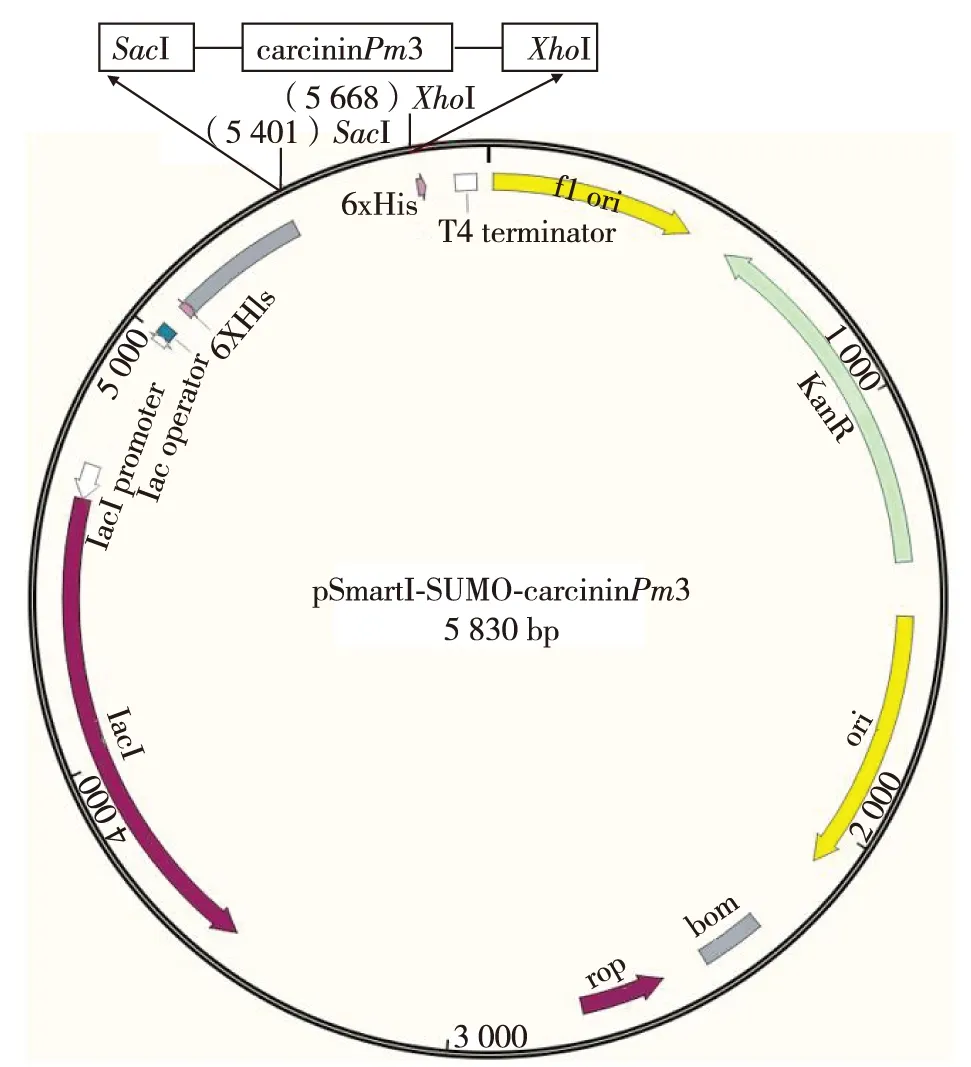
图1 重组质粒的构建
2.2 carcininPm3的表达及可溶性检测
将正确的重组质粒转入E.coliBL21(DE3)进行表达,并对carcininPm3的可溶性进行检测,结果如图2.由图2可见,诱导6 h后融合蛋白carcininPm3得以正常表达,分子质量约为25 kDa,并且存在于上清中,表明融合蛋白carcininPm3是可溶性蛋白.
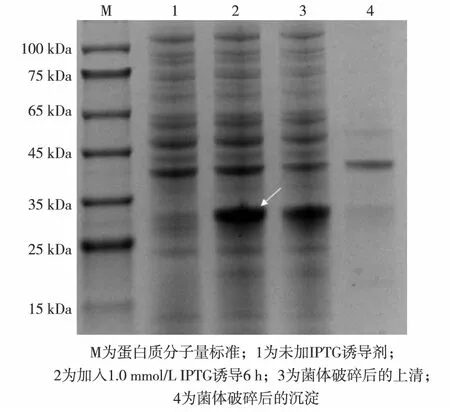
图2 carcininPm3的原核表达及可溶性分析
2.3 获得carcininPm3最优表达条件
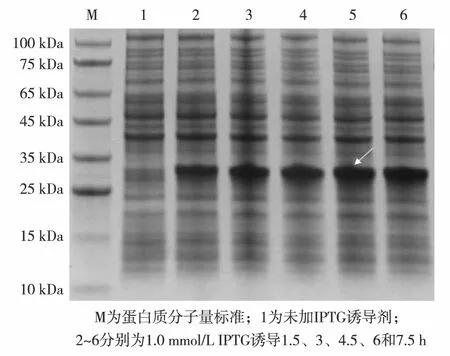
图3 carcininPm3原核表达的时间优化
图3为carcininPm3原核表达及可溶性分析结果.由图3可见,在1 mmol/L的IPTG诱导下,融合蛋白carcininPm3的表达量逐渐增大,6 h及之后的表达量接近.图4的电泳结果显示,在不同终浓度IPTG条件下诱导6 h,0.2、0.8和1.0 mmol/L诱导蛋白的表达量相差不大,又因IPTG具有毒性,会危害表达宿主,所以选择IPTG终浓度为0.2 mmol/L.因此,可确定融合蛋白carcininPm3的在IPTG终浓度为0.2 mmol/L条件下诱导6 h表达量最大.
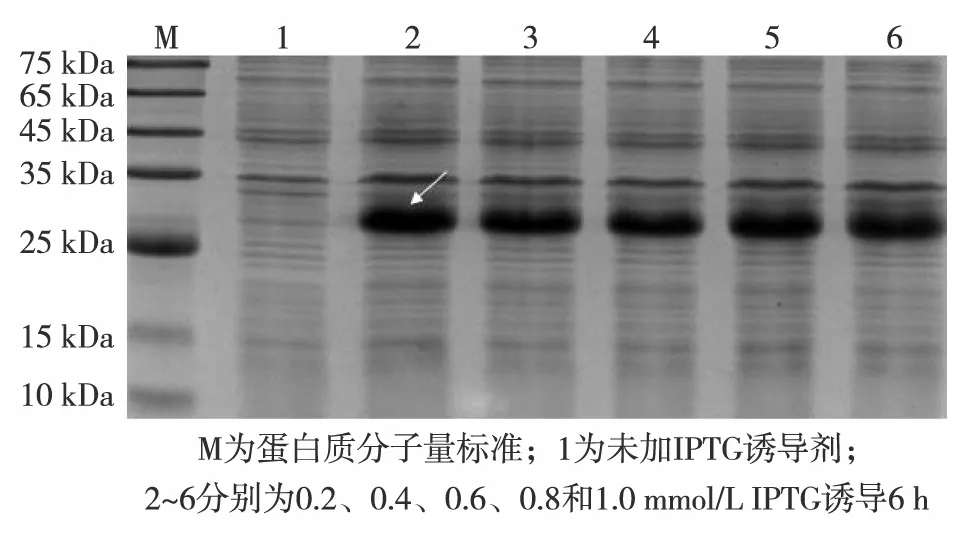
图4 carcininPm3原核表达的IPTG浓度优化
2.4 carcininPm3的分离纯化
将重组菌破碎后的上清先过滤去除杂质,再上亲和层析柱进行纯化,经过杂蛋白洗脱、目的蛋白收集、透析和SDS-PAGE电泳检测等步骤获得目的蛋白.由图5可见,收集到的融合蛋白在25 kDa附近有一清晰条带,说明用200 mmol/L和500 mmol/L咪唑均能收集到目的蛋白.
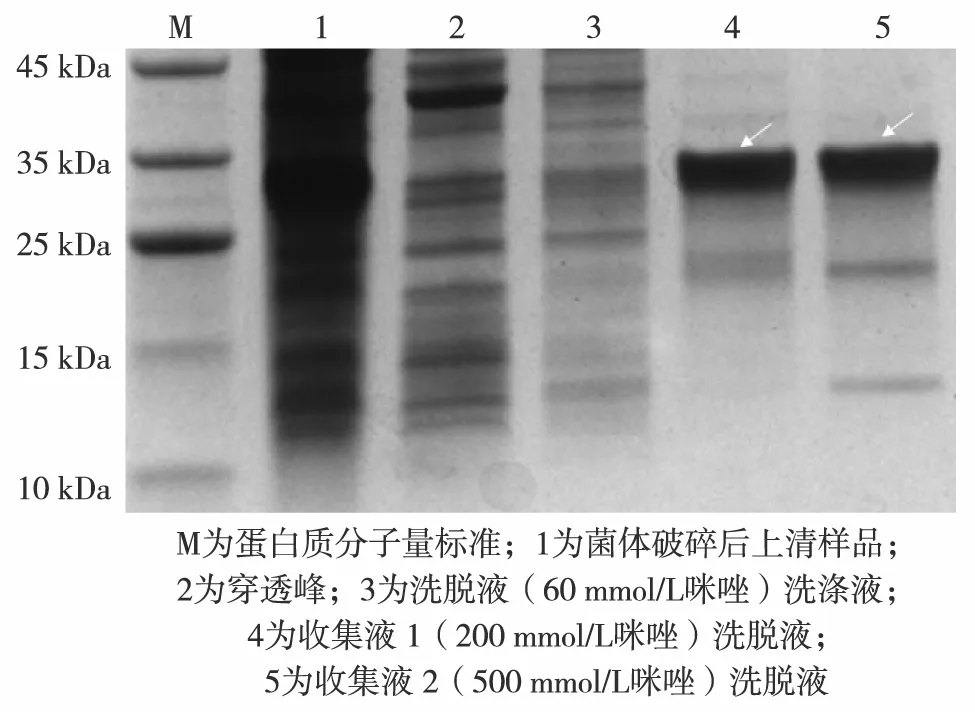
图5 carcininPm3蛋白的分离纯化
2.5 融合蛋白carcininPm3浓度测定
用Bradford法对纯化和透析后的融合蛋白carcininPm3进行质量浓度测定,测得质量浓度为195 μg/mL.随后将蛋白溶液分装成10 mL/管,通过冷冻真空干燥获得蛋白粉末,于-80 ℃存储备用.
2.6 tag-free 抗菌肽的获得
SUMO蛋白酶是一种具有较高活性的半胱氨酸蛋白酶,仅识别SUMO蛋白的3级结构.在双Gly残基C端精确切割,能高效且特异性地将SUMO蛋白从重组蛋白上切割下来,从而获得目的蛋白,图6(a)为SUMO蛋白酶酶切示意图.
取1管融合蛋白carcininPm3冻干粉,加入超纯水,溶解至质量浓度为500 μg/mL.取5个1.5 mL离心管,分别标号1、2、3、4和5,每个EP管都加入50 μg carcininPm3蛋白,然后分别加入0.5、1.0、1.5和2.0 U(1 U=16.67 nmol/s=16.67 nkatal)的SUMO酶,于4 ℃酶切16 h.图6(b)为切除SUMO的蛋白电泳图.结果显示,50 μg带有SUMO标签的融合蛋白只需要0.5 U的SUMO酶就能除去SUMO标签.经过SUMO酶切割,获得去除SUMO蛋白的抗菌肽,该抗菌肽是tag-free多肽,无外源氨基酸,不影响多肽的正确折叠,分子质量为10.2 kDa,符合预期.最终,确定1 U SUMO酶能有效切割100 μg融合蛋白.
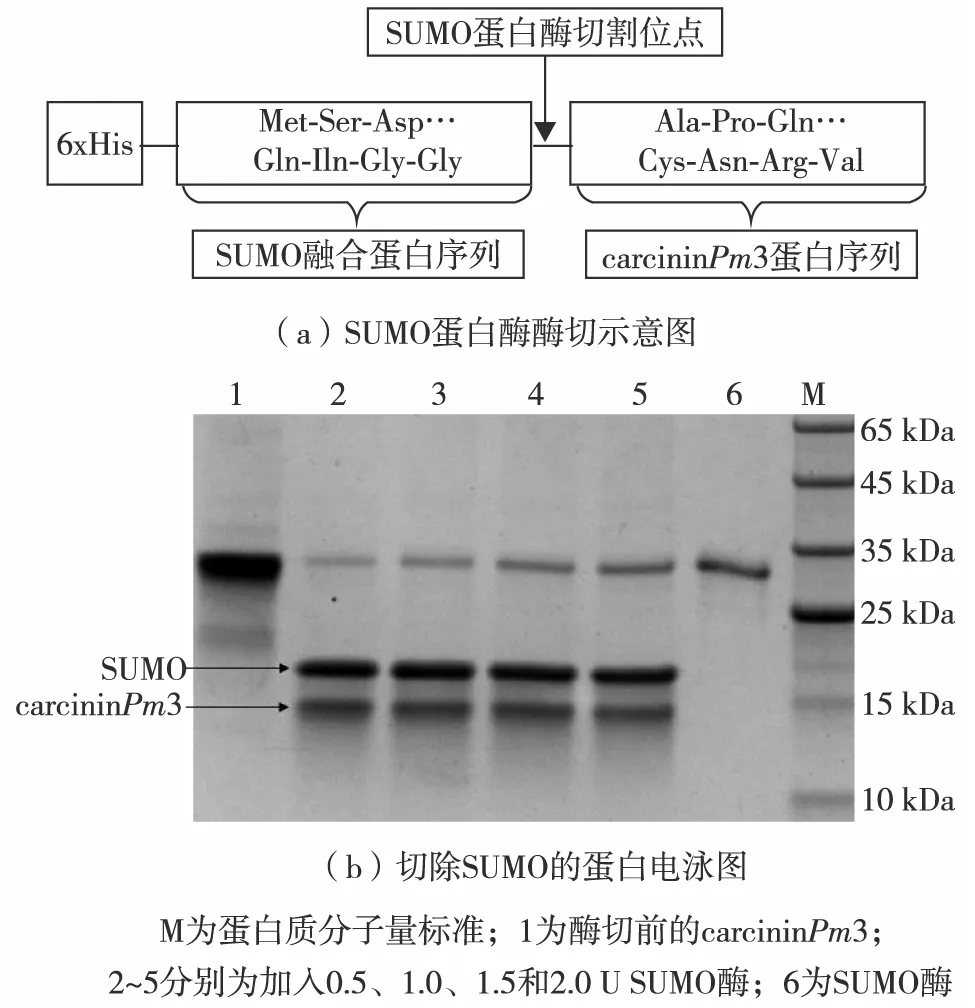
图6 SUMO融合标签的分离
2.7 确认carcininPm3蛋白的表达

图7 carcininPm3蛋白的质谱鉴定
通过LC-MS鉴定目的蛋白,利用Mascot软件与已知carcininPm3 的氨基酸序列进行比对.由图7可见,共检测到两条肽段,氨基酸序列覆盖率达到36%,方框里是质谱鉴定到的氨基酸,检测到的氨基酸序列是目的蛋白N端的17个氨基酸和中间肽段的17个氨基酸,表明原核表达获得了融合蛋白,成功通过SUMO酶切除标签,获得没有外源氨基酸的carcininPm3蛋白.
3 讨 论
如今抗生素滥用日趋严重,已产生了越来越多的耐药性菌株,严重威胁人类健康[18].绿色、环保的抗菌肽是替代抗生素的最佳选择,在养殖业尤其是水产养殖中具有重要的开发潜力.然而,天然的抗菌肽广泛存在于动植物体内,含量少且提取难度大.虽然可通过化学合成抗菌肽,但步骤复杂且成本昂贵.所以,利用基因工程技术获得抗菌肽是最佳选择,它成本低廉、操作简便且易于规模化生产,具有广阔的应用前景[19-20].
抗菌肽最成功的表达系统是大肠杆菌表达系统.但是,表达抗菌肽往往对其本身产生毒性且会将其杀死[21],导致表达失败.大肠杆菌表达量大,且易形成包涵体,需通过复杂的复性过程才能获得有活性蛋白[22].因此,采取与可溶性高的蛋白一起融合表达,更容易获得高活性的蛋白.作为蛋白融合表达的标签有很多,SUMO因其可作为分子伴侣,高重组蛋白的表达量,促进蛋白的正确折叠,且其分子量小,方便切割,且不影响多肽序列和活性,被广泛用于融合蛋白的表达[23].抗菌肽的异源表达往往带有各种标签(表1),能促进可溶表达或方便纯化[24-26],但也会影响抗菌肽的正确折叠,不利于抗体的制备,影响抗菌活性等.抗菌肽一般仅有15~50个氨基酸,几个外源氨基酸的存在也可能影响其功能[27].因此,获得无外源氨基酸的“纯净”抗菌肽对其功能研究意义重大.本研究通过利用SUMO酶切除了带有组氨酸标签的SUMO蛋白,获得了无外源氨基酸的抗菌肽.该技术既能实现目的蛋白的高表达,又能最终获得“纯净”的抗菌肽,可为进一步研究该抗菌肽的功能提供可靠的活性蛋白.

表1 非tag-free抗菌肽表达的分析
抗菌肽富含疏水性氨基酸,原核表达往往容易形成包涵体[28],本研究通过优化表达条件获得了可溶性蛋白.然而,无论是融合表达的抗菌肽还是切除融合标签的抗菌肽,均未能对金黄葡萄球菌和溶藻弧菌等产生显著抑杀效果,仅对副溶血弧菌和嗜水气单胞菌等有一定的抑制作用.抗菌肽往往具有α-螺旋和β-折叠,α-螺旋富于疏水性的C端往往插入细胞膜中,β-折叠则需要二硫键来维系其稳定性.影响抗菌肽抗菌活性的主要因素包括疏水性、两亲性、螺旋度和正电荷数等[29].虽然原核表达系统得到的蛋白量大,但较难得到正确折叠的产物,因此也能影响蛋白活性.下一步将利用本实验室建立的莱茵衣藻表达系统表达抗菌肽,有助于蛋白形成正确的结构,以获得高活性的抗菌肽.
结 语
本研究利用SUMO融合表达技术,在E.coli成功表达了源于斑节对虾的抗菌肽carcininPm3,经过镍柱亲和层析纯化获得大量且纯度较高的重组蛋白,通过SUMO酶酶切和分离后,可获得高纯度的tag-free抗菌肽分子,为carcininPm3的功能研究和应用奠定了基础.
