柯萨奇病毒B5基因组进化存在结构蛋白编码区VP4的重组
2019-12-13答嵘,王伟
答 嵘,王 伟
(西安交通大学第一附属医院:1.检验科;2.神经外科,陕西西安 710061)
柯萨奇病毒B5(CVB5)是小核糖核酸病毒科中肠病毒属的成员,并且已鉴定出4种不同的血清型(A~D)[1]。 CVB5 与心肌炎和脑炎具有密切关系[2-5],因此了解CVB5进化的机制将改善相关疾病的控制。CVB5是单正链RNA病毒,基因组长7.3 kb,包含5′UTR和3′UTR,P1、P2和P3,衣壳蛋白VP1~4由P1区域编码。最初在脊髓灰质炎病毒中发现了重组,并且已经提出并通过体外研究证明了复制(模板转换)和非复制(链断裂和重新连接)机制[6-7]。对已经测序的肠道病毒完整基因组序列的分析表明,重组在非脊髓灰质炎肠道病毒的进化中亦具有重要意义[8-10]。 对CVB5的研究表明,重组可能发生在2C结构域内[11],这与肠道病毒重组热点首选在非编码基因组区域一致。然而,最近的一项研究表明,在埃可病毒30的VP4区域中存在重组基因位点,这是第一次在肠道病毒的蛋白质编码区中发现重组事件[12],为了阐明CVB5中是否存在这种现象,本研究通过分析GenBank数据库中已测序的CBV5基因组序列以研究在蛋白编码基因组区域例如VP4中是否存在类似的重组事件。
1 资料与方法
1.1一般资料 本研究使用的多个已报道的CVB5病毒分离株的5′UTR和VP1核苷酸序列在GenBank中以FASTA格式检索。 每个病毒分离株都标记了登录号、位置和分离年份。 这些序列用于通过最大似然法确定5′UTR和VP1区域的系统发育和通过序列相似性作图(Similarity plot)分析重组事件。
1.2方法
1.2.15′UTR与VP1进化分析 共检索到53个CVB5同时含有5′UTR和VP1核苷酸序列,HE998771序列因为在其5′UTR区域中仅有198 nt用于比对,故未纳入本研究分析。 其他52个序列使用Clustal W进行多序列比对。对于5′UTR和VP1,根据AF114383的序列,所选区域跨越468 nt至742 nt和2 447 nt至3 295 nt。通过使用基于Tamura-Nei模型的最大似然法推断进化[13]。在分支旁显示了相关分类群聚集在一起的树的百分比。通过将Neighbor-Join和BioNJ算法应用于使用最大复合似然(MCL)方法估计的成对距离矩阵,然后选择具有优越对数似然值的拓扑,自动获得用于启发式搜索的初始树。 树按比例绘制,分支长度以每个位点的取代数测量。该分析涉及52个核苷酸序列。所有包含缺口和缺失数据的位置都被消除。进化分析使用MEGA X中构建[14]。
1.2.2基因重组的检测 长度为845 nt的亚基因组序列(根据AF114383.1从257 nt到1 101 nt)覆盖部分5′UTR(257 nt至742 nt),VP4(743 nt至949 nt)和VP2的5′端来自所有可用的32个CVB5序列的(950 nt至1 101 nt)用于检测重组事件。SimPlot软件(3.5版本)用于查询序列和基于5′UTR和VP1簇分组的不同簇中的其他序列之间的相似性图和步进扫描分析,使用200 nt的滑动窗口大小和20 nt的步长[15]。
1.2.3基因重组的分析 为了确认重组事件,序列480 nt(长度为625 nt至1 104 nt,根据AY875692.1)覆盖部分5′UTR(625 nt至745 nt),VP4(746 nt至952 nt)和5′VP2的末端(953nt至1104nt)用于与相关CVB5进行重组分析。 此外,笔者使用Nucleotide BLAST程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)搜索了AY875692.1的5′UTR(625 nt至745 nt)和VP2(953nt至1104 nt)在GenBank中最匹配的核苷酸序列,其中JX976772.1/Shandong/2010和KT285000.1/Hong Kong/1972是与AY875692.1最相似的序列用于进行重组分析。
2 结 果
2.1VP1进化分析 将基于CVB5的VP1区序列构建的系统发育树聚类成3组,其进一步聚类成16个谱系(a至p)。 每个谱系包含1个或多个病毒分离株,并用相同形状和颜色的相同符号标记。第1组仅包含1个谱系作为a;第2组包含8个谱系,包括b至i;其他谱系包括j至p构成第3组。几乎所有来自亚洲地区的病毒分离株(除了病毒分离株KP233830和病毒分离株KT285018)聚集在第2组中,而来自欧洲、美洲和澳大利亚地区的大多数病毒分离株聚集到第3组。大多数具有相似分离时间的病毒分离株聚集成相同的谱系。基于VP1的系统发育树见图1。
2.25′UTR进化分析 用于VP1系统发育分析的相同病毒分离株同时用于5′UTR区域分析,见图2。树被标记为与VP1树中的谱系一致。 该树可分为2个分支,4个病毒分离株包括AY875692、MG845892、KF311743和MG845891,改变了谱系分布。 谱系a和b与谱系j~p聚类,但MG845892与谱系h反向聚类。 此外,病毒分离株AY875692显示出与谱系c分开的簇。其他谱系显示出与VP1树相似的分布。
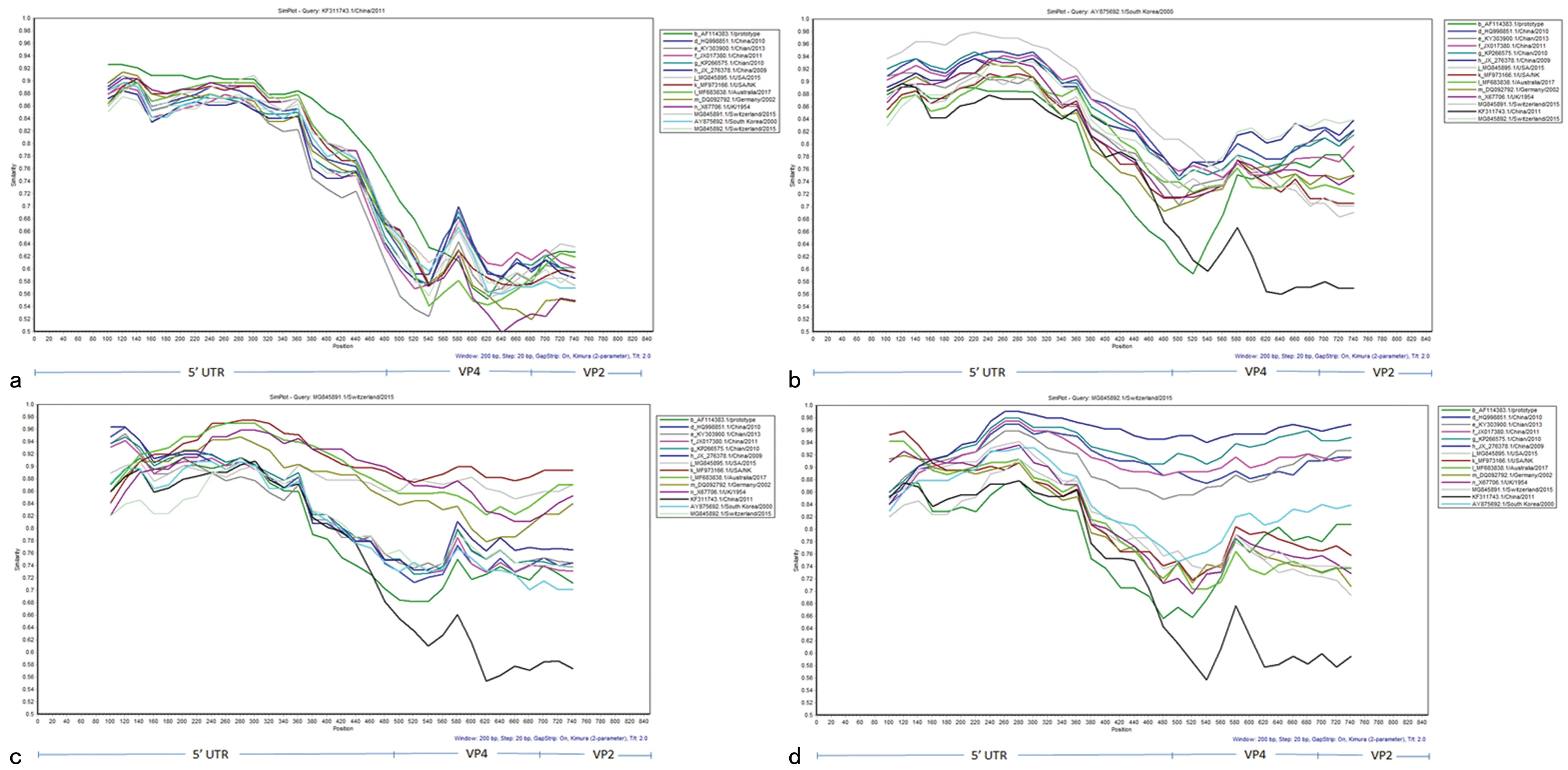
2.3基因重组检测 通过Similarity plot进一步分析了5′UTR和VP1的系统发育树的上述不一致的重组事件。使用亚基因组跨部分5′UTR-VP4-5′VP2来检测重组事件,病毒分离株KF311743.1/China/2011的序列与5′UTR区域的其他谱系高度相似,VP4区域有明显的相似性降低,VP2区域相似性低。AY875692.1 /Korea/ 2000与j_MG845895.1/USA/2015的5′UTR和MG845892.1/Switzerland/2015毒株VP2的5′末端具有更高的相似性(图3b)。MG845891.1/Switzerland/2015和MG845892.1/Switzerland/2015的另一序列的图均显示这些病毒分离株聚集成3组,并且在具有较低相似性的组中5′UTR-VP4连接处的深度下降(图3c和3d)。
2.4基因重组分析 为了阐明病毒分离株AY875692.1 /Korea/ 2000的VP4区域中的重组事件,进行序列分析,基因组区域覆盖部分5′UTR(625 nt至745 nt)、VP4(746 nt至952 nt)和具有相关序列的VP2的5′末端(953 nt至1 104 nt)。病毒分离株序列与5′UTR中的MG845893.1/Switerland/2015和VP2的5′末端的MG845892.1/Switerland/2015高度相似。这3个核苷酸序列之间的比较显示可能的重组基因座是803 nt和804 nt,见图4。 JX976772.1/Shandong/2010和KT285000.1/Hong Kong/1972的两个病毒分离株匹配部分5′UTR和5′end VP2核苷酸序列,相似性分别是93%(E值:4e-40)和92% (E值:3e-52),见图5。这3个核苷酸序列之间的比较显示可能的重组基因座是873 nt和874 nt。可能的2个重组位点均位于VP4区域。
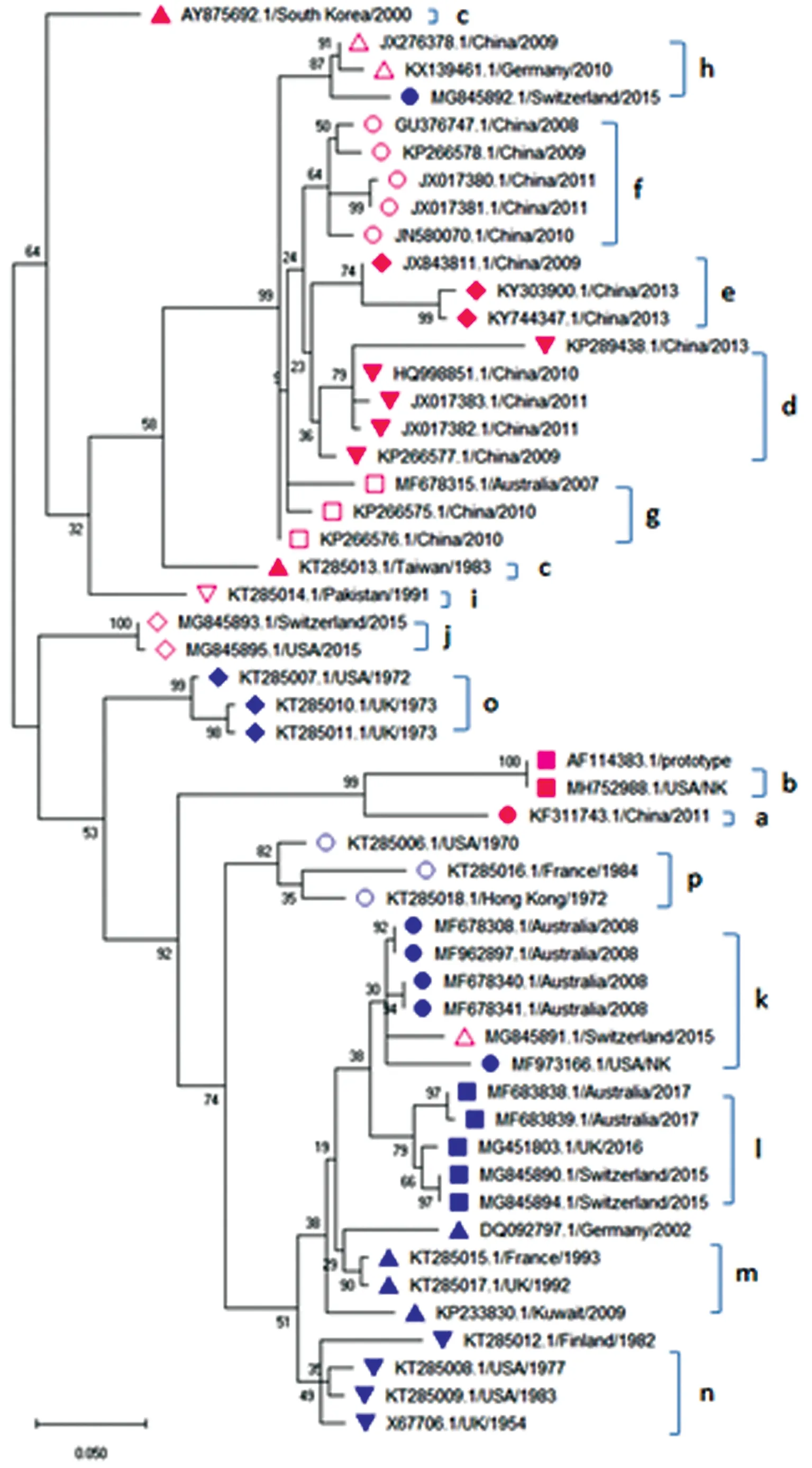
图2 CVB5 5′UTR区的系统发育分析树
注:a为KF311743.1/China/2011;b为AY875692.1 /Korea/ 2000;c为MG845891.1/Switerland/2015;d为MG845892.1/Switerland/2015
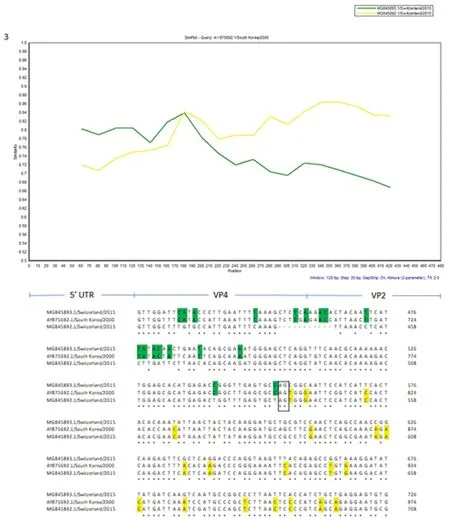
图4 AY875692.1 /Korea/ 2000与其他CVB5的重组分析
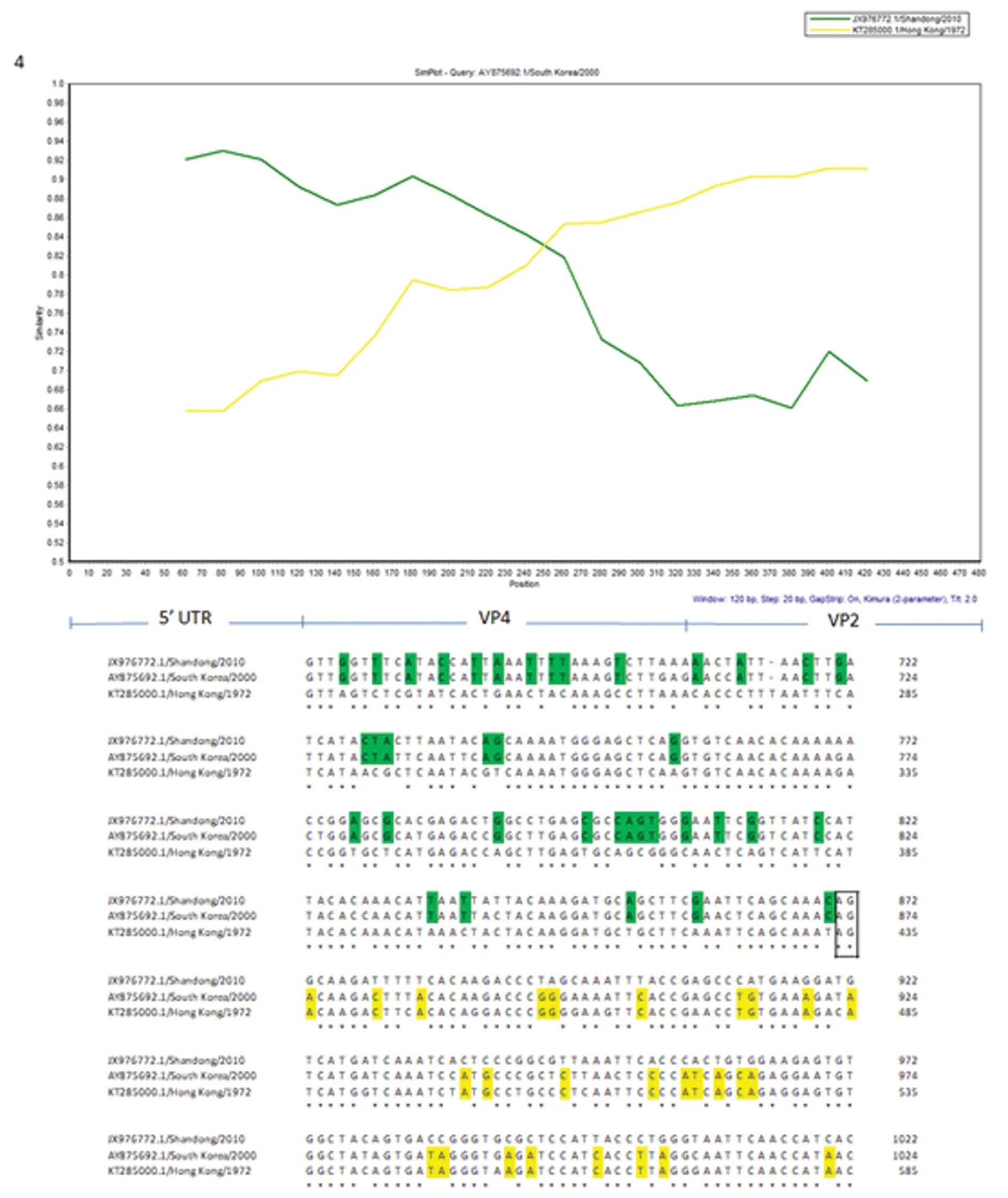
图5 AY875692.1 /Korea/ 2000与其他肠道病毒的重组分析
3 讨 论
肠道病毒通过突变和重组实现自身进化,但由于缺乏校对活性而且每个新合成的基因组中几乎有1个突变,误插入率一直很高[16]。 VP1和5′UTR的系统发育树均显示从同一大陆收集的病毒分离株大多数都有聚集到同一组的趋势,但收集年份比收集的病毒分离株的国家更重要,而那些相似时期的病毒分离株倾向于集群成同一个世系。此前的系统发育分析也表明,在不同年份分离的希腊病毒分离株之间存在25%的差异,这表明肠道病毒类型随时间演变而不是地理分布[3]。本研究中的趋势与上述结果类似,支持分离时间在肠道病毒进化中起关键作用,可能原因是肠道病毒总是作为准种存在[17]。
通过比较系统发育,VP1和5′UTR的树不一致表明CVB5病毒分离株中两个区域之间可能的重组。肠道病毒基因组的不同部分可能因其独特的作用而独立发展,5′UTR含有起始位点和内部核糖体进入位点,而在P1区内编码的衣壳负责病毒进入宿主细胞和组织嗜性。 此外,衣壳也是宿主免疫应答的重要靶标[18]。造成不一致谱系的4个病毒分离株使用亚基因组序列跨越部分5′UTR-VP4-5′VP2进行Similarity plot分析,发现在病毒分离株AY875692.1 /Korea/ 2000中发生重组。然而,病毒分离株KF311743.1/China/2011是一种独特的病毒分离株,BLAST比对后GenBank中没有病毒分离株序列与其VP1区域相似。在5′UTR和VP1树中具有不一致谱系的另外两个病毒分离株均来自瑞士的未处理污水,但在Similarity plot分析中未显示重组。
因为只有不超过0.01%的肠道病毒能够被分离获取,所以很难系统地研究肠道病毒的进化[19]。可获得的数据表明重组通常发生在B组肠道病毒的基因组中的几个基因区域[20]。并且作为HEV-B的成员,在通过系统发育分析证实的CVB5基因型C的进化研究中,其基因组中频繁重组[21]。CVB5病毒分离株AY875692.1 /Korea/ 2000的重组发生在VP4区域。Similarity plot分析表明,5′UTR的JX976772.1/Shandong/2010和VP2的5′端的KT28000.1 /Hong Kong/ 1972均具有高于0.9的相似值,这表明这两个病毒分离株可能是这种重组的供体。该研究中的重要性是重组发生在P1区域,其是CVB5的衣壳编码区,尽管先前的报道表明在除P1区域之外的所有基因组区域中检测到重组断裂点[10]。此外,本研究的结果表明,CVB5病毒分离株AY875692.1 /Korea/ 2000是埃可病毒25和猪水疱病病毒的接受者,但是基因组非结构区域内的重组不同(P2和P3),他们仅在同一物种的成员中被观察到[19]。肠道病毒衣壳蛋白暴露在病毒体表面,但是VP4蛋白排列在衣壳内部,提示本研究中观察到的VP4区域中的重组对于依赖于VP1蛋白的功能影响很小。
4 结 论
5′UTR和VP1之间的进化不一致,以及详细的系统发育分析表明,VP4作为结构蛋白编码区可能是CVB5进化过程中的候选重组基因座。
