中国地鼠口腔鳞状细胞癌相关的长链非编码RNA差异表达谱建立及应用
2019-12-04王晓堂肖兰飞高继萍梁宇翔轩瑞晶闫晓如宋国华
王晓堂,肖兰飞,高继萍,梁宇翔,轩瑞晶,闫晓如,高 莉,宋国华
(山西医科大学实验动物中心,实验动物与人类疾病动物模型山西省重点实验室,太原 030001)
口腔癌作为近年来频发的肿瘤,在全身恶性肿瘤中约占3%~5%,是全球范围内常见的第11位癌症,具有转移性高、手术切除难度大、术后易复发等特点[1]。口腔鳞状细胞癌(Oral squamous cell carcinoma,OSCC)的发病率约占口腔恶性肿瘤的90%,且发病率较高、恶化程度极强,患者5年存活率在50%左右[2]。已有证据指出,口腔鳞状细胞癌的形成与患者的多种不良生活习惯有关。此外,遗传因素、慢性炎症及病毒感染也是该病的诱因[3]。世界卫生组织预计,未来几十年口腔癌的发病率将持续上升,成为一种严重的世界公共卫生问题[4]。因此,需进一步研究口腔鳞状细胞癌发病进程中的相关机制,以便更好地进行癌前诊断与预后治疗。
近年来,大量研究表明长链非编码RNA(long non-coding RNA,LncRNA)在多层次调控生物体的基因表达,参与生物体内多种重要的生物学过程[5]。最新研究指出,LncRNA具有复杂的生物学功能与作用调控机制,通过介导基因表达、转录、翻译以及组织分化等途径调控基因表达,引起细胞功能紊乱、代谢失调,导致多种疾病和癌症的形成[6-7]。目前,有研究指出LncRNA在多种肿瘤发生过程中异常表达,参与调控肿瘤细胞的增殖、迁移、侵袭与凋亡过程,在肿瘤发生发展中发挥致癌或抑癌作用,是肿瘤发生的重要因素之一[8-10]。但是,LncRNA在口腔鳞状细胞癌中作用机制的研究相对较少,局限于未能获得足够的临床样本。因此,建立与人类口腔鳞状细胞癌相似的疾病动物模型模拟OSCC的发生发展过程,深入研究其发病机制至关重要。
中国地鼠(Cricetulusbarabensisgriseus)具有独特的颊囊组织结构,口腔黏膜与人类的相似性较高,可用其构建口腔癌实验动物模型,模拟人类口腔癌癌变过程[11-13]。本课题组在前期研究中已成功建立中国地鼠口腔鳞状细胞癌动物模型,本文将高通量LncRNA测序技术与生物信息学技术相结合,筛选中国地鼠口腔鳞状细胞癌组织样本与正常组织样本中显著差异表达的LncRNA,对其进行靶基因预测,并对靶基因进行GO功能分析与KEGG信号通路分析,预测差异表达的LncRNA可能具有的功能,深入探讨其对中国地鼠口腔鳞状细胞癌发生发展的调控机制,为口腔鳞状细胞癌的临床研究提供数据与理论基础。
1 材料和方法
1.1 实验动物
清洁级中国地鼠60只,雄性,8~10周龄,体重(20 ± 2)g,购自山西医科大学实验动物中心[SCXK (晋)2015-0001]。中国地鼠饲养于山西医科大学实验动物中心屏障环境[SYXK (晋)2015-0001],温度25℃,湿度40%~70%,光暗循环12 h/12 h)。严格按照山西医科大学实验动物管理委员会的要求进行全部动物实验【IACUC号:2017016】,所有实验均遵循3R原则。
1.2 主要试剂与仪器
RNA提取试剂盒(北京天根生物科技有限公司),普通PCR与荧光定量PCR相关试剂(日本 Takara 公司),Nanodrop微量分光光度计(美国Thermo Fisher公司),Qubit荧光定量仪(美国Thermo Fisher公司),Agilent 2100生物芯片分析系统(美国Thermo Fisher公司)。
1.3 实验方法
1.3.1 组织样本中RNA提取
本课题组前期采用二甲基苯并蒽涂抹的方式成功制备口腔鳞状细胞癌动物模型[12-15]。在课题组前期制备的中国地鼠口腔鳞状细胞癌模型组与正常组中分别随机选取3只动物,取适量的颊囊组织样本用液氮充分研磨,采用RNA提取试剂盒提取不同组织中的总RNA。
1.3.2 高通量LncRNA测序
RNA提取完成后,对其进行质检。首先,采用琼脂糖凝胶电泳检测RNA完整性、降解程度以及是否被污染。RNA纯度采用Nanodrop微量分光光度计(美国Thermo Fisher公司)进行检测,浓度采用Qubit荧光定量仪(美国Thermo Fisher公司)进行精确定量。采用Agilent 2100生物芯片分析系统(美国Thermo Fisher公司)精确检测RNA的纯度与完整性。完成质检后,对质检合格的RNA进行片段化、反转录成cDNA、末端修复、加接头、PCR扩增等一系列操作后完成cDNA文库构建[16-17]。筛选片段大小为200 bp左右的cDNA文库,按有效浓度及目标上机数据量的需求进行Illumina测序[17]。
1.3.3 测序数据处理
测序获得的原始序列中存在低质量数据,本研究采用FASTQC软件[18]处理原始数据并进行质量控制后得到高质量数据。质控完成后,使用RSEM比对软件[19]将高质量数据比对到中国地鼠参考基因组上进行序列联配。RSEM比对软件使用自身脚本对联配的结果进行表达量提取,生成基因的读数、FPKM和TPM表达矩阵[20-21]。最后,筛掉低表达量的基因(平均数值>5),分离mRNA和LncRNA对应的基因。
1.3.4 差异表达LncRNA筛选及靶基因预测
首先,将RSEM估算得到的各个LncRNA的读数作为输入数据输入到DESeq2软件[20-21]进行差异鉴定,软件通过自行标准化与模型拟合,通过Wald检验[22-23]鉴定两组样本间基因的差异显著性。其次,采用生物统计学与生物信息学相结合的方法筛选与OSCC密切相关的显著差异表达的LncRNA,以log2(差异倍数)1(|log2(fold change)|1),P<0.05为筛选条件。本研究通过表达量相关性分析的方法预测差异表达LncRNA的反式作用靶基因,选取P<0.05的基因作为反式作用候选基因。
1.3.5 GO功能富集分析与KEGG信号通路分析
差异表达LncRNA的靶基因富集分析包括GO功能(Gene Ontology)分析与KEGG信号通路(Kyoto Encyclopedia of Genes and Genomes pathway)分析[24]。本研究采用GO功能注释对中国地鼠口腔鳞状细胞癌组织样本中差异表达的LncRNA的靶基因进行功能分析,使用生物统计学方法筛选差异表达基因富集的生物学功能,以P<0.05表示具有统计学意义的功能条目。采用KEGG信号通路富集分析,以达到对差异表达LncRNA的靶基因生物通路注释的目的,筛选出与OSCC相关的信号调控网络,以P<0.05表示具有统计学意义的信号通路条目。
1.4 统计学方法
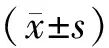
2 结果
2.1 中国地鼠口腔鳞状细胞癌组织样本与正常组织样本差异表达基因
采用高通量LncRNA测序技术对组织样本进行测序分析,结果表明与中国地鼠正常颊囊组织样本相比口腔鳞状细胞癌组织样本中共筛选出54个显著差异表达的LncRNA(P<0.05,|log2(差异倍数)|>1),31个基因表达上调,23个基因表达下调,并绘制了显著差异表达的LncRNA的火山图(图1)。进一步采用t检验分析实验数据,筛选出10个差异具有极显著统计学意义的LncRNA(P<0.01,|log2(差异倍数)|>3),分别为LOC100754872、LOC103160606、LOC107978163、LOC103163417、LOC103162225、LOC103161306、LOC107979530、LOC103161117、LOC103164378、LOC103161380。其中7个基因表达上调,3个基因表达下调(表1)。

表1 两组织样本中显著差异表达前十的LncRNA
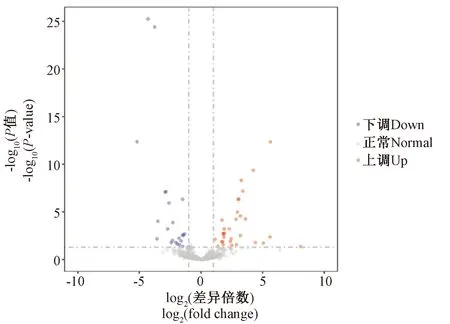
注:X轴代表差异表达倍数的log2值,纵坐标代表各个基因的经过标准化后的P值。显著上调的基因(|log2(差异倍数)|>1)为红色标识,显著下调的基因为蓝色标识,表达量没有变化的基因为灰色标识。
2.2 中国地鼠口腔鳞状细胞癌组织样本与正常组织样本差异表达的LncRNA靶基因预测
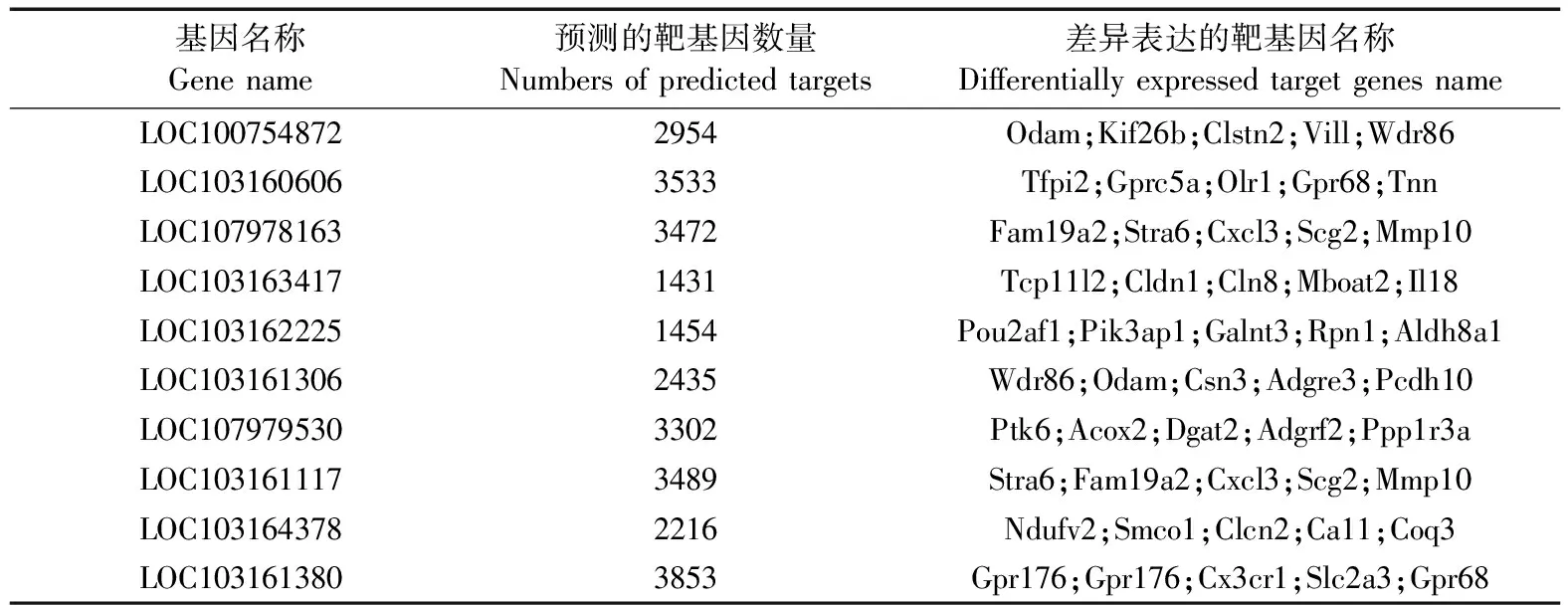
表2 显著差异表达LncRNA靶基因预测结果统计
采用靶基因预测软件预测显著差异表达的54个LncRNA反式作用靶基因。本研究对极显著差异表达(P<0.01)的10个LncRNA预测的靶基因数目进行统计,不同LncRNA位于染色体上不同位置,发挥不同生物学功能,因此预测的靶基因数目不同(表2)。进一步使用生物统计学方法分别统计了极显著差异表达的LncRNA相关性最高的前5个靶基因(表2)(相关性>0.85,P<0.01)。推测组织样本中极显著差异表达的LncRNA可能通过与其靶基因之间相互调控发挥作用,从而调控口腔鳞状细胞癌的发生进程。
2.3 中国地鼠口腔鳞状细胞癌组织样本与正常组织样本差异表达LncRNA的靶基因GO分析
采用GO功能注释方法分析54个显著差异表达的LncRNA的靶基因富集的生物学功能。GO注释结果中共获得73个条目,70条与生物过程相关,3条与细胞位置相关,未发现与分子功能相关的条目。采用生物统计学方法筛选显著富集的GO条目(P<0.05),其中10条与生物过程相关,3条与细胞位置相关,该结果表明差异表达基因的靶基因主要分布在细胞外区域,参与调控硫酯、酰基辅酶A、辅酶、辅因子、脂质代谢过程,调节细胞死亡、细胞黏附、组织发育及组织形态发生等过程(图2A)。以P<0.01为筛选条件,筛选出极显著富集的GO条目,8条与生物过程相关(图2B、2C),3条与细胞位置相关(图2D、2E)。推测以上生物学功能的紊乱或异常可能引起口腔鳞状细胞癌的恶化。
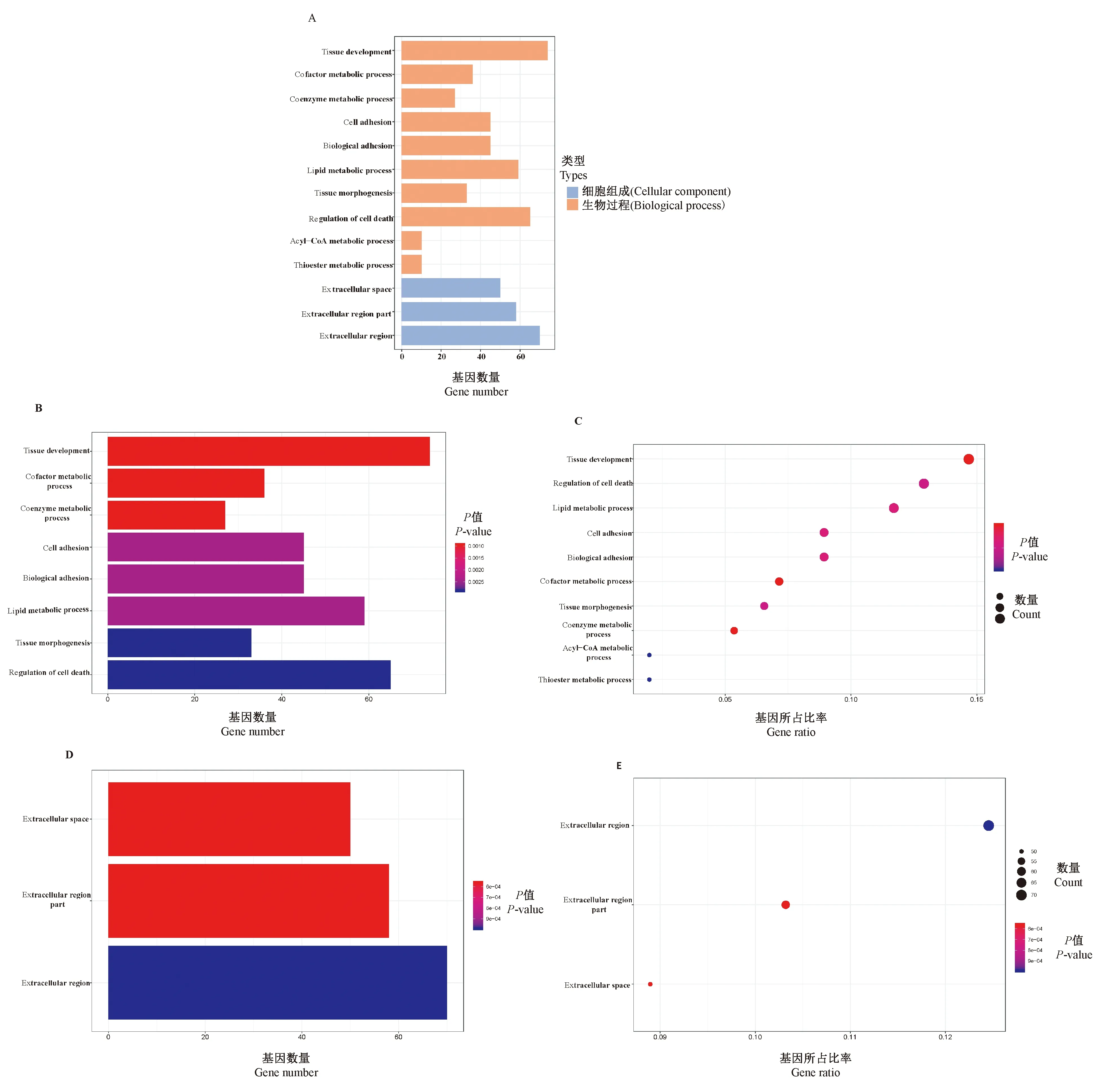
注:A:GO分类所有条目柱状图,横坐标为富集的基因数量,纵坐标为GO条目,不同的颜色代表不同的GO分类。B:生物过程相关的GO条目的柱状图。C:生物过程相关的GO条目的气泡图。D:细胞组成相关的GO条目的柱状图。E:细胞组成相关的GO条目的气泡图。
2.4 中国地鼠口腔鳞状细胞癌组织样本与正常组织样本差异表达LncRNA的靶基因KEGG分析
KEGG通路富集分析结果表明,差异表达的LncRNA的靶基因共富集了25条信号通路(P<0.05)。采用生物统计学方法,筛选极显著富集的信号通路(P<0.01),共筛出12条极显著的信号通路(图3A、3B,表3)。主要包括细胞因子-细胞因子受体相互作用信号通路(cge04060)、粘着力信号通路(cge04510)、轴突指导信号通路(cge04360)、钙信号通路(cge04020)与TNF信号通路(cge04668),这些通路多与肿瘤的发生发展密切相关。此外,差异表达的LncRNA的靶基因还参与调控ARVC信号通路(cge05412)、DCM信号通路(cge05414)、HCM信号通路(cge05410)、心肌细胞的肾上腺素信号传导通路(cge04261)、造血细胞谱系信号通路(cge04640)、萜类骨架的生物合成(cge00900)及ECM-受体相互作用通路(cge04512),这些通路大多与心脏疾病密切相关。推测在口腔鳞状细胞癌中这些信号通路在不同程度上发挥调控作用,影响口腔鳞状细胞癌的发生发展。

注:A:KEGG信号通路柱状图。B:KEGG信号通路气泡图。
3 讨论
口腔癌是经过一系列组织病理学发展进程演变而来,病损因轻到重,最后发展为原位癌或口腔鳞状细胞癌[25-26]。近年来,口腔鳞状细胞癌的发病者趋于年轻化,发病率不断上升[27]。由于缺少针对性的早期筛查方法,就诊患者大多为晚期病人。因此,寻找口腔鳞状细胞癌早期筛查与诊断预后的生物标记物成为近年来临床研究热点。长链非编码RNA是长度大于200 bp的非编码RNA,不编码蛋白质,在生物体生长发育、生殖分化过程中发挥重要作用,是目前生物学与医学的热点研究领域[28-29]。大量研究报道,LncRNA在表观遗传与基因表达过程中扮演重要角色,参与细胞增殖、分化、凋亡进程[30-32]。LncRNA与其靶mRNA的3’非翻译区部分序列通过互补配对的方式结合,调控靶mRNA的表达,导致多种肿瘤的发生,在一定程度上影响肿瘤细胞增殖,参与调控细胞病变与癌症恶化过程,在肿瘤增生及患者生存期不同的病理生理特点中发挥作用,表明LncRNA可能作为潜在的癌症诊断标记物[33-34]。高通量LncRNA测序技术能在较短的时间内对大量LncRNA基因组信息进行分析,预测其可能具有的生物学功能,有利于加速新的肿瘤标记物的产生,推动肿瘤的临床研究与治疗。
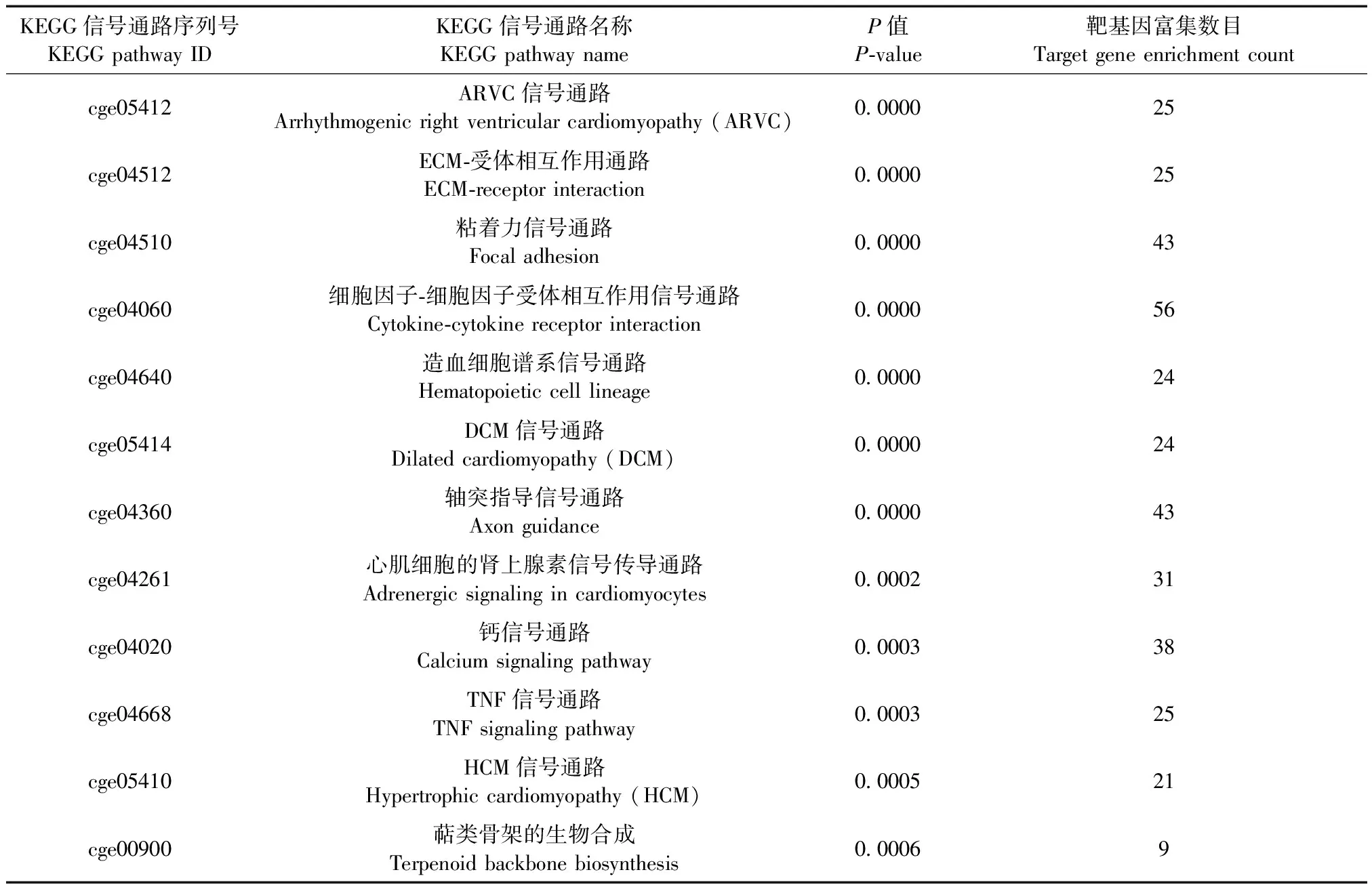
表3 极显著差异KEGG pathway统计表
大量临床数据表明,口腔癌患者体内异常表达的LncRNA参与调控肿瘤的发生发展[35-36]。有研究者对人类第一个口腔黏膜及其癌前病变的LncRNA表达谱进行研究,结果表明在癌组织中超过60%的异常表达的LncRNA与口腔癌相关[37]。孔祥盼等[38]研究指出LncRNAFOXCUT在口腔癌组织及鳞癌细胞中呈高表达状态,其表达下调后会抑制口腔鳞状细胞癌细胞的增殖、克隆与迁移。付丛等[39]研究表明,LncRNAAFAP1-AS1在口腔鳞癌组织中相对表达水平是正常组织的5.16倍,并与肿瘤临床分期、分化与转移相关。刘速等[40]研究指出LncRNAMALAT1敲低表达后抑制癌细胞的迁移、侵袭能力。Tang等[41]检测了口腔鳞状细胞癌中6种常见的LncRNA的表达情况,结果表明癌组织与正常组织相比MALAT-1、HOTAIR、NEAT-1、HULC、UCA1的表达显著升高,MEG-3表达水平显著降低。基于已有研究,推测LncRNA在口腔鳞状细胞癌的病变过程中发挥重要作用。因此,阐明新发现的LncRNA与已知的LncRNA在肿瘤形成过程中具有的生物学功能,将有助于癌症的早期诊断和预后治疗。目前,口腔鳞状细胞癌患者的组织样本较难获取,且耗时很长,严重阻碍了对其发病机制的研究。基于以上原因,本课题组在前期研究中采用化学诱导法成功建立中国地鼠口腔鳞状细胞癌动物模型,模拟人类口腔鳞状细胞癌发生的病变过程[12-15]。该动物模型的建立可以在较短的时间内获得实验所需的口腔鳞状细胞癌组织样本,以弥补临床样本的不足。大量临床研究采用基因芯片技术对肿瘤组织样本中差异表达基因进行筛选,本研究则选用高通量LncRNA测序技术进行研究。与基因芯片技术相比,采用高通量测序技术筛选模型动物组织样本中LncRNA差异表达谱可发现一些新的LncRNA,为揭示更多新发现的LncRNA在疾病中的作用调控机制提供数据基础。
本实验采用高通量LncRNA测序技术对中国地鼠口腔鳞状细胞癌组织样本与正常样本进行测序分析,揭示了LncRNA在两种组织样本中的差异表达,成功建立了中国地鼠口腔鳞状细胞癌相关的长链非编码RNA差异表达谱。采用聚类分析的方法对实验组与对照组差异表达基因进行研究,共筛选出54个显著差异表达的LncRNA(P<0.05)。对显著差异表达LncRNA的靶基因进行功能性分析发现,OSCC相关的GO条目有73条,以P<0.01为条件,筛选出极显著富集的GO条目,8条与生物过程相关,3条与细胞位置相关。GO功能分析表明,差异表达LncRNA的靶基因主要位于细胞外区域,参与调控参与调控硫酯、酰基辅酶A、辅酶、辅因子、脂质代谢过程,调节细胞死亡、细胞黏附、组织发育及组织形态发生过程。此结果与先前的研究报道一致[42-44]。KEGG信号通路富集分析结果指出,差异表达LncRNA的靶基因主要参与调控25条信号通路(P<0.05),其中极显著富集的信号通路12条(P<0.01)。主要包括细胞因子-细胞因子受体相互作用信号通路、粘着力信号通路、轴突指导信号通路、钙信号通路和TNF信号通路。这些信号通路在其他癌症发生发展中的作用机制已有报道,但其在口腔鳞状细胞癌中的报道相对较少[45-47]。因此,深入研究口腔鳞状细胞癌发生进程中的生物学功能变化与相关信号通路的作用机制,有利于更好的揭示口腔鳞状细胞癌的发病机制。
综上所述,本研究以中国地鼠为实验动物,利用其独特优势成功构建成功构建了符合口腔鳞状细胞癌病理生理过程的中国地鼠模型,实验操作简单,造模成功率高。应用此模型对口腔鳞状细胞癌进行基础研究,可为其临床治疗提供新思路。此外,本实验主要基于生物信息学技术对动物模型中差异表达LncRNA进行筛选,对其靶基因的功能进行预测,并未进行大量的实验验证,在接下来的研究中还有许多问题有待解决。因此,在下一步的研究中,将深入探讨测序结果筛选出的极显著差异表达LncRNA参与调控口腔鳞状细胞癌发生发展的具体调控机制,阐明其与靶基因的相互关系以及在口腔鳞状细胞癌相关的信号通路中发挥的作用,以期望为口腔鳞状细胞癌的癌前诊断与预后治疗提供新的肿瘤标记物。
注:中国地鼠(Cricetulusbarabensisgriseus)是仓鼠科仓鼠亚科动物,在公开出版物中使用仓鼠作为名称更为科学。但鉴于目前我国实验动物国家标准中提到的动物的种类都是地鼠,作为专业人员能够清楚该物种在动物分类中地位,并不妨碍科学研究的成果和使用。中文名称后面的拉丁文不会对该研究的主体产生歧义,故本文在发表时仍沿用“中国地鼠”,待国标修订后再统一命名为“仓鼠”。
