青稞叶绿体基因RNA编辑位点的预测、鉴定与比较分析
2019-11-29任苗苗聂小军
昌 西,裴 悦,胡 凡,任苗苗,王 彤,聂小军
(1.西藏农牧学院植物科学与技术学院,西藏林芝 860000;2.西北农林科技大学农学院,陕西杨凌 712100)
RNA编辑(RNA editing)是指DNA在转录形成mRNA过程中,由于核苷酸的插入、缺失或替换而使得原有的遗传信息发生改变的生物学现象[1]。大量研究发现,RNA编辑是一种常见的RNA水平修饰,是增加基因转录和功能多样性的重要形式,一个基因序列通过编辑修饰,可以合成多个不同的蛋白质,从而参与控制不同的性状或者生物学过程[2-3]。自1986年,RNA编辑现象首次在锥虫的线粒体中被发现以来[4],人们围绕RNA编辑已经开展了大量的研究工作,发现RNA编辑现象广泛存在于各种生物体中,包括动物、植物、真菌和细菌,表明RNA编辑可能是生物在长期进化过程中形成的一种保守的遗传信息扩展机制[5-8]。据报道,真核生物大约35%的基因存在基因编辑现象,不仅细胞核中三类RNA编码基因(mRNA、tRNA和rRNA)存在RNA编辑现象,细胞器(包括线粒体和叶绿体)基因组也存在大量的RNA编辑现象,并在转录后水平调控基因转录、翻译效率及时序特异性表达等方面发挥了重要作用[9-10],如玉米叶绿体基因rpl2,其起始密码子为ACG,必须发生C到T的编辑才能形成正确的起始密码子ATG,起始转录和翻译[11]。
叶绿体是植物细胞所特有的细胞器,是植物光合作用的主要器官,其具有自身的基因组并半自主地进行转录和翻译,在很多生理生化过程中发挥了重要作用[12-13]。因此,对植物叶绿体基因组中的RNA编辑进行系统分析和研究不仅有利于探究RNA编辑发生的分子机制和进化意义,也有利于解析叶绿体RNA编辑参与植物生长发育调控的生物学功能。1991,Hoch等[11]首次在植物叶绿体基因组中发现了RNA编辑现象。随后,研究者们对陆生植物叶绿体RNA编辑开展了大量研究工作,尤其是对ndhB基因研究较为深入[14-15]。研究发现,在高等被子植物中,ndhB基因存在约15个编辑位点,但低等植物中其编辑位点显著减少,比如在松叶蕨类植物中只有1个位点,在地钱类植物中仅有2个位点,在蕨类植物中有4个位点。在被子植物叶绿体基因组中,通常具有约30个编辑位点[16-17],Corneille等[18]在水稻叶绿体中发现了21个编辑位点,在玉米叶绿体中发了26个编辑位点,其中18个位点为两者共有;Kerstin等[19]利用RT-PCR技术对黑麦叶绿体的编辑位点进行了分析,共从33个转录本中鉴定发现31个编辑位点,其中5个是黑麦特有的;邓李坤等[20]通过对小麦返白系叶绿体RNA编辑位点进行分析,发现了分布于14个基因的28个位点。
青稞(HordeumvulgareLinn.var.nudum Hoork.f.)又称裸大麦,是青藏高原一种重要的传统粮食作物,具有悠久的栽培历史[21-22]。其在分类学上属于禾本科大麦属,是大麦的一种特殊类型,因其脱粒时内外颖和颖果容易分离,籽粒为裸露,故称裸大麦。青稞作为青藏高原的特色作物,现已成为藏区种植业、粮食生产与加工领域中占有支柱地位的主要作物[23]。青稞不仅是藏民族的主要粮食,同时也是酿造(青稞酒)的原料及牲畜的饲料,由于其籽粒蕴含丰富的矿物质、蛋白质、赖氨酸和β-葡聚糖,青稞逐渐成为新兴保健食品的主要原料[24]。与其他禾本科作物相比,青稞表现出更强的抗旱、抗寒、耐盐碱和耐瘠薄等优良特性。因此,青稞不仅为藏区人民的经济发展做出了非常大的贡献,而且为作物抗逆遗传改良提供了重要的基因库[25]。目前,青稞全基因组测序已经完成,为开展青稞基因组学研究提供了必要的序列资源和信息,推动青稞基础研究进入到组学时代[26]。同时,青稞的叶绿体基因组也已测定[27],为鉴定青稞叶绿体基因组RNA编辑位点奠定了基础。鉴于目前有关青稞叶绿体RNA编辑的研究还未见报道,本研究首先通过生物信息学方法对青稞叶绿体基因组中的RNA编辑位点进行预测,然后结合RT-PCR技术对预测结果进行验证,最后将青稞与栽培大麦、野生大麦及其他麦类作物叶绿体RNA编辑位点进行比较,以期从RNA编辑视角揭示青稞的起源和进化关系,为研究青稞叶绿体RNA编辑的生物学功能奠定基础,也为进一步揭示RNA编辑的作用机制提供有益信息。
1 材料与方法
1.1 材 料
本研究以青稞主栽品种藏青2000为试验材料。将其室内发芽后,正常培养至三叶期,采集幼嫩叶片,并保存在-80 ℃冰箱中备用。
1.2 方法
1.2.1 青稞及栽培大麦、野生大麦叶绿体基因组RNA编辑位点的预测
首先,从NCBI数据库中下载青稞的叶绿体基因组序列(序列号为KT962228.1),根据注释信息,提取其蛋白质编码基因并去冗余,得到76个蛋白质编码基因;然后将这些序列提交到Prep-Cp数据库[28],对其RNA编辑位点进行预测。同时下载栽培大麦(EF115541.1)和野生大麦(KC912689.1)的叶绿体基因组序列,提取所有蛋白质编码序列并去除冗余后,也采用同样方法进行RNA编辑位点的预测。
1.2.2 青稞DNA及总RNA的提取
取0.2 g叶片,在液氮中研磨成精细粉末,采用改良的CTAB法[29]提取样品的DNA;采用TRizol法(Life technology,USA)提取样品的总RNA,经紫外分光光度计测定样品浓度及琼脂糖凝胶电泳检测后-80 ℃保存备用。
1.2.3 青稞总RNA的纯化及cDNA的合成
将提取的RNA样品经DNaseⅠ(TaKaRa,大连)37 ℃处理30 min,去除其中可能存在的DNA污染,然后加入等体积的酚/氯仿(苯酚∶氯仿=25∶24)抽提,获得纯化后的总RNA,并取 1 μL用于cDNA的合成。反转录采用PrimeScriptTMRT Reagent Kit进行(TaKaRa,大连),具体步骤参见说明书。最后,将得到的反转录产物保存于-20 ℃备用。
1.2.4 青稞叶绿体基因RNA编辑位点的PCR验证
为检验预测到的编辑位点准确性,将所有预测的分布于15个基因的编辑位点进行RT-PCR验证。根据编辑位点上下游序列,利用Primer 5.0设计相应的引物(见表1)进行PCR扩增。PCR体系为20 μL,包括10 μL 2×Mix,8 μL ddH2O,1 μL上述步骤得到的cDNA,目标基因上下游引物各0.5 μL。PCR程序为:94 ℃ 5 min;94 ℃ 30 s,50~60 ℃ 30 s,72 ℃ 1 min,共34个循环;最后72 ℃ 5 min。同时以青稞DNA为模板,采用同样引物和程序进行PCR扩增。PCR产物用纯化回收试剂盒(天根,北京)回收后直接进行双向测序(生工,上海)。用Seqman 11.2软件对测序结果进行拼接,用 BioEdit 7.2.6工具比对每个基因的DNA和cDNA序列,最后手工校对和分析其中的RNA编辑位点。

表1 RT-PCR和PCR扩增所用引物序列信息
1.2.5 RNA编辑对蛋白结构的影响
选取验证后发生编辑的叶绿体基因,将其编辑前后的蛋白序列分别采用在线工具TMHMM Server V.2.0(http://genome.cbs.dtu.dk/services/TMHMM/)和NPS(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=/NPSA/npsa_server.html)对其蛋白质跨膜区段和蛋白质二级结构域进行预测和比较,初步分析RNA编辑对其编码蛋白结构的影响。
1.2.6 禾本科作物叶绿体基因RNA编辑位点间的比较
将预测获得的青稞叶绿体RNA编辑位点,与已报道的小麦[20]、野生二粒小麦[30]、粗山羊草[31]、黑麦[19]、大麦[3]、玉米[32]、水稻[18]、甘蔗[33]等禾木科作物的叶绿体编辑位点进行比较,分析麦类及其他禾本科作物叶绿体编辑位点的异同,鉴定保守位点。
2 结果与分析
2.1 青稞叶绿体基因RNA编辑位点的预测与 鉴定
将从NCBI数据库下载的青稞76个非冗余叶绿体基因组蛋白质编码基因递交到Prep-Cp数据库进行RNA编辑位点的预测(表2)。结果发现,在青稞叶绿体基因组中共预测到35个编辑位点,所有的编辑位点均发生了从C到U的变化;在76个蛋白编码基因中,发生RNA编辑的有15个基因;所有的编辑均引起了氨基酸的变化,氨基酸转变类型有7种,分别为H→Y、L→F、P→S、P→L、S→F、S→L、T→M,其中S→L和P→L两种转变形式出现次数最多,这与其他禾本科叶绿体基因组RNA编辑的组成特征相似[30-31]。在发生编辑的所有基因中,ndhB的编辑位点数最多,达9个;之后依次是rpoC2,有5个位点;rpoB和ndhA分别有4个位点,ndhF和rpoB分别有2个位点,其余的基因均为1个编辑位点(表2)。对编辑发生的密码子位置进行分析发现这些编辑位点中有4个发生于于密码子的第一位,剩余的31个均位于密码子的第二位,在密码子第三位没有编辑现象的发生。
2.2 青稞叶绿体基因RNA编辑位点的PCR验证
为了验证所预测的编辑位点是否为真实发生的编辑,采用PCR和RT-PCR方法对所预测的35个位于15个基因的青稞叶绿体RNA编辑位点进行了扩增及测序(图1)。结果发现,其中的10个基因中的18个位点DNA和cDNA碱基不一致,被证实确实发生了RNA水平的序列改变,即RNA编辑(图2)。所有这18个被验证的位点均被前期生物信息分析方法所预测到,没有发现新的未被预测到的编辑位点(表2),表明保守位点的生物信息预测的结果具有一定的准确性。其中ndhB基因预测的9个编辑位点均证实为真实存在的编辑位点(表2),暗示RNA编辑对nhdB基因生物学功能的正常发挥可能具有重要的调控作用。
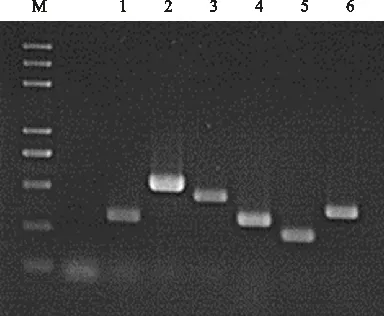
M:DL2000; 1~6分别是基因ndhF、petB、rpl2、rpl20、rpoA和ycf3的扩增产物。
M:DL2000 marker; 1-6 indicate PCR products ofndhF,petB,rpl2,rpl20,rpoAandycf3,respectively.
图1 部分基因的RT-PCR电泳图谱
Fig.1 Electrophoresis of the PT-PCR proclucts of 6 chloroplast genes in hulless barley
2.3 RNA编辑对蛋白质结构的影响效应分析
为了明确RNA编辑对蛋白质结构的影响,我们进一步对未编辑和编辑后其所编码蛋白质的二级结构进行了预测。结果表明,在所有可能发生RNA编辑的基因中,基因ndhD、rpl2、atpA、atpB发生编辑后的α螺旋减少;而基因petB、rps8、ndhA的α螺旋则相对增多;其中基因rpl2和atpB的无规卷曲数在发生编辑后有所减少,其他5个基因的无规卷曲数量均有增加;另外,基因rpl2、petB、rps8的延伸链数量在RNA编辑后有所增加,其余4个基因的延伸链数量减少(图3)。以上预测结果说明,RNA编辑的发生会影响蛋白质的二级结构,因此在编辑发生后,由于蛋白质结构的变化,可能导致相应的生理生化反应发生变化从而最终影响基因生物学功能的表达。对基因跨膜结构域的预测结果表明,基因ndhA与ndhB在RNA编辑发生后,均发生了较显著的变化,即分别多出了3处和1处跨膜结构域(图4)。
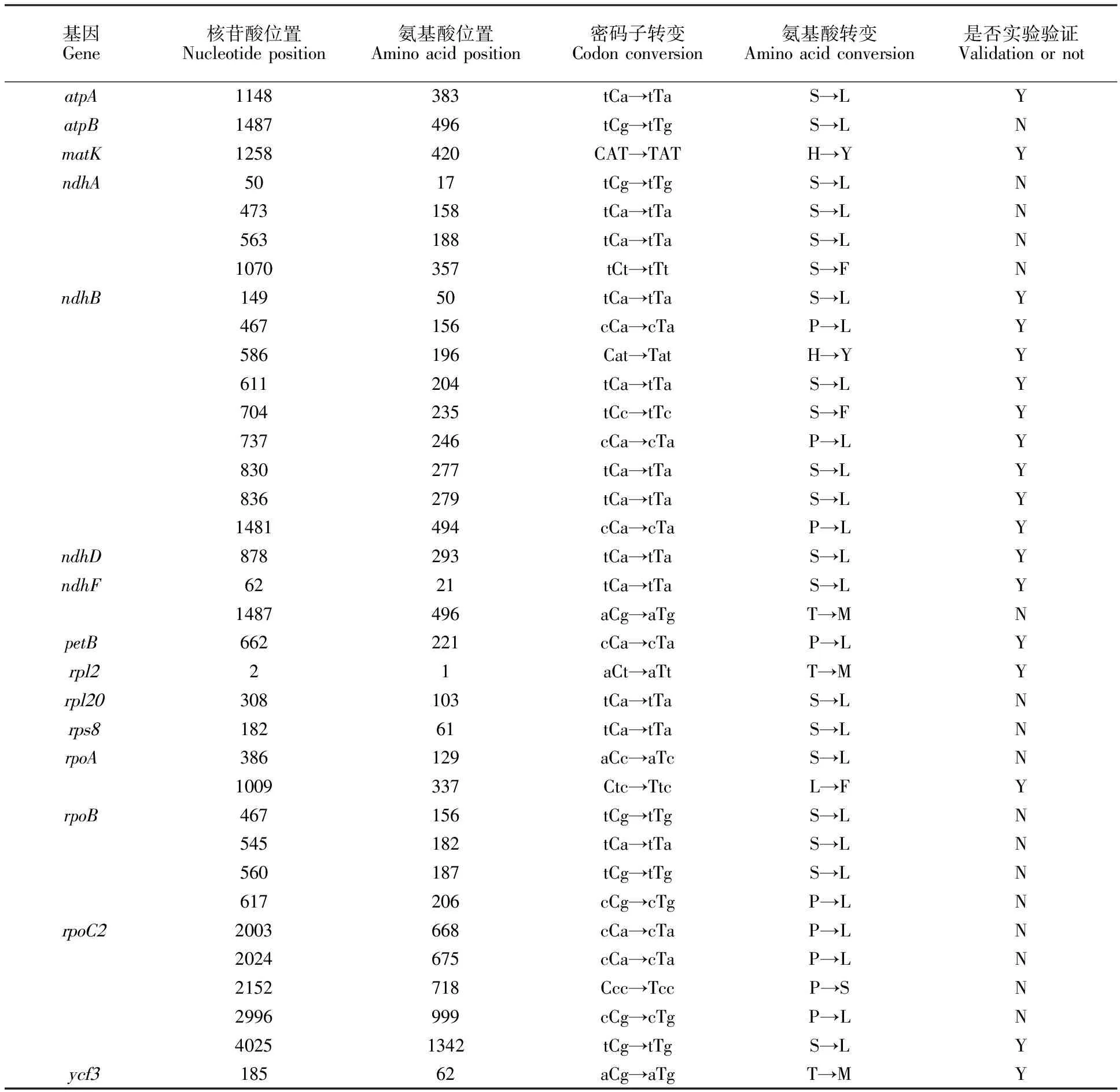
表2 青稞叶绿体基因RNA编辑位点预测与验证
Y表示实验验证的编辑位点;N表示实验验证未发生编辑的位点。
Y represents the validated RNA editing sites by RT-PCR while N represents the non-validated site.
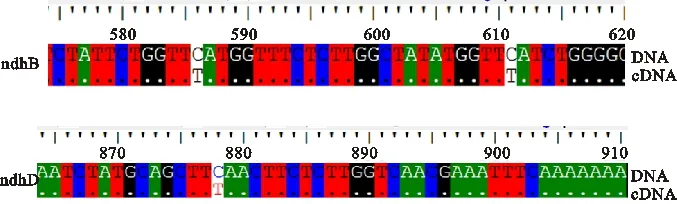
图2 青稞ndhB和ndhD基因的序列比对及RNA编辑位点的鉴定

A,B,C,D,E,F,G分别为编辑前的ndhD,rpl2,atpA,atpB,petB,rps8和ndhA基因编码蛋白的二级结构; a,b,c,d,e,f,g分别为编辑后的ndhD,rpl2,atpA,atpB,petB,rps8和ndhA基因编码蛋白的二级结构。
A to G indicate secondary structures of coding proteins before editing ofndhD,rpl2,atpA,atpB,petB,rps8andndhArespectively; a to g indicates secondary structures of coding proteins after editing ofndhD,rpl2,atpA,atpB,petB,rps8andndhA,respectively.
图3 RNA编辑对基因编码蛋白二级结构的影响
Fig.3 Predicted secondary structures of seven proteins encoded by seven edited genes
2.4 青稞与其他禾本科物种叶绿体基因RNA编辑位点间的比较
比较分析青稞与栽培大麦以及野生大麦的RNA编辑位点的异同,发现青稞与栽培大麦的RNA编辑位点分布和组成完全一致,均有分布于15个基因中的35个编辑位点,而野生大麦只有34个RNA编辑位点;相比于栽培大麦和青稞,野生大麦中缺少了ndhA-563编辑位点(表3)。结合前人研究结果,比较青稞与其他七个禾本科物种的叶绿体RNA编辑位点的异同,发现在这8个物种中有atpA-1、ndhA-2、ndhB-2等13个位点均发生了完全编辑,暗示这些位点在禾本科物种进化中具有较高的保守性;而ndhK-1只在黑麦中发生了编辑,其他7个物种中均未发生编辑,表明其为黑麦所特异的编辑位点(表3)。
3 讨 论
作为一种非常重要的转录后基因表达调控方式,RNA编辑极大地丰富了生物体的遗传信息。在高等植物中,RNA编辑主要在叶绿体等细胞器基因组中发生。大量研究发现,RNA编辑在植物生长发育、逆境响应等生理生化过程中起着重要的调控作用。发现和鉴定RNA编辑位点是开展RNA编辑生物学功能相关研究的前提。本研究首先利用生物信息学方法,采用Prep-Cp数据库预测得到了青稞叶绿体基因组中可能发生了RNA编辑的位点,然后基于预测结果,通过分子克隆方法对这些预测的编辑位点进行了实验验证。预测发现,青稞叶绿体基因组的76个蛋白编码基因中,有15个发生了RNA编辑,共包含35处编辑位点,而其中18个被实验证实,暗示生物信息学预测虽然为RNA编辑的发掘与鉴定提供了一个高效、简便的方法,但对预测结果需要进一步的实验验证才能保证结果的准确性[30-31]。同时发现,所有编辑位点均为胞嘧啶到尿嘧啶的变换,而且编辑位点主要发生于密码子第二位碱基,第一位碱基变换频率次之,而第三位没有发现编辑位点,这与前人对禾本科植物叶绿体RNA编辑组成特征分析的结果相一致[34]。进一步分析还发现,RNA编辑的发生会造成相应的氨基酸发生改变;最常见的改变是从丝氨酸(Ser,S)到亮氨酸(Leu,L)的转变,这与早前高等植物的叶绿体RNA编辑的特征一致[35-36]。同时,这也表明由RNA编辑所导致的基因个体间翻译的蛋白质可能存在差异,从而使得基因组具有了遗传多样性。RNA编辑引起相应的蛋白结构发生变化,由此或将造成蛋白功能发生变化,而蛋白质功能的变化如何影响或调控植株的生长发育,还有待我们更深入的研究。
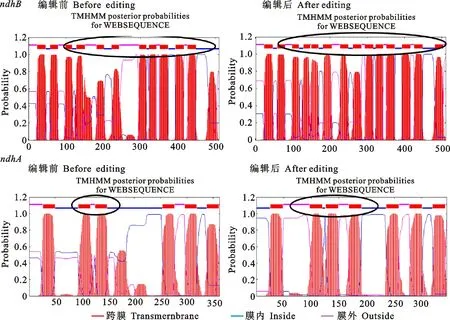
图4 RNA编辑对基因ndhA和ndhB编码蛋白的跨膜结构域的影响
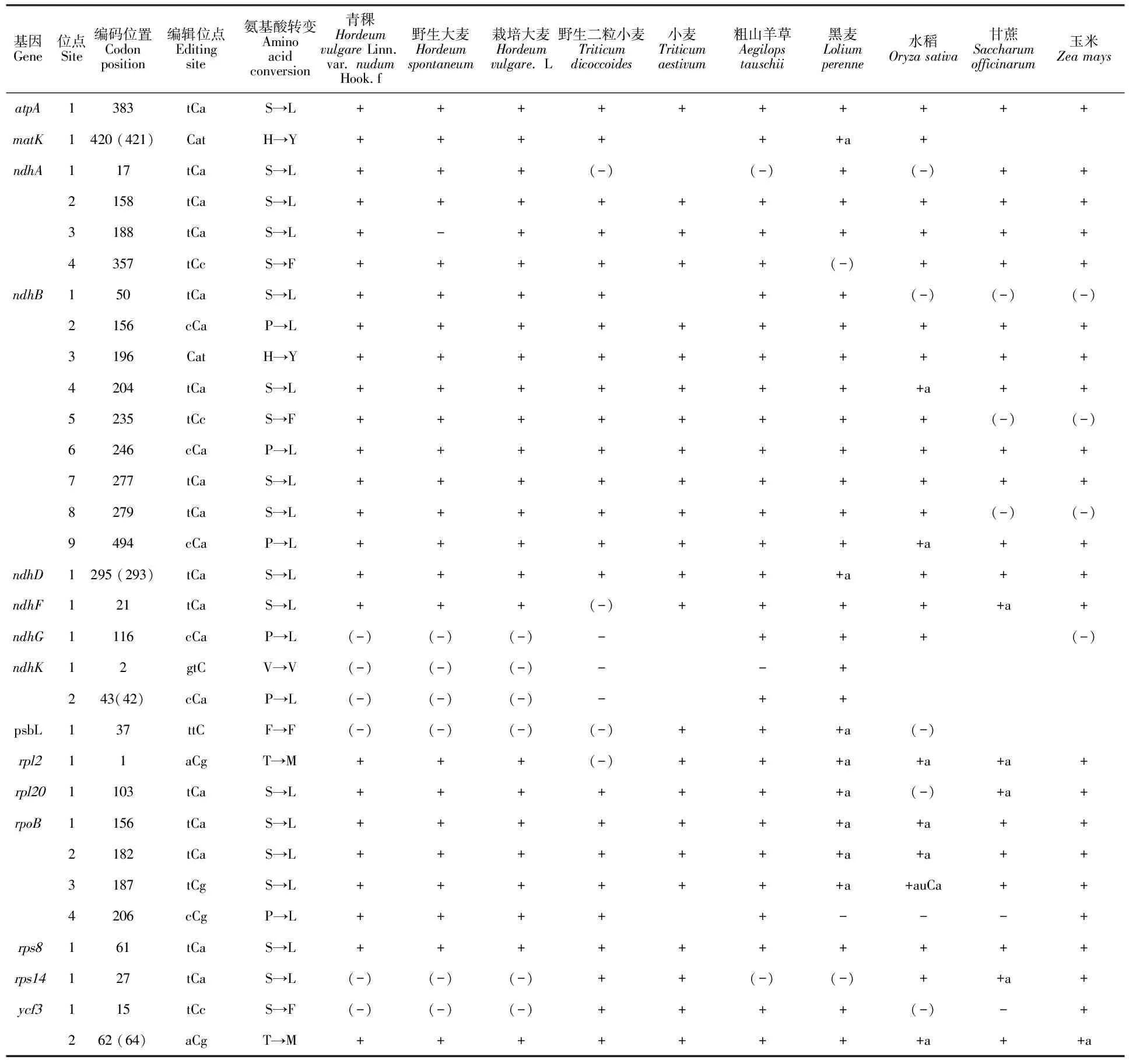
表3 青稞与野生大麦、栽培大麦及其他7种禾本科植物叶绿体基因RNA编辑位点的比较
编辑位点列中大写字母代表该碱基发生了编辑;+表示完全编辑;-表示未发生编辑,DNA序列中为C;(-)表示未发生编辑,DNA序列中为T;+a表示部分编辑;空格表示编辑情况未知。
Capital letters in editing site indicate edited bases;+ indicates complete editing;- indicates no editing and C encoded in DNA;(-)indicates no editing and T encoded in DNA;+a indicates partial editing;Blank indicates unknown editing.
