利用SRAP与ISSR分子标记鉴别牛樟种质
2019-11-28周艳平郑惠成彭金彬方福钟邹双全
王 巧, 周艳平, 郑惠成, 彭金彬, 方福钟, 邹双全
(1.福建农林大学林学院,福建 福州 350002;2.莆田市荔城区西天尾镇林业站,福建 莆田 351131;3.福建省林业调查规划院,福建 福州 350003;4.泉州市洛江区林业局,福建 泉州 362011;5.漳州市林业科学研究所,福建 漳州 363000)
牛樟(CinnamomumkanehiraeHayata)属樟科(Lauraceae)樟属(Cinnamomum)植物,又名黑樟,原产地为我国台湾[1]。牛樟主要分布于海拔200~2 000 m的山区,尤其以台湾的新竹、花东及中南部较多[2]。老龄腐朽的牛樟树干内壁寄生了极具药用价值的牛樟芝,因而深受医疗保健市场的欢迎[3]。由于过度砍伐,目前牛樟天然林仅存在于交通不便的中、高海拔地区,且多为单株分散的高龄老树,结实量少,树高体大不容易攀爬,母树间授粉不易,采种也极其困难[4]。现有的大量民间引种未经系统鉴别,导致种源混乱,阻碍了牛樟的良种保存与繁育。为加强牛樟种质资源的保护与满足市场需求,需建立有效的鉴别技术。
分子标记技术已在作物遗传多样性分析、品种鉴定及遗传图谱构建等方面得到广泛应用。相关序列扩增多态性(sequence-related amplified polymorphism, SRAP)具有遗传信息丰富、可检测的多态性强、DNA纯度要求不高、条带清晰等特点[5],已广泛应用于水稻[6]、桃[7]及芹菜[5]等研究中。简单重复序列区间(inter-simple sequence repeat, ISSR)具有遗传信息丰富、可检测的多态性强、试验操作简单并且成本较低等特点[8]。朱丽萍[8]采用ISSR分子标记技术检测牛樟、冇樟和樟树三者的族群遗传结构表明,牛樟为台湾的特有种,并认为牛樟是由樟树演变而来的。孟红岩等[9]探索了台湾牛樟总RNA的提取方法;Hung et al[10]在樟属植物中分离出了15个微卫星序列,可用于牛樟等樟属植物的鉴定;曾焕中等[11]根据樟脑素、松油醇和黄樟素含量判别是否为牛樟。目前尚未见利用SRAP与ISSR分子标记技术研究牛樟种质。福建省为适宜牛樟生长的主要地区,为此本研究在福建选取10个牛樟种质,通过SRAP与ISSR分子标记技术分析种质间的遗传多样性,以期为牛樟优质种源的保存与推广提供依据。
1 材料与方法
1.1 试验材料
10个牛樟种质分别取自:①福建农林大学植物园(农大牛樟);②南安市蓬华镇苏厝村将军山(南安牛樟);③永春县东平镇冷水村(永春牛樟1);④永春县东平镇冷水村(永春牛樟2);⑤德化县龙浔镇高阳村(德化牛樟);⑥福建省林业科技试验中心(南靖牛樟);⑦漳浦县扬基生物科技有限公司(漳浦牛樟);⑧漳州市龙海林下国有林场(林场牛樟);⑨永安市贡坪采育场(永安牛樟1);⑩永安市大西坑永林种苗中心(永安牛樟2)。其中,农大牛樟与漳浦牛樟由种植人从台湾引进,并已通过台湾专家鉴定。
1.2 DNA提取与检测
2016年7月初采集发育良好且无病虫害的牛樟嫩叶,采用改进的十六烷基三甲基溴化铵法(cetyltrimethyl ammonium bromide, CTAB)提取DNA[12]。用Nano Drop ND-1000核酸蛋白检测仪(Nano Drop Technologies Inc,美国)检测DNA浓度,通过1.5%琼脂糖凝胶电泳检测DNA质量[13]。
1.3 SRAP-PCR扩增与引物筛选
从16个正向和16个反向引物组合的256对引物中筛选出10对具有丰富多态性的SRAP引物组合(表1),进行SRAP-PCR扩增试验。程序基本采用 Zhou et al[14]的方法,并稍作改良。
1.4 ISSR-PCR扩增与引物筛选
从哥伦比亚大学公布的100个ISSR引物中筛选出10个具有丰富多态性的ISSR引物(表2),进行ISSR-PCR扩增试验。试验基本参照刘玉香[15]的ISSR-PCR反应体系。
1.5 统计与分析
筛选出SRAP与ISSR引物,观察其扩增结果的电泳图。在电泳图谱中,迁移性一致的DNA条带即具有同源性。采用人工读图的方式,将电泳图上有清晰条带的记为“1”,没有条带或者条带不清晰的记为“0”。根据每对引物组合扩增出来的条带计算多态性位点数目(number of polymorphic bands, NPB)、总扩增位点数目(total number of bands, TNB)和多态性位点百分比(percentage of polymorphic bands, PPB)。公式:PPB=NPB/TNB。使用NTSYS 2.1统计分析软件中Similarity程序计算10个牛樟种质间的遗传相似系数(genetic similarity, GS),再用非加权组平均法(unweighted pair-group method using arithmetic average algorithm, UPGMA)进行聚类分析[16]。

表1 10对SRAP引物组合名称和序列

表2 10个ISSR引物名称和序列1)
1)R=(A,G),Y=(C,T),B=(C,G,T),D=(A,G,T),H=(A,C,T),V=(A,C,G)。
2 结果与分析
2.1 牛樟种质资源的多态性分析

表3 10对SRAP引物组合的扩增结果
(1)分别采用10对SRAP引物组合对10个牛樟种质进行DNA扩增。结果表明,片段大小主要集中在50~2 000 bp,10对引物总共扩增出251条条带,其中220条为多态性条带,多态性条带百分比为87.6%(表3)。Em8-Me9和Em10-Me2这两对引物组合产生的多态性条带百分比最多,均达到100%(图1)。(2)分别采用10个ISSR引物对10个牛樟种质进行DNA扩增。结果表明,扩增出来的片段大小主要集中在100~2 000 bp。10个引物总共扩增出237条条带,其中210条为多态性条带,多态性条带百分比为88.6%(表4)。UBC895和UBC900这两个引物产生的多态性条带百分比最多,均达到100%(图2)。

A.Em8-Me9;B.Em10-Me2。1~10分别为:农大牛樟、南安牛樟、永春牛樟1、永春牛樟2、德化牛樟、南靖牛樟、漳浦牛樟、林场牛樟、永安牛樟1、永安牛樟2。
2.2 牛樟种质资源的遗传相似性分析

表4 10个ISSR引物的扩增结果
分别根据SRAP、ISSR结果,用NTSYS 2.1软件计算10个牛樟种质的遗传相似系数(表5、6)。从表5可见,牛樟种质间的遗传相似系数变化范围在0.355~0.882之间,平均相似系数为0.606。从表6可见,牛樟种质间的遗传相似系数变化范围在0.371~0.814之间,平均相似系数为0.606。
2.3 牛樟种质资源的聚类分析
分别根据SRAP、ISSR扩增结果和10个牛樟种质间的遗传相似性,利用UPGMA法建立系统聚类分支树状图(图3、4)。从图3可见,在遗传相似系数为0.43时可将10个牛樟种质分成两大类,即Ⅰ类包括农大和漳浦牛樟,Ⅱ类包括南安、永春1、德化、永春2、南靖、林场、永安2和永安1牛樟。从图4可见,在遗传相似系数为0.41时可将10个牛樟种质分成两大类,即Ⅰ类包括农大和漳浦牛樟,Ⅱ类包括南安、德化、永春1、永春2、永安2、南靖、永安1和林场牛樟。
2.4 SRAP与ISSR的结果比较
2.4.1 遗传相似系数 (1)SRAP标记得到的10个牛樟种质间的遗传相似系数变化范围在0.355~0.882之间,平均相似系数为0.606,农大牛樟和永春牛樟1的遗传相似系数(0.355)最小,永春牛樟1和德化牛樟(0.882)最大。将农大牛樟与其余9个牛樟种质相比,农大牛樟与漳浦牛樟的遗传相似系数(0.755)最大。

A.UBC895;B.UBC900。1~10分别为:农大牛樟、南安牛樟、永春牛樟1、永春牛樟2、德化牛樟、南靖牛樟、漳浦牛樟、林场牛樟、永安牛樟1、永安牛樟2。
表5 10个牛樟种质间的遗传相似系数(SRAP)
Table 5 Genetic similarity coefficient of 10C.kanehiraebased on SRAP amplification profiles

牛樟农大南安永春1永春2德化南靖漳浦林场永安1永安2农大1.000南安0.3821.000永春10.3550.8731.000永春20.4950.7050.6501.000德化0.3640.8450.8820.6771.000南靖0.4140.6860.6770.7820.7051.000漳浦0.7550.4450.4360.4680.4090.4051.000林场0.4360.7180.6730.7860.6730.8320.4821.000永安10.3680.6230.6590.5910.6320.6270.4860.6231.00永安20.3730.7000.6550.7770.6910.8230.4270.8360.6591.00

表6 10个牛樟种质间的遗传相似系数(ISSR)
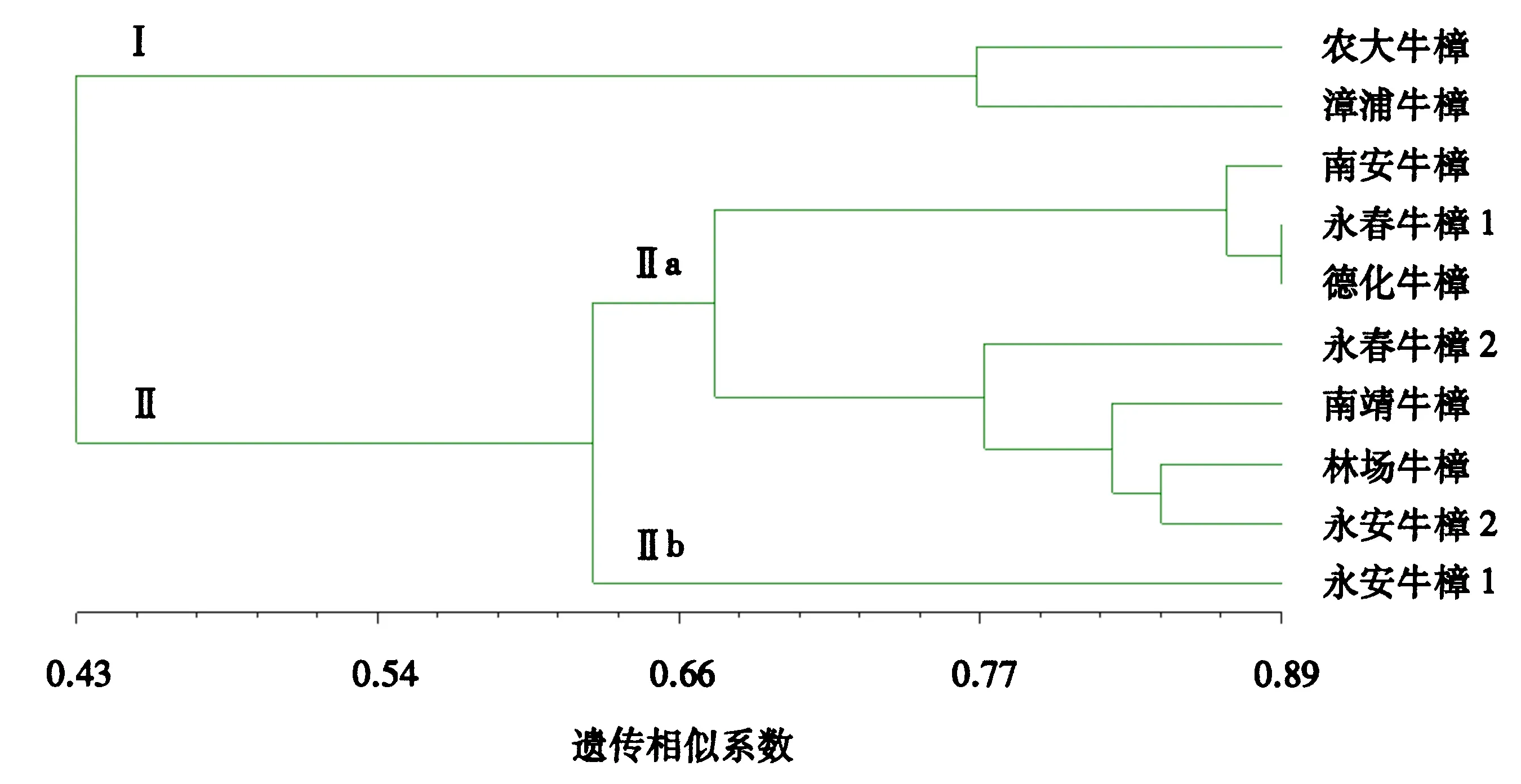
图3 10个牛樟种质的SRAP聚类图

图4 10个牛樟种质的ISSR聚类图
(2)ISSR标记得到的10个牛樟种质间的遗传相似系数变化范围在0.371~0.814之间,平均相似系数为0.606。永春牛樟2和漳浦牛樟的遗传相似系数(0.371)最小,南安牛樟和德化牛樟的遗传相似系数(0.814)最大。将农大牛樟与其余9个牛樟种质相比,农大牛樟与漳浦牛樟的遗传相似系数(0.743)最大。将SRAP与ISSR数据整合得到的遗传相似系数变化范围在0.412~0.847之间,平均相似系数为0.603,所得结果均与SRAP一致。
2.4.2 聚类分析 SRAP和ISSR分子标记的聚类分析图略有不同,具体表现为SRAP将永安牛樟1单独分在Ⅱb类,而ISSR将林场牛樟单独分在Ⅱb类。将SRAP与ISSR数据整合得到的聚类分析结果与SRAP的结果更具有一致性,除了林场牛樟和永安牛樟2顺序稍有不同,其他分类几乎相同。
利用Mantel检验SRAP、ISSR和SRAP+ISSR三者相似系数的相关性,SRAP与ISSR之间的相关系数为0.864;SRAP与SRAP+ISSR之间的相关系数为0.977;ISSR与SRAP+ISSR之间的相关系数为0.952。以上表明,SRAP和ISSR遗传相似系数都与SRAP+ISSR数据整合得到的遗传相似系数的相关性较高,SRAP标记更接近于两标记的综合遗传多样性。
3 讨论
遗传相似系数对鉴别牛樟种质有着重要意义。本研究表明,SRAP分析中农大牛樟与漳浦牛樟的遗传相似系数为0.755,与其余8个种质遗传相似系数均低于0.500。ISSR分析中,农大牛樟与漳浦牛樟的遗传相似系数为0.743,与其他牛樟均低于0.530。农大牛樟与漳浦牛樟已鉴定为台湾牛樟[8,17],本研究结果也证明了两者为同一种源。
本研究通过SRAP与ISSR分子标记技术将10个牛樟种质完全鉴别出来,说明这两种分子标记技术对植物的鉴别均具有一定的可靠性。两者也存在一定的差异性,由于SRAP分子标记是对开放阅读框进行特异性的扩增[18],而ISSR分子标记检测的是位于反向排列的简单重复序列间的基因组DNA片段[19]。SRAP、ISSR均与SRAP+ISSR相关性较强,说明利用2种或2种以上的分子标记相结合,具有较高的可信性和一致性[20]。SRAP+ISSR的聚类分析结果与SRAP更具一致性,除林场牛樟和永安牛樟2顺序稍有不同,其他分类几乎相同。因此,在经济条件有限的情况下,仅使用SRAP分子标记便可达到良好的鉴别效果。
目前关于牛樟分子标记技术体系的研究甚少,本文首次采用SRAP与ISSR分子标记鉴定牛樟种质,为有效鉴别牛樟与杂樟奠定基础。
