基于SSR分子标记的药用黄芪遗传多样性与遗传结构分析
2019-11-07刘亚令耿雅萍解潇冬张鹏飞
刘亚令,耿雅萍,解潇冬,王 芳,张鹏飞
(1. 山西农业大学生命科学学院,山西 太谷,030801; 2. 山西农业大学园艺学院,山西 太谷,030801)
药用黄芪(AstragaliRadix)为豆科植物蒙古黄芪(Astragalusmembranaceus(Fisch.) Bge.var.mongholicus(Beg.) Hsiao)与膜荚黄芪(Astragalusmembranaceus(Fisch.) Bge.)的干燥根[1]。其中野生蒙古黄芪主要分布于山西、内蒙古、甘肃、青海等高海拔、半干旱地区,道地药材产区以山西浑源、天镇等北部地区产的蒙古黄芪为优,其次是内蒙古的固阳县、武川县等地的蒙古黄芪[2]。野生膜荚黄芪不为主流商品,在中国西南、西北地区以及东北地区的黑龙江、辽宁、吉林地区均有分布[3]。黄芪味甘、性温,具有补气、利水、排毒等功效,为常用大宗药材之一,已拥有2000多年的药用历史,是中医方剂中常用的补气药物[4]。目前黄芪不仅用于药用,还在食疗及保健的开发和利用方面也得到了广泛应用[5]。但随着黄芪资源市场需求量的增大,造成近几年野生黄芪的过度采挖,野生黄芪资源几近枯竭,大面积的成规模的野生黄芪分布已十分少见,大多只是零星分布[2],人工栽培黄芪成为了市场上的主力军,但人工栽培黄芪种质资源混杂,规范化种植管理和生产水平较低,常会造成其质量及药用疗效的不稳定,因此亟需进行规范化育种以保护黄芪种质资源。研究遗传多样性有助于人们更清楚地理解生物多样性的起源和进化,并为植物分类提供一定的理论依据,进而为植物育种和保护策略的制定奠定基础[6-7]。
目前,随着分子标记技术的发展,多种分子标记已被广泛的用于植物遗传多样性分析[8-9]、亲缘关系鉴定[10]、核心种质构建[11]、遗传图谱的构建[12]等,其中简单重复序列(Simple Sequence Repeat,SSR)分子标记为共显性标记,因其具有操作方便、重复性好、多态性高等优点[13],成为理想有效的分子标记之一,现已经成功应用在甘草(GlycyrrhizaL.)[14-15]、党参(Codonopsis)[16]、黄芩(Scutellariabaicalensis)[17]等多种药用植物的遗传多样性研究及育种中。
因此,本试验利用SSR分子标记对黄芪主产区山西、内蒙、吉林不同产地的两种药用黄芪进行遗传多样性及居群遗传结构分析,了解不同居群不同物种药用黄芪的遗传多样性水平、遗传分化、变异程度以及居群结构组成,为今后黄芪种质选育利用及保护策略的制定提供一定的参考。
1 材料与方法
1.1 材料的收集
本试验共采集了380份药用黄芪材料,其中蒙古黄芪285份,膜荚黄芪85份,涵盖山西(A)、内蒙古(B)、辽宁(C)、吉林(D)、黑龙江(E)5个省份17个产地共22个自然居群,每个居群进行GPS定位,并对收集到的每个样本嫩叶保存于装有硅胶的自封袋中,带回实验室进行DNA提取。地理分布图及采样详细信息见图1及表1。

图1 黄芪采样地理分布图
1.2 基因组总DNA提取
采用十六烷基三甲基溴化铵(hexadecyltrimethylammonium bromide,CTAB)法[18]对所有居群中的每个单株样本材料分别进行基因组总DNA的提取,利用核酸蛋白检测仪及琼脂糖凝胶电泳对所提取的DNA浓度及质量进行检测,将DNA终浓度为调至50 ng·ul-1,并置于—20℃保存以备后用。

表1 黄芪材料来源信息
1.3 PCR扩增及检测
本实验根据SSR通用性特征,选用本实验组所开发的同科植物甘草的64对SSR引物[19],进行多态性的筛选,最终确定10对多态性高、重复性好的SSR引物用于试验(表2),引物合成由上海生工生物工程有限公司完成。PCR扩增体系参照刘亚令前期研究[20],扩增产物进行Fragment AnalyzerTM全自动毛细管电泳检测。

表2 SSR引物序列信息
1.4 SSR数据分析
对毛细管电泳检测结果进行扩增片段大小的判读并记录。利用PowerMarker V3.25软件[21]计算SSR位点的多态信息含量(PIC),利用GenAlEx 6.503软件[22]进行遗传多样性及遗传分化分析:主要参数有等位基因数(Na)、有效等位基因数(Ne)、Shannon’s信息指数(I)、Nei’s遗传多样性(H)、居群内近交系数(Fis)、总居群近交系数(Fit)、遗传分化系数(Fst)、基因流(Nm)、期望杂合度(He)、观测杂合度(Ho);利用GenAlEx 6.503对居群内及居群间分子方差检验(AMOVA)及主坐标分析(PCoA);利用MEGA 7.0软件[23]基于居群间Nei’s遗传距离[24]构建UPGMA聚类树;使用Structure V2.3.4[25]软件分析居群的遗传结构,其中群体数目(K)设为2~10,对每个K值模拟运算10次,设不作数迭代(length of burn-in period)开始时的马尔科夫链蒙特卡洛(markov chain monte carlo,MCMC)为10 000次,不作数迭代后的MCMC为100 000次,将Structure的运行结果导入在线程序STRUCTURE HARVESTER(http://taylor0.biology.ucla.edu/structureharvest/)进行最佳群体数的预测[26]。
2 结果与分析
2.1 SSR扩增产物的多态性
本试验利用筛选出的10对SSR引物对380个黄芪样本的基因组DNA进行SSR-PCR扩增,通过毛细管电泳检测,扩增结果在150~500 bp之间(图2)。对黄芪10对SSR位点的遗传多样性进行分析,其结果如表3所示:10对引物均具有较高的多态性,其多态信息含量范围(PIC)为0.698(SSR4)~0.930(SSR6),平均为0.827;等位基因数(Na)为5.455(SSR1)~11.182(SSR6,SSR9),平均为7.818;有效等位基因数(Ne)为3.063~6.527,平均为4.153;Shannon’s信息指数(I)为1.262(SSR2)~2.040(SSR9),平均值为1.573;Nei’s遗传多样性指数(H)为0.622(SSR2)~0.820(SSR9),均值为0.706,以上结果均表明筛选的10对SSR引物具有较高的多态信息含量。
2.2 物种遗传多样性分析
在物种水平上,分别对蒙古黄芪与膜荚黄芪进行遗传多样性分析(表4),结果显示:蒙古黄芪与膜荚黄芪Shannon’s信息指数(I)分别为I=2.241,I=1.982,平均为2.112;Nei’s遗传多样性指数(H)分别为H=0.804,H=0.757,均值为0.781,总体来说两种黄芪具有较高的遗传多样性,其中蒙古黄芪的遗传多样性高于膜荚黄芪的遗传多样性。此外蒙古黄芪、膜荚黄芪的居群观测杂合度均小于期望杂合度,且固定指数F均大于零,表明两种黄芪居群存在一定的杂合子缺陷。
注:等位基因数(Na)、有效等位基因数(Ne)、Shannon’s信息指数(I)、Nei’s遗传多样性(H)、期望杂合度(He)、观测杂合度(Ho)、多态信息含量(PIC)
Note:Na:Average allele number,Ne:Average effective allele number,I:Average shannon′s information index,H:Average Nei′s genetic diversity index,He:Average expected heterozygosity,Ho:Average observed heterozygosity,PIC:Polymorphism information content
注:等位基因数(Na)、有效等位基因数(Ne)、Shannon’s信息指数(I)、Nei’s遗传多样性(H)、观测杂合度(Ho)、期望杂合度(He)、固定指数(F)
Note:Na:Average allele number,Ne:Average effective allele number,I:Average shannon's information index,H:Average Nei's genetic diversity index,Ho:Average observed heterozygosity,He:Average expected heterozygosity,F:Fixation index
2.3 物种遗传分化分析
分别对两种黄芪进行遗传分化分析(表5),结果显示:蒙古黄芪与膜荚黄芪居群内近交系数(Fis)分别为0.350±0.132,0.387±0.141;总居群近交系数(Fit)分别为0.425±0.122,0.421±0.133;居群间遗传分化系数(Fst)分别为0.134±0.016,0.058±0.006,表明蒙古黄芪居群间相比膜荚黄芪居群间具有较大程度的遗传分化,而两种黄芪种内遗传分化系数(Fst)分别为0.134±0.016,0.058±0.006,表明蒙古黄芪居群内的遗传分化(13.4%)高于膜荚黄芪居群内的遗传分化(5.8%);此外,对两种黄芪22个居群总的遗传分化分析得到居群间的遗传分化系数(Fst)为0.154±0.020,表明黄芪遗传分化有84.6%来自居群内,有15.4%来自居群间;黄芪居群总的基因流Nm为1.719(Nm>1),表明黄芪居群间基因流处于较高水平。

表5 黄芪种遗传分化分析
注:居群内近交系数(Fis)、总居群近交系数(Fit)、遗传分化系数(Fst)、基因流(Nm)
Note:Fis:Inbreeding coefficient within population,Fit:Inbreeding coefficient in total population,Fst:Genetic differentiation coefficient,Nm:Gene flow
2.4 居群聚类分析及Mantel检测
利用MEGA 7.0软件基于Nei’s遗传距离对黄芪22个居群进行了UPGMA聚类分析(图3),结果显示:蒙古黄芪与膜荚黄芪首先按物种在0.92处分为两大类,其中蒙古黄芪这一类中在0.65处又以地域特征分为两组,第一组中主要是来自内蒙古地区的样本,第二组则主要是来自山西地区的样本。

图3 基于Nei’s遗传距离的黄芪物种22个居群UPGMA聚类分析
2.3 居群主坐标分析
主坐标分析(Principal Coordinates Analysis,PCoA)是基于居群间遗传距离得到的一系列的特征值和特征向量进行排序后,绘制的二维或三维坐标图,从而研究数据相似性或差异性的一种可视化方法。为了进一步了解黄芪不同居群间在遗传上的差异及相似性,本试验基于Nei’s遗传距离对黄芪22个居群进行PCoA主坐标分析,结果得出占总变异贡献率较多的三个主坐标特征值共为69.59%,其中第一主坐标占总变异的36.5%,第二主坐标解释了总变异的22%,第三主坐标解释了总变异的11.09%。二维坐标图(图4)结果显示:黄芪22居群被分为两大组,其中,膜荚黄芪5个居群分为一组,蒙古黄芪的17个居群分为另一大组,这与UPGMA聚类结果一致。对其前三个主坐标作三维主坐标分析图(图5),可以看出其分类结果与二维主坐标分析结果一致,但在二维的基础上增加了第三主坐标,可以从空间位置上更直观的了解22个居群间亲缘关系的远近。
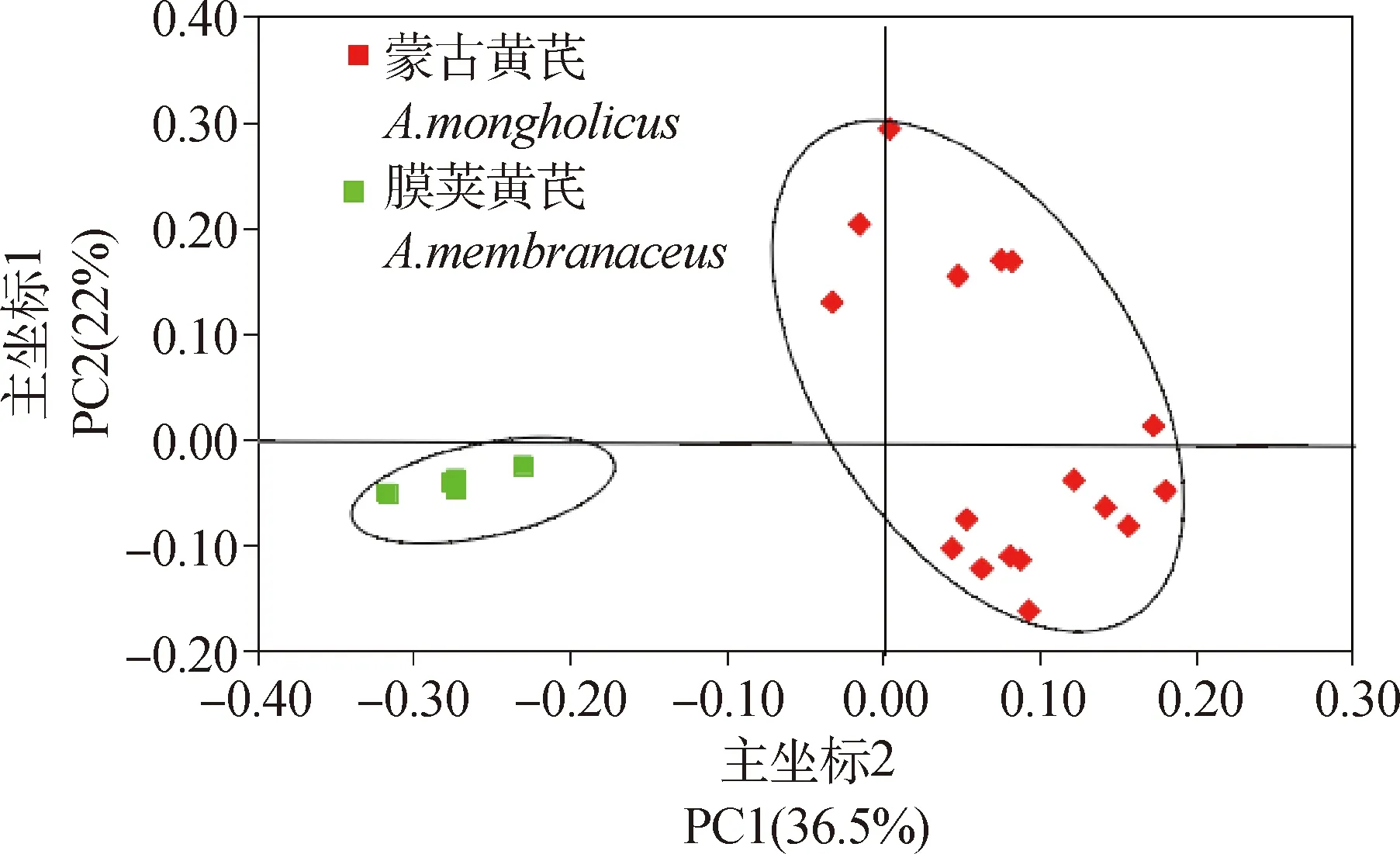
图4 黄芪22居群主坐标分析的二维散点图

图5 黄芪22居群主坐标分析的三维散点图
2.3 居群遗传变异分析
遗传变异水平能够在一定程度上反映群体在面对不同气候及生存环境下的适应能力。本试验利用GenAlEx 6.503软件对蒙古黄芪及膜荚黄芪居群间、居群内的遗传变异进行分子方差分析(Analysis of Molecular Variance,AMOVA)(表6),结果得出:黄芪种间遗传变异为9%,种内居群间的遗传变异为7%,居群内的遗传变异为83%,表明遗传变异主要发生在居群内。这与其遗传分化分析结果一致(表4),即居群间的遗传分化(15.4%)小于居群内的遗传分化(84.6%)。
2.4 居群遗传结构分析
利用STRUCTURE软件分析黄芪22个居群遗传结构(图6),其中将分组数K设为2-10,每个K值重复10次,并对其作K与Δ K的关系图(图6-A),当Δ K达到最大值时(Δ K=737.218),K=3,表明可以将黄芪22个居群分为3个亚群(图6-B),其中膜荚黄芪为一个亚群(绿色),蒙古黄芪被分为两个亚群,一亚群主要包括内蒙古地区的的7个居群以及山西地区的3个居群(红色),第二亚群为山西地区的4个居群(GPJT,GZQL,GXXT,GDXY)及内蒙的两个居群(GHDM,GMZL)(蓝色)。此结果与UPGMA聚类及主坐标分析PCoA结果一致。

表6 黄芪种间及居群分子方差分析(AMOVA)

图6 黄芪22个居群的遗传结构分析
3 讨论
多态信息含量(PIC)是衡量微卫星位点多态性高低的较好指标,一般来说,当PIC>0.5时,具有高度多态性;当0.25 评价药用黄芪种质资源的遗传多样性,对充分利用开发及保护黄芪种质具有重要意义,前人曾利用不同的分子标记进行了不同产地黄芪的遗传多样性分析,如张茹[28]利用ISSR对14个产地共183个黄芪样进行遗传多样性分析,得出蒙古黄芪的遗传多样性(H=0.336,I=0.497)略高于膜荚黄芪(H=0.311,I=0.466);王傲[29]利用SSR分子标记对162个蒙古黄芪与186个膜荚黄芪的遗传多样性结果显示:蒙古黄芪遗传多样性(H=0.822)高于膜荚黄芪(H=0.689);王含彦[30]利用RAPD技术对来自四川、河北、山西、甘肃四个省份的共15份黄芪的遗传多样性得出平均Shannon’s信息指数(I)为0.225。本试验对黄芪主产区山西、内蒙、吉林等地区22个居群共380个样进行遗传多样性分析,结果同样得出黄芪具有丰富的遗传多样性(H=2.112,I=0.781),蒙古黄芪的遗传多样性(I=2.241,H=0.804)要高于膜荚黄芪遗传多样性(I=1.982,H=0.757),表明了蒙古黄芪作为膜荚黄芪的变种,在不断地生长进化中,积累了丰富的等位变异基因,比膜荚黄芪有较高的环境适应能力,具有丰富的遗传育种资源及进化潜力。而本试验得到的黄芪遗传多样性相比前人的研究数值都较高,可能是由于本试验选用的试验材料多,产地跨域广,使其具有高的遗传多样性水平。 本试验基于Nei’s遗传距离,采用非加权算数平均法(UPGMA法)进行了物种(居群)间聚类分析,结果得出药材黄芪首先是按物种明显划分,此聚类结果与对其表型观测结果一致,而在同种物种中居群间的聚类较混杂,没有完全的按地理来源进行聚类,如蒙古黄芪中来自山西的3个居群(GSJP,GLCW,GPJY)与来自内蒙的7个居群聚为一类,另外来自山西的4个居群(GPJT,GZQL,GXXT,GDXY)及内蒙的2个居群(GHDM,GMZL)聚为一类。可能是因为内蒙及山西地区都是药材黄芪的道地产区,由于人为原因等使两地区种质间会发生一定的种质交流,且两地间的地理环境差异较小,在生长过程中可能会发生同等分化及变异。 蒙古黄芪及膜荚黄芪物种间(内)、居群间(内)进行分子方差分析结果显示,遗传变异主要发生在居群内(83%)。根据在黄芪传粉特性的研究中表明,蒙古黄芪和膜荚黄芪均为自交不亲和的异花授粉植物[31-32],这一特性使得黄芪在生长繁育的过程中,居使得群内部发生较多的基因交流及变异,因此,这可能是导致黄芪遗传变异主要发生在居群内的主要原因。 本试验所选用的10对黄芪同科植物的SSR引物能够有效的显示黄芪基因多态性,可用于黄芪遗传多样性研究。对蒙古黄芪、膜荚黄芪22个居群共380个样本进行遗传多样性与遗传结构分析,结果得出黄芪具有较高程度的遗传多样性水平,其中蒙古黄芪遗传多样性水平要高于膜荚黄芪,遗传分化程度高于膜荚黄芪。两种黄芪的遗传背景相对单一,且遗传变异主要发生在居群内;UPGMA聚类分析及STRUCTURE群体遗传分析可将其分为3组,分类结果具有一定的地域性。以上研究结果对药用黄芪种质资源的有效利用、遗传多样性的保护及育种开发优质黄芪种质资源提供一定的理论基础。4 结论
