兰格湖裸鲤肌肉生长抑制素基因(MSTN)的克隆及生物信息学分析
2019-10-24张驰刘飞王万良周建设
张驰,刘飞,王万良,周建设
(西藏自治区农牧科学院水产科学研究所,西藏 拉萨 850000)
兰格湖裸鲤Gymnocypris chui 隶属鲤形目Cypriniformes鲤科 Cyprinidae 裂腹鱼亚科Schizothoracinae,裸鲤属Gymnocypris,主要分布于青藏高原海拔4 300m 以上的浪措、公珠措和玛旁雍措等湖泊,极其适应于青藏高原水域的生物和非生物条件,是青藏高原鱼类三大类群之一[1]。兰格湖裸鲤以底栖动植物为食,在青藏高原淡水湖泊生态系统的食物链中具有重要的地位。同其他高原鱼类相似,兰格湖裸鲤具有生长期短、生长缓慢、性成熟迟、以及繁殖力低等典型生物学特性。目前,关于兰格湖裸鲤的研究主要集中在形态分类,对其功能基因的研究属于空白。
肌肉生长抑制素(Myostatin,MSTN)又称转化生长因子(growth differentiation factor 8,GDF-8),是转化生长因子(Transforming growth factor β,TGF-β)超家族成员之一,对骨骼肌的生长具有负调控作用。Mcpherron 等[2]首先从小鼠Mus musculus 骨骼肌cDNA 文库中克隆得到MSTN 基因,缺失该基因的小鼠与正常小鼠体重存在明显差异。Grobet 等[3-5]发现,牛Bos Taurus MSTN 基因外显子存在自然缺失与变异,导致MSTN 活性消失,使肌肉增生。随后Alexander 等[6]克隆出了人Homo sapiens、大鼠Rattus norvegicus、猪Sus scrofa、绵羊Ovis aries、鸡Gallus gallus、狒狒Papio hamadryas 以及斑马鱼Danio rerio 等的cDNA 序列,发现脊椎动物不同物种间MSTN 基因非常保守。
目前越来越多鱼类[7-16]的MSTN 基因已被克隆出来,与其他动物类似,鱼类MSTN 基因都是由3个编码分泌性N-端信号序列的外显子和2 个内含子组成;内含子-外显子边界都显示了保守性,符合GT-AG 规则。相关研究也发现:脊椎动物中MSTN 有不同的转录本。Roberts 和Goetz[7]首次在大马哈鱼Oncorhynchus keta 不同组织里发现MSTN蛋白的2 个异构体,二者在核酸水平的同源性为92%。Tong 等[8]在青海湖裸鲤Gymnocypris przewalskii 不同组织中也发现两种转录本,各个组织广泛表达MSTN1 型转录本,而MSTN2 型转录本仅在肌肉和脑组织中表达,两种转录产物在胚胎发育早期阶段表达水平也有差异。
鉴于MSTN 基因对动物肌肉生长发育的调控作用及其潜在的应用价值,本研究通过克隆MSTN基因,并进行生物信息学分析,探讨兰格湖裸鲤MSTN 基因结构组成以及种间的遗传分化,以期为高原湖泊鱼类的遗传资源保护、开发与利用提供分子遗传学基础。
1 材料与方法
1.1 材料
兰格湖裸鲤采集于西藏自治区日喀则市昂仁县浪措(29°12'33.45''N,87°23'5.46''E),又名兰格湖,位于西藏自治区昂仁县东南部。湖泊长轴呈东西向延伸,湖面海拔4 300m,长6 900m,最大宽2 300m,平均宽1 750m,面积约12.1km2。
2017 年7 月—2018 年5 月,以三层刺网(网目5cm)于浪措近岸水域采集兰格湖裸鲤样本15 尾,取鱼鳍样品,置于5mL 离心管中,75%酒精保存运输,-80℃冰箱保存备用。
图1 浪措的地理位置Fig.1 The geographical position of the Langcuo
1.2 方法
1.2.1 DNA 的提取
取尾鳍样品约20mg,剪碎或者置于有液氮的研钵中磨碎,然后置于1.5mL 离心管中。用OMEGA DNA tissue kit 试剂盒,按照说明书步骤提取总DNA。
1.2.2 MSTN 基因PCR 扩增和测序
根据GenBank 注释的青海湖裸鲤的MSTN 基因序列(序列号:KP277104),设计4 对引物(表1),以基因组DNA 为模板扩增兰格湖裸鲤MSTN 基因序列,PCR 扩增引物由上海生工合成。PCR 12.5μL反应体系为6×Master Mix 6.25μL,dd H2O 4.75μL,上下游引物各0.25μL,DNA 模板1μL。PCR 扩增程序为94℃预变 性5min;94℃变性30s;62℃退火30s;72℃延伸30s;共计35 个循环;最后72℃继续延伸5min。PCR 产物经1.5%琼脂糖凝胶电泳采用紫外成像仪拍照。

表1 兰格湖裸鲤MSTN 基因克隆及分析引物Tab.1 Primers for MSTN clone and analysis in Gymnocypris chui
1.2.3 PCR 产物的回收、连接、转化和测序
用OMEGA Gel Extraction Kit DNA 凝胶回收试剂盒回收纯化PCR 产物,具体步骤按照说明书操作。回收的DNA 在T4 连接酶的作用下,16℃4h,连接到T-载体pMD18-T 上(TaKaRa 公司)。连接产物转化后筛选阳性克隆,提取质粒并送至生工生物工程(上海)有限公司测序。
1.2.4 数据分析
采用DNAMAN 8.0(https://www.lynnon.com)软件进行基因序列拼接;在线使用Blastx 程序(https://blast.ncbi.nlm.nih.gov/Blast.cgi)进行基因序列相似性搜索;MEGA7.0 软件构建邻接(Neighbor-joining,NJ)系统发育树[9];DNAstar7.1 进行序列组成及遗传变异位点分析(https://www.dnastar.com)。
2 结果与分析
2.1 兰格湖裸鲤MSTN 基因PCR 扩增结果
从兰格湖裸鲤鳍条样本中提取出完整的基因组DNA,用MSTN 基因4 对引物进行扩增,得到特异性条带,产物电泳结果如图2 所示。将测序所得序列比对后,得到MSTN-1a 的长度为1101bp,MSTN-1b 为1 179bp,MSTN-2a 为1 111bp,MSTN-2b 为908bp,4 条序列均去除了两端引物且与目的片段大小基本相同。
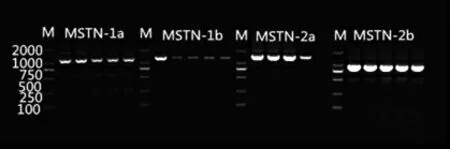
图2 PCR 扩增目的条带电泳图Fig.2 Electrophoresis map of amplification of the purpose bands by PCR
2.2 兰格湖裸鲤MSTN 基因结构
利用DNAMAN 软件对4 对引物的测序结果进行序列拼接,获得长为3 526bp 的兰格湖裸鲤MSTN 基因序列,并分析了其碱基组成A(29.6%);C(21.7%);G(20.6%);T(28.1%)。采用比较基因组方法与青海湖裸鲤MSTN 基因的mRNA 序列进行比较,确定兰格湖裸鲤MSTN 基因结构。兰格湖裸鲤MSTN 基因序列,包含该基因的全部外显子和内含子序列以及部分5'-侧翼区、3'-侧翼区序列。兰格湖裸鲤MSTN 基因结构与人、牛、山羊、小鼠、鸡等各物种相同,均由3 个外显子和2 个内含子组成,内含子剪接符合GT-AG 法则。其中,3 个外显子大小分别为376bp、71bp 和381bp,外显子Ⅰ包含起始密码子ATG,外显子Ⅲ含有终止密码子TGA;内含子Ⅰ和内含子Ⅱ大小分别为665bp 和912bp。使用DNASTAR 软件对兰格湖裸鲤MSTN 基因与其他物种的MSTN 基因序列进行比较分析,确定该基因CDS 序列全长为1 128bp,编码375 个氨基酸,Gen-Bank 序列号MK497253。

表2 物种信息表Tab.2 Summary of species used in this study
2.3 兰格湖裸鲤MSTN 基因CDS 序列种间相似性分析
将兰格湖裸鲤MSTN 基因全编码区核苷酸序列与GenBank 数据库中的青海湖裸鲤、黄河裸裂尻、鲤、金线鲃、斑马鱼、大西洋鲑、兰州鲇、人、小鼠、牦牛和猪等11 个物种相应基因序列进行序列比对分析,计算不同物种编码序列间的相似性(表2)。由表2 可知,兰格湖裸鲤与青海湖裸鲤、黄河裸裂尻间相似性最高,均为98.9%。两种裸鲤MSTN 基因的基因编码区中存在12 个差异位点,其中5 个位点(11bp、236bp、795bp、838bp 和1 011bp)处存在AG 碱基转换;5 个位点(432bp、477bp、924bp、942bp和1 065bp)发生了TC 碱基转换;两个位点(614bp和930bp)处发生了GT 碱基颠换。这些碱基变异导致这两种裸鲤该基因的4 个位点编码蛋白产生差异。除了与同为裂腹鱼亚科的两种鱼最为相似外,兰格湖裸鲤与鲤、大鳞副泥鳅、斑马鱼、大西洋鲑、兰州鲇、人、小鼠、牦牛和猪的种间相似性依次为97.0%、93.1%、89.8%、80.5%、77.5%、65.1%、64.9%、64.6%和63.6%,结果与生物进化的历程相一致,说明该基因在物种间保守性很强,可能是影响生物进化、生长发育的重要因子。
2.4 兰格湖裸鲤与其他5 种鲤科鱼类MSTN 基因氨基酸序列间比较分析
利用DNASTAR 软件将获得的兰格湖裸鲤MSTN 编码序列转换成氨基酸序列,并与NCBI 数据库的青海湖裸鲤、黄河裸裂尻、鲤、斑马鱼和大鳞副泥鳅的氨基酸序列进行排列比较(图3),计算氨基酸序列之间的相似性,分别为99.2%、98.9%、96.5%、94.7%和93.1%,裂腹鱼亚科的3 种鱼类氨基酸序列相似性明显高于其他物种。
2.5 兰格湖裸鲤MSTN 基因系统发育分析
基于兰格湖裸鲤MSTN 编码序列和GenBank公布的其他物种的相应序列,通过MEGA7 基于最大似然法参数模型,用NJ 法构建了12 种脊椎动物MSTN 基因系统发育树(图4)。从图4 中可以看出,系统发育树将脊椎动物分成2 个主要族群,鱼类与哺乳动物分别聚成一支。兰格湖裸鲤等3 种裂腹鱼亚科鱼类的系统发育关系与其地理分布区系相一致。
3 讨论
MSTN 基因已受到动物遗传育种研究者的广泛关注,相继克隆了牛、羊、猪、鸡、大黄鱼Larimichthys crocea、罗非鱼Oreochromis niloticus[10-14]等多种经济动物的MSTN 基因,研究了该基因功能[15-17]。CRISPR/Cas9 系统是近几年研究较多的基因编辑工具,以MSTN 基因为靶位点,利用CRISPR/Cas9 基因编辑技术获得了猪、牛、羊、兔等多种基因编辑动物[18-21]。遗传修饰可以加速改良物种的生长性状、抗病性能和肉质品质,可以进行MSTN 基因的敲除或者人工突变,获得MSTN 基因修饰的鱼类新品种。
本研究克隆了兰格湖裸鲤的MSTN 基因,获得了该基因1 128bp 的CDS 全序列,通过比较基因组和生物信息学方法预测和分析MSTN 基因编码产物,为揭示兰格湖裸鲤MSTN 基因的遗传特性提供了有价值的理论依据。本研究获得的兰格湖裸鲤MSTN 基因结构与鲤科鱼类的结构基本一致,均包含3 个外显子和2 个内含子,编码区包含1 128 个核苷酸,共编码375 个氨基酸,其中前22 个氨基酸为MSTN 的信号肽。不同科鱼类物种间MSTN 基因转录产物差异较大,而在哺乳动物中MSTN 基因却相当保守,鱼类MSTN 基因的表达方式可能与哺乳类动物不同。
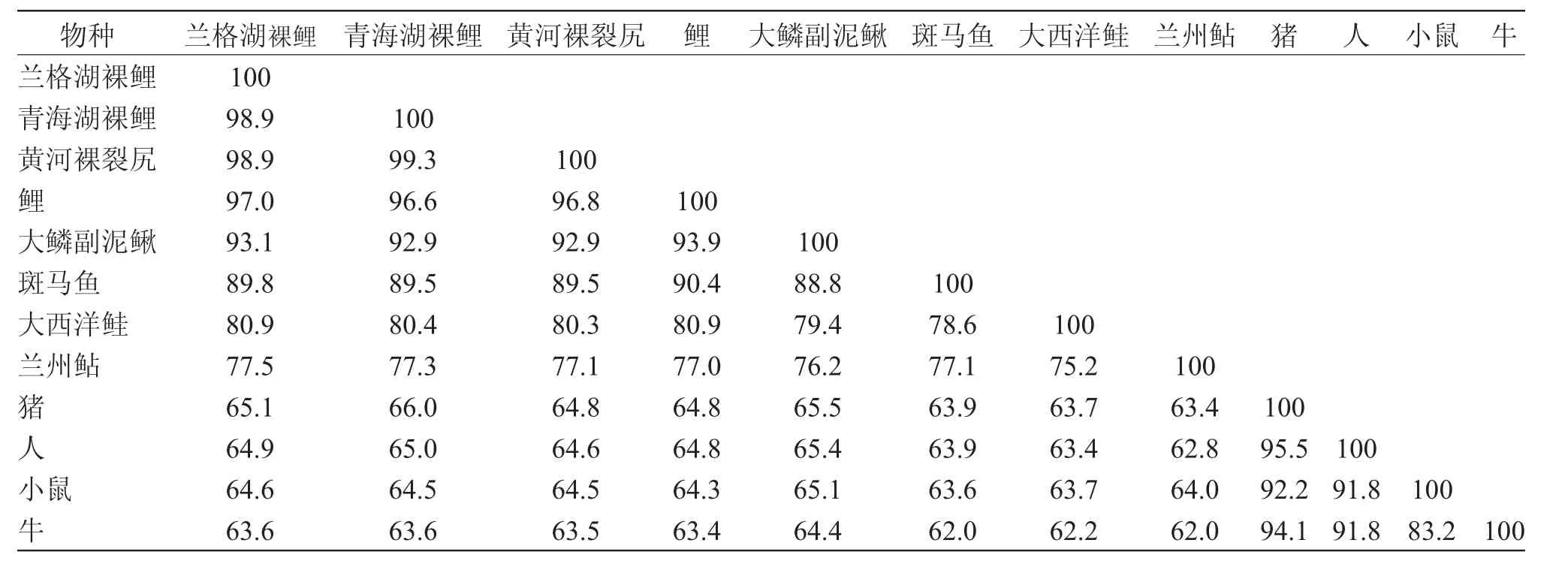
表3 兰格湖裸鲤与11 个物种间MSTN 基因核苷酸序列编码区相似性Tab.3 Homology of nucleotide sequences of encoding region of MSTN gene between Gymnocypris chui and other 11 species
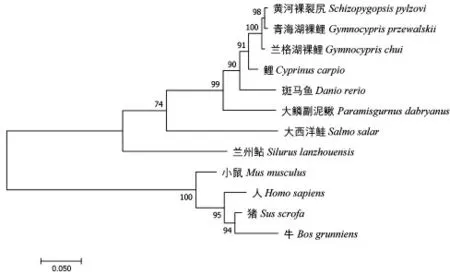
图4 基于MSTN 基因编码序列构建的部分脊椎动物系统发育树Fig.4 Phylogenetic tree of representative vertebrates based on MSTN CDS sequences
序列同源性分析发现,兰格湖裸鲤MSTN 与鲤科鱼类,尤其是裂腹鱼亚科的鱼类同源性较高,与鲇鱼形目鱼类的同源性次之,与哺乳动物的同源性最低,这与Song 等[22]的研究结果相一致。MSTN 基因种间分化明显,构建的系统发育树与传统动物进化分类相一致,不同物种的MSTN 序列同源性基本反应了它们的亲缘关系,符合物种的分子进化理论。
本研究成功克隆了兰格湖裸鲤MSTN 基因序列,分析其基因结构特点,为进一步开展兰格湖裸鲤MSTN 基因的表达调控、进化和多态性研究奠定基础。
