基于好氧甲烷氧化菌的反硝化效能及微生物群落研究
2019-10-23李彦澄杨娅男刘邓平
李彦澄,杨娅男,刘邓平,李 蕾,李 江
基于好氧甲烷氧化菌的反硝化效能及微生物群落研究
李彦澄*,杨娅男,刘邓平,李 蕾,李 江
(贵州大学资源与环境工程学院,贵州 贵阳 550025)
采用气体循环序批式生物膜反应器(gcSBBR),构建反硝化型甲烷好氧氧化(AME-D)系统.考察了进水氮负荷的影响,发现氮负荷为0.075kg/(m3·d)时,硝酸盐氮去除率达到98.93%,其反硝化速率为74.25mg/(L·d),系统的甲烷日平均消耗量为35.91%(初期为50%);扫描电子显微镜(SEM)分析结果显示,系统中的微生物主要以短杆菌(12~18 μm)为主,并存在少量的丝状菌(长150~200 μm);16S rRNA高通量测序结果显示,该系统中的甲烷氧化菌为、、和_ unclassified,反硝化菌为、、、和,其中主要的功能微生物为、和,系统对氮的去除是由好氧甲烷氧化菌与反硝化菌协同实现.此外,系统中存在大量以甲醇和甲基胺类物质为生长基质的_ uncultured(30.4%).
反硝化型甲烷好氧氧化(AME-D);甲烷氧化菌;反硝化;gcSBBR;功能微生物
甲烷氧化菌能以甲烷作为唯一的碳源和能源物质,广泛分布于自然环境中,如生活垃圾卫生填埋场[1]、泥炭沼泽[2]、煤矿坑[3]、污水处理厂污泥[4]、土壤[5]、水库与河流沉积物[6]和深海沉积物[7]等,在碳循环、氮循环和氧循环中起重要作用.相关研究发现,甲烷氧化菌在氧化甲烷的同时能实现氮的去除,该过程分为反硝化型甲烷好氧氧化(AME-D)[8]和反硝化型甲烷厌氧氧化(ANME-D)[9],如薛松等[10]在研究中构建出反硝化型甲烷厌氧氧化系统,发现甲烷转化率为34.7%,并对系统中的微生物进行了分析; Modin等[11]在膜生物反应器中成功富集了具有脱氮能力的甲烷氧化菌,培养过程中采用的气体为沼气,并得出了脱氮和甲烷利用之间的关系为0.25~0.36molN/molCH4,发现了生物膜表面的优势菌群为I型甲烷氧化菌. AME-D和 ANME-D过程均能有效实现氮的去除,但AME-D过程需要氧的参与.此外,反硝化型甲烷氧化能实现对甲烷的资源化利用,减少甲烷的排放,减缓甲烷排放导致的温室效应.由上述可知,反硝化型甲烷氧化具有显著的经济和环境效益,日益受到关注.
目前,关于反硝化型甲烷好氧氧化过程(AME- D)的作用机理存在两种解释:①反硝化过程由好氧甲烷氧化菌实现,相关研究发现,部分甲烷氧化菌的基因中含有脱氮基因、和等[12];②反硝化过程由好氧甲烷氧化菌和反硝化细菌协同实现,甲烷氧化菌氧化甲烷会产生中间产物,如甲醇、甲醛、乙酸盐和柠檬酸盐等,所产生的中间产物为反硝化细菌提供碳源[8].AME-D的反应过程可通过表1中的化学方程式进行描述.
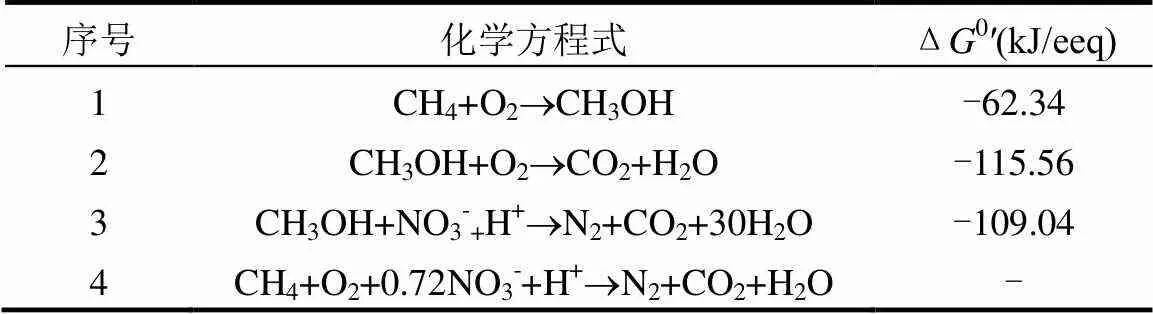
表1 反硝化型甲烷好氧氧化(AME-D) 化学方程式[8]
为了探究反硝化型甲烷好氧氧化过程(AME-D)的脱氮效能及功能微生物,研究采用气体循环序批式生物膜反应器(gcSBBR),构建反硝化型甲烷好氧氧化系统,考察了进水氮负荷的影响,采用气相色谱仪(GC)分析反应器中甲烷气体的变化,通过扫描电子显微镜(SEM)分析系统中微生物的形态,并采用16S rRNA高通量测序分析微生物种群结构,识别主要的好氧甲烷氧化菌与反硝化菌.研究结果可为甲烷与氮的有效控制提供理论依据,并能为脱氮工艺的改进提供技术支持.
1 材料与方法
1.1 试验装置
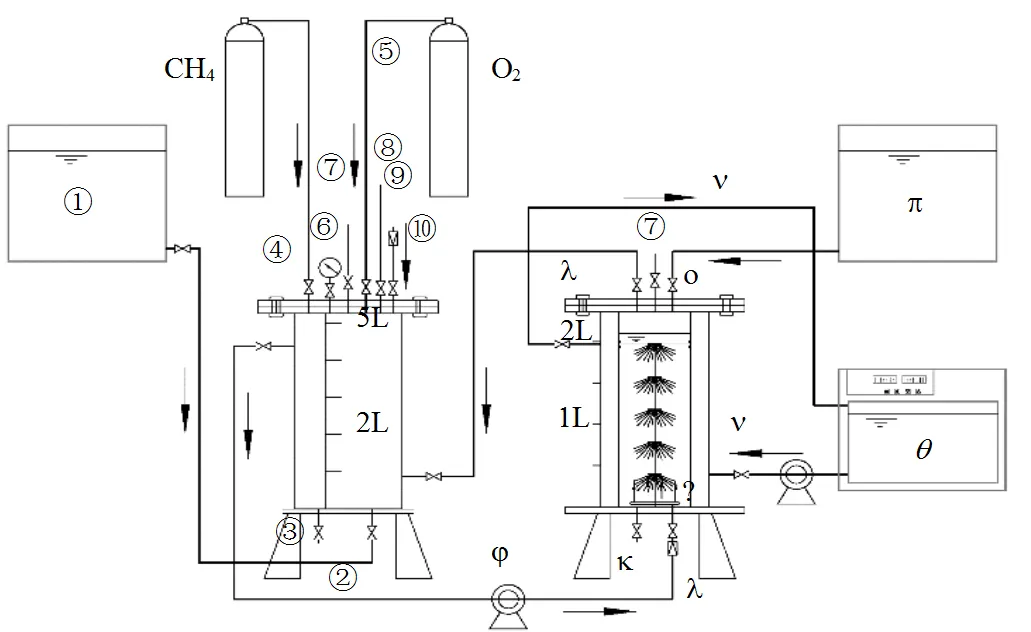
图1 气体循环的序批式生物膜反应器(gcSBBR)
① 水箱; ②进水; ③出水; ④ CH4进气口; ⑤ O2进气口; ⑥正-负压力表; ⑦通气口; ⑧取样口; ⑨空气进气口; ⑩单向阀;⑪蠕动泵;⑫出水及取样口;⑬气体循环管;⑭曝气头;⑮热水循环管;⑯NMS 培养液及废水进入口;⑰进水箱;恒温水浴锅
采用气体循环序批式生物膜反应器(gcSBBR),该试验装置由气罐(有效容积为5L)和生物膜反应器(有效容积为2.25L,纤维丝柔性填料,填充率为45%)两部分组成(图1),使反应器中同时形成厌氧、缺氧和好氧环境.气体在气罐和生物膜反应器之间通过蠕动泵进行循环,进水从反应器的顶部重力加入,通过循环热水控制温度.
1.2 试验水质
试验进水采用人工配水,其中氮污染物采用KNO3模拟,其他营养物质与Nitrite Mineral Salt (NMS)培养液[13]相同,其浓度分别为(mg/L): MgSO4·7H2O,1000;CaCl·2H2O,200;Fe-EDTA,0.38;Na2MoO4·H2O,0.26;FeSO4·7H2O,0.90;MnCl2·4H2O,0.02;CoCl2·6H2O,0.05;H3BO3,0.015;Na-EDTA,0.25;ZnSO4·7H2O,0.40;NiCl2·6H2O, 0.01;CuSO4·5H2O,1.25; Na2HPO4·12H2O,71.60;KH2PO4·7H2O,26.00.
1.3 试验方法
反应器接种污水处理厂浓缩池污泥,接种的干污泥量为8g/L,加入混合气体为甲烷:氧气=1:1,即甲烷比例为50%,控制反应器的温度为30℃,采用间歇式运行的方式,运行周期为“进水0.5h-反应23h-出水 0.5h”,排水比为1/2,进水的同时更新反应器中的混合气体.反应器运行初期的进水硝酸盐氮浓度为150mg/L(即氮负荷:0.075kg/(m3·d)),待反应器运行稳定后,调节进水硝酸盐氮浓度分别为300mg/L(即氮负荷:0.15kg/(m3·d))和500mg/L(即氮负荷:0.25kg/ (m3·d)).采集每个周期的进气和出气,采用气相色谱仪分析甲烷的含量,并检查出水的DO、pH值、硝酸盐氮和亚硝酸盐氮.待反应器运行稳定时,通过扫描电子显微镜分析微生物的形态,并对反应器中的微生物开展16S rRNA高通量测序分析.
1.4 分析方法
DO和pH值采用HACH便携式多参数数字化分析仪分析,硝酸盐氮的分析采用紫外分光光度法(实行)(HJ/T 346-2007),亚硝酸盐氮的分析采用N-(1-萘基)-乙二胺分光光度法[14].
甲烷气体通过气相色谱仪(GC-2010Plus, SHIMADZU, Japan)进行检测,采用的色谱柱型号为Rt-Q-BOND (30m × 0.53mm × 20μm),配置TCD检测器,采用氮气作为载气,总流量为36.9mL/min,进样量1μL[15].
扫描电子显微镜分析(SEM):取适量的生物膜,采用4%的多聚甲醛固定1h后,使用0.9%生理盐水清洗2次;再依次使用浓度分别为30%、50%、70%、80%、90%和100%的乙醇脱水处理;然后依次使用浓度分别为50%、70%、90%、95%和100%的叔丁醇进行置换处理;将样品放在样品托上镀金处理200s后,采用扫描电子显微镜(S-3400N, Hitachi, Japan)进行观察并拍照.
16S rRNA高通量测序分析:采集反应器中的微生物,立即在-40℃条件下保存,然后送至微生物分析公司进行16S rRNA基因的V3-V4之间的高变区测序,选用细菌通用引物338F(5’-ACTCCTACGG- GAGGCAGCAG-3’)和806R(5’-GGACTACHVGG- GTWTCTAAT-3’).微生物分析公司接收到样品后,提取微生物的DNA,提取的合格DNA进行PCR扩增和产物纯化,扩增反应体系为:5×FastPfu Buffer 4μL,2.5mmol/L dNTPs 2μL,引物(2.5mmol/L)各0.8μL,FastPfu聚合酶0.4μL,BSA 0.2μL,模板DNA 10ng,ddH2O补至20μL.采用ABI GeneAmp® 9700型PCR扩增仪,扩增程序为:95 ℃预变性3min,95 ℃变性30s,55 ℃退火30s,72 ℃延伸45s,27个循环, 72℃延伸10min.扩增结束后,采用2%琼脂糖凝胶电泳检测PCR产物,上样量为3μL.然后对PCR产物进行定量和均一化,构建Illumina平台文库,进行Illumina平台测序,然后对所得到的数据进行生物信息分析,包括OTU聚类、物种注释及分类、群落组成等分析.
2 结果与讨论
2.1 反硝化型甲烷好氧氧化系统构建
系统构建过程中对甲烷的消耗量如图2所示,在前20d内,系统对甲烷的消耗量呈缓慢增长趋势,从第21d开始,系统对甲烷的消耗量迅速增加,运行至第37d时,系统对甲烷的消耗量达到30.10%,从第38d开始,系统对甲烷的消耗量趋于稳定,反应器运行第37~98d的甲烷日平均消耗量为35.91%.由此可知,采用gcSBBR构建出甲烷氧化菌系统的时间约为37d,该系统构建时间与Pfluger等[16]的研究结果类似,Pfluger等采用流化床富集培养甲烷氧化菌,当运行至35d时,出现了大量的优势甲烷氧化菌spp. Strains.
如图2所示,当进水中硝酸盐氮的浓度升高至300mg/L[0.15kg/(m3·d)]时,系统对甲烷的日平均消耗量变为41.72%,而进水中硝酸盐氮的浓度升高至500mg/L[0.25kg/(m3·d)]时,系统对甲烷的日平均消耗量变为44.65%,说明系统的甲烷消耗量随氮负荷的升高而增加.通过分析系统出水的pH值和DO(表2),发现不同氮负荷条件下的pH值均呈中性,而DO随氮负荷的升高而有一定程度的降低,主要是由于系统对甲烷的消耗量增加后,在氧化甲烷的过程中消耗了更多的DO.
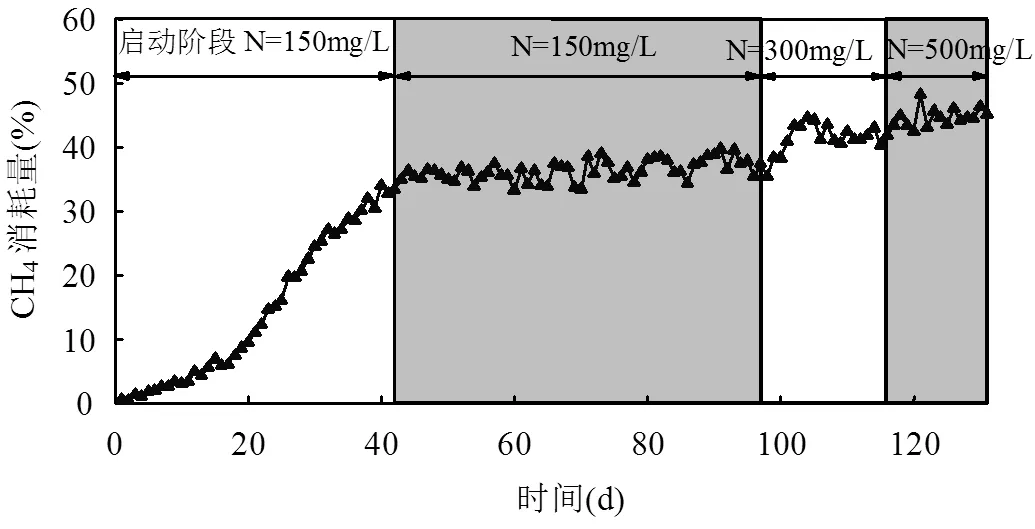
图2 甲烷消耗量日变化
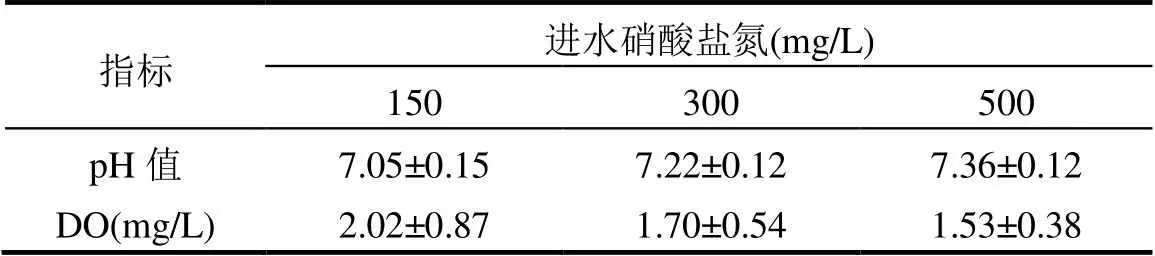
表2 系统出水的pH值和DO
不同氮负荷条件下系统出水中的总氮、硝酸盐氮和亚硝酸盐氮如图3所示,结果显示,随着氮负荷的增加,系统对硝酸盐氮的去除量逐渐增加,当氮负荷为0.25kg/(m3·d)时,硝酸盐氮去除量达到333.98mg/L,其最大反硝化速率为167.01mg/(L·d);随着氮负荷的增加,系统对硝酸盐氮去除率逐渐降低,当氮负荷为0.075kg/(m3·d)时,硝酸盐氮去除率达到98.93%,其反硝化速率为74.25mg/(L·d).相关研究如Sun等[17]采用膜生物反应器(MBfR)成功构建出好氧甲烷氧化-脱氮系统,发现进水硝酸盐氮为30mg/L时,硝酸盐氮去除率可以达到97%; Kampman等[18]采用膜生物反应器中成功培养出了具有脱氮能力的甲烷氧化菌,实现的最大脱氮容积速率为36mg/(L·d);周祥玉等[19]在序批式反应器研究了微氧条件下氮的去除,发现硝酸盐氮和亚硝酸盐氮的去除率分别为3.69mg/(L·d)和18.04mg/(L·d);卢培利等[20]研究了亚硝酸盐型厌氧甲烷氧化,实现的最大脱氮速率为73.10mg/(L·d).通过对比分析可知,相比已有研究所报道的系统,采用gcSBBR构建出的好氧甲烷氧化菌反硝化系统具有较高的反硝化脱氮速率和硝酸盐氮去除率,其原因可能为研究所采用的gcSBBR系统提供了一个甲烷与微生物充分接触的环境,使系统中富集了更多的功能微生物,提高了系统的脱氮速率.此外,系统运行稳定后,出水中存在一定的COD浓度,且不同氮负荷下的COD浓度相差不大,其平均COD浓度为35±4mg/L,说明出水中含有一定浓度的有机物,其原因可能为甲烷被微生物利用过程中产生的中间产物、酶或者脱落的菌体引起.

图3 不同硝酸盐氮浓度条件下反应器出水TN、NO3--N和NO2—N
2.2 微生物的形态分析

图4 生物膜的扫描电子显微镜(SEM)分析
由Semrau等[21]对甲烷氧化菌的综述可知,甲烷氧化菌的细胞形态存在椭圆状、球状、杆状、球杆状、梭状、弯曲球状、棒状和梨状等,也有的甲烷氧化菌为丝状[22].通过扫描电子显微镜(SEM)分析反应器中的微生物形态,如图4所示,系统中的微生物主要以短杆菌(12~18μm)为主,并存在少量的丝状菌(长150~200μm),在细菌周边还存在大量似胞外聚合物(EPS)[23]的物质.
2.3 微生物群落结构分析
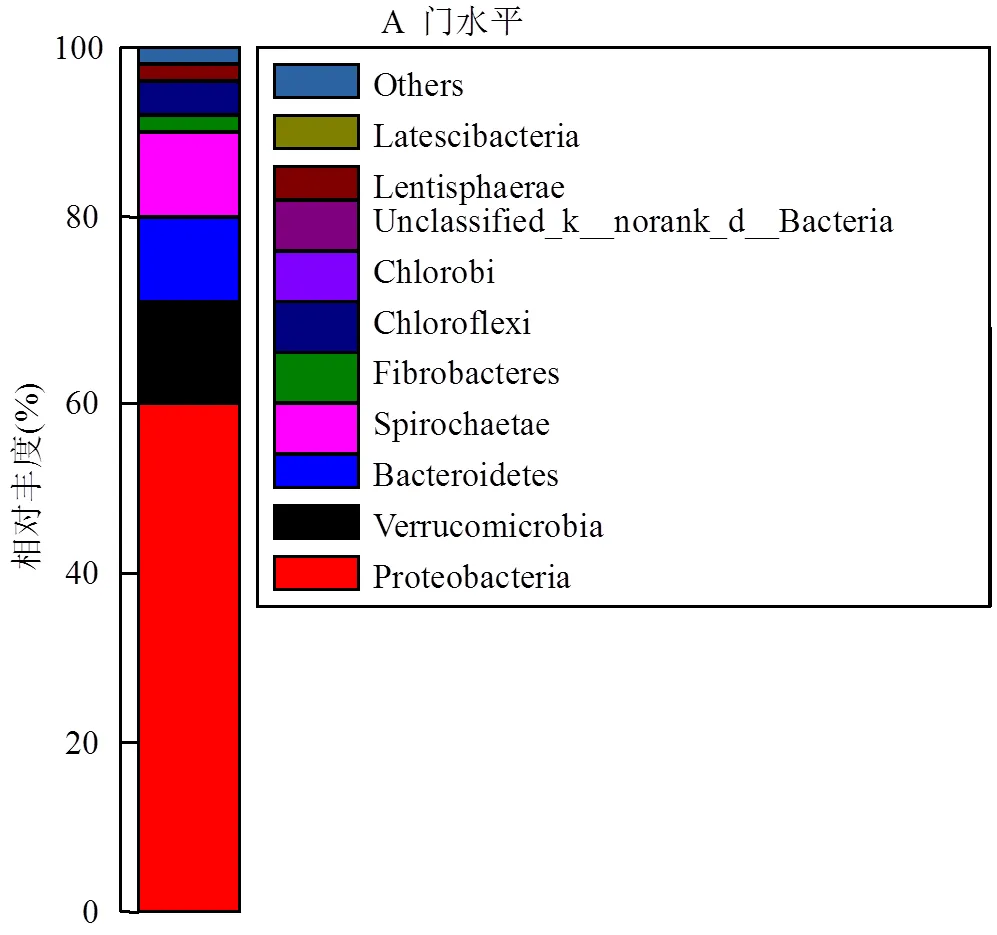
对反应器中的生物膜进行16S rRNA高通量测序,获得41685条序列,并按照97%相似性对非重复序列(不含单序列)进行OTU聚类,得到307个OUT.进一步对OTU代表序列进行分类学分析,共发现30门、112科和156属.在门水平上(图5A),主要以变形菌门(Proteobacteria)为主,其相对丰度高达59.47%,变形菌门为革兰氏阴性细菌的最大的组成部分,早在1994年就有研究学者发现用于水处理的微生物主要为变形菌门[24],且大部分甲烷氧化菌和反硝化细菌均属于变形菌门.此外,其他的分类主要为疣微菌门(Verrucomicrobia)(11.16%)、拟杆菌门(Bacteroidetes) (11.10%)和螺旋菌门(Spirochaetae) (8.94%).
在科水平上(图5B),嗜甲基菌科(Methylophilaceae)和甲基球菌科(Methylococcaceae)的相对丰度最高,分别为30.74%和11.70%.其中,甲基球菌科属于I型甲烷氧化菌[25],具有氧化甲烷的能力,通过产生的酶pMMO或sMMO将甲烷氧化成甲醇[26],甲基球菌科也可直接利用甲醇作为生长基质[27].而嗜甲基菌科是一种甲基营养菌,主要发现于活性污泥、河道和湖泊等,不能直接利用甲烷[28],主要以甲醇和甲基胺类物质为生长基质[29],嗜甲基菌科在系统中大量存在,可能是由于甲烷氧化过程的中间产物主要为甲醇、甲醛、甲酸等有机物[8],Xia等[30]在富集甲烷氧化菌时,分离出的最优势微生物也属于嗜甲基菌科,表明甲烷氧化菌氧化甲烷后产生的有机物可被其他微生物利用[31].此外, Kalyuhznaya等[32]研究发现嗜甲基菌科在湖泊沉积物的反硝化脱氮过程中具有一定的作用.其他相对丰度较高的为FukuN18淡水组(FukuN18_ freshwater_group)(10.93%)、细螺旋体科(Leptospiraceae) (5.68%)、多囊粘菌科(Polyangiaceae) (4.46%)和腐螺旋菌科(Saprospiraceae)(3.89%).其中丰度较高的FukuN18淡水组(FukuN18_freshwater_ group)被广泛发现存在于湖、河的底泥中[33-34],该类微生物还不能进行纯培养,其生存环境中主要富含腐殖酸等有机物, FukuN18淡水组可能主要以甲烷氧化菌产生的中间产物为营养物质.
在属水平上(图6),甲烷氧化菌属包括(10.49%)、(0.35%)、(0.34%)和_ unclassified (0.53%)[25],其中的相对丰度最高, 是该系统中的主要甲烷氧化菌, 属于甲基球菌科(),其形态为杆状或球状,仅能产生pMMO,此外,也只能产生pMMO,而可以产生sMMO和pMMO[21],说明系统中起主要作用的酶为pMMO.此外,系统中的反硝化菌属包括(1.21%)、(0.76%)、(0.052%)、(0.017%)和(0.014%)[35],其中常见于城镇污水处理厂的活性污泥中,可利用硝酸盐和有机物作为营养物质,将化合态的氮还原成N2O和N2[36].是氢营养型反硝化细菌,存在于污水处理厂活性污泥[37]、地下水环境[38]和试验装置[39],由于和的丰度最高,说明这两类微生物是该系统的主要反硝化细菌.
此外,属于嗜甲基菌科的主要为_ uncultured(30.40%)和(0.34%)[28],这两类微生物丰度相对较高的原因可能为系统中甲烷氧化过程中产生了大量含甲基的中间产物,也间接说明系统进气中甲烷比例偏高.
由上述可知,系统中同时存在大量好氧甲烷氧化菌和反硝化菌,丰度最大的甲烷氧化菌为,丰度较多的反硝化菌为和,说明这3类微生物是系统中最主要的功能微生物.系统对氮的去除是由好氧甲烷氧化菌与反硝化菌协同实现.
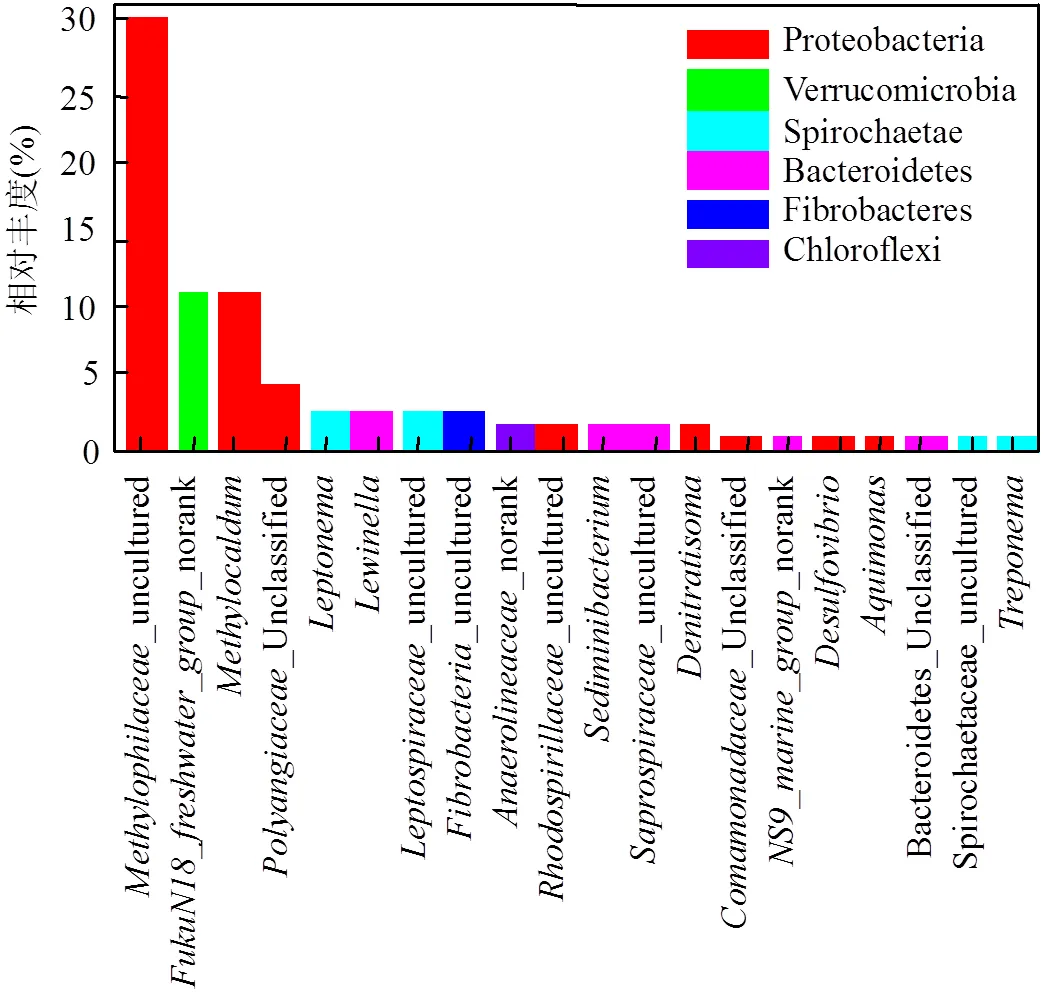
图6 属水平上的微生物分类
3 结论
3.1 采用气体循环序批式生物膜反应器(gcSBBR)构建出反硝化型甲烷好氧氧化系统,实现较高的反硝化脱氮速率(74.25mg/L·d)和硝酸盐氮去除率(98.93%),并考察了不同氮负荷对脱氮效率和甲烷消耗量的影响.
3.2 扫描电子显微镜(SEM)分析结果显示,反应器中的微生物主要以短杆菌为主,并存在少量的丝状菌.
3.3 高通量测序结果显示,系统中起主要作用的甲烷氧化菌为,主要的反硝化菌为和. 系统对氮的去除是由好氧甲烷氧化菌与反硝化菌协同实现.
[1] 郭 敏,何品晶,吕 凡,等.垃圾填埋场覆土层Ⅱ型甲烷氧化菌的群落结构[J]. 中国环境科学, 2008,28(6):536-541. Guo M, He P J, Lv F, et al. Type II methanotrophs community structure in the cover soils of landfill [J]. China Environmental Science, 2008,28(6):536-541.
[2] Raghoebarsing A A, Smolders A J P, Schmid M C, et al. Methanotrophic symbionts provide carbon for photosynthesis in peat bogs [J]. Nature, 2005,436(7054):1153-1156.
[3] Han B, Chen Y, Abell G, et al. Diversity and activity of methanotrophs in alkaline soil from a Chinese coal mine [J]. FEMS Microbiology Ecology, 2009,70(2):40-51.
[4] Ho A, Vlaeminck S E, Ettwig K F, et al. Revisiting methanotrophic communities in sewage treatment plants [J]. Applied and Environmental Microbiology, 2013,79(8):2841-2846.
[5] Belkhelfa S, Labadie K, Cruaud C, et al. Complete genome sequence of the facultative methylotroph methylobacterium extorquens TK 0001isolated from soil in poland [J]. Genome Announcements, 2018, 6(8).
[6] 刘 洋,陈永娟,王晓燕,等.水库与河流沉积物中好氧甲烷氧化菌群落差异性研究[J]. 中国环境科学, 2018,(5):1844-1854. Liu Y, Chen Y J, Wang X Y, et al. Microbial communities differences between aerobic methanotrophs in Miyun Reservoir and North Canal [J]. China Environmental Science, 2018,(5):1844-1854.
[7] Ruff S E, Felden J, Gruber-Vodicka H R, et al. In situ development of a methanotrophic microbiome in deep-sea sediments [J]. The ISME Journal, 2019,13(1):197-213.
[8] Zhu J, Wang Q, Yuan M, et al. Microbiology and potential applications of aerobic methane oxidation coupled to denitrification (AME-D) process: A review [J]. Water Research, 2016,90:203-215.
[9] Haroon M F, Hu S, Shi Y, et al. Anaerobic oxidation of methane coupled to nitrate reduction in a novel archaeal lineage [J]. Nature, 2013,501(7468).
[10] 薛 松,张梦竹,李 琳,等.甲烷厌氧氧化协同硝酸盐还原菌群驯化及其群落特征[J]. 环境科学, 2018,39(3):1357-1364. Xu S, Zhang M Z, Li L, et al. Acclimatization and community structure analysis of the microbial consortium in nitrate-dependent anaerobic methane oxidation [J]. Environmental Science, 2018,39(3): 1357-1364.
[11] Modin O, Fukushi K, Nakajima F, et al. Nitrate removal and biofilm characteristics in methanotrophic membrane biofilm reactors with various gas supply regimes [J]. Water Research, 2010,44(1):85-96.
[12] Stein L Y, Klotz M G. Nitrifying and denitrifying pathways of methanotrophic bacteria [J]. Biochemical Society Transactions, 2011, 39(6):1826-1831.
[13] 江 皓.甲烷氧化混合菌群的富集培养及其治理瓦斯工艺研究[D]. 北京:清华大学, 2010. Jiang H. Enrichment culture of mixed Methanotrophic community and its application in coal mine gas control [D]. Beijing: Tsinghua University, 2010.
[14] 国家环境保护总局.水和废水监测分析方法[M]. 第4版.中国环境科学出版社, 2002. State Environmental Protection Administration. Water and wastewater monitoring and analysis methods [M]. 4th Ed. China Environmental Science Press, 2002.
[15] Sheets J P, Ge X, Li Y. Effect of limited air exposure and comparative performance between thermophilic and mesophilic solid-state anaerobic digestion of switchgrass [J]. Bioresource Technology, 2015, 180:296-303.
[16] Pfluger A R, Wu W, Pieja A J, et al. Selection of Type I and Type II methanotrophic proteobacteria in a fluidized bed reactor under non-sterile conditions [J]. Bioresource Technology, 2011,102(21): 9919-9926.
[17] Sun F, Dong W, Shao M, et al. Aerobic methane oxidation coupled to denitrification in a membrane biofilm reactor: Treatment performance and the effect of oxygen ventilation [J]. Bioresource Technology, 2013, 145:2-9.
[18] Kampman C, Temmink H, Hendrickx T L G, et al. Enrichment of denitrifying methanotrophic bacteria from municipal wastewater sludge in a membrane bioreactor at 20°C [J]. Journal of Hazardous Materials, 2014,274:428-435.
[19] 周祥玉,赵云飞,李 东,等.微氧条件下以甲烷为碳源的反硝化实验研究[J]. 环境科学学报, 2017,(5):1704-1710. Zhou X Y, Zhao Y F, Li D, et al. Experimental study on denitrification using methane as carbon source under microaerobic condition [J]. Acta Scientiae Circumstantiae, 2017,(5):1704-1710.
[20] 卢培利,唐荧霜,丁阿强,等.亚硝酸盐型厌氧甲烷氧化过程强化新视角:排泥及其微生物机制研究[J]. 环境科学学报, 2019:1-9. Lu P L, Tang Y S, Ding A Q, et al. New insight into the enhancement of nitrite-dependent anaerobic methane oxidation: withdrawing sludge and its microbial mechanism [J]. Acta Scientiae Circumstantiae, 2019: 1-9.
[21] Semrau J D, Dispirito A A, Yoon S. Methanotrophs and copper [J]. FEMS Microbiology Reviews, 2010,34(4):496-531.
[22] Stoecker K, Bendinger B, Schoning B, et al. Cohn's Crenothrix is a filamentous methane oxidizer with an unusual methane monooxygenase [J]. Proceedings of The National Academy of Sciences of the United States of America, 2006,103(7):2363-2367.
[23] Sheng G, Yu H, Li X. Extracellular polymeric substances (EPS) of microbial aggregates in biological wastewater treatment systems: A review [J]. Biotechnology Advances, 2010,28(6):882-894.
[24] Manz W, Wagner M, Amann R, et al. In-situ characterization of the microbial consortia active in 2 waste-water treatment plants [J]. Water Research, 1994,28(8):1715-1723.
[25] Bowman J P. The Family Methylococcaceae [J]. The Prokaryotes: Gammaproteobacteria, 2014:411-440.
[26] Kalyuzhnaya M G, Puri A W, Lidstrom M E. Metabolic engineering in methanotrophic bacteria [J]. Metabolic Engineering, 2015,29:142-152.
[27] Bowman J. The Methanotrophs - The Families Methylococcaceae and Methylocystaceae [M]//The Prokaryotes: Volume 5: Proteobacteria: Alpha and Beta Subclasses, New York, NY: Springer New York, 2007: 266-289.
[28] Doronina N, Kaparullina E, Trotsenko Y. The family methylophilaceae [J]. The Prokaryotes: Alphaproteobacteria and Betaproteobacteria, 2014:869-880.
[29] Beck D A C, Mctaggart T L, Setboonsarng U, et al. The expanded diversity of methylophilaceae from Lake Washington through cultivation and genomic sequencing of novel ecotypes [J]. PLOS ONE, 2014,9(e1024587).
[30] Xia F, Zou B, Shen C, et al. Complete genome sequence of Methylophilus sp TWE2 isolated from methane oxidation enrichment culture of tap-water [J]. Journal of Biotechnology, 2015,211:121-122.
[31] Beck D A C, Kalyuzhnaya M G, Malfatti S, et al. A metagenomic insight into freshwater methane-utilizing communities and evidence for cooperation between the Methylococcaceae and the Methylophilaceae [J]. PEERJ, 2013,1(e23).
[32] Kalyuhznaya M G, Martens-Habbena W, Wang T, et al. Methylophilaceae link methanol oxidation to denitrification in freshwater lake sediment as suggested by stable isotope probing and pure culture analysis [J]. Environmental Microbiology Reports, 2009, 1(5):385-392.
[33] Yang Y, Li S, Gao Y, et al. Environment-driven geographical distribution of bacterial communities and identification of indicator taxa in Songhua River [J]. Ecological Indicators, 2019,101:62-70.
[34] Newton R J, Kent A D, Triplett E W, et al. Microbial community dynamics in a humic lake: differential persistence of common freshwater phylotypes [J]. Environmental Microbiology, 2006,8(6): 956-970.
[35] 李振灵,丁彦礼,白少元,等.潜流人工湿地基质结构与微生物群落特征的相关性[J]. 环境科学, 2017,(9):3713-3720. Li Z L, Ding Y L, Bai S Y, et al. Correlations between substrate structure and microbial community in subsurface flow constructed wetlands [J]. Environmental Science, 2017,(9):3713-3720.
[36] Fahrbach M.gen. nov., sp. nov., a 17 beta-oestradiol-degrading, denitrifying betaproteobacterium [J]. International Journal of Systematic and Evolutionary Microbiology, 2006,56(7):1547-1552.
[37] Jørgensen K S, Pauli A S. Polyphosphate accumulation among denitrifying bacteria in activated sludge [J]. Anaerobe, 1995,1(3): 161-168.
[38] Moreno B, Gómez M A, González-López J, et al. Inoculation of a submerged filter for biological denitrification of nitrate polluted groundwater: a comparative study [J]. Journal of Hazardous Materials, 2005,117(2/3):141-147.
[39] Mansell B O, Schroeder E D. Hydrogenotrophic denitrification in a microporous membrane bioreactor [J]. Water Research, 2002,36(19): 4683-4690.
Denitrification efficiency and microbial community research in an aerobic methanotroph-based system.
LI Yan-cheng*, YANG Ya-nan, LIU Deng-ping, LI Lei, LI Jiang
(College of Resources and Environmental Engineering, Guizhou University, Guiyang 550025, China)., 2019,39(10):4387~4393
An aerobic methane oxidation coupled to denitrification (AME-D) was constructed using a gas circulation sequencing batch biofilm reactor (gcSBBR), and nitrogen loading in influent was observed. It was found that the removal rate of nitrate nitrogen reached 98.93% and the denitrification rate 74.25mg/(L·d) when the nitrogen load was 0.075kg/(m3·d), and the average daily consumption of methane was 35.91% (at the initial stage: 50%). According to scanning electron microscopy (SEM) results, microorganisms in the system were mainly(12~18μm), dotted with a few filamentous bacteria (150~200μm). The high-throughput sequencing of 16S rRNA indicated that methanotroph were,and, the denitrifying bacteria,,,and, and the main functional bacteria,andin this system. Nitrogen was removed by the synergism of aerobic methanotroph and denitrifying bacteria. In addition, a large volume of(30.4%), which utilized methanol and methylamine as growth substrates, was found.
An aerobic methane oxidation coupled to denitrification (AME-D);methanotroph;denitrification;gcSBBR;functional microorganisms
X172
A
1000-6923(2019)10-4387-07
李彦澄(1989-),男,重庆人,讲师,博士,主要从事废水处理理论与技术方向研究.发表论文5篇.
2019-03-26
贵州省科技计划项目(黔科合基础[2019]1079号);贵州省教育厅青年科技人才成长项目(黔教合KY字[2018]118);贵州大学培育项目(黔科合平台人才[2017]5788)
* 责任作者, 讲师, flywithwin08@163.com
