转录组测序研究兰州百合抗冻关键基因及途径
2019-10-10田雪慧郁继华颉建明
田雪慧 ,郁继华,颉建明
(1.杨凌职业技术学院生态环境工程分院,陕西 杨凌 712100;2.甘肃农业大学园艺学院,甘肃 兰州 730070)
【研究意义】百合(Lilium)是国家卫生部首批公布的药食同源植物,也是著名的观赏植物[1]。食用百合是营养价值很高的特种蔬菜,其鳞茎含碳水化合物、蛋白质、果胶、脂肪、钾、磷以及钙、铁、百合甙等抗癌性植物碱和多种维生素,兼具保健功能,产品畅销中国及东南亚地区,在世界范围享有盛誉,市场前景十分广阔。兰州百合〔Lilium davidiiDuch.var.unicolor(Hong) Cotton〕微甜、口感好,是食用百合中的精品,生长在 103°54´ 54´´ E、36°06´ 12´´ N、海拔2 000多米的七里河区,是食用百合中的耐寒品种代表[2]。温度是植物生长最重要的环境因子之一,通过对兰州百合抗冻转录组的研究,筛选出百合抗冻的基因及抗冻相关通路,为后续兰州百合SSR标记开发、抗寒性相关基因的发掘及分子育种提供借鉴。
【前人研究进展】目前,对百合抗寒性研究集中于亚洲百合[3]、东方百合[4]、卷丹百合[5]等观赏百合品种,对百合耐寒的研究主要集中在细胞膜透性和保护酶系统方面,相对电导率、脂膜氧化产物丙二醛、抗氧化酶类(SOD、CAT、POD)活性通常被作为植物耐寒性生理指标[6-7]。HOSHI等[8]对百合在农杆菌介导下转录组进行研究,建立了东方百合杂交品种的转基因生产体系;杜方等[9]对百合不同器官的转录数据进行测序并开发了SSR标记。
【本研究切入点】关于抗寒食用百合代表——兰州百合的抗寒转录因子研究至今仍是空白。本研究运用先进的Illumina高通量测序平台的转录组测序技术,分别对正常温度条件下和零下超低温条件下生长的兰州百合苗期叶片的转录产物mRNA进行测序,比较常温与零低温下差异表达的基因及通路,了解兰州百合超低温胁迫下的基因表达状况。【拟解决的关键问题】进一步对差异表达基因进行GO、KEGG(Kyoto Encyclopedia of Genes and Genomes)注释和富集分析,以期进一步筛选出与兰州百合抗冻相关的基因[10]。
1 材料与方法
1.1 试验材料
在兰州市七里河区采集露地自然生长成熟的兰州百合种球,贮藏于2℃冰箱110 d[11]后,种植于15 cm口径的花盆中,置于20℃智能低温人工气候箱(RXZ-0288型)中培养,相同的水肥条件管理。将生长至15 cm左右的百合幼苗放在智能低温人工气候箱中进行-4℃低温处理[12],降至目标温度后维持24 h[13-14],以室温20℃作为对照,每个处理3次重复。
1.2 试验方法
1.2.1 转录测序 分别取3份20℃与-4℃的兰州百合叶片样品,采用TRIzol法提取总RNA[15],并对RNA浓度、纯度、完整性进行检测。样品检测合格后,用带有Oligo (dT)的磁珠富集真核生物mRNA[16]。随后加入fragmentation buffer将mRNA打断成短片段,以打断后的 mRNA为模板合成一链cDNA,再合成二链cDNA。然后纯化、修复,再用AMPure XP beads进行片段大小选择,最后进行PCR扩增,纯化PCR产物,得到最终文库。质检合格后使用Illumina 4000高通量测序平台(HiSeq/MiSeq)进行测序[16]。由Illumina 4000高通量测序平台测序所得的数据称为 raw reads或raw data。然后进行转录结果组装与拼接,并进行基因功能注释与表达水平分析。
1.2.2 实时荧光定量PCR验证 随机选取10个差异性表达的基因进行qRT-PCR分析验证。以20 ℃与-4 ℃的兰州百合叶片RNA为材料,采用HiScriptⅡQRT SuperMix for qPCR试剂盒反转录合成cDNA,设计引物(表1)进行qRT-PCR,以Actin基因作为内参[17]。各处理均设3次重复,计算基因的相对表达量。

表1 定量PCR引物序列Table 1 Primer sequences for qRT-PCR
2 结果与分析
2.1 测序结果与Trinity拼接
兰州百合20℃转录组共得到56 882 837 raw reads,56 456 634 clean reads,Q20值为94.56%,Q30值为87.32%,序列碱基GC含量为44.84%;-4℃转录组共得到58 092 923 raw reads,57 800 442 clean reads,Q20值为94.51%,Q30值为87.32%,序列碱基GC含量为44.54%。表明兰州百合转录组测序质量较高,可为后续的数据组装与分析提供良好基础。经过Trinity 转录本拼接和CD-HIT去冗余序列处理结果见表2。Unigene序列长度<300的最多(图1),说明转录拼接较好,利于后续分析研究。
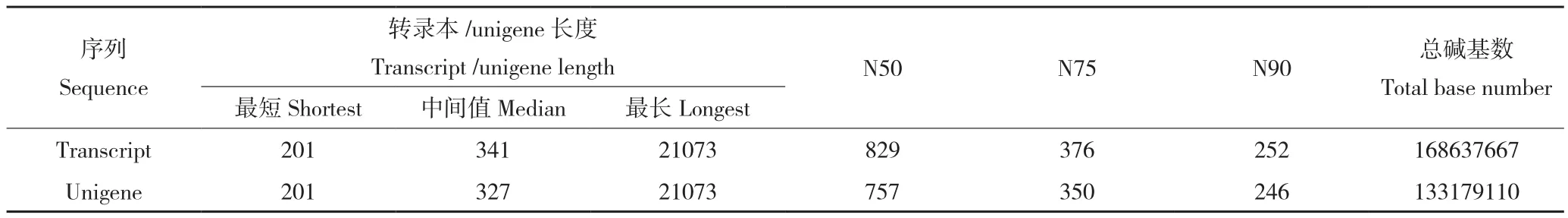
表2 处理前后序列数量、N50、N75、N90长度和总碱基数统计Table 2 Sequence number, length of N50, N75, N90 and total base number by different data processing

图1 拼接后Unigene 长度分布Fig.1 Length distribution of all unigenes after splicing
2.2 基因功能注释结果
对获得的转录组序列进行数据库比对注释可知,注释的Unigene数量共238 301个,Nr(NCBI 蛋白质非冗余数据库)注释的Unigene数量为65 314个、占27.41%,KOG(真核生物直系同源基因数据库)注释的Unigene数量为42 138个、占17.68%,GO库注释的Unigene数量为35 244个、占14.79%,Swiss-Prot注释的Unigene数量为46 337个、占19.44%,Pfam注释的Unigene 数量为58 605个、占24.59%(表3)。
2.2.1 GO 分类 对Unigene进行GO注释后,按照成功注释到生物学过程(Biological Process,BP)、细胞组分(Cellular Component,CC)和分子功能(Molecular Function,MF)的基因进行统计。其中生物学过程中较多Unigene注释有高分子代谢过程28 326个、初级代谢过程16 483个、细胞代谢过程38 883个;分子功能中较多Unigene注释有转移酶活性5 090个、水解酶活性5 153个、跨膜转运功能3 069个;细胞组分中较多Unigene注释有细胞壁8 663个、细胞膜7 832个、细胞器9 581个(图2)。

表3 Unigenes 注释结果统计Table 3 Annotation statistics for all Unigenes
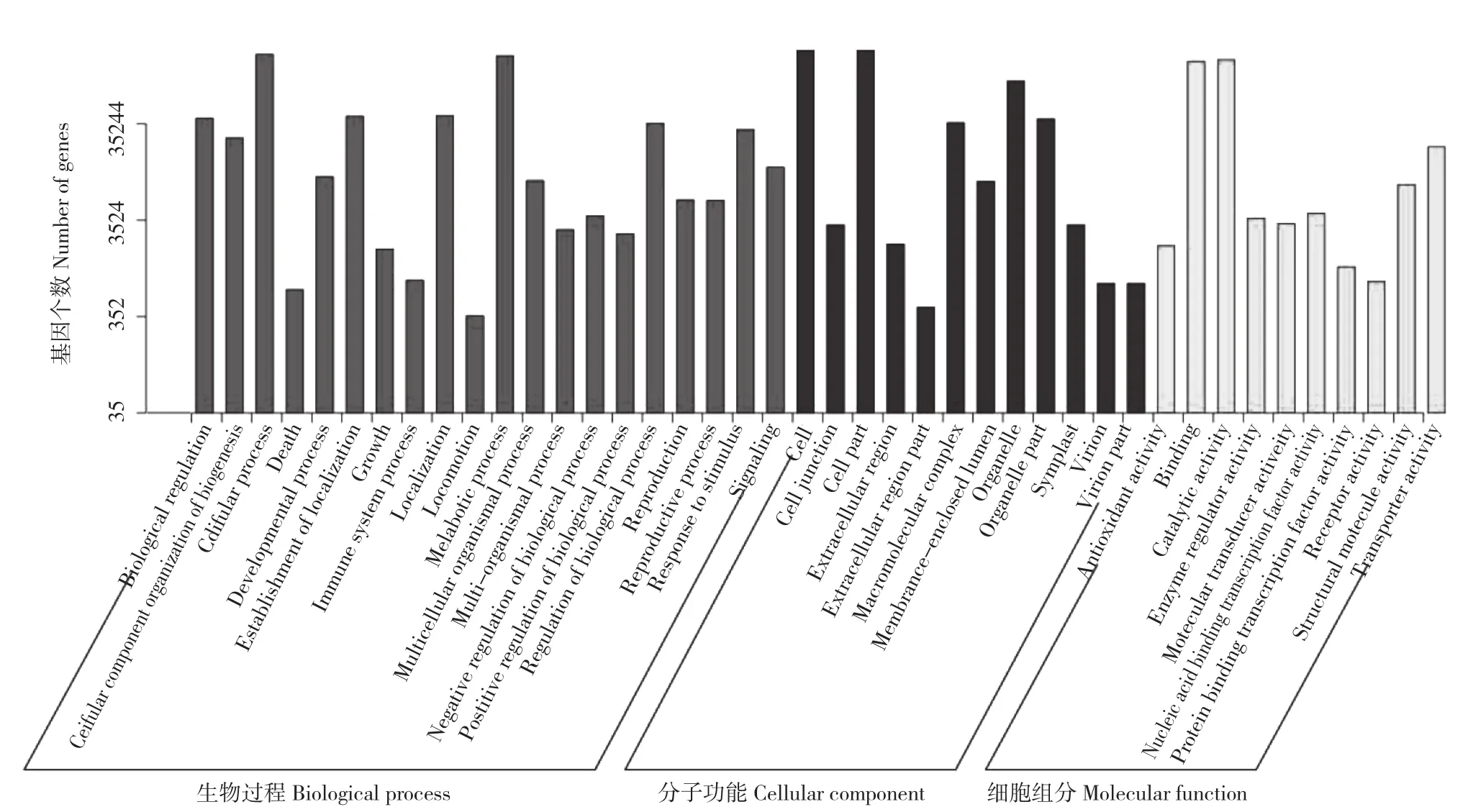
图2 GO分类Fig.2 GO function classification
2.2.2 KEGG分类 由表3可知,兰州百合测序结果有26 291条Unigene注释到KEGG数据库,涉及5大类代谢通路。其中代谢作用中注释4 914条、占40.66%;有机系统中注释905条、占7.49%;细胞过程中注释923条、占7.64%;遗传信息处理中注释3 609条、占29.86%;环境信息处理中注释1 734条、占14.35%。
2.3 基因表达差异分析结果
2.3.1 差异基因GO富集分析 将所有差异表达基因映射到Gene Ontology 数据库的各个功能分类中,计算每个分类的基因数目,与整体基因组背景相比,找出在差异表达基因中显著富集的功能分类。
对上调的差异基因进行富集分析,结果(图4)发现,在细胞成分中,差异基因富集效果较明显的有细胞核,基因占比13.81%;细胞内细胞器,基因占比16.06%;面上细胞器,基因占比16.06%。分子功能中差异基因富集效果较明显的功能有钙离子结合,基因占比3.52%;离子束缚,基因占比8.81%;阳离子绑定,基因占比8.81%;金属离子结合,基因占比8.81%。生物过程中上调基因富集效果较明显的功能有水响应,基因占比1.27%;非生物刺激反应,基因占比1.37%;磷酸化作用,基因占比8.62%。结果表明,含量增加的差异基因在细胞成分、分子功能、生物过程三方面主要调节物质的合成代谢。
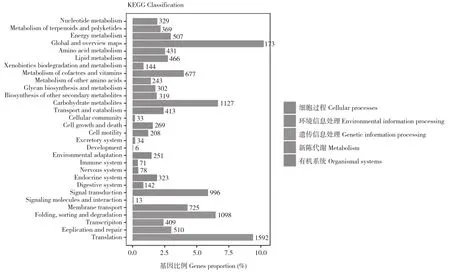
图3 KEGG 代谢通路Fig.3 KEGG metabolic pathway
对下调的差异基因富集分析,结果(图4)发现,差异基因主要富集在生物过程中。其中富集较为明显的是光合作用、光收获,基因占比1.65%;光合作用、光反应,基因占比1.65%。结果表明,含量相对减少的差异基因主要调节生物过程中的光合作用过程。
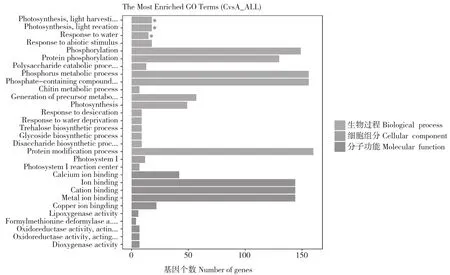
图4 差异基因(20℃ Vs -4℃)的GO富集结果Fig.4 GO enrichment result of differential genes (20℃ Vs -4℃)
2.3.2 差异基因KEGG富集分析 差异基因KEGG富集结果显示,常温(20℃)与超低温(-4℃)下差异表达基因共有5 848个,其中上调的差异基因有3 478个,下调的差异基因有2 370个(图5)。经分析后发现,与蛋白激酶、蛋白磷酸酶、碳代谢以及ABA等相关的基因显著变化;此外,吲哚-3-甘油磷酸合成酶、蛋白磷酸酶、已糖激酶、钙结合蛋白、叶绿素a/b结合蛋白等相关基因显著上调或下调。
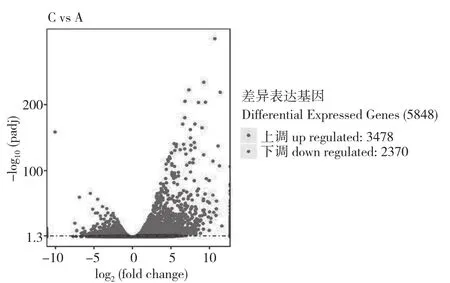
图5 差异表达基因(20℃ Vs -4℃)火山图Fig.5 Differential expressed genes(20℃ Vs -4℃)
Pathway显著性富集分析以KEGG Pathway为单位,应用超几何检验,找出差异基因相对于所有注释的基因显著富集的Pathway。处理后含量增加的差异基因显著富集的通路有植物病原菌互作、氨基酸和核苷酸糖代谢、植物昼夜节律、亚油酸代谢等(图6)。如上调的差异基因c156798_g2、c163371_g2 (calcium-dependent protein kinase,EC:2.7.11.1)、c166423_g2、c145559_g1 (respiratory burst oxidase,EC:1.6.3.- 1.11.1)、c162064_g1、c164545_g2、c166855_g1(3.1027)c145934_g1(8.2812) c101051_g1(4.9356)、c144017_g1(Inf) c144017_g2(Inf) c159000_g1(2.9702)c140553_g1(9.9651) c170503_g1(Inf)、c140013_g1(3.7728)、c154815_g1(Inf) c173122_g1(7.8456)c168962_g1(Inf) c163915_g1(Inf)、c156536_g2(2.9685) c156536_g1(1.2021)(calmodulin)、c147877_g2(mitogen-activated protein kinase kinase 4/5,EC:2.7.12.2)主要调节植物病原互作,病原互作相关基因与植物的抗性息息相关;上调的差异基因c154320_g1、c125968_g1(5.6972)c171764_g2(5.6213)、c172816_g3(5.9018)、c237479_g1(3.5347) c161991_g1(2.2688)、c172816_g4(2.2231)、c113669_g1(5.4648) c161991_g2(1.9362)、c106411_g1调节氨基酸和核苷酸糖代谢;上调的差异基因c151010_g1、c156146_g2(2.1536) c171563_g1(2.3117)、c156146_g1、c167910_g3、c152959_g1、c131089_g1(Inf)c144654_g1(6.9885)主要调节植物昼夜节律。
处理后下调的差异基因显著富集的通路有光合作用、光合作用-天线蛋白、卟啉和叶绿素代谢、光合生物体中的碳固定、脂肪酸生产等。如下调的差异基因c237057_g1、c13699_g1、c237057_g1、c76799_g1、c158296_g1、c152833_g1、c152881_g1、c158839_g1、c134148_g1、c140011_g1、c153193_g1、c131998_g1、c48634_g1、c71692_g1、c42744_g1、c153649_g1调节光合作用;下调的差异基因c149517_g1、c139230_g1、c152435_g1、c149445_g1、c153860_g1、c115377_g1、c164577_g1、c155535_g1、c174985_g1、c157047_g1调节光合作用-天线蛋白;下调的差异 基 因 c167743_g1、c167947_g1、c167743_g1、c71809_g1、c165450_g1c168519_g1、c169028_g1c133188_g1、c123480_g1、c185147_g2、c157598_g1、c157598_g1、c155686_g1、c166557_g2(-1.4097) c166557_g1(-1.5029)调节卟啉调节叶绿素代谢;下调的差异基因c104889_g2(-1.5635)c174574_g3(-3.7395)、c104889_g1、c71483_g1、c159323_g1、c172966_g1、c170442_g1、c162112_g2、c154502_g4、c43883_g1、c198353_g1、c168133_g3、c164307_g1调节光合生物体中的碳固定;下调的差异基因c134603_g2、c164323_g1、c166224_g1(-1.1149) c150017_g1(-1.3389)、c163278_g2、c148702_g1、c164323_g2、c134778_g1、c117604_g1调节脂肪酸伸长(图7)。
2.4 qRT-PCR 验证
为验证RNA-seq测序结果的可信度,随机选取10个差异表达基因,其中含上调的5条差异基因(c166605_g1、c199368_g1、c171769_g1、c164813_g2、c153721_g1)和下调的5条差异基 因(c76799_g1、c153860_g1、c149445_g1、c115377_g1、c155535_g1),以Actin为内参进行qRT-PCR验证。由图8可知,10个基因在低温胁迫下的表达程度不同,但表达趋势与高通量测序结果基本一致,即表明测序结果真实可靠。
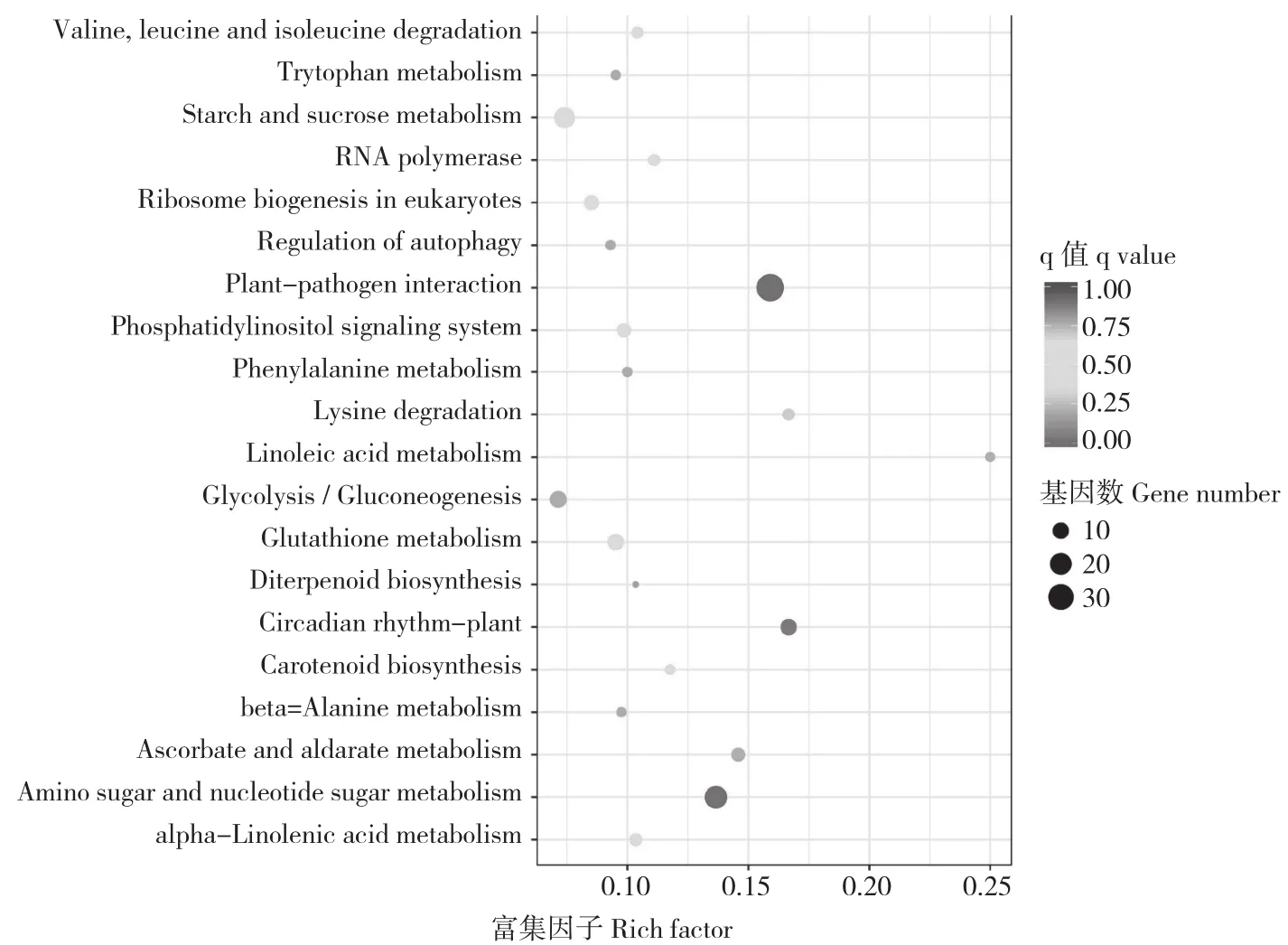
图6 上调差异基因(20℃ Vs -4℃)参与的KEGG代谢通路富集结果Fig.6 Differential up-regulation genes(20℃ Vs -4℃)assignment to KEGG metabolic pathway

图7 下调差异基因(20℃ Vs -4℃)参与的KEGG代谢通路富集结果Fig.7 Differential down-regulation genes(20℃ Vs -4℃)assignment to KEGG metabolic pathway
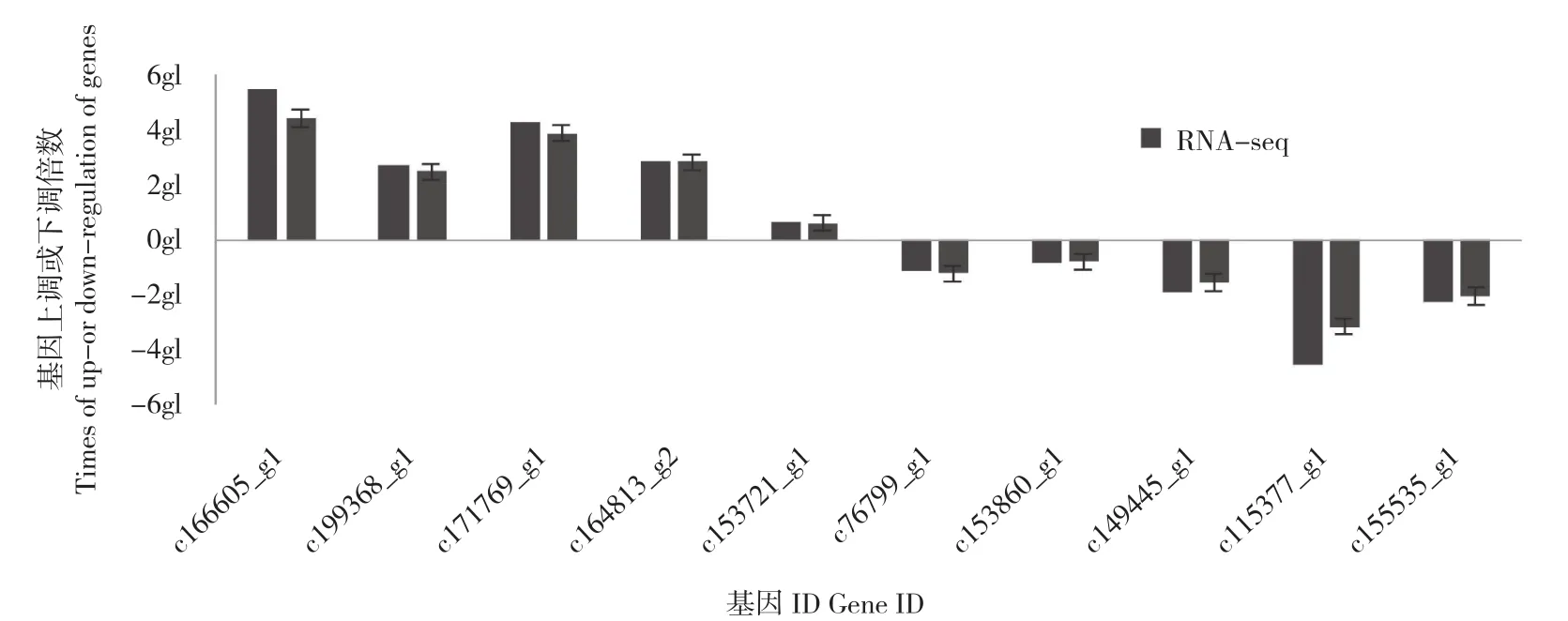
图8 差异基因的qRT-PCR验证Fig.8 Validation of DEGs by qRT-PCR
3 讨论
前人关于百合抗冻分子机制研究较少,不良生长环境尤其是零下低温胁迫是植物生长发育中常遇到的自然灾害。本研究采用先进的测序平台Illumina 4000高通量测序平台对常温(20℃)与零下低温(-4℃)处理的兰州百合进行测序,确保测序数据的准确性和序列的高度丰富性[18]。测序结果在NCBI数据库中共注释到了238 301条Unigene,但注释到每个库中的基因最大不超过27.41%,说明还有很多基因没有被注释到NCBI库中,原因可能是数据库信息不足、测序结果中有新基因和基因片段短等。因此,在后续的研究中,我们可以进一步比对测序结果中差异极显著的基因片段,进一步深入发掘兰州百合抗冻转录新基因。
根据KEGG富集分析结果,可以看出共有25条差异基因参与病原互作途径,10条差异基因参与氨基酸和核苷酸糖代谢途径,8条差异基因参与植物昼夜节律途径,15条差异基因参与卟啉调节叶绿素代谢途径,16条差异基因参与光合作用途径,10条差异基因调节光合作用-天线蛋白途径,13条差异基因调节光合生物体中的碳固定途径,9条差异基因调节脂肪酸伸长途径。因此我们可以深入研究以上通路以进一步揭示兰州百合抗冻的分子机制。另外也可以将兰州百合相关的代谢产物作为研究对象进行深入探究。
4 结论
本研究对兰州百合转录组测序后通过KEGG通路分析,结果检测到24 465个差异表达的基因,筛选后获得8 558条差异表达基因,共占差异表达基因总数的10.27%。经富集分析后发现,与蛋白激酶、蛋白磷酸酶、碳代谢以及ABA等相关的基因可以作为研究兰州百合寒冷响应机制的主要研究对象。qRT-PCR分析结果表明,随机选出的10个差异性表达基因的表达趋势与高通量测序结果相一致。此外,还候选了吲哚-3-甘油磷酸合成酶、蛋白磷酸酶、已糖激酶、钙结合蛋白、叶绿素a/b结合蛋白等基因作为与兰州百合零下低温响应相关的应答候选基因,为揭示兰州百合抗冻分子机制奠定了基础。
