肝癌转移中脂代谢-免疫相关基因的筛选
2019-10-08王永坤李保国
王永坤 李保国


摘 要: 肝癌转移是肝癌患者治疗失败和死亡的主要原因,寻找与肝癌发生转移相关的基因对于研究肝癌转移的分子机制,及时诊断、防治肝癌转移具有重要的生物学意义。我们在前期研究中已经发现脂肪代谢可能与免疫微环境存在关联。本研究从GEO数据库下载27例伴有高低转移的肝细胞癌样本的表达谱信息,比较高低转移样本之间基因表达谱的差异,从中筛选出91个脂代谢相关的差异表达基因。通过脂代谢基因的表达谱数据,我们计算了22种免疫细胞在这27例转移样本中的免疫细胞占比,并通过皮尔森相关性分析筛选出脂代谢-免疫相关基因,进一步探究脂代谢与与免疫微环境之间的关系。最后,通过KEGG通路Pathway功能富集分析以及预后分析发现CYP4A11、CYP3A5、CYP2C8、ALDH2、BDH1、ALDH7A1等脂代谢-免疫相关基因可能在肝癌侵袭和转移中发挥重要作用。
关键词: 肝癌转移;脂代谢;免疫细胞;表达谱
【Abstract】: Hepatocellular carcinoma (HCC) has a poor prognosis due to its rapid development and early metastasis. Finding genes related to metastatic HCC may bring important biological significance for studying the molecular mechanism and intervention targets of prevention and cure of liver cancer. In this study, 27 samples of hepatocellular carcinoma with high and low metastasis were downloaded from the GEO database, 91 differentially expressed genes related to lipid metabolism were screened out by comparing gene expression profiles between high and low metastasis samples. Furthermore, the proportions of 22 immune cells in 27 samples were calculated by CIBERSORT, the lipid metabolism genes which are highly correlated with HCC metastasis and immune microenvironment were further explored by Pearson correlation analysis. Eventually, we found that CYP4A11, CYP3A5, CYP2C8, ALDH2, BDH1, ALDH7A1 lipid metabolism-immunity-related genes may play an important role in liver cancer invasion and metastasis by KEGG pathway functional enrichment and prognostic analysis. Not only might these molecules allow more accurate prediction of prognosis for patients with HCC, but they may also provide targets for potential therapeutic agents.
【Key words】: Liver cancer metastasis; Lipid metabolism; Immune cells; Expression profile
0 引言
原发性肝癌是最常见的恶性肿瘤之一,居全球癌症患者致死率的第三位,并在全世界范围内呈现逐年上升趋势[1]。肝細胞肝癌(Hepatocellular carcinoma,HCC)是最常见的原发性肝癌,癌细胞的扩散和转移是HCC患者的主要死因[2]。代谢是肝癌的标志,其有助于肝癌发生和发展,一方面,致癌因素破坏机体的代谢平衡,诱发代谢重组,细胞发生癌变;另一方面,重组后的代谢系统介导多种生物学行为,参与癌细胞的增殖、侵袭和转移[3-4]。脂代谢是机体代谢中的重要一环,肝脏是脂代谢的重要场所。肝癌转移是一个包含肝癌细胞脱离原发瘤、在循环系统中存活并种植生长于靶器官的复发的过程,其中的每一步都受到肝癌微环境中的非肝癌细胞的影响,炎症在肝癌的发生发展、侵袭转移过程中起重要作用[5]。肿瘤微环境(Tumor microenvi ronment,TME)中的免疫抑制被认为是基于免疫细胞和肿瘤细胞的相互代谢需求[6]。肿瘤细胞中不仅发生脂质代谢异常,而且TME显示脂质代谢异常,进一步募集免疫细胞。HCC中免疫微环境、脂代谢异常以及肝癌侵袭转移之间的作用机制目前尚不清楚。因此,深入了解三者之间的复杂关系对于肝癌的综合治疗具有重要意义。
近几年,随着数据类型的增多,EXCEL、SPSS等常用软件已经不能满足科研需求了。R语言以其方便、快捷的优势,迅速获得许多科研人员的青睐,例如R语言的limma包可以有效分析FPKM数据的表达值,计算差异表达基因,ggplot2包和heatmap包可以将表达情况以热图的方式直观展示出来。Python语言中的re板块可以快速匹配多套数据,sys模块可以在外部向程序内部传递参数,还可以使用SVM--Lasso等,训练模型,画出AUC曲线。但不是所有的科研人员可以熟练的使用编程语言,因此越来越多的分析软件被研发出来应用于生物信息的研究,例如CIBERSORT是一种利用组织RNA混合物表征细胞异质性的软件,采用基因表达谱数据[7,8],以1为基数,计算22种免疫细胞在某个样本中的相对比例。该软件通过线性支持向量回归(SVR)和机器学习方法对混合物进行去卷积,有助于对细胞生物标记物和治疗靶标的RNA混合物进行大规模分析[20-24],即从基因表达谱数据中中鉴定复杂组织细胞组成的工具。g:Profiler是一个公共Web服务器,用于表征和操作由挖掘高通量基因组数据产生的基因列表,进行KEGG和GO等多种分析。STRING是已知和预测的蛋白质-蛋白质相互作用的数据库。
GEO数据库[9]是由美国国立生物技术信息中心(NCBI)建立的基因表达资源库。GEO数据库中已收录了不同组织来源的各个组学水平的数据,可作为肝癌转移研究的主要数据来源。利用GEO数据库中肝癌转移数据及相关的工具和算法,能够全面分析肝癌脂质代谢与免疫微环境对转移的影响。本课题通过基因表达谱筛选与免疫微环境中免疫细胞占比显著相关的脂代谢基因,为肝癌转移相关重要基因和标志物的筛选提供另一种角度。
1 材料与方法
1.1 材料
通过GEO数据库检索界面的GEO DataSets搜索HCC Metastasis,共搜集到856篇相关文献或数据。精读文献最终确定研究号为GSE77314[10]的肝癌转移基因表达谱数据(https://www.ncbi.nlm.nih. gov/geo/query/acc.cgi?acc=GSE77314)为研究数据。研究对象为27例肝癌转移样本,其中13例伴有高转移,14例伴有低转移。
1.2 方法
(1)筛选脂代谢相关差异表达基因
为了全面筛选在肝癌转移过程中改变的脂代谢相关基因,通过查阅200多篇脂代谢相关文献以及KEGG数据库中整合代谢通路中的脂代谢相关基因,使用R语言计算差异表达基因(p<0.05),进而通过Python语言的re模块对两者进行匹配从而筛选出脂代谢相关基因。
(2)免疫细胞占比及皮尔逊相关性分析
本文使用CIBERSORT软件计算22种免疫细胞在27例转移样本中的免疫细胞占比。为探讨高低转移组中脂代谢基因与免疫微环境之间的关系,对脂代谢基因表达值与免疫细胞占比进行了皮尔逊相关性检验,筛选既与脂代谢相关又与免疫细胞占比显著相关(P<0.05)的候选基因以供后续的分析。筛选标准:高/低转移样本中与22种免疫细胞中的至少一种细胞显著相关。
(3)KEGG通路富集和基因预后分析
利用本组开发的OmicsBean-Cancer (http://www. omicsbean.com:XX/)多组学数据分析系统,针对多种类型组合的组学实验设计方案进行数据分析。对与脂代谢-免疫细胞占比显著相关的候选基因进行KEGG富集分析,比较高低转移组脂代谢通路及通路上脂代谢基因的差异。为了探究脂代谢基因对患者预后的影响,采用Omicsbean-Cancer软件对脂代谢通路上富集的基因和高低转移组中共有的基因分别作预后分析。筛选条件设定为:Log-rank检验P<0.05,用于筛选目标基因。
(4)蛋白质相互作用网络(protein protein interaction network,PPI network)
通过STRING数据库[11]将与肝癌预后分析显著性相关的目标基因进行蛋白质相互作用网络分析,筛选出目标基因的中心节点。
2 结果与分析
2.1 脂质代谢相关基因的筛选
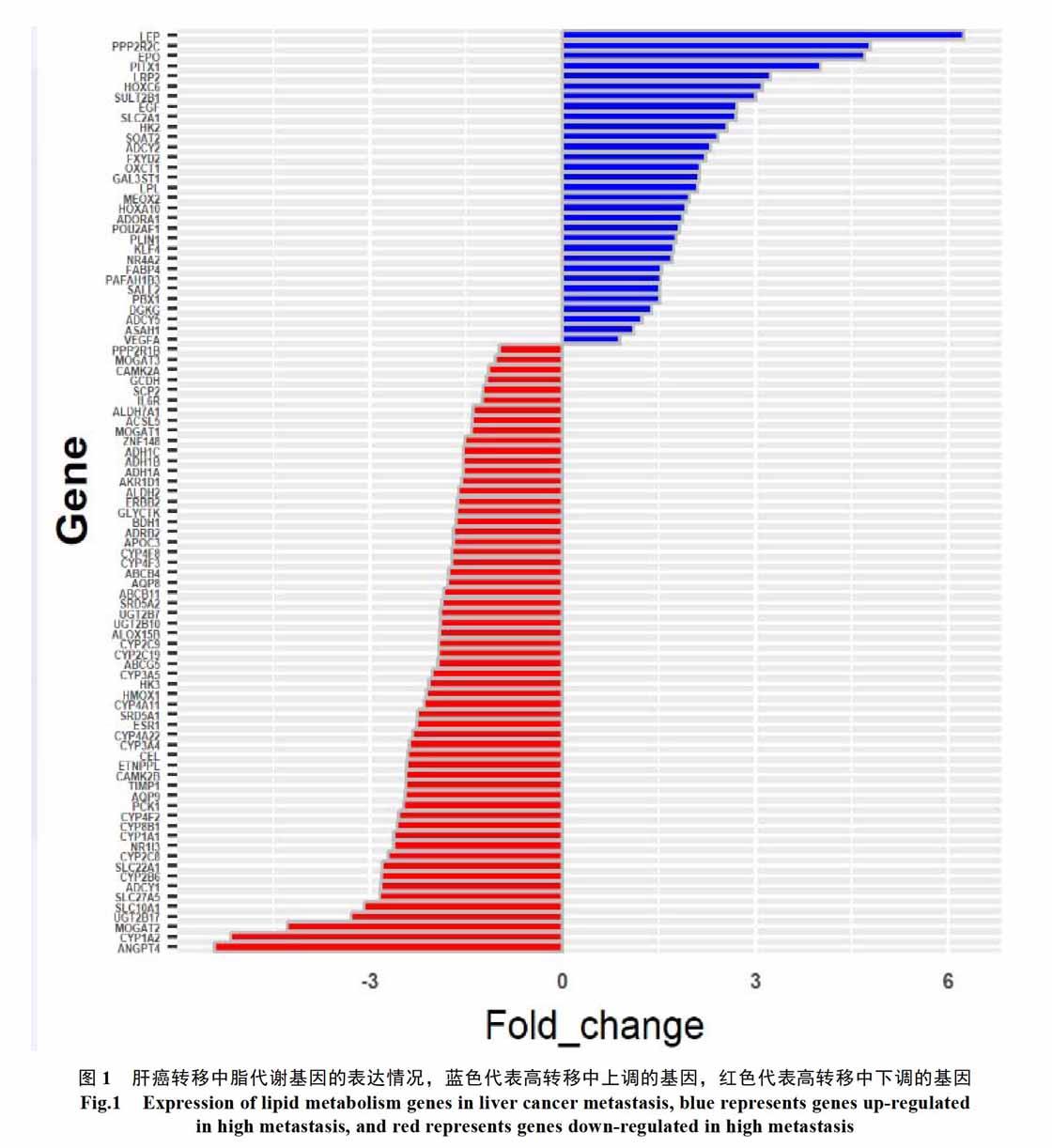
通过查阅文献以及KEGG数据库中收集的脂代谢相关基因1009个对1789个差异表达基因进行比对,最终筛选出91个差异表达的脂代谢相关基因。将91个脂代谢基因作为主的研究对象,其中明显表达上调的基因35个,明显表达下调的基因56个,如图1所示。
2.2 免疫细胞占比及皮尔逊相关性检验
通过使用CIBERSORT软件分别计算高转移组和低转移组中的22种免疫细胞占比,发现在高/低转移样本中幼稚B细胞、巨噬细胞M1、树突状休眠细胞占比最高(见图2)。
肿瘤微环境中的炎症细胞有肿瘤相关巨噬细胞(Tumer Associated Macrophage,TAM)、树突状细胞(Dendritic cells,DC)和淋巴细胞。研究表明,TAM被认为是肿瘤细胞侵袭转移中重要的参与者,TAM
分泌的某些分子,不仅能够促血管形成机制促进原发肿瘤发展,也介导了肿瘤的转移过程[12]。肿瘤组织中TAM缺乏,不能明显地改变癌前病变,但却能很明显地推迟肿瘤侵袭能力的出现,减少肿瘤转移的形成[12-13]。DC诱导的自体T淋巴细胞增殖分化为细胞毒性T细胞(CLT)能明显降低AFP mMRNA阳性率,并相应减少肝癌转移的发生[14]。在本文的研究中,在高转移组中TAM的平均占比为0.12略高于低转移组的平均占比0.11,高转移组DC的平均占比0.1略低于低转移组的平均占比0.12,与文献中的报道相一致[12-14]。
为了进一步探讨脂代谢与免疫微环境之间的关系,对脂代谢基因表达值与免疫细胞占比进行了皮尔逊相关性检验,得到既与脂代谢有关又与免疫细胞占比显著性相关的基因(P<0.05)。其中,高转移组中基因47个,低转移组基因18个,其中9个基因同时存在于高/低转移样本中。在高转移样本中的脂代谢-免疫相关基因中,ALDH2及CYP450家族基因(CYP4A11、CYP3A5等)呈显著下调(图1),这与CYP3A5基因表达水平低能够促进肝癌的转移相一致[15]。另外,Hou G,Chen L等[16]研究表明在迁移能力较强的肿瘤组织中,HCC患者的预后较差与ALDH2基因表达水平降低有关。
2.3 KEGG通路富集分析
上述的研究中,发现高/低转移组中与免疫细胞占比显著相关的脂代谢基因相差很大,为了进一步
探討转移过程中脂代谢通路的改变,对高/低转移组中的脂代谢-免疫相关基因分别作KEGG通路富集分析,结果显示:高转移组中的47个脂代谢基因主要富集在脂肪酸降解、酮体的合成和降解、原发性胆汁酸生物合成、类固醇激素生物合成、甘油脂代谢、花生四烯酸代谢六种脂代谢通路中,而低转移组中的18个脂代谢基因仅有SRD5A1, SRD5A2两个基因富集在类固醇激素生物合成通路中(表1)。高低转移脂代谢通路的不同,对肝癌转移程度产生很大的影响,例如,高转移组的花生四烯酸代谢通路,其代谢途径有四个子过程:磷脂酶A2(PLA2)催化产生花生四烯酸;通过各种酶生产下游产物;环加氧酶(COXs),脂氧合酶(LOXs)产生白三烯和氢过氧二十碳四烯酸(HETEs);以及细胞色素P450(CYP450)环加氧酶产生几种HETE和环氧二十碳三烯酸(EETs),这四个子过程与炎症的发生和发展关系密切,以促炎或者抑炎的方式参与炎症过程的调节[17]。对于HETE和EET的产生,CYP450家族基因起重要作用。在花生四烯酸代谢通路中CYP450家族基因显着下调,表明HETES和EETs产生的功能减少。CYP4A11合酶的主要作用是使花生四烯酸发生ω酶氧化,同时CYP4A11也是转移风险的重要基因[18]。
2.4 脂代謝基因对预后的影响
为了研究脂质代谢的预后影响,特别是高/低转移组脂代谢通路上显著富集的基因。为了研究脂质代谢对肝癌病人的预后影响,特别是高/低转移组脂代谢通路上显著富集的基因。我们从UCSC(Uni versity of California Santa Cruz)数据库中下载TCGA Liver Cancer数据集(https://xenabrowser.net/dat a pages/?cohort=TCGA%20Liver%20Cancer%20(LIHC)&removeHub=https%3A%2F%2Fxena.treehouse.gi.ucsc.edu%3A443),从中提取出既有基因表达数据又有完整的总生存期(OS)和无复发生存期(RFS)的样本342例。根据基因表达量的中值把病人分成两组,用Log-Rank检验估计两组的存活时间之间的差异。筛选出具有显著预后影响的脂质代谢相关基因。其中高转移中CYP4A11、ALDH7A1、ADH1C、ALDH2、ADH1B、ADH1A、BDH1、SCP2、SLC27A5、
CYP3A5、SRD5A2、DGKG、CYP4A11、CYP2C8等基因预后较显著(P<0.05),低转移中仅有SRD5A2基因预后较显著。高低转移中基因预后差别较大,可能是导致转移高低的原因。对高低转移共有的基因作预后分析,结果发现AQP9,SRD5A2,ABCG5,BDH1较显著。根据STRING数据库对预后较显著的基因构建蛋白质-蛋白质相互作用网络(PPI)分析,发现除了已经报道的CYP4A11和ALDH2外[17-18],CYP3A5、CYP2C8、BDH1、ALDH7A1是被识别的网络中心节点(图3),且其预后效果比较好(图4),如酮体合成与降解通路中的BDH1基因,其为促进酮体产生的限速酶,在乳腺癌中生酮成纤维细胞促进肿瘤的生长与转移[19],这与本研究的结果相一致,为下一步的研究提供了依据。这些分子可能是HCC的潜在预后生物标志物,在肝癌转移过程中可能发挥了非常重要的作用。
3 结论
肝癌的复发转移已经严重影响肝癌病人生存率,寻找促使肝癌发生转移的基因改变 ,探明肝癌转移的分子机制,预防或阻断肝癌转移成为进一步提高肝癌治疗效果的关键,并已成为近年肝癌研究的热点。虽然过去的许多研究对肝癌转移分子机制进行了有益的探索,并且已经发现了一些与肝癌转移相关的基因[18]。但这些研究大多集中在寻找信号通路相关基因。近年来生物信息分析软件在肿瘤研究中的应用越来越广泛,本文通过R语言的limma包比较分析27例伴有高低转移的肝癌之间基因表达谱的差异,CIBERSORT分析免疫细胞占比与肝癌转移的相关性,g:Profiler软件聚焦筛选了与肝癌转移密切相关的脂代谢-免疫基因,并使用STRING软件分析了这些基因的功能以及它们之间相互作用的关系,最终使用Log-Rank检验筛选出CYP4A11、CYP3A5、CYP2C8、ALDH2、BDH1、ALDH7A1等6个基因进一步与肝癌预后相关。本文研究结果为筛选可阻断肿瘤侵袭和转移的新的分子标志物提供了思路和方法。
参考文献
[1]Pascual S, Herrera I, Irurzun J. New advances in hepa tocellular carcinoma[J]. World J Hepatol, 2016, 8(9): 421- 438.
[2]Kim J U, Shariff M I F, Crossey M M E, et al. Hepatocellular carcinoma: Review of disease and tumor biomarkers[J]. World Journal of Hepatology, 2016, 8(10): 471.
[3]Deberardinis R J, Lum J J, Hatzivassiliou G, et al. The Biology of Cancer: Metabolic Reprogramming Fuels Cell Growth and Proliferation[J]. Cell Metabolism, 2008, 7(1): 0-20.
[4]Yang G, Wang Y, Feng J, et al. Aspirin suppresses the abnormal lipid metabolism in liver cancer cells via disrupting an NFκB-ACSL1 signaling. [J]. Biochemical & Biophysical Research Communications, 2017, 486(3): 827.
[5]Shi Y, Qin N, Zhou Q, et al. Role of IQGAP3 in metastasis and epithelial–mesenchymal transition in human hepatoce llular carcinoma[J]. Journal of Translational Medicine, 2017, 15(1): 176.
[6]Tahmasebi Birgani M, Carloni V . Tumor Microenvironment, a Paradigm in Hepatocellular Carcinoma Progression and Therapy[J]. International Journal of Molecular Sciences, 2017, 18(2): 405.
[7]Chen B, Khodadoust M S, Liu C L, et al. Profiling Tumor Infiltrating Immune Cells with CIBERSORT[J]. Methods in Molecular Biology, 2018, 1711: 243.
[8]Li Taiwen, Fan Jingyu, Wang Binbin, et al. TIMER: A Web Server for Comprehensive Analysis of Tumor-Infiltrating Immune Cells[J]. Cancer Research, 2017, 77(21): e108.
[9]Barrett T, Troup D B, Wilhite S E, et al. NCBI GEO: archive for functional genomics data sets--10 years on[J]. Nucleic Acids Research, 2013, 39(Database issue): 1005-10.
[10]Liu G, Hou G, Li L, et al. Potential diagnostic and prognostic marker dimethylglycine dehydrogenase (DMGDH) suppre sses hepatocellular carcinoma metastasis in vitro and in vivo. [J]. Oncotarget, 2016, 7(22): 32607-32616.
[11]Li L, Lei Q, Zhang S, et al. Screening and identification of key biomarkers in hepatocellular carcinoma: Evidence from bioinformatic analysis[J]. Oncology Reports, 2017.
[12]Tan H, Wang N, Feng Y . Autophagy mediated activation of Relb/p52 responsible for the reprogramming of tumour associated macrophages[J]. European Journal of Cancer, 2016, 61: S49-S49.
[13]许晶, 管晓翔. 肿瘤微环境的组成及其在肿瘤转移中的作用[J]. 癌症进展, 2014, 12(2): 144-148.
[14]孙建聪. 1. 负载肝癌干细胞RNA树突状细胞疫苗诱导的抗肝癌免疫应答研究;2. EDIL3的表达和肝癌患者预后的关系[D]. 中山大学, 2010.
[15]Jiang F. CYP3A5 Functions as a Tumor Suppressor in Hepa tocellular Carcinoma by Regulating mTORC2/Akt Signa ling[J]. Cancer Research, 2015, 75(7): 1470-1481.
[16]Hou G, Chen L, Liu G, et al. Aldehyde dehydrogenase‐2 (ALDH2) opposes hepatocellular carcinoma progression by regulating AMP-activated protein kinase signaling in mice[J]. Hepatology, 2017, 65(5): 1628.
[17]Li J, Li O, Kan M . Berberine induces apoptosis by suppressing the arachidonic acid metabolic pathway in hepatocellular carcinoma.[J]. Molecular Medicine Reports, 2015, 12(3): 4572.
[18]Chen P, Wang F, Feng J, et al. Co-expression network analysis identified six hub genes in association with meta stasis risk and prognosis in hepatocellular carcinoma[J]. Oncotarget, 2017, 8(30): 48948-48958.
[19]Martinez-Outschoorn U E, Lin Z, Whitaker-Menezes D, et al. Ketone body utilization drives tumor growth and meta sta sis[J]. Cell Cycle, 2012, 11(21): 3964-3971.
[20]蒲杰方, 盧荧玲. 基于聚类算法和神经网络的客户分类模型构建[J]. 软件, 2018, 39(4): 130-136.
[21]吴亚熙, 岑峰. 基于卷积神经网络的多层级目标检测方法[J]. 软件, 2018, 39(4): 164-169.
[22]阮阳, 刘禹, 韩港成, 等. 基于爬虫的定向数据检索系统[J]. 软件, 2018, 39(5): 118-120.
[23]岳立言, 欧阳昇, 翟宝蓉. 嵌入式Linux远程监控系统的设计与开发[J]. 软件, 2018, 39(6): 20-22.
[24]张敏, 徐启华. 基于改进BP的神经网络模型参考自适应控制[J]. 软件, 2015, 36(7): 118-123.