噬菌弧菌N1对淡水和海水养殖水体细菌群落影响PCR-DGGE分析
2019-09-25陈慧黠韩民泳于佳豪
陈慧黠,韩民泳,于佳豪,刘 琳
噬菌弧菌N1对淡水和海水养殖水体细菌群落影响PCR-DGGE分析
陈慧黠1,韩民泳1,于佳豪1,刘 琳2
(1. 大连海洋大学水产与生命学院,海洋生物资源恢复与生境修复重点实验室,辽宁 大连 116023;2 青岛农业大学生命科学学院, 山东省应用真菌重点实验室,山东 青岛 266109)
【】研究噬菌弧菌sp. N1在一个投放周期内对淡水和海水养殖环境中细菌、弧菌总数及细菌群落结构的影响。自市售微生态制剂分离蛭弧菌N1并进行分子鉴定,测定其裂解效果,制备高浓度N1菌液分别投放至淡水红鲤鱼(red carp)和海水仿刺参()养殖水体,采用细菌平板计数法及PCR-变性梯度凝胶电泳(PCR-DGGE)技术分析48 h内水体环境中细菌、弧菌总数及细菌群落结构变化。经鉴定蛭弧菌N1为噬菌弧菌,其对大肠杆菌()、溶藻弧菌()、黄海希瓦氏菌()、灿烂弧菌()、哈氏弧菌()和金黄色葡萄球菌()均有裂解效果。将噬菌弧菌N1投放进淡水和海水养殖环境的12~24 h,其能显著降低两种环境中弧菌含量(< 0.05)。DGGE分析显示添加噬菌弧菌N1后淡水组优势菌群弧菌属(,a1)和不可培养杆菌属(Uncultured bacterium, a5)在 12 h 以后含量有明显的减少,假单胞菌属(, a3)和红杆菌科属(, a6)菌含量增加。海水组优势菌群不可培养杆菌属(c2)菌在 12 h 时变成非优势菌群,而白杆菌属(,c1)增加成为优势菌群。噬菌弧菌N1在水中含量在24 h时降至最低。噬菌弧菌N1对海水和淡水环境中的弧菌属和不可培养杆菌属菌群有明显的裂解作用导致其含量下降,但也使假单胞菌属(),红杆菌科属和白杆菌属()菌群含量增加,但生物效应不明。为维持噬菌弧菌N1对弧菌的控制,需要24 h左右重新补充投放。
噬菌弧菌N1;养殖水体;PCR-DGGE分析;细菌群落;弧菌;不可培养杆菌属
蛭弧菌及其类似物(-and-like organisms,BALOs),简称蛭弧菌,是一个革兰阴性(G-)细菌类群,菌体呈弧状或短杆状,具有寄生性,能够裂解一些革兰阴性(G-)细菌,如大肠杆菌、鼠伤寒沙门氏菌、幽门螺旋杆菌等[1]。蛭弧菌包括蛭弧菌科(Bdellovibrionaceae)和噬菌弧菌科(Bacteriovoracaceae),噬菌弧菌科分噬菌弧菌属(,Ba)和吞菌弧菌属(,Per)。蛭弧菌普遍存在于环境中[2-4],对环境微生物群落结构产生影响。
在水产养殖领域,随着我国高密度规模化养殖方式的普及,水产细菌性病害问题日益凸显,抗生素等药物的滥用导致药物残留加重,对食品安全问题造成威胁,直接对水产养殖业造成严重经济损失[5-6]。蛭弧菌因其特有的裂解特性、广泛的分布及种类繁多等优点,在生物防治领域具有一定的应用价值[7-9]。在应用蛭弧菌防治细菌性疾病上,早期Nakamura等[10]利用蛭弧菌治疗弗氏志贺菌()感染的兔眼角结膜炎。Nayak等[11]报道噬菌蛭弧菌成功防治稻田水中由黄单胞菌()引起的水稻白枯病的。国内关于蛭弧菌防治的研究在水产养殖应用方面报道较多,如应用淡水蛭弧菌防治嗜水气单胞菌和维氏气单胞菌等取得一定得成效[12-13]。目前国内外对噬菌弧菌的研究主要集中在其裂解机制方面[14-15],及对凡纳滨对虾弧菌病的防治问题[16],但噬菌弧菌对环境微生物群落结构影响目前尚未见报道。本研究主要以淡水(红鲤鱼)养殖环境和海水(仿刺参)养殖环境为实验条件,利用变性梯度凝胶电泳(DGGE)技术研究了噬菌弧菌N1在一个投放周期内对淡水红鲤鱼水体和海水刺参养殖水体中细菌群落结构的影响,以期了解噬菌弧菌N1在淡水和海水养殖的实际应用效果,为噬菌弧菌的细菌性疾病防治应用开发奠定理论基础。
1 材料与方法
1.1 材料
仿刺参,体长10 cm左右,体质量12 g左右,取自农业部北方海水增养殖重点实验室;红鲤鱼苗,体长5 cm左右,购于观赏鱼市场,且产地均为辽宁省大连市。
噬菌弧菌N1(sp. N1)分离自市售微生态制剂(淡、海水均适用)。
1.2 方法
1.2.1 蛭弧菌的分离及裂解能力测试
1.2.1.1 蛭弧菌的分离纯化 样品预处理:将蛭弧菌微生态制剂用0.45 μm微孔滤膜粗过滤以除去大部分杂菌和颗粒物质,滤出液经0.22 μm微孔滤膜收集蛭弧菌菌体。由生理盐水洗脱悬浮备用。
蛭弧菌的分离:将大肠杆菌作为宿主菌()进行扩大培养,吸取1 mL宿主菌和0.5 mL样品混合后加入4 mL冷却至40~50 ℃的质量分数0.7%琼脂培养基中,均匀混合后,立即灌注到预先制备的质量分数1.5%琼脂平板上,待凝固后于28 ℃正置培养3~5 d。
蛭弧菌的纯化:取单一噬菌斑以大肠杆菌为宿主菌在28 ℃、160 r/min进行摇床扩大培养48 h。同分离培养方法,再次双层琼脂平板培养,培养3次以上直到平板上出现形态一致的噬菌斑。将纯化后蛭弧菌培养液在4 ℃、3 000 r/min低速离心15 min,上清中即含有大量蛭弧菌细胞,该纯化菌命名为蛭弧菌N1。蛭弧菌浓度测定采用计数噬菌斑法获得。
1.2.1.2 蛭弧菌N1 16S rDNA序列扩增及序列分析 以噬菌蛭弧菌16S rDNA区特异性引物Bac676F和Bac1442R进行扩增。PCR反应体系(50 μL):10×DNA buffer 5 μL,dNTPs(2.5 mmol/L)4 μL,引物(10 μmol/L)2 μL,模板DNA 5 μL,Taq酶(2.5 U/μL)0.5 μL,ddH2O补足至50 μL。PCR反应条件为:94 ℃,5 min;94 ℃,30 s;53 ℃,30 s;72 ℃,30 s; 40个循环;72 ℃,7 min。PCR产物进行送样测序,测序结果用NCBI数据库的Nuclotide BLAST进行比对分析。
1.2.1.3 蛭弧菌N1裂解能力的测试 用实验室保存的6株菌作为宿主菌进行分离蛭弧菌裂解能力的测试。革兰氏阴性菌有大肠杆菌()、溶藻弧菌()、黄海希瓦氏菌()、灿烂弧菌()、哈氏弧菌(),革兰阳性菌为金黄色葡萄球菌()。将大肠杆菌等革兰氏阴性菌置于30 ℃恒温摇床,将金黄色葡萄球菌置于37 ℃的恒温摇床中培养,转速160 r/min,培养16~24 h。将培养液在6 000 r/min、4 ℃条件下离心15 min,弃上清,沉淀用灭菌生理盐水悬浮,4 ℃保存备用。采用双层琼脂平板法对分离的蛭弧菌进行裂解能力测试。每个宿主3个重复,记录噬菌斑数量,并在结束时测量噬菌斑最大直径。
1.2.2 蛭弧菌N1对淡水和海水养殖环境微生物的影响
1.2.2.1 实验分组 实验设置对照组、淡水组和海水组:淡水组养殖红鲤鱼(10尾),海水组(盐度31~32)养殖仿刺参(10头),两组均添加50 mL纯化的蛭弧菌N1菌液,致添加后终浓度为1×103PFU/mL,每个水槽注水量为50 L,实验开始时(0 h)加入20 mL含2 g蛋白胨和0.5 g酵母粉的无菌营养液[17],对照组不添加噬菌弧菌N1菌液,所有组均2个平行。室内养殖,保证温度一致(25 ℃左右),每个水槽放置一个气石,暂养期为7 d,不换水,实验期为48 h。
1.2.2.2 样品采集和处理 在添加蛭弧菌N1菌液前30 min内采集第一次水样,即0 h时刻,加入蛭弧菌N1后4、12、24、48 h分别采集水样1 L。所有水样均在当天处理完成:各取500 mL水样用0.22 μm微孔滤膜在无菌条件下抽滤,无菌勺刮取滤膜上的菌体,并用TE缓冲液冲洗,溶于总计4 mL缓冲液中,用于总细菌、弧菌和蛭弧菌的计数,剩余样品-20 ℃保存备用,用于DNA的提取。
1.2.2.3 微生物计数 将过滤收集的细菌进行梯度稀释,计数总细菌数量时,海水样品采用2216E固体平板培养,淡水样品采用NB固体培养基培养。弧菌计数则用TCBS培养基检测。蛭弧菌计数用双层自来水琼脂平板进行计数。以上各稀释梯度样品均设置两个重复。
1.2.2.4 水环境中细菌基因组DNA提取 取适量样品于12 000 r/min离心10 min,取沉淀,加入180 μL溶菌酶充分混匀,置于37 ℃水浴锅温浴30 min,使用细菌基因组DNA提取试剂盒篇[天根生化科技(北京)有限公司]提取基因组DNA。取5 μL DNA样品用10 mg/mL的琼脂糖凝胶电泳检测,其余存于-20 ℃备用。
1.2.2.5 16S rDNA V3 区扩增及变性梯度凝胶电泳(DGGE) 分别提取的五批次40个样品(编号1-40)总基因组DNA作为模板,用V3区特异性引物进行PCR扩增,选择细菌16S rRNA V3区引物357F-40GC5′-CGCCCGCCGCGCCCCGCGCCCGGCCCGCCGCCCCCGCCCCCCTACGGGAGGCAGCAG-3′)和518R(5′-ATTACCGCGGCTGCTGG-3′)作为引物[18],扩增产物片段大小约为230 bp。使用Bio-Rad DcodeTM system(Bio-Rad )对16S rDNA PCR扩增产物进行DGGE分析。在变性梯度为30% ~ 60%的质量分数8% 聚丙烯酰胺凝胶(丙烯酰胺和甲叉双丙烯酰胺的质量比为 37.5∶1)上,电泳5 h。凝胶成像仪上拍照,分析V3区电泳条带差异。
对DGGE中部分优势电泳条带通过无菌刀片切胶回收、纯化,纯化产物分别用 pEASY-T1 载体连接,转化DH5α,每个条带各选取2个阳性克隆送生工生物工程(上海)股份有限公司测序,测序引物M13F-47和M13R-48。
1.2.2.6 数据分析 PCR-DGGE图谱采用Quantity One(Bio-Rad)软件分析。将优势条带和变化明显的条带割胶回收后进行测序,测序所得的16S rDNA序列经GenBank数据库比对分析给出结果。
2 结果与分析
2.1 蛭弧菌N1分离与鉴定
蛭弧菌N1在双层琼脂平板上生长48 h后形成清晰可见的圆形噬菌斑,30 ℃培养4 d之后菌斑直径可达4 ~ 5 mm(图1所示)。PCR扩增产物测序分析得知,N1的16S rDNA基因片段大小为766 bp,测序结果表明N1菌株为噬菌弧菌属菌株,与JS5具有95 %的相似性,命名为噬菌弧菌N1(sp. N1)。
2.2 噬菌弧菌N1的裂解能力
实验表明,噬菌弧菌N1对选取的6株宿主菌均有较好的裂解能力,除大肠杆菌外,最大噬菌斑直径均可达到4 mm,且出斑时间短,在24 ~ 48 h即可形成清晰可见的噬菌斑,最大噬菌斑直径达4.5 mm,见表1。裂解效价为哈氏弧菌最高,达到了600 PFU/mL,最小的为灿烂弧菌,裂解效价为188 PFU/mL。

a,纯化前噬菌斑;b,纯化后噬菌斑
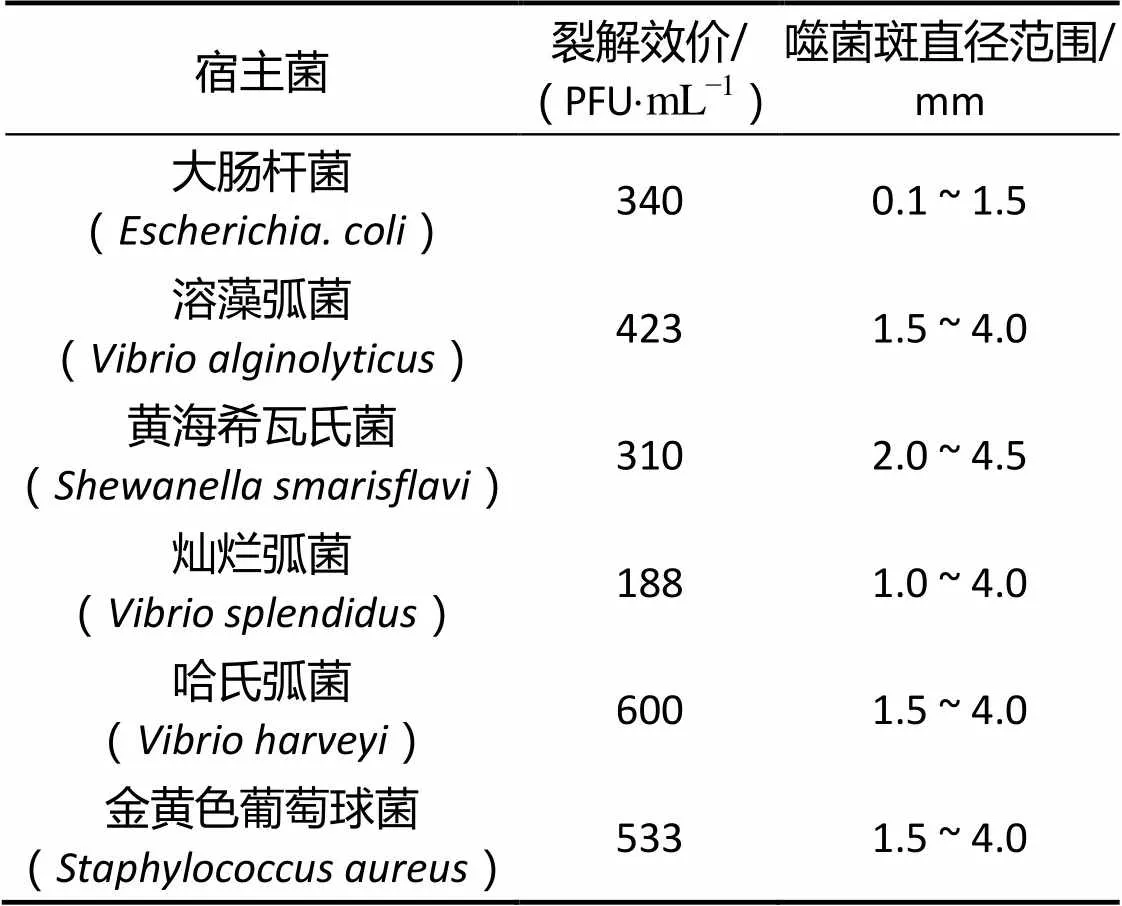
表1 Bacteriovorax sp. N1对不同宿主菌的裂解效价
2.3 噬菌弧菌N1对淡水和海水养殖水体微生物总量的影响
淡水组中,实验组和对照组中总可培养细菌总含量和弧菌含量的变化趋势较一致。弧菌含量在0 ~ 4 h呈迅速上升趋势,随后含量逐渐减少至峰值的一半,在第24 h后又趋于增长趋势,实验组弧菌含量在12、24 h显著低于对照组(< 0.05)(图2a)。实验组中蛭弧菌含量变化明显,在0 ~ 4 h时由初始浓度1×103PFU /mL增长至1.5×104PFU /mL,而随后含量持续降低,到24 h时降低到8×103PFU /mL,到48 h几乎检测不到(图2 b)。总可培养细菌含量在第1 d内迅速增长,在第24 h时达到峰值,在第2 d内含量逐渐下降至接近初始水平,两组间差异不显著(0.05)(图2 c)。
海水组中,实验组和对照组总可培养细菌数和弧菌数变化趋势较一致。实验组弧菌含量也在12、24 h显著低于对照组(< 0.05)(图3d)。蛭弧菌含量变化趋势与淡水组基本一致,但是蛭弧菌浓度高于淡水环境,最高可达2.5×104PFU /mL,而且在48 h后水中蛭弧菌的含量可达到8×103PFU /mL(图3e)。可培养细菌总数在48 h内总体呈增长趋势,实验组在12 ~ 24 h之间有明显下降趋势(< 0.05),弧菌含量也有此现象(< 0.05)(图3f)。
综上分析可知,添加噬菌弧菌N1菌液会使淡水和海水养殖水体中异氧细菌和弧菌含量有所下降,表现为具有一定的防控作用,但对弧菌的防控效果显示在24 h效果显著。

a为弧菌数;b为蛭弧菌数;c为可培养总细菌数

a为弧菌数;b为蛭弧菌数;c为可培养总细菌数
2.4 淡水及海水环境中基因组DNA提取和PCR扩增
PCR扩增结果经质量分数2%琼脂糖凝胶电泳检测,40个样品均得到大小为200 bp左右的扩增基因片段,且条带较亮。
2.5 PCR-DGGE分析噬菌弧菌N1对海水和淡水养殖环境细菌群落的影响
DGGE分析结果如图4、5所示。DGGE图谱上不同条带代表不同的微生物种类,条带亮度大小代表微生物相对数量的多少,越亮则代表微生物数量越多。由图4、5可以看出,添加噬菌弧菌N1后不同取样时间淡水组实验组和海水实验组条带较对照组均存在一定的差异。淡水组和海水组DGGE显示优势条带(a1 ~ a6, c1 ~ c2)和部分差异条带(b5, d1)被切胶回收和克隆测序,结果见表2,主要变化的微生物群落有弧菌属()、不可培养杆菌属(uncultured bacterium)、假单胞菌属()、红杆菌科属()等。
随着时间的延长淡水实验组菌群丰度下降较明显,而对照组基本维持在同一水平;淡水组加入噬菌弧菌N1后与加入噬菌弧菌N1前(0 h)菌群数量及优势类群出现部分差异变化。0 h和4 h以弧菌(, a1)和不可培养杆菌属(uncultured bacterium, a5)为主要优势类群,在12、24、48 h时这两个优势类群有明显的减少,弧菌属(a1)甚至变为非优势类群,随之新增了假单胞菌属(, a3)和红杆菌科属(, a6)为优势类群。在4 h时新增的差异条带b5经测序检测为噬菌弧菌N1,但因含量较低,在12 h之后不再有明显可见的条带出现。在24 h时实验组微生物菌群丰度值达到最低,即菌群数量最少。
海水实验组和对照组随时间变化其微生物群落丰度值逐渐增加后回落。海水组微生物菌群结构除部分优势类群有一定变化,其他非优势类群变化较少。白杆菌属(,c1)在0 h和4 h并不完全是优势类群,而在12 h及之后以绝对优势菌群存在,不可培养杆菌属(uncultured bacterium, c2)在0 h为主要的优势菌群,在12 h时变成非优势菌群,随后又成为优势菌群。d1经检测为噬菌弧菌N1,且因为含量较低,在之后并没有明显条带出现。其他非优势类群在海水实验组和对照组中数量均有所增加,而没有出现如淡水实验组中明显减少现象。
a1―a6为优势菌群;b1―b10为差异菌群
a1―a6 are dominant microflora; b1—b10 are different from microflora
图4 噬菌弧菌N1对淡水组水体细菌菌群结构影响的DGGE分析及电泳识别
Fig. 4 DGGE of the effect of Bacteriovorax sp. N1 on bacterial community structure in freshwater
c1,c2是优势菌群;d1―d5是差异菌群
c1, c2 are dominant microflora; d1―d5 are different from microflora
图5 噬菌弧菌N1对海水组水体细菌菌群结构影响的DGGE分析及电泳识别
Fig. 5 DGGE of the effect of Bacteriovorax sp. N1 on bacterial community structure in seawater
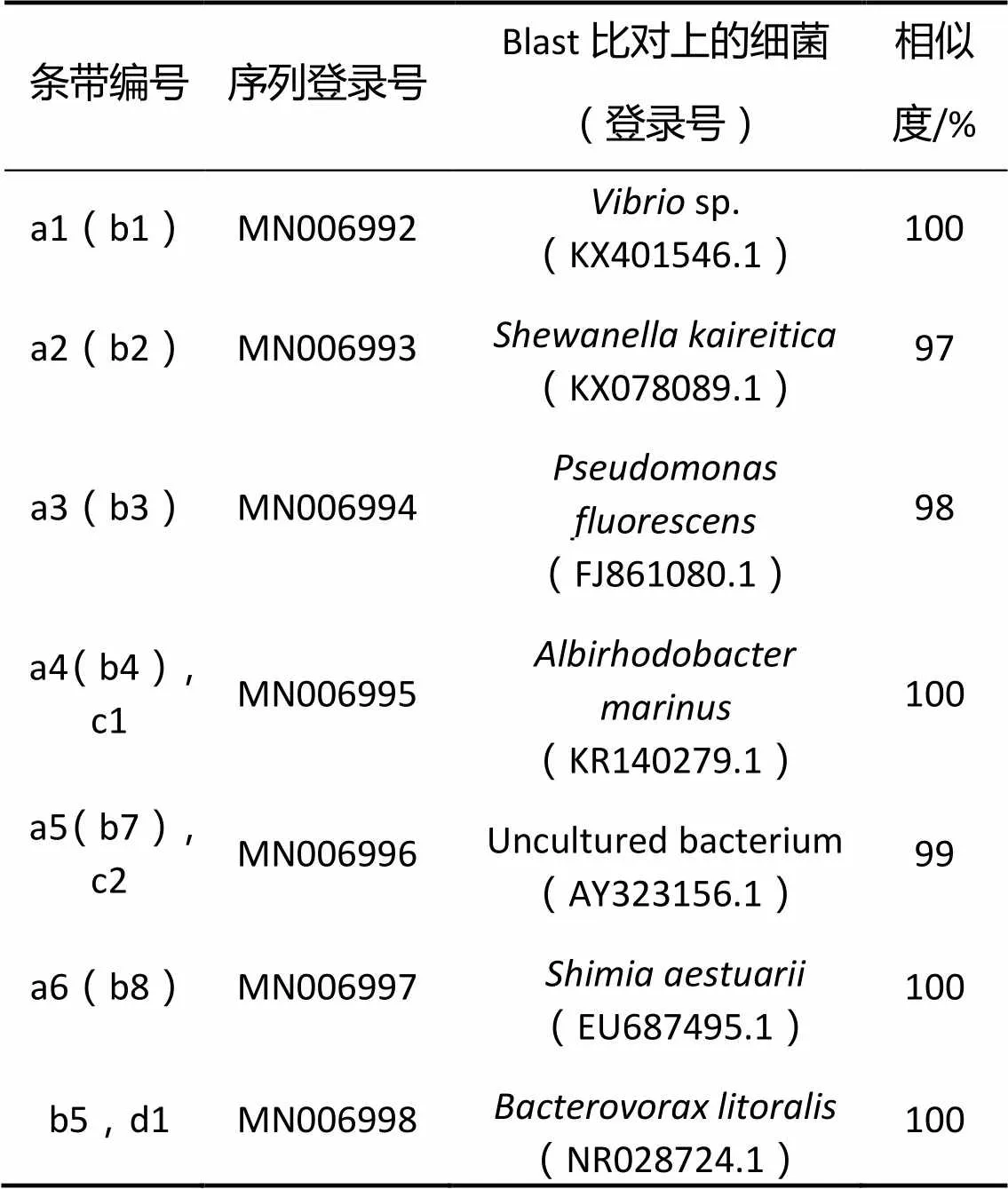
表2 DGGE回收条带序列分析
3 讨论
蛭弧菌对环境细菌数量、群落结构和丰度的影响主要取决于蛭弧菌裂解特性[19-23]。水产养殖环境中大多数病原菌都是革兰氏阴性菌,有研究[24]报道蛭弧菌更易裂解环境中的弧菌。本研究主要研究噬菌弧菌N1在一个投放周期内对养殖环境中的微生物总数及群落结构或丰度的影响,DGGE结果显示淡水组加入噬菌弧菌N1后菌群数量及优势类群出现部分差异变化,原优势群落弧菌属(,a1)和不可培养杆菌属(uncultured bacterium, a5)12 h后有明显的减少,弧菌(a1)甚至变为非优势类群,该结果与平板培养结果一致,而增加了假单胞菌属(,a3)和红杆菌科属(, a6)两个优势类群。海水组的优势菌群不可培养杆菌属(uncultured bacterium, c2)在12 h后明显减少,在24 h后又恢复为优势菌群,而白杆菌属(A)在12 h后增加成为优势菌群。平板培养实验显示噬菌弧菌N1对海水中的弧菌也有较明显的裂解效果,尤其是12~24 h,实验组弧菌含量显著下降(< 0.05),但在DGGE实验中没有发现这一现象,原因可能是在该海水养殖环境中弧菌还未形成优势菌群,因此在分析中忽略了弧菌条带的变化。综合16S rDNA V3区扩增及DGGE分析可知,噬菌弧菌N1主要对弧菌和不可培养杆菌属等细菌具有明显的裂解作用致其含量下降。温崇庆等[25]关于噬菌弧菌DA5对对虾池育苗水体中细菌群落结构变化的研究表明较高含量的DA5在一定时间内对育苗水体中的弧菌和异养细菌有较明显的削减效果,这与本研究所得结果具有一致性。
本研究发现噬菌弧菌N1也可使淡水中假单胞菌属()和红杆菌科属微生物含量增加,使海水中的白杆菌属()细菌含量增加,而这些微生物对养殖动物的生物学效应还需要进一步研究。尤其假单胞菌属的部分种在一定条件下具有致病性,噬菌弧菌N1作为微生态制剂应用在红鲤鱼养殖环境中,假单胞菌属菌含量增加引起的效应还属未知,因此噬菌弧菌N1的长期微生物环境效应需要进一步研究。此外,本研究中噬菌弧菌N1在24 h时数量降至最低,因此施用频率应该为每24 h补充一次噬菌弧菌N1,才能较好得使噬菌弧菌N1对弧菌的控制效应得以累积。
单一蛭弧菌的裂解范围是有限的,而养殖水体中微生物菌群结构复杂,因此可选用不同裂解范围的蛭弧菌混合使用,可能会达到协同作用效果[26]。蛭弧菌也可与其他诸如增加营养,改善水质或增强免疫等的功能菌组合使用,以实现多目的同时进行,优势互补。水体中微生物群落结构的研究准确性很大程度上依赖于方法和技术的进步[27]。DGGE能够直观的比较和分析微生物群落结构、丰度变化规律,但分析图谱只能反映可视条带的数量和变化,对含量极低的群落很难呈现出来,对这部分微生物影响的研究可用高通量测序的方法进行。此外,噬菌弧菌N1对养殖环境中水质指标的影响,以及其与微生物群落变化间的关系还需要进行进一步的研究,本文研究了一个施用周期噬菌弧菌N1对环境微生物群落的影响,而其长时间施用对环境乃至刺参肠道微生物群落组成及功能的影响也需要考虑。
[1] DWIDAR M, MONNAPPA A K, MITCHELL R J. The dual probiotic and antibiotic nature of[J]. BMB Reports, 2012, 45(2): 71-78.
[2] DAVIDOV Y, JURKEVITCH E. Diversity and evolution of-and-like organisms (BALOs), reclassification ofasgen. nov., comb. nov., and description of theclade as. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2004, 54(5): 1439-1452.
[3] 韩民泳, 陈慧黠, 斯晗, 等. ARDRA分型测定刺参养殖环境中蛭弧菌多样性[J]. 微生物学通报, 2016, 43(2): 254-261.
[4] 韩民泳, 陈慧黠, 斯晗, 等. 刺参消化道中蛭弧菌类的生物多样性分析[J]. 微生物学杂志, 2015, 36(1): 44-48.
[5] 赵玲敏, 左妍斐, 黄力行. 水产健康养殖中病害防控研究进展[J]. 安徽农业科学, 2018, 46(28): 18-21; 52.
[6] HAO R Z, ZHAO R T, QIU S F, et al. Antibiotics crisis in China[J]. Science, 2015, 348(6239):1100-1101.
[7] DEFOIRDT T, SORGELOOS P, BOSSIER P. Alternatives to antibiotics for the control of bacterial disease in aquaculture[J]. Current Opinion in Microbiology, 2011, 14(3): 251-258.
[8] SARA R, Bacterial arms race revs up[J]. Nature, 2015, 521(7553): 402-403.
[9] ELIZABETH S R, GAREY L.as therapeutic agents: a predatory renaissance[J]. Nature Reviews Microbiology, 2004, 2(8): 669-675.
[10] NAKAMURA M. Alteration of Shigella pathogenicity by other bacteria[J]. The American Journal of Clinical Nutrition. 1972, 25(12): 1441-1451.
[11] NAYAK N, PANI B K, DAS S R. Biological control of bacterial blight of rice (pv) by[J]. Research in Plant Disease, 2002, 17(2): 381-383.
[12] 李永文, 朱文漓, 顾继锐, 等. 大口鲶病原菌蛭弧菌的分离及其生物学特性研究[J]. 淡水渔业, 2006, 36(2): 9-12.
[13] CAO H, HOU S, HE S, et al. Identification of asp. isolate as a potential biocontrol bacterium against snakehead fish-pathogenic[J]. Journal of Fish Diseases, 2014, 37(3): 283-289.
[14] KANESHIRO E S, HUNT S M, WATANABE Y.proliferation and predation without sphingophosphonolipids[J]. Biochemical and Biophysical Research Communications, 2008, 367(1): 21-25.
[15] JAYASIMHULU K, HUNT S M, KANESHIRO E S, et al. Detection and identification ofUKi2 sphingophosphonolipid molecular species[J]. Journal of the American Society for Mass Spectrometry, 2007, 18(3): 394-403.
[16] WEN C Q, XUE M, LIANG H F, et al. Evaluating the potential of marinesp. DA5 as a biocontrol agent against vibriosis inlarvae[J]. Veterinary Microbiology, 2014, 173(1/2): 84-91.
[17] 温崇庆, 丁贤, 薛明, 等. 一株海洋蛭弧菌类生物对海水细菌的生物控制和水质的影响[J]. 中山大学学报(自然科学版), 2008, 47(3): 85-88.
[18] MUYZER G, DE WAAL E C, UITIERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16SrRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
[19] 陆友云, 温崇庆, 李志桦, 等. 不同感染复数下海洋蛭弧菌 DA5 对溶藻弧菌的控制效率[J]. 广东海洋大学学报, 2018, 38(2): 30-36.
[20] KOVAL S F, BAYER M E. Bacterial capsules: no barrier against[J]. Microbiology, 1997, 143: 749-753.
[21] YAIR S, YAACOV D, SUSAN K, et al. Small eats big: ecology and diversity ofand like organisms, and their dynamics in predator-prey interactions[J]. Agronomie, 2003, 23(5/6): 433-439.
[22] ROGOSKY A M, MOAK P L, EMMERT E A. Differential predation by109J[J]. Current Microbiology, 2006, 52(2): 81-85.
[23] SCHOEFFIELD A J, WILLIAMS H N. Efficiencies of recovery offrom brackish-water environments by using various bacterial species as prey[J]. Applied and Environmental Microbiology, 1990, 56(1): 230-236.
[24] WILLIAMS F N, PIÑEIRO S. Ecology of the predatory-and-like organisms[J]. E. Jurkevitch Predatory Prokaryotes.: Springer-Verlag Berlin Heidelberg, 2007, 4: 213-248.
[25] 温崇庆, 梁华芳, 丁贤, 等. 海洋蛭弧菌类生物 DA5 对凡纳滨对虾育苗期幼体和水质的影响[J]. 热带海洋学报, 2010, 29(6): 147-152.
[26] 陈康勇, 钟为铭, 高志鹏. 蛭弧菌在水产养殖中应用研究进展[J]. 水产科学, 2018, 37(2): 283-288.
[27] 夏围围, 贾仲君. 高通量测序和DGGE分析土壤微生物群落的技术评价[J]. 微生物学报, 2014, 54(12): 1489-1499.
Effect ofsp. N1 on the Bacterial Community in the Freshwater and Seawater Environment Using PCR-DGGE
CHEN Hui-xia1,HAN Min-yong1,YU Jia-hao1,LIU lin2
(1.,,,, 116023. 2.,,,266109,)
【】To study the effects ofsp. N1 on the total number of bacteria, Vibrio and bacterial community structure within an addition cycle in seawater and freshwater aquaculture environments. 【】N1 was isolated from commercially available microecological preparations. Molecular identification was carried out and its lysis effect was preliminarily determined. In addition, high concentration of N1 strain was splashed evenly into freshwater tank and seawater tank containing the red carprespectively. The number of bacteria, Vibrio and structure of bacterial community in the water environment within 48 h were analyzed by plate counting and Polymerase Chain Reaction-Denaturing Gradient Gel Electrophoresis (PCR-DGGE) respectively.【】The strain N1 was identified assp.. It had lysis effect on,,,,and. WhenN1 was added to the fresh and marine culture environments for 12―24 hours, the content of Vibrio in both environments was significantly reduced (< 0.05). Results from DGGE showed that by addingsp. N1, the dominant(a1) and uncultured bacterium (a5) counts decreased significantly after 12 h in freshwater group, but(a3) and(a6) counts were increased. The dominant bacterium uncultured bacterium (c2) became a non-dominant species after 12 h in the seawater group, however,(c1) has become the new dominant bacterium. The content ofsp. N1 decreased to the lowest level within 24 hours. 【】sp. N1 can split Vibrio, uncultured bacterium, and also increase the number of microflora of,andin freshwater and seawater aquaculture environment. At present, the biological effects of these newly increased microflora are not known. In order to control the outbreak of Vibrio in fish cluture,sp. N1 should be replenished in 24 hours.
sp. N1; aquaculture environment; PCR-DGGE analysis; bacterial community; vibrio; uncultured bacterium
Q939.99
A
1673-9159(2019)05-0008-08
10.3969/j.issn.1673-9159.2019.05.002
2019-05-07
辽宁省教育厅计划项目(L2012255)
陈慧黠(1980-),女,博士,讲师,研究方向为微生物学及微生物应用。E-mail:chenhuixia@dlou.edu.cn
刘琳(1979-)女,博士,讲师,研究方向为微生物应用。E-mail:liulin@qau.edu.cn
陈慧黠,韩民泳,于佳豪,等. 噬菌弧菌N1对淡水和海水养殖水体细菌群落影响PCR-DGGE分析[J]. 广东海洋大学学报,2019,39(5):8-15.
(责任编辑:刘朏)
