新疆哈密地区原驼乳中乳酸菌多样性分析
2019-09-24张苗苗倪永清
张苗苗 倪永清

摘要[目的]对新疆哈密地区原驼乳中乳酸菌的多样性进行分析。[方法]采用菌落培养及Rep-PCR指纹图谱、16S rRNA基因测序和系统发育关系相结合的方法研究原驼乳内乳酸菌的菌群分布。[结果]从9份原驼乳中分离出65株乳酸菌,分别归属于Lactococcus、Streptococcus、Enterococcus、Lactobacillus和Leuconostoc 5个属,其中Leuconostoc所占比例最大,达到26%。[结论]新疆哈密地区原驼乳中蕴含丰富的乳酸菌菌种资源,为开发新疆地区益生乳酸菌提供了依据。
关键词 原驼乳;乳酸菌;16S rRNA基因测序
中图分类号 TS207.3文献标识码 A
文章编号 0517-6611(2019)15-0163-04
doi:10.3969/j.issn.0517-6611.2019.15.046
开放科学(资源服务)标识码(OSID):
Abstract[Objective]To analyze the diversity of lactic acid bacteria in raw camel milk in Hami area of Xinjiang.[Method]The diversity of lactic acid bacteria was analyzed by using pure culture,repetitive genomic fingerprinting (RepPCR) analysis patterns and 16S rRNA gene sequence and phylogenetic relationship in the raw camel milk.[Result]A total of 65 strains of lactic acid bacteria were obtained from nine raw camel milk samples.The results showed that the isolated lactic acid bacteria mainly comprised five genera:Lactobacillus,Enterococcus,Streptococcus, Lactococcus, and Leuconostoc.The dominant genus was Leuconostoc,which is up to 26%.[Conclusion]The abundant diversity of lactic acid bacteria in the raw camel milk of Hami area in Xinjiang provided rich resources for exploiting probiotic lactic acid bacteria products.
Key words Raw camel milk;Lactic acid bacteria;16S rRNA gene sequence
作者简介 张苗苗(1988—),女,新疆石河子人,硕士研究生,研究方向:食品生物技术。
*通信作者,博士,教授,从事食品微生物、生物技术研究。
收稿日期 2019-03-22;修回日期 2019-05-07
随着经济水平的不断提高,人们更加关注健康问题。乳制品不再仅限于牛乳,营养丰富的羊乳和骆驼乳等也渐渐进入人们的视野[1]。骆驼,早在几千年以前就被驯化,作为一种交通工具,它为物品的运输和文化的传播做出了不可磨灭的贡献[2]。当前我国有25万峰的骆驼存栏数,新疆作为一个畜牧大区,骆驼存栏数就达到了16万峰,居全国之首[3]。
骆驼奶有“沙漠软白金”和“长寿奶”的美誉[4],其味道微甜,而且与人奶中的营养成分很接近,比牛奶的营养价值还高[5]。骆驼乳中的乳蛋白、乳脂肪、乳糖、干物质含量比牛乳略高,与牛乳相比含有丰富的矿物质元素,钙、磷、钾的含量更高,镁的含量相对較低[1]。据现代药理研究表明,驼乳具有抗氧化、保肝、消炎、抗菌的作用[6],常喝驼乳可以滋养美容,增强抵抗力[7],也可用于治疗糖尿病、结核病和女性疾病,防止儿童佝偻病等[8]。驼奶作为新疆的主要特色乳,其研究和利用价值极其重要。目前,随着我国骆驼数量的逐渐下降,国家出台了一系列鼓励和支持特种乳发展的产业政策,新疆具有得天独厚的自然环境以及丰富的饲草资源[9],一些少数民族自古就有饲养骆驼的传统,饮用驼乳及加工驼乳制品也有非常悠久的历史。在长期的自然演变中[10],对人类有益的微生物群落在传统发酵乳制品中被大量保留下来,特别是乳酸菌[11]。这些乳酸菌资源是探索未知有益乳酸菌的绝佳宝库[12]。新疆原奶资源丰富,采用古老、传统的方法,将发酵性能好、生命力强、传代好、具有地方特色的乳酸菌菌株从当地牧民生产的发酵奶制品中分离出来具有重要意义。这为今后研发和加工驼乳积累了经验[13]。目前,国内外对于驼奶的研究主要集中于成分、微生态、医药价值等方面[14],在我国有关驼奶和酸驼奶的报道极少[15]。为此,笔者对新疆哈密地区原骆驼奶乳酸菌进行初步分离、鉴定和遗传多样性分析,以促进原料骆驼奶和酸骆驼奶的开发和研究并筛选出具有良好发酵性能的菌株,旨在为生产乳酸菌发酵剂提供理论依据。
1 材料与方法
1.1 材料
1.1.1 主要试剂和仪器。
用于PCR扩增的全套试剂及扩增引物购自TaKaRa公司;PCR 仪为德国Biometra公司Tprofessional;电泳仪为美国BioRad公司PowerPac Universal;凝胶成像系统为法国Vilber多功能成像系统;生物显微镜为徕卡DM3000;高速冷冻离心机为Fresco21型(德国Thermo公司)。
1.1.2 样品和培养基。
该研究9份原驼奶于2017年9月采自哈密地区(HMT)各草原牧民家里。无菌收集约40 mL样品于50 mL无菌离心管中,置于4 ℃的车载冰箱,24 h内运回实验室,在无菌操作台上立即处理。实验人员在无菌条件下收集样品,后由实验人员立即将装有25 mL奶样的无菌离心管放置在4 ℃的车载冰箱内,24 h内运回实验室,-20 ℃保存,并尽快进行乳酸菌的分离。
MRS培养基:蛋白胨10.0 g,牛肉膏10.0 g,酵母提取物5.0 g,K2HPO4 2.0 g,柠檬酸二铵2.0 g,乙酸钠5.0 g,葡萄糖20.0 g,吐温-80 1 mL,MgSO4·7H2O 0.58 g,MnSO4·4H2O 025 g,琼脂15.0 g,蒸馏水1 000 mL,pH 6.2~6.4。
M17培养基和乳酸杆菌选择性培养基:青岛高科园海博生物技术有限公司。
1.2 方法
1.2.1 菌株分离纯化。
参考Lee等[16]的方法,在无菌条件下吸1 mL 样品于9 mL的无菌生理盐水中,振荡混匀,采用梯度稀释液涂板法在MRS固体培养基、乳杆菌选择培养基、M17琼脂培养基上37 °C倒置培养48 h。从3种选择性培养基中挑取菌落颜色、大小、形态等不同的单菌落,接种于 MRS 琼脂培养基上,连续转接培养3次后,观察记录菌落形态并进行接触酶试验。挑取过氧化氢酶试验为阴性菌落接种于MRS肉汤培养基中,经24 h培养后进行革兰氏染色,在显微镜下观察记录细胞形态及排列方式,最终将革兰氏染色阳性、过氧化氢酶试验阴性的菌株确定为疑似乳酸菌,在新鲜培养基中补充25% 的甘油重悬菌体置于-80 ℃ 保藏。
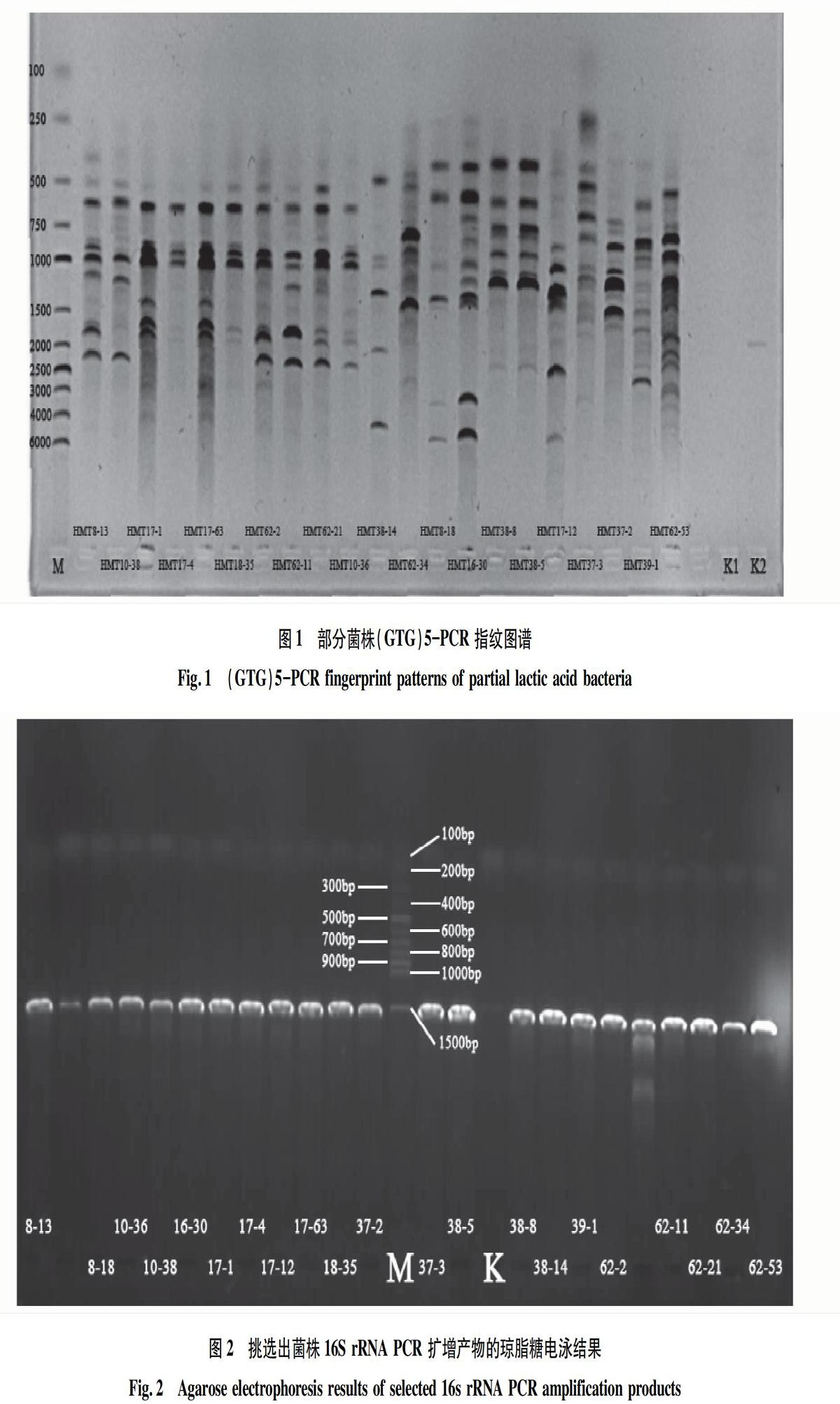
1.2.2 乳酸菌Rep-PCR指纹图谱分析。参考Justé等[17]的方法提取菌株DNA,选用(GTG)5(5′-GTGGTGGTGGTGGTG-3′)对疑似乳酸菌菌株进行Rep-PCR的扩增。将PCR产物用1.5%的琼脂糖凝胶进行检测,在1×TAE Buffer中100 V电泳1.5 h。用凝胶成像系统观察并进行拍照分析[18]。以(GTG)5(5′-GTGGTGGTGGTGGTG-3′)为引物,以筛选出乳酸菌的DNA为模板,进行Rep-PCR的扩增。将PCR的扩增产物在1.5%(W/V)的琼脂糖凝胶上上样,电泳液为1×TBE Buffer,电压100 V,电泳30~60 min,待样品移至3/4位置时停止电泳。在紫外凝胶成像仪中观察电泳结果并进行拍照,用软件Gel Compar Ⅱ 对DNA图谱进行聚类分析,电泳后条带完全一致的菌株通常被认为是属于同一个属或者同一物种,因此可将分离筛选到的菌株对照指纹图谱归为若干组种系型,然后从每组中选取至少1~2株代表菌株进行测序分析。
1.2.3 16S rRNA 基因的系统进化。
选取1~2株指纹图谱带型相同菌株
作为代表,采用通用引物27f/1492r进行16S rRNA扩增[19]。扩增产物送至上海生物工程(上海)股份有限公司测序,将测序得到的16S rRNA序列與GEN-BANK数据库中的标准菌株进行BLAST同源性比对分析。从数据库获得相关属、种的16S rRNA基因序列,运用MEGA 5.0软件构建系统发育进化树[20]。
2 结果与分析
2.1 菌株分离纯化
依据菌落特征、细胞形态特征从9份样品中初步分离纯化得到176株纯培养物,经革兰氏染色、过氧化氢酶试验得到69株疑似乳酸菌(表1)。其中大部分菌株菌落颜色为白色、灰白色,边缘整齐的圆形凸起,表面较湿润,易挑起,直径大部分在0.5~1.0 mm。
2.2 菌株Rep-PCR指纹图谱
根据指纹图谱条带大小和数量差异,从69株疑似乳酸菌中挑出21株代表菌株,指纹图谱如图1。
47卷15期张苗苗等 新疆哈密地区原驼乳中乳酸菌多样性分析
2.3 16S rRNA 测序结果
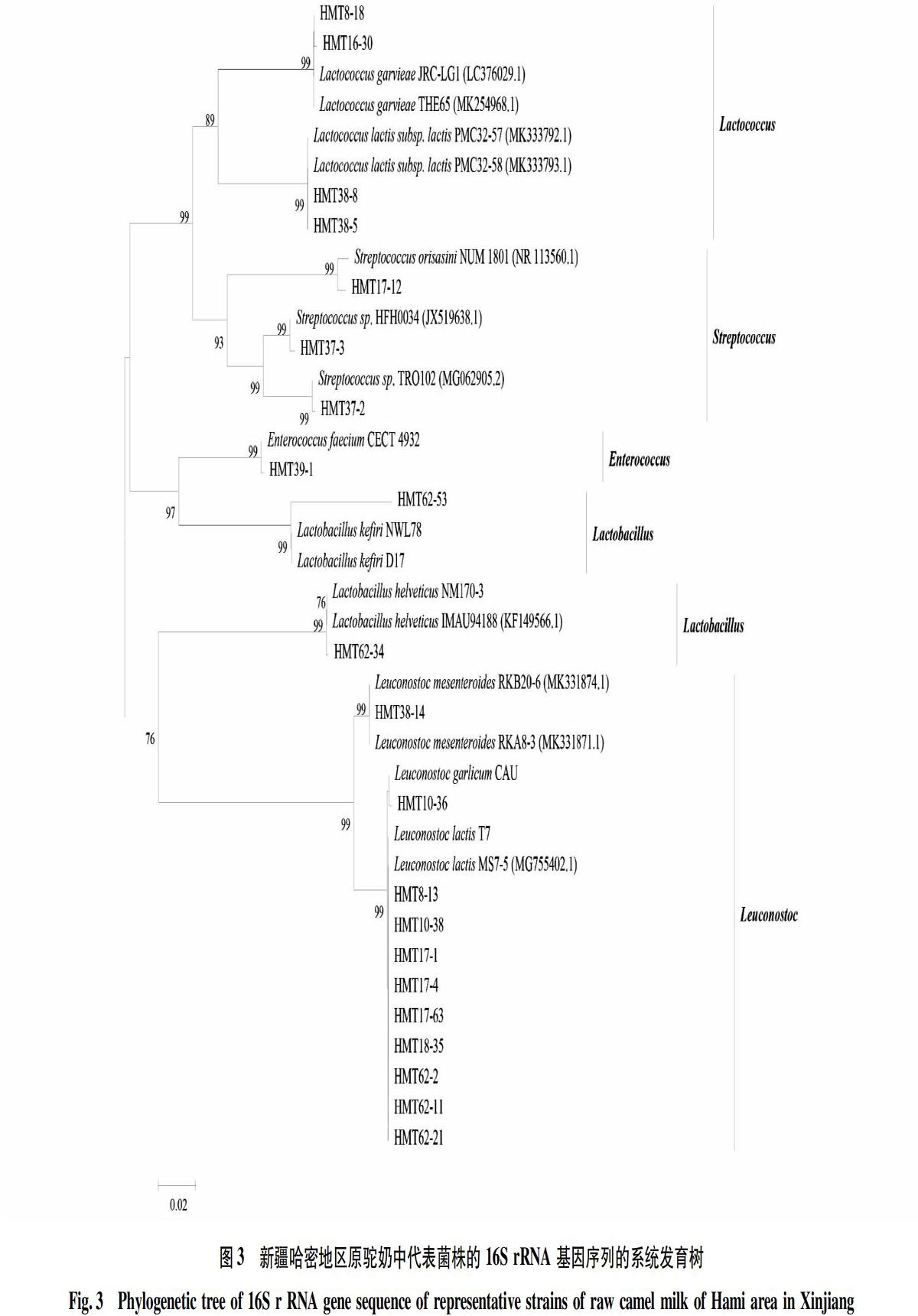
将提取出来的DNA以27F和1492R为引物经过PCR扩增,产物可通过电泳检测是否有DNA,扩增产物的电泳结果如图2所示。
由图2可以清楚看到,在1 500 bp处有一条明亮的条带,说明挑选出的菌株16S rRNA基因扩增的产物可以满足送去测序的基本要求。笔者将出现弥漫现象和不满足测序的菌株剔除,最终将21株满足测序要求的菌株低温保存运往上海生工生物工程技术服务有限公司进行测序分析。
2.4 系统发育树的构建和乳酸菌的鉴定结果 将测序结果通过BLAST在线分析,与NCBI数据库中的已知序列进行对比,选取同源性大于98%的序列建立系统发育树(图3)。
由图3可以看出,21株代表菌株分别被归属于Lactococcus、Lactobacillus、Leuconostoc、Streptococcus 和 Enterococcus 5个属,Lactococcus garvieae (2株)、Leuconostoc lactis(9株)、Lactobacillus helveticus(1株)、 Lactococcus lactis(2株)、Streptococcus orisasini(1株)、Streptococcus sp(2株)、Enterococcus faecium(1株)、Lactobacillus kefiri(1株)、Leuconostoc mesenteroides(1株)、Leuconostoc garlicum(1株)10个种。
2.5 原驼乳中乳酸菌优势菌种的分析
将从新疆哈密地区采集回来的9份驼奶样品,通过生理生化鉴定以及Rep-PCR指纹图谱分析并结合16S rRNA 基因序列鉴定分析,确定的乳酸菌有65株。
[12]王一迪.新疆伊犁地區原牛乳中乳酸菌的分离鉴定与系统发育研究[D].石河子:石河子大学,2017.
[13]徐敏,陆东林,罗晓红,等.新疆双峰驼驼乳成分及特性分析[J].中国奶牛,2014(15):49-52.
[14]孙天松,王俊国,张列兵,等.中国新疆地区酸马奶中乳酸菌生物多样性研究[J].微生物学通报,2007,34(3):451-454.
[15]马兆瑞,秦立虎.现代乳制品加工技术[M].北京:中国轻工业出版社,2010.
[16]LEE S,LEE J,JIN Y I,et al.Probiotic characteristics of Bacillus strains isolated from Korean traditional soy sauce[J].LWTFood Science and Technology,2017,79:518-524.
[17]JUST A,THOMMA B P H J,LIEVENS B.Recent advances in molecular techniques to study microbial communities in foodassociated matrices and processes[J].Food microbiology,2008,25(6):745-761.
[18]GEVERS D,HUYS G,SWINGS J.Applicability of repPCR fingerprinting for identification of Lactobacillus species[J].FEMS Microbiology Letters,2001,205(1):31-36.
[19]倪亚雯,兰国伟,杨尚娇,等. 新疆不同地域发酵乳品中 Lactobacillus 多样性的研究[J]. 现代食品科技,2016,32(6):104-109.
[20]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular biology and evolution,2011,28(10):2731-2739.
[21]李伟程,侯强川,于洁,等.传统发酵乳制品中微生物多样性研究[J].食品工业科技,2018,39(1):131-136.
[22]李远,巴吐尔,张小燕,等.新疆哈萨克族传统发酵驼乳中乳酸菌的分离鉴定[J].中国食物与营养,2011,17(1):54-58.
[23] ERCOLINI D,RUSSO F,FERROCINO I,et al.Molecular identification of mesophilic and psychrotrophic bacteria from raw cow's milk[J].Food microbiology,2009,26(2):228-231.
[24]TAMURA K,PETERSON D,PETERSON N,et al.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J].Molecular biology &evolution,2011,28(10):2731-2739.
