基于线粒体COI基因的南海浮游介形类斜突浮萤(Proceroecia procera)单倍型与种群遗传结构研究
2019-09-24徐磊季莹莹黎红杜飞雁
徐磊, 季莹莹, 黎红, 杜飞雁, *
基于线粒体COI基因的南海浮游介形类斜突浮萤()单倍型与种群遗传结构研究
徐磊1, 3, 季莹莹1, 2, 黎红1, 3, 杜飞雁1, 3, *
1. 中国水产科学研究院南海水产研究所, 广州 510300 2. 上海海洋大学海洋科学学院, 上海 201306 3. 广东省渔业生态环境重点实验室, 广州 510300
介形类是一类小型的双壳甲壳类动物, 其海洋浮游种类就超过200余种, 是海洋浮游动物中主要组成部分, 也是海洋生态系统中物质循环与能量流动的关键环节。以线粒体细胞色素氧化酶亚基I(mtCOI)基因为标记, 分析南海浮游介形类中的广布种斜突浮萤()的单倍型多样性与种群遗传结构在空间上的分布, 并结合环境选择压力对种群的遗传多样性与结构分化的影响。结果显示, 38个个体共检出18种单倍型, 广布的单倍型在6个种群中都有分布, 说明种群可以实现远距离扩散, 最远超过700 km。种群呈现中度的遗传分化(平均ST= 0.186)。种群间遗传分化系数与地理距离无相关性(=0.17,=0.15), 种群未呈现空间距离隔离。远距离分布的单倍型并没有带来强劲的基因流, 相邻种群间甚至呈现出明显的遗传分化。RDA分析结果显示, 空间与环境并不是决定种群遗传结构的主要因素, 推测历史上种群扩展带来的拓殖隔离可能是主要解释。
介形类; 遗传分化; 空间距离隔离; COI基因; 南海
0 前言
南海是一个半封闭的深水海盆, 其跨度大约为北纬0°至23°、东经99°至121°, 面积约达 350万平方公里, 平均水深逾1800米, 最深达5420米。南海通过吕宋海峡、台湾海峡、民都洛海峡和巴拉巴克海峡、邦加海峡、加斯伯海峡和卡里马塔海峡、马六甲海峡分别与西太平洋、中国东海、苏禄海、爪哇海以及印度洋相联; 海域岛屿众多, 海底地形复杂, 既有宽广的大陆架, 又有险峻的大陆坡和辽阔的深海盆地[1]。因此, 南海独特的环境对分布在该海域的浮游生物种类多样性与种群遗传结构格局都有着显著的影响。
介形类(甲壳动物亚门, 介形纲)作为一个分化较多的类群[2], 分布较广, 种类多, 在海水、淡水、半咸水中均能生活, 其海洋浮游种类就超过200种[3], 中国海域及其邻近海域的浮游介形类共183种(含亚种)[4], 它们分别隶属于 2亚目、4科、8亚科、52属[5-10]。其中, 南海种数最多, 多达122种[11]。海洋浮游介形类是重要的浮游生物类群, 作为鱼类的饵料, 使得其在海洋物质循环, 能量流动中起到重要作用和关键环节。同时, 介形类对水温和盐度等环境因子较敏感, 使其可以成为潜在的气候环境变化的指示种类[12-13]。尽管浮游介形类占海洋浮游生物丰度比例较大(仅次于桡足类)[14-15], 但在浮游动物群落中的作用意义以及种类多样性仍被低估[16], 这主要是由于缺乏有效的形态分类特征, 难以直接通过显微镜直接进行分类鉴定, 此外, 具备丰富海洋浮游介形类形态鉴定经验的专家为数较少。近年来, 随着分子条码技术(DNA barcode)的普及, 海洋浮游介形类的种类多样性和隐种的分化得到进一步认识[17-18]。然而, 关于海洋浮游介形类广布种的种群遗传结构研究却没有进一步深入研究。对一个具体的介形类广布种类而言, 我们通常是以现生种群为对象, 通过测定水体中现生种群的遗传信息来推断其遗传结构。一个种群的遗传结构主要受基因流(gene flow), 突变(mutation)、自然选择(natural selection)、和遗传漂变(genetic drift)共同作用。当一个小种群发生时, 基因频率容易发生偏差。介形类在多数情况下依靠无性繁殖来快速扩张其种群数量, 此时如果缺少(来自外部的)基因流时, 受到选择压力的小种群会通过快速的无性生殖导致水体现生种群的遗传多样性下降[19-22]。而此时如果存在强劲的洋流影响, 种群间大量的基因流使得种群分化的可能减小, 遗传结构均一化, 通过构建种群遗传结构、单倍型关系, 及基因频率、单倍型频率在不同区域间的分布, 将很好的揭示这一问题。斜突浮萤()作为海洋浮游介形类的常见种, 在我国东海、台湾海峡、巴士海峡以及南海等海域都有分布[8]。
本研究以线粒体细胞色素氧化酶亚基I(mtCOI)基因为标记, 分析南海浮游介形类中的广布种斜突浮萤()的单倍型多样性与种群遗传结构在空间上的分布, 并结合环境选择压力对生种群的遗传多样性与结构分化的影响。
1 材料与方法
1.1 样品采集与DNA提取
2017年4月—5月分别于南海的不同海域进行浮游生物样品采集, 具体采样位点和信息见图1和表1。样品现场采集使用中型浮游生物网(网孔直径0.077 mm)垂直采集水深200—0 m范围内浮游生物。现场用无水乙醇固定, 4 ℃低温保存。回到实验室后, 样品在解剖镜下根据壳型、第1触角e刚毛、棒状器等形态特征, 参照《中国海洋浮游介形类》[9]进行种类鉴定。对鉴定好的样品进行单个个体DNA提取前, 先将样品放入到加有80 μL磷酸缓冲液的破碎管中, 加入破碎珠后于破碎仪(Tissue Cell-Destroyer)内进行震荡破碎, 然后使用DNeasy Blood&Tissue Kit试剂盒(QIAGEN,德国)提取DNA, 具体方法参见试剂盒说明书。提取的DNA于-20℃保存。
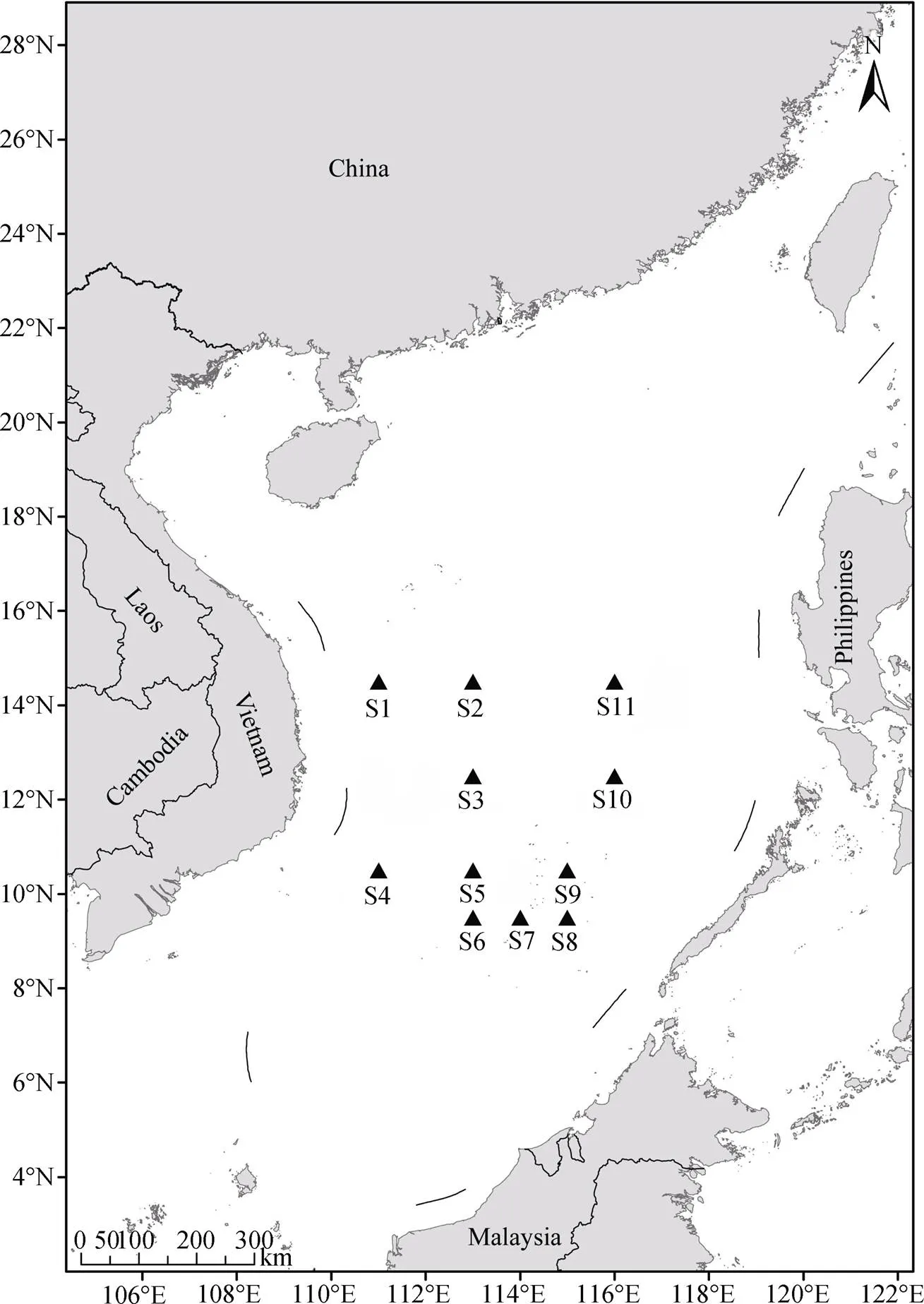
图1 采样点分布示意图
Figure 1 Location of sampling sites in the South China Sea
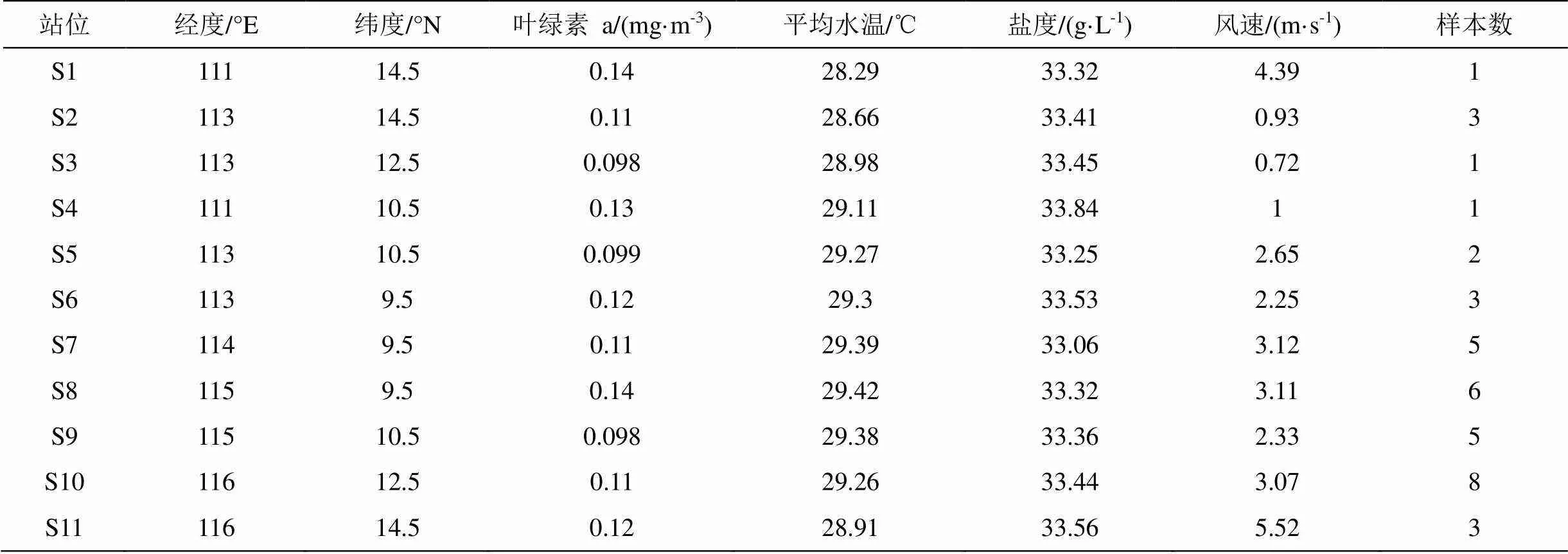
表1 15个采集站点的基本信息
1.2 COI序列扩增与测序
采用通用引物对COI目的基因进行PCR扩增, 正向引物为LCO1490(5’GGT CAA CAA ATC ATA AAG ATA TTG G3’); 反向引物为HCO2198(5’TAA ACT TCA GGG TGA CCA AAA AAT CA3’)[23]。引物序列送英潍捷基(上海)贸易有限公司合成。PCR扩增总体系为30μL, 包括3 μL 10× Ex Taq Buffer(Mg2+plus), 1.6 μL dNTP Mixture, 0.15 μL TaKaRa Ex Taq (5 U/μL), 3 μL模板DNA和引物(20 μM)各0.4 μL, ddH2O补充至30 μL。扩增程序设置为: 95℃预变性5 min, 94 ℃变性30 s, 51 ℃退火30 s, 72 ℃延伸45 s, 共35个循环, 最后72 ℃充分延伸5 min, 4 ℃保存。PCR产物经琼脂糖凝胶电泳检测, 选择700 bp左右且亮度清晰的PCR产物送天一辉远(广州)纯化并采用双向引物测序。
1.3 数据处理
所有测序获得的COI序列均在NCBI数据库中进行BLAST搜索比对以保证序列的可靠性(http:// blast.ncbi.nlm.nih.gov/Blast.cgi)。利用BioEdit7软件[24]及其中所附带Clustal W比对软件进行同源比对, 人工校正, 并去除序列两端引物结合位点的缺失序列, 比对分析获得COI序列通过HAPLOVIEWER[25]构建单倍型网络图。使用Arlequin 3.5[26]计算基于线粒体基因数据的种群遗传距离FST。在R程序中的vegan软件包中将种群间的空间距离与遗传距离进行mantel检验。为了进一步分解空间与环境对种群遗传差异的影响, 使用莫兰特征向量(Moran eigenvector maps, MEM), 环境数据(水温、叶绿素a、风速、盐度等)在R程序下进行RDA (Redundancy analysis)变差分析。分解环境与空间变量对种群遗传结构的解释率。
2 结果与分析
2.1 单倍型多样性
本文共获得38条线粒体COI基因序列, 经对比及剪切得到序列长度为665 bp, 核苷酸组成为A: 35.78%, G: 20.88%, C: 16.45%, T: 26.88%, 其中A+T含量(62.66%)高于G+C(37.34%)含量, 符合线粒体基因组碱基组成的特点, 多态位点为47个, 平均转换/颠换(si/sv)值为2.0。11个种群共检出18个单倍型(图2)。其中, 6个单倍型分布于2个以上种群(H3, H4, H5, H7, H8, H9), 部分单倍型只局限于局部种群, 但也发现广布的单倍型, 在6个种群中都有分布(H3), 跨度超过700 km。S10种群单倍型多样性最高, 8种单倍型中有5个为独有单倍型。所有线粒体COI单倍型序列都提交至GenBank, 序列登录号为MH557822-MH557839。
2.2 种群间遗传分化
根据线粒体COI基因数据分析种群遗传结构显示,种群呈现中度的遗传分化(种群间ST为0.02—0.628, 平均ST= 0.186)。Mantel检验发现种群遗传距离与地理距离无相关性(Mantel检验0.17,=0.15)(图3), 因此种群未呈现空间距离隔离(isolation-by-distance)。
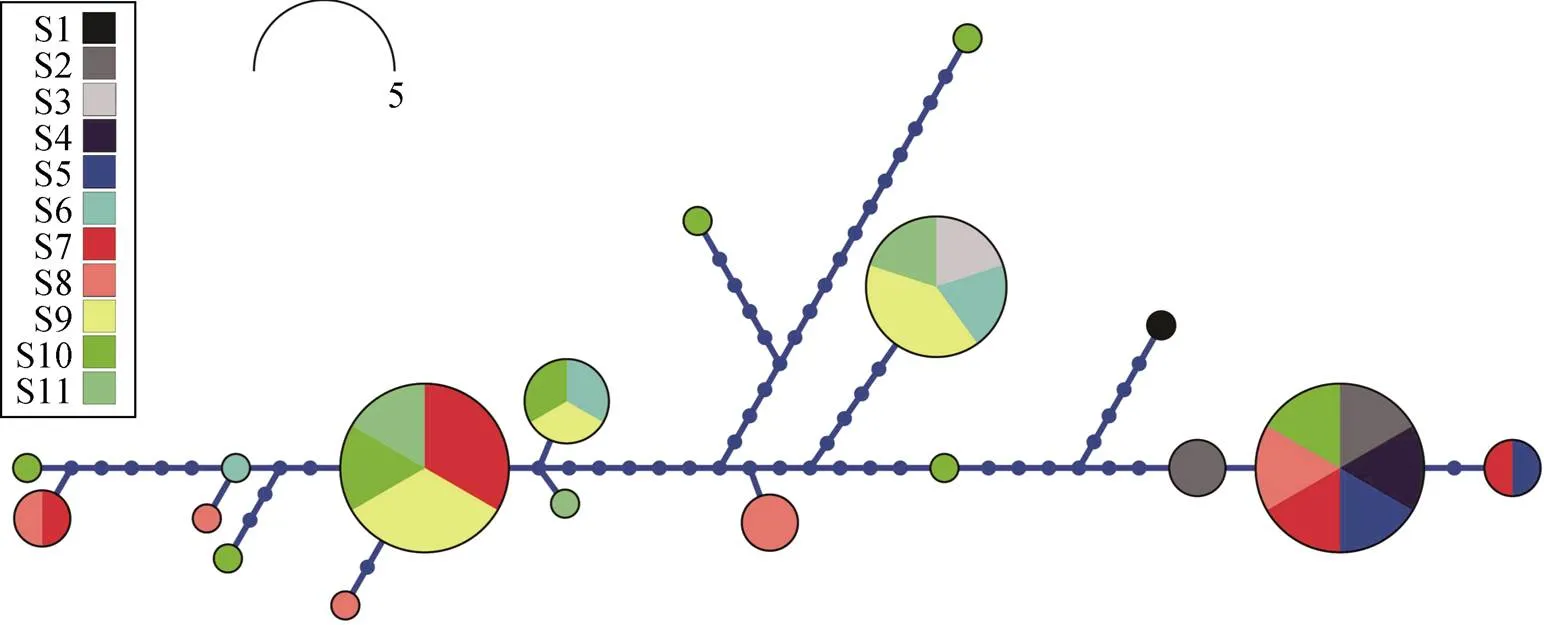
图2 P. procera的单倍型网络图
Figure 2 Haplotype network forbased on the gene of cytochrome coxidase subunit I (COI)
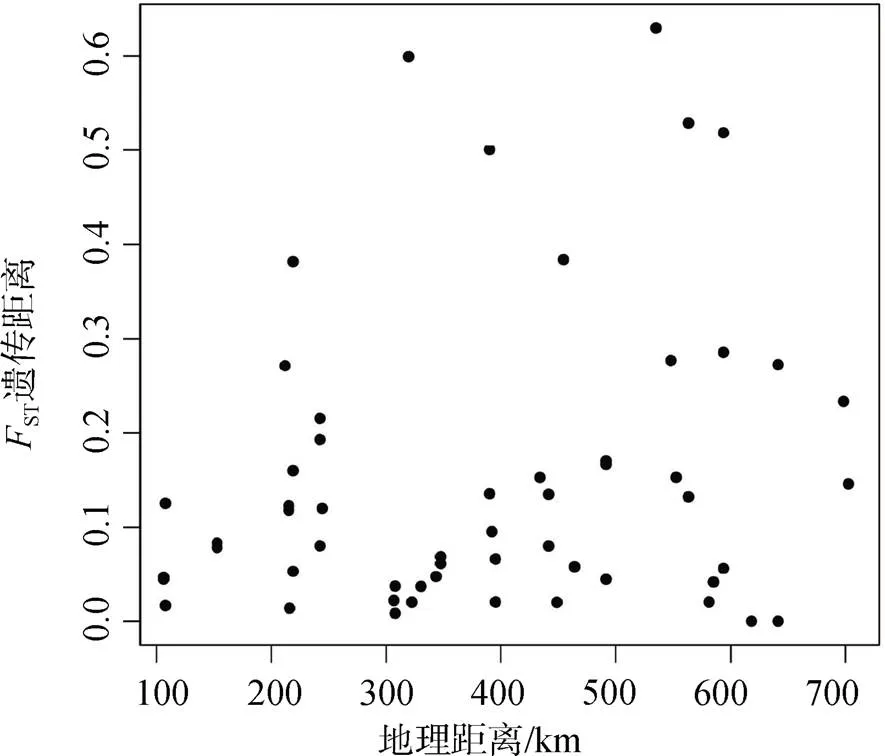
图3 遗传距离与空间距离相关性
Figure 3 The relationship between genetic differentiation coefficient (ST) and geographic distances (km)
RDA分析显示, 没有显著空间莫兰特征向量(MEM)被选中(>0.05)(表2); 当纳入环境变量后, 单纯空间(S|E)对遗传结构的解释量为0(>0.05), 说明空间变量不对种群遗传结构的变异做出解释。环境变量RDA矩阵中, 环境因子的总解释量为49.1%, 当纳入空间变量后, 单纯环境因子(E|S)对遗传结构的解释为3%(<0.05), 环境因子中, 盐度与风速成为显著影响种群遗传结构的环境变量(=0.02;=0.05)。空间变量与环境因子(S+E)共同对遗传结构的解释量为15.6%。环境和空间变量对种群遗传结构不能解释的部分高达84.4%。
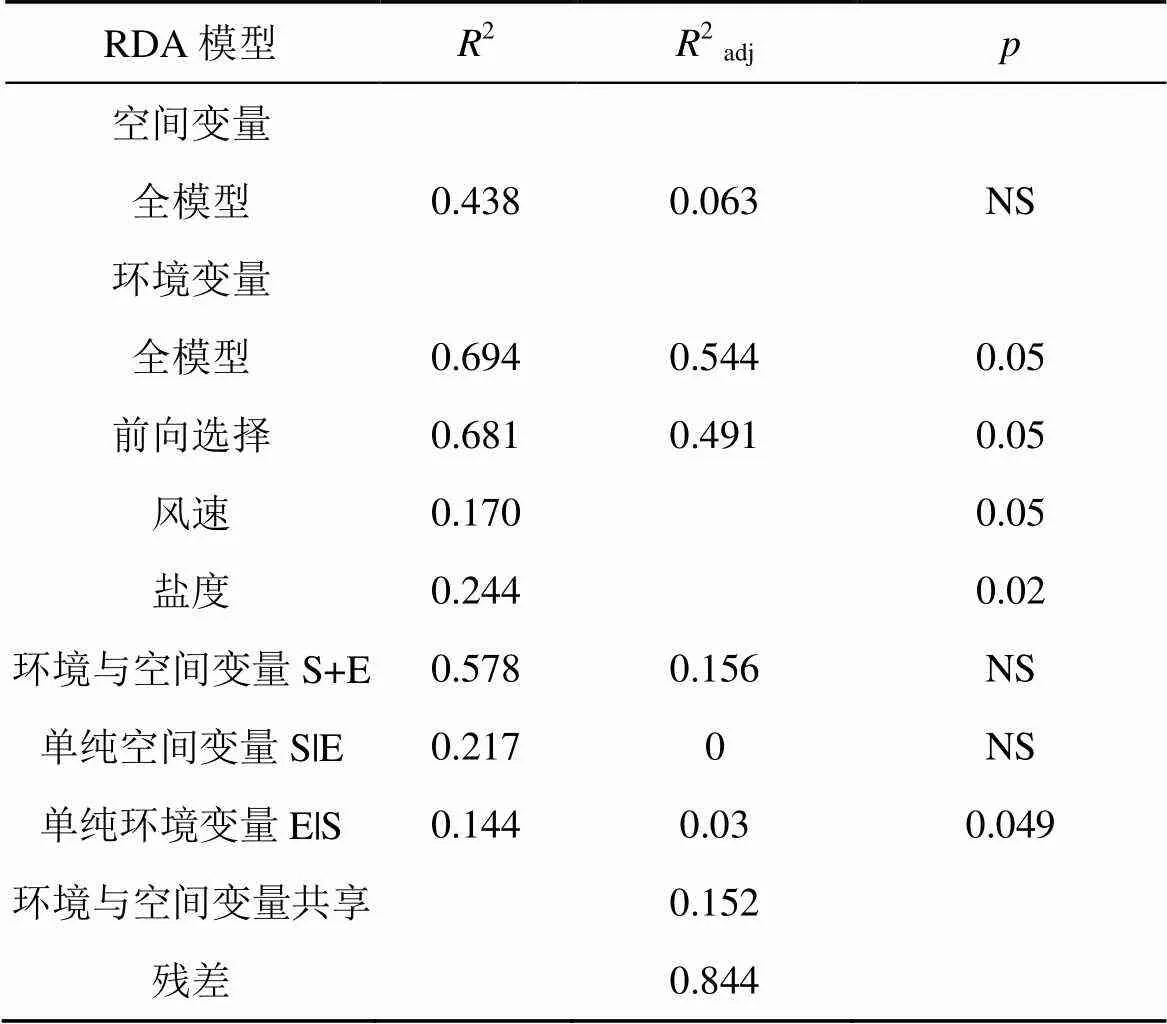
表2 RDA变差分解结果
3 讨论
海洋景观遗传学(seascape genetic)与景观遗传学(landscape genetic)相似, 都是从基于零假设框架下描述种群遗传结构转变为识别影响种群遗传结构的不同驱动力, 实现定量研究具有空间分布的种群遗传结构与多样性的维持机制[27-28]。相对于陆地生物, 海洋生物通常被认为没有明显的地理分化, 这主要是人们认为海洋生态系统缺乏明显的物理屏障, 阻碍种群间在“开放”海域中的基因交流。另外, 海洋生物的一些生物学特性也削弱了种群的分化, 例如具有较大的种群数量和较强的繁殖能力削弱了小种群遗传漂变的可能, 种群广泛的分布范围和成体或幼体阶段较强的扩散能力等进一步加强了种群间的基因流[29-32]。然而, 近20年的研究证明, 尽管海洋生态系统缺乏明显的物理屏障并呈现出有较高的连通性, 以及海洋生物具有较强的主动与被动扩散的能力, 但种群间的遗传结构依旧存在明显的分化(空间尺度与时间尺度的分化)。海洋生物种群遗传结构总体呈现出适中的结构化与强基因流相结合的形式。事实上, 影响海洋生物种群遗传结构的因素要比陆地生物复杂许多, 种群间尽管表现出潜在高水平的基因流, 然而实际上真实存在的基因流水平往往并不高。在众多因素(历史事件, 环境选择, 距离隔离和物种的快速进化等)的影响下, 即使缺乏明显的物理障碍, 种群间的基因流依然受到很大的限制[33-38]。
对于本研究而言, 浮游介形类种群呈现较高的单倍型多样性, 11个种群中共检出18种单倍型(图2), 其中12种单倍型为独有单倍型, 种群间单倍型组成分化明显。这一现象在其他海洋甲壳类物种中也有发现, 如粗腿厚纹蟹()鹰爪虾()远海梭子蟹()和锈斑蟳()[39-42]。从单倍型的空间分布来看, 种群间的个体扩散对种群单倍型结构有显著影响, 在6个种群中都有分布(H3), 扩散距离超过700 km, 横跨南海南部到南海中部。虽然我们无法直接测定种群间个体扩散的主要机制, 但我们认为南海海流是种群间扩散最有可能的解释, 海流在海洋生物物种形成与种群遗传结构分化中的影响早已得到证实[43]。南海处于东亚季风影响之下[44], 夏季西南季风一般于 5 月中旬在南海南部及中部生成, 并在6月时迅速扩展至整个南海, 而在南海北部风向较偏南。9月时东北季风开始在南海北部出现, 10月扩展至南海中部, 而至11月时则遍及全南海, 直到翌年4月冬季季风逐渐消失。南海的表层海水, 因受季风的吹拂等原因, 冬夏的流向亦有所不同。我们认为这种季节性的局部海流变化, 使得种群间的个体扩散非常复杂, 从而形成了现有的单倍型分布模式。然而, Lord[45]等人(2012年)在对科摩罗群岛附近海域分布的鰕虎鱼()种群遗传结构研究发现, 不同地理分布的种群间存在持续的基因流动, 因此一些学者认为海流不是解释海洋生物种群间扩散的唯一原因[46]。虽然海洋浮游生物的高被动扩散能力已经得到证实[47-49], 但我们的结果显示,种群呈现中度的遗传分化(平均ST= 0.186)。Mantel检验发现种群遗传距离与地理距离无相关性(=0.17,=0.15)(图3), 种群未呈现空间距离隔离(isolation-by-distance)。远距离分布的单倍型并没有带来强劲的基因流, 相邻种群间甚至呈现出明显的遗传分化。RDA结果显示, 空间与环境并不是决定种群遗传结构的主要因素, 因而历史上种群扩展带来的拓殖隔离(isolation- by-colonization)可能是主要解释[50]。这一现象在许多海洋生物中都得到证实[51-54], 即使海洋中缺乏明显的物理障碍, 海洋生物种群间的基因流依然受到很大的限制, 独特的历史事件在种群分化与物种形成的过程中起着至关重要的作用。因而, 我们推测今天所观测到的的种群遗传结构很可能早已形成, 而很少受现实的基因流影响。但要证实我们的推测还需进一步深入研究, 获得更多的种群遗传结构信息。
[1] 杨海军, 刘秦玉. 南海海洋环流研究综述[J]. 地球科学进展, 1998, 13(4): 364–368.
[2] ANGEL M. Planktonic oceanic ostracods-historical, present and future[J]. Proceedings of The Royal Society of Edinburgh Section B (biology), 1972, 73(22): 213–228.
[3] ANGEL M V. A review on the progress of research on halocyprid and other oceanic planktonic ostracods 1972- 1982[M]//Maddocks R F. Applications of Ostracoda. Houston: University of Houston, 1983: 529–548.
[4] 徐兆礼. 中国海洋浮游动物研究的新进展[J]. 厦门大学学报: 自然科学版, 2007, 45(A02): 16–23.
[5] 陈瑞祥. 台湾海峡西部沿岸浮游介形类的分布[J]. 海洋学报, 1982, 4(5): 617–626.
[6] 陈瑞祥, 林景宏. 南海中部海域浮游介形类的生态研究[J].海洋学报, 1993, 15(6): 91–98.
[7] 陈瑞祥, 林景宏, 黄亚如.南海北部大陆架水域浮游介形类与水系的相关性研究[J]. 海洋学报, 1996, 18(6): 93–98.
[8] 陈瑞祥, 林景宏. 中国海及其邻近海域浮游介形类大尺度生态研究Ⅲ.浮游介形类的物种与生态类群多样性[J]. 生物多样性, 1997, 05(4): 18–23.
[9] 陈瑞祥, 林景宏. 中国海洋浮游介形类[M]. 北京: 海洋出版社, 1995.
[10] 陈华, 徐兆礼. 长江口及邻近海域浮游介形类的分布与季节变化[J]. 应用与环境生物学报, 2009, 15(1): 72–77.
[11] 赵汉取, 贾晓平, 李纯厚等. 中国海洋浮游介形类研究进展[J]. 南方水产, 2005, 1(6): 66–71.
[12] 林景宏, 陈明达, 陈瑞祥.东海浮游介形类对海流、水团的指示作用[J]. 海洋学报, 1996, 18(3): 86–91.
[13] ANGEL M V, BLACHOWIAK-SAMOLYK K, DRAPUN I, et.al. Changes in the composition of planktonic ostracod populations across a range of latitudes in the North-east Atlantic[J]. Progress in Oceanography, 2007, 73(1): 60–78.
[14] 尹健强, 陈清潮. 南沙群岛海区海洋动物区系和动物地理研究专集[M]. 北京: 海洋出版社, 1991.
[15] 杜飞雁, 王雪辉, 谷阳光. 南沙群岛西南大陆斜坡海域浮游动物的垂直分布[J]. 海洋学报, 2014, 36(6): 94–103.
[16] BUCKLIN A, ORTMAN B D, JENNINGS R M, NIGRO L M, SWEETMAN C J, COPLEY N J. A “Rosetta Stone” for zooplankton: DNA barcode analysis of holozooplankton diversity of the Sargasso Sea (Northwest Atlantic Ocean) [J]. Deep-Sea Res II, 2010 57: 34–47.
[17] NIGRO L M, ANGEL M V, BLACHOWIAK-SAMOLYK K, et al. Identification, discrimination, and discovery of species of marine planktonic ostracods using DNA barcodes[J]. Plos One, 2016, 11(1): e0146327.
[18] TANAKA H, OHTSUKA S. Historical biogeography of the genus(Ostracoda: Myodocopa: Cladocopina), with the description and DNA barcode of the second Indo-Pacific species from the Seto Inland Sea[J]. 2015, Marine Biodiversity. 46: 625–640.
[19] SPARK P. Temporal changes in the genetic structure of thespecies complex in Tjeukemeer, with evidence for backcrossing[J]. Heredity, 1996, 76(5): 539–548.
[20] SPAAK P, DENK A, BOERSMA M, et al. Spatial and temporal patterns of sexual reproduction in a hybridspecies complex[J]. Journal of Plankton Research, 2004, 62(26): 625–635.
[21] XIANG Xianling, I Yilong, WEN Xinli, et al. Patterns and processes in the genetic differentiation of thecomplex, a passively dispersing freshwater zooplankton[J]. Molecular Phylogenetics and Evolution, 2011, 59(2): 386–398.
[22] XIANG Xianling, XI Yilong, WEN Xinli, et al. Genetic differentiation and phylogeographical structure of thecomplex in eastern China[J]. Molecular Ecology, 2011, 20(14): 3027–3044.
[23] FOLMER O, BLACK M, HOEH W, et al. DNA primers for amplification of mitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrates[J]. Molecular Marine Biology and Biotechnology, 1994, 3(5): 294–299.
[24] HALL T A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for Windows 95/98/NT[J]. Nucleic Acids Symposium Series, 1999, 41(41): 95–98.
[25] SALZBURGER W, EWING G B, VON H A. The performance of phylogenetic algorithms in estimating haplotype genealogies with migration[J]. Molecular Ecology, 2011, 20(9): 1952–1963.
[26] EXCOFFIER L, LISCHER H E L. Arlequin suite ver 3.5: a new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10(3): 564–567.
[27] MANEL S, HOLDEREGGER R. Ten years of landscape genetics[J]. Trends in Ecology and Evolution, 2013, 28(10): 614–621.
[28] WAGNER H H, FORTIN M J. A conceptual framework for the spatial analysis of landscape genetic data[J]. Conservation Genetics, 2013, 14(2): 253–261.
[29] PALUMBI S R. Marine speciation on a small planet[J]. Trends in Ecology and Evolution, 1992, 7(4): 114–118.
[30] NIELSEN E E, KENCHINGTON E. A new approach to prioritising marine fish and shellfish populations for conservation: a useful concept? [J]. Fish and Fisheries, 2001, 7: 328–343.
[31] LUTTIKHUIZEN P C, DRENT J, BAKER A J. Disjunct distribution of highly diverged mitochondrial lineage clade and population subdivision in a marine bivalve with pelagic larval dispersal[J]. Molecular Ecology, 2003, 12(8): 2215–2229.
[32] KENCHINGTON E L, PATWARY M U, ZOUROS E, et al. Genetic differentiation in relation to marine landscape in a broadcast spawning bivalve mollusc ()[J]. Molecular Ecology, 2006, 15(7): 1781–1796.
[33] WARD R D, WOODWARK M, SKIBINSK D O F. A comparison of genetic diversity levels in marine, fresh-water, and anadromous fishes[J]. Journal of Fish Biology, 1994, 44(2): 213–232.
[34] WAPLES R S. Separating the wheat from the chaff: Patterns of genetic differentiation in high gene flow species[J]. Journal of Heredity, 1998, 89(5): 438–450.
[35] DEWOODY J A, AVISE J C. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals[J]. Journal of Fish Biology, 2000, 56(3): 461–473.
[36] BAUS E, DARROCK D J, BRUFORD M W. Gene-flow pattern in Atlantic and Mediterranean populations of the Lusitanian sea star[J]. Molecular Ecology, 2005, 14(11): 3373–3382.
[37] HAYE P A, SEGOVIA N I, MUNOZHERRERA N C, et al. Phylogeographic structure in benthic marine invertebrates of the southeast pacific coast of Chile with differing dispersal potential[J]. Plos One, 2014, 9(2): e88613.
[38] ELLIS C D, HODGSON D J, DANIELS C L, et al. Population genetic structure in European lobsters: implications for connectivity, diversity and hatchery stocking[J]. Marine Ecology Progress, 2017, 563: 123–137.
[39] CASSONE B J, BOULDING E G. Genetic structure and phylogeography of the lined shore crab,, along the northeastern and western Pacific coasts[J]. Marine Biology, 2006, 149(2): 213–226.
[40] HAN Zhiqiang, ZHU Wenbin, ZHENG Wei, et al. Significant genetic differentiation between the Yellow Sea and East China Sea populations of cocktail shrimprevealed by the mitochondrial DNA COI gene[J]. Biochemical Systematics and Ecology, 2015, 59: 78–84.
[41] MA Hongyu, MA Chunyuan, LI Chenhong, et al. First mitochondrial genome for the red crab () with implication of phylogenomics and population genetics[J]. Scientific Reports, 2015, 5: 11524.
[42] REN Guijing, MIAO Guidong, MA Chunyan, et al. Genetic structure and historical demography of the blue swimming crab () from southeastern sea of China based on mitochondrial COI gene[J]. Mitochondrial DNA Part A, 2018, 29(2): 192–198.
[43] WHITE C, SELKOE K A, WATSON J, et al. Ocean currents help explain population genetic structure [J]. Proceedings: Biological Sciences, 2010, 277(1688): 1685– 694.
[44] KYLE C J, BOULDING E G. Comparative population genetic structure of marine gastropods (spp.) With and without pelagic larval dispersal[J]. Marine Biology, 2000, 137(5/6): 835–845.
[45] LORD C, LORION J, DETTAI A, et al. From endemism to widespread distribution: phylogeography of three amphidromousspecies (Teleostei:Gobioidei: Sicydiinae)[J]. Marine Ecology Progress, 2012, 455: 269–285.
[46] 梁必骐. 南海热带大气环流系统[M]. 北京: 气象出版社, 1991: 244.
[47] PROVAN J, BEATTY G E, KEATING S L, et al. High dispersal potential has maintained long-term population stability in the North Atlantic copepod[J]. Proceedings of the Royal Society B: Biological Sciences, 2009, 276(1655): 301–307.
[48] HELLBERG M E. Gene flow and isolation among populations of marine animals[J]. Annual Review of Ecology Evolution and Hellberg, Michael E . Gene Flow and Isolation among Populations of Marine Animals[J]. Annual Review of Ecology Evolution and Systematics, 2009, 40(1):291-310.
[49] CLAREMONT M, WILLIAMS S T, BARRACLOUGH T G, et al. The geographic scale of speciation in a marine snail with high dispersal potential[J]. Journal of Biogeoraphy, 2011, 38(6): 1016–1032.
[50] ORSINI L, VANOVERBEKE J, SWILLEN I, et al. Drivers of population genetic differentiation in the wild: isolation by dispersal limitation, isolation by adaptation and isolation by colonization[J]. Molecular Ecology, 2013, 22(24): 5983–5999.
[51] BARATTI M, MESSANA G G. High level of genetic differentiation in the marine isopod(Crustacea Isopoda Sphaeromatidae) as inferred by mitochondrial DNA analysis[J]. Journal of Experimental Marine Biology and Ecology, 2005, 315(2): 225–234.
[52] HORNE J B, VAN H L, CHOAT J H, et al. High population connectivity across the Indo-Pacific: congruent lack of phylogeographic structure in three reef fish congeners[J]. Molecular Phylogenetics and Evolution, 2008, 49(2): 629–638.
[53] WINTERS K L, VAN H L, CHOAT J H, et al. Phylogeography of the Indo-Pacific parrotfish: isolation generates distinctive peripheral populations in two oceans[J]. Marine Biology, 2010, 157(8): 1679–1691.
[54] CASTELIN M, FEUTRY P, HAYTECOEUR M, et al. New insight on population genetic connectivity of widespread amphidromous prawn(fabricius, 1798) (Crustacea: Cecapoda: Palaemonidae)[J]. Marine Biology, 2013, 160(6): 1395–1406.
Genetic structure and haplotype pattern of marine planktonic ostracodfrom the South China Sea based on the mitochondrial COI gene
XU Lei1,3, JI Yingying1,2, LI Hong1,3, DU Feiyan1,3
1. South China Sea Fisheries Research Institute, Guangzhou 510300, China 2. College of Marine Sciences, Shanghai ocean university, Shanghai 201306, China 3. Guangdong Provincial Key Laboratory of Fishery Ecology and Environment, Guangzhou 510300, China
Ostracods (Crustacea, Ostracoda) are small bivalved crustaceans, contributing over 200 described species to the marine zooplankton community. They are widely distributed and are relatively abundant components of the mesozooplankton, playing an important role in the transport of organic matter to deep layers. In this study, based on the mitochondrial COI gene analysis, we explored the population genetic structure and haplotype pattern ofwhich was the dominant species of ostracods in the South China Sea. We aimed to detect the population genetic structure of ostracods at medium spatial scales in the absence of physical barriers. Our data provided evidence of the importance of both long-distance dispersal as well as genetic isolation in determining the seascape genetic structure of this species. Our data suggested thatcould achieve long distance dispersal and specific haplotypes were successful in colonizing habitats from south area to central area in South China Sea. A total of 18 haplotypes were defined from 38 individuals, with most of them being existing only one time. The dominant haplotype was found in six sampling sites. The largest distance between two sampling sites harbouring this haplotype was more than 700 km. Our findings of long distance dispersal in the South China Sea combined with mild genetic differentiation among populations (averageST= 0.186) were in line with a scenario where population genetic structure was strongly impacted by colonization patterns. The seascape genetic structure ofin the South China Sea reflects both the importance of long distance dispersal as well as of reduced levels of gene flow, likely caused by colonization events followed by demographic expansions.
ostracods; genetic differentiation; isolation-by-distance; COI; South China Sea
10.14108/j.cnki.1008-8873.2019.05.003
180; 3135
A
1008-8873(2019)05-015-08
2018-10-10;
2018-12-12
中国水产科学研究院南海水产研究所中央级公益性科研院所基本科研业务费专项资金资助(2017YB26)
徐磊 (1982—), 男, 助理研究员, 主要研究方向为浮游动物分子生物学, E-mail:cloud_xu@hotmail.com
杜飞雁(1974—), 研究员, 主要研究方向为海洋生态学研究, E-mail:feiyanegg@163.com
徐磊, 季莹莹, 黎红, 等. 基于线粒体COI基因的南海浮游介形类斜突浮萤()单倍型与种群遗传结构研究[J]. 生态科学, 2019, 38(5): 15-22.
XU Lei, JI Yingying, LI Hong, et al. Genetic structure and haplotype pattern of marine planktonic ostracodfrom the South China Sea based on the mitochondrial COI gene[J]. Ecological Science, 2019, 38(5): 15-22.
