甜菜夜蛾核多角体病毒VP39基因的克隆与生物信息学分析
2019-09-19李赛男赵海洲姜玉霞
李赛男,李 萌,赵海洲,姜玉霞
(肇庆学院 生命科学学院, 广东 肇庆 526061)
杆状病毒是一类特异性感染节肢动物的病源微生物,具有宿主特异性,每种病毒只限感染一种或少数几种亲缘关系较近的昆虫,对人畜无害,已被认为是最安全的生物杀虫剂之一[1-2]。此外,杆状病毒还被用于在昆虫细胞中表达外源重组蛋白[3]、基因治疗[4]以及作为重组抗原表位的展示系统[5]。迄今为止,已从600多种昆虫中分离出杆状病毒,主要来自鳞翅目(如蝴蝶和蛾)、膜翅目(如蜻蜓)和双翅目(如蚊子)[6]。杆状病毒基因组DNA呈双链闭合环状,大小约为80~180 kbp,预测编码89~183个ORF[7]。自苜蓿丫纹夜蛾核多角体病毒(Autographacalifornicamultiplenucleopolyhedrovirus,AcMNPV)全基因组[8]测序被完成以来,已有超过80株杆状病毒基因组序列被测定。最新的杆状病毒分类方法根据完成全基因组测序的杆状病毒中的29个核心基因编码的氨基酸序列,将杆状病毒科分成4个属,分别为Alphabaculovirus、Betabaculovirus、Gammabaculovirus和Deltabaculovirus[9]。根据对多角体蛋白基因的进化分析,Alphabaculovirus又可以进一步分为Group I和Group II NPV[10]。
甜菜夜蛾核多角体病毒(SpodopteraexiguaMNPV,SeMNPV)是Group II NPV的成员,其基因组全序列测定已经完成并已发表[11]。VP39是昆虫杆状病毒的核心基因之一,在感染的晚期表达,其编码蛋白VP39叠环排列在核蛋白核心(nucleoprotein core)的周围[12],是杆状病毒核衣壳的最主要结构蛋白[13-15]。肌动蛋白(actin)在杆状病毒入侵以及在细胞内复制过程中具有重要作用[16-19]。细胞中,actin以两种形式存在,即单体(Globular actin,G-actin)和多聚体(Filamentous actin,F-actin)。研究发现,VP39与G-actin和F-actin都可以结合,但更倾向于结合G-actin[20]。在体外,纯化的中国棉铃虫单粒包埋型核多角体病毒VP39蛋白能与G-actin以1∶5的比例结合,因此提出VP39蛋白在体外诱导G-actin形成F-actin索的模型:首先,VP39与G-actin以1∶1的分子比例结合形成核,然后这个核诱导其他4个G-actin聚集在它的周围形成六聚体,接着许多六聚体互相结合形成微丝,最后微丝扭曲形成索状结构[21]。但具体体内VP39结合G-actin所发挥的作用目前尚不明确。此外,有研究表明,AcMNPV VP39能与它本身以及其他病毒蛋白,如多角体蛋白、ODV-EC27、ODV-E56、IE1、38K和ODV-E66,以及宿主的kinesin-1具有相互作用[22-25],推测VP39参与了病毒入侵以及在宿主细胞内复制过程。
本文选取了国内外尚未进行研究的SeMNPV VP39(Sevp39)基因作为研究对象,利用生物信息学方法,预测了Sevp39基因编码的Sevp39蛋白的理化特性、序列模式特性及二级结构,并进行了Sevp39及其同源蛋白的序列比对,为今后研究Sevp39蛋白在病毒入侵和在细胞内复制过程中所担负的功能,探讨宿主细胞蛋白与VP39蛋白间的相互作用机制,深入研究宿主与杆状病毒之间的相互作用关系等提供依据。
1 材料和方法
1.1 材料
克隆载体pMD18-T和Premix Ex Taq Hot Start Version PCR 试剂盒购自TaKaRa公司;限制性内切酶购自Thermo Fisher公司;X-gal、IPTG和氨苄青霉素购自Sigma公司;质粒小量提取试剂盒和DNA凝胶回收试剂盒购自Omega公司;DNA分子质量购自东盛公司;其他试剂为国产分析纯。
甜菜夜蛾核多角体病毒毒株SeMNPV-SeUS1为中山大学有害生物控制与资源利用国家重点实验室赠送(其引自美国加州大学Riverside分校B.A.Federici教授实验室);大肠杆菌(Escherichia coli) DH5α为肇庆学院生命科学学院保存。
1.2 SeMNPV基因组DNA的提取
SeMNPV基因组DNA的提取参照文献[26]的方法进行。
1.3 引物设计及PCR
利用DNASTAR软件,参照SeMNPV基因组全序列[11](Genbank accession No.NC_002169)中的Sevp39全长序列,设计一对扩增Sevp39基因全长的引物,由深圳华大基因股份有限公司合成。上游引物P1:5’-GAATTCATGGCTCTGACGCCTGTCTCCTCGA-3’,下划线部分为引入的EcoRI酶切位点;下游引物P2:5’-GGTACCCTAGACGACGGCCGTGGGCCGAAG -3’,下划线部分为引入的KpnI酶切位点。PCR模板为SeMNPV基因组DNA,反应体系为50 μL。PCR反应设计为:98 ℃,3 min;98 ℃,1 min,50 ℃,1 min,72 ℃,1 min,30个循环;72 ℃,10 min;4 ℃保存。扩增结束后,进行琼脂糖凝胶电泳检测,回收Sevp39基因的PCR产物。
1.4 质粒酶切分析及序列测定
将回收的PCR产物与克隆载体pMD18-T进行连接,转化DH5α,挑取阳性克隆进行培养,提取质粒T-Sevp39,用EcoRI和KpnI进行双酶切鉴定,并在深圳华大基因股份有限公司对插入的DNA片段进行测序。将测序结果与Sevp39基因序列进行比较。
1.5 生物信息学分析
将克隆并测序得到的Sevp39基因序列及其编码的氨基酸序列作为原始数据,利用网络服务器或生物学分析软件,有针对性地分析了该基因及其编码蛋白的氨基酸结构特点、理化性质等。一级结构采用DNASTAR软件根据Sevp39基因序列推导出Sevp39蛋白的氨基酸序列,预计编码的Sevp39蛋白的预测相对分子质量、等电点、氨基酸组成、化学特性、蛋白质结构域与模式的预测用DNASTAR软件、EBI (http://www.ebi.ac.uk)和ExPASy (http://www.expasy.org)等网站上的ProtParam和MotifScan (https://myhits.isb-sib.ch/cgi-bin/PFSCAN),用Psipred (http://bioinf.cs.ucl.ac.uk/psipred/)等在线软件进行蛋白质二级结构分析。在GenBank/EMBL数据库中用BLAST搜索引擎寻找Sevp39同源序列,用Clustal W软件进行多序列比较,用GeneDoc软件进行编辑。
2 结果与分析
2.1 Sevp39基因的扩增
以SeMNPV基因组DNA为模板,从SeMNPV基因组中用PCR方法扩增了Sevp39基因,扩增片段大小为981 bp (图1),与预期大小相符。回收PCR产物,连入pMD18-T载体,将连接产物转化DH5α,挑取阳性克隆进行培养,提取质粒,得到质粒T-Sevp39,用EcoRI和KpnI进行双酶切鉴定,获得981 bp的目的片段和2692 bp的载体片段,与预期大小一致(图2)。
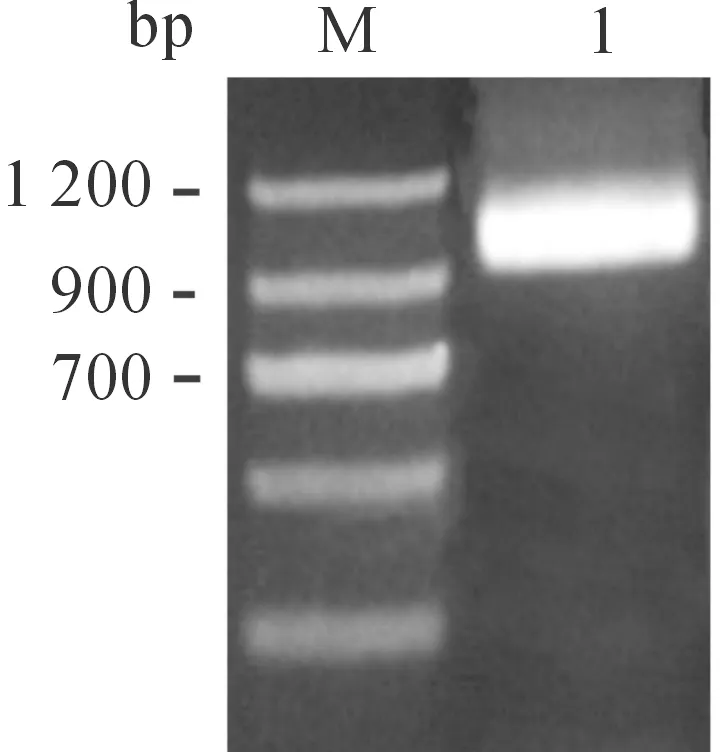
M.DNA分子量标准; 1.PCR产物
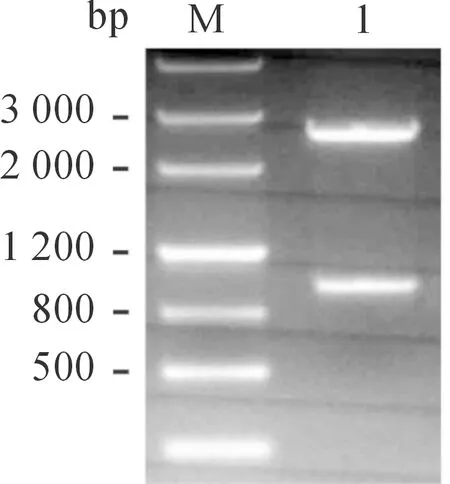
M.DNA分子量标准; 1.重组质粒T-Sevp39的EcoRI/KpnI双酶切
2.2 T-Sevp39重组体测序
将酶切鉴定正确的T-Sevp39重组体进行测序。结果表明:除Sevp39第308位的碱基由G突变成T (图3,红色标记碱基),其余所有碱基与Genbank上公布的SeMNPV基因组全序列(Genbank accession No.NC_002169)中的Sevp39序列完全一致(图3)。虽然进行了多次重复,测序结果均显示在Sevp39第308位的碱基由G突变成T,原因可能是病毒毒株SeMNPV-SeUS1经过多次传代培养,在该位点发生了变异。该位点的突变导致该编码氨基酸的密码子由GGT变为GTT,从而氨基酸由甘氨酸(Gly)变成缬氨酸(Val) (图3,红色方框标注),但是在培养病毒过程中,并没有观察到SeMNPV病毒的形态发生受到影响,因此该位点突变不影响Sevp39的功能。

图3 Sevp39核苷酸序列和推导的氨基酸序列
2.3 生物信息学分析
运用在线软件ProtParam和DNASTAR软件分析Sevp39编码的氨基酸序列的基本特征,结果如表1所示。Sevp39编码326个氨基酸,预测相对分子质量为37 ku,蛋白等电点为5.859;氨基酸组成中带负电荷氨基酸(Asp、Glu) 35个,占10.7%;带正电荷氨基酸(Arg、Lys) 32个,占9.8%;亲水性氨基酸128个,占39.3%,疏水性氨基酸176个,占54.0%;Sevp39蛋白在大肠杆菌中的半衰期大于10 h,蛋白的不稳定指数为42.31,是不稳定蛋白(蛋白的不稳定指数大于40.00就为不稳定蛋白)。
运用MotifScan分析发现(表2),Sevp39蛋白序列中包含3个可能的N-糖基化位点(153-156、174-177、223-226)、2个可能的酪蛋白激酶Ⅱ磷酸化位点(40-43、110-113)、6个可能的蛋白激酶C磷酸化位点(11-13、53-55、83-85、175-177、226-228、300-302)、1个可能的N-豆蔻酰化位点(267-272)和1个可能的酪氨酸激酶磷酸化位点(146-152)。
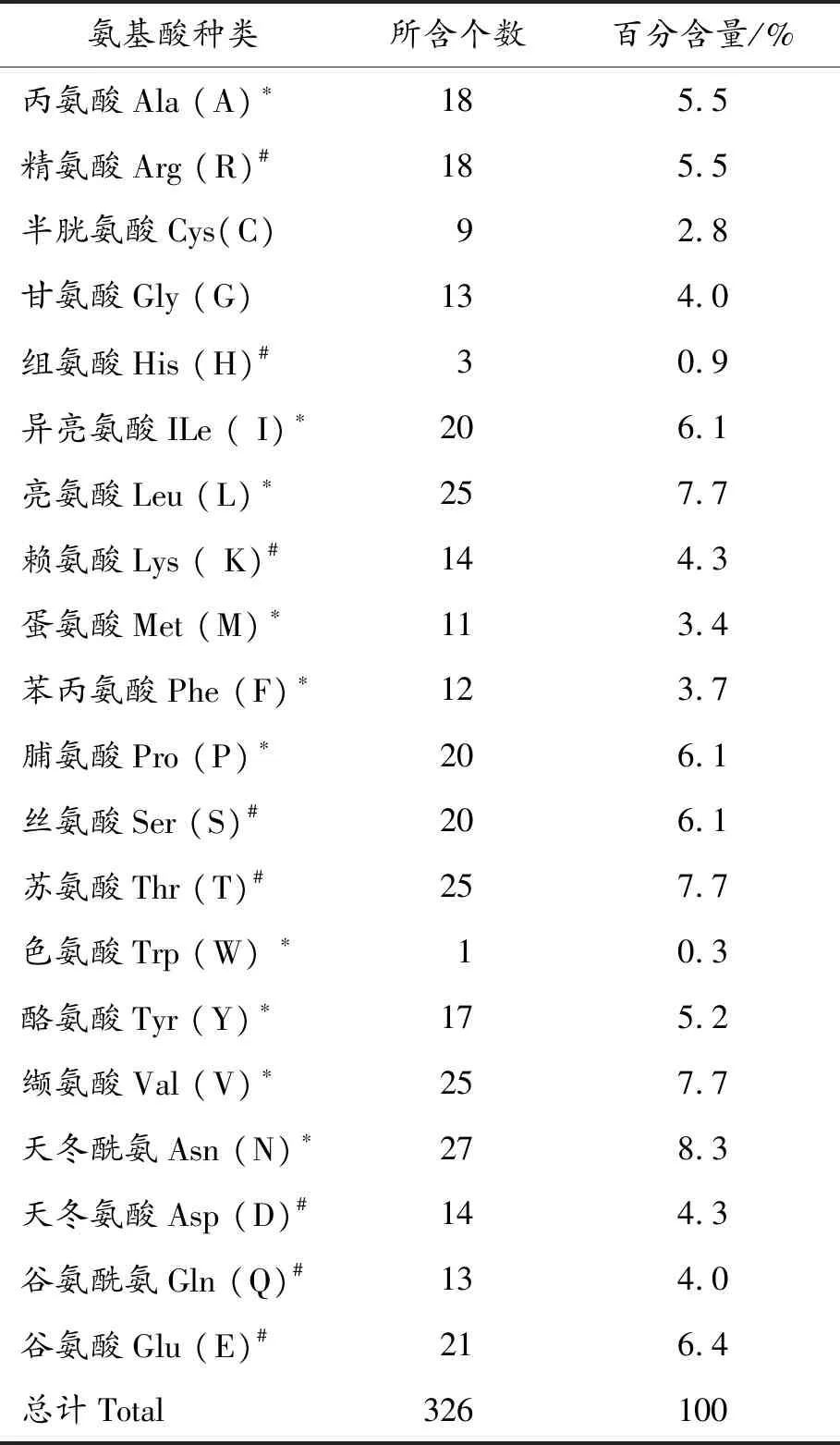
表1 Sevp39编码蛋白氨基酸含量
注:* 疏水氨基酸, #亲水氨基酸
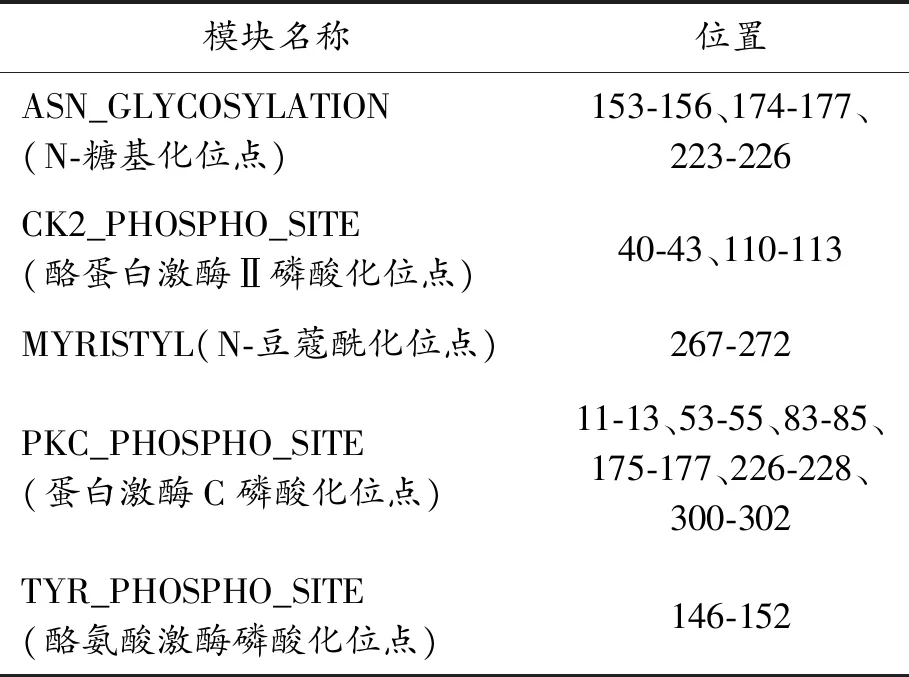
表2 Sevp39蛋白序列模式分析结果
BLAST搜索Sevp39的同源蛋白,并用Clustal W软件比较其与同源蛋白的序列相似性。分析发现,Sevp39除与Neodiprionabietis(Neab)NPV、Neodiprionlecontii(Nele)NPV、Neodiprionsertifer(Nese)NPV 和Culexnigripalpus(Cuni)NPV的VP39蛋白序列相似性较低,介于12%~18%之间,与其余同源蛋白的序列相似性均大于30%,保守性适中。
从每个杆状病毒属中选择Sevp39同源蛋白的氨基酸序列进行比对,结果显示出6个高度保守的半胱氨酸残基(Cys) (图4),其中:Cys17、Cys48、Cys131存在于在所有的同源蛋白中;Cys28、Cys35存在于除NeabNPV、NeleNPV、NeseNPV、CuniNPV外的其他杆状病毒的同源蛋白中;Cys168则存在于除CuniNPV外的其他杆状病毒的同源蛋白中。
用Psipred分析Sevp39蛋白的二级结构,结果显示Sevp39可能形成7个α螺旋和14个β折叠(图5)。通过氨基酸序列比对所确定的保守Cys在图中以星号标出以指示其在二级结构中的分布情况,发现上述6个保守的Cys中只有Cys17参与构成β折叠。
3 讨论
在SpltMNPV中,VP39基因的转录在病毒感染后6 h即可检测到,并且转录起始于保守的杆状病毒晚期启动子ATAAG中的第2个碱基A。Western blotting分析表明:VP39蛋白存在于BV和ODV的核衣壳中,因此VP39基因被认为是一个杆状病毒晚期结构基因[27]。最近,有研究表明,家蚕核多角体病毒VP39基因的突变导致在培养细胞及家蚕幼虫体内的感染性芽生型病毒粒子和包涵体的产生显著下降[28]。因此,VP39在病毒复制过程中具有重要作用。
为揭示VP39的作用机理,本文运用DNASTAR等生物学软件分析了Sevp39基因及其编码蛋白Sevp39。结果表明:Sevp39蛋白在大肠杆菌中的半衰期大于10 h,是不稳定蛋白。Sevp39蛋白有多个位点可以发生糖基化修饰和磷酸化修饰。蛋白质的脱磷酸化和磷酸化在细胞信号传导过程中具有重要作用,糖基化、磷酸化位点可能是Sevp39蛋白完成其生理功能的结构基础。
同源性分析表明,Sevp39与绝大部分杆状病毒同源蛋白的序列相似性均大于30%,保守性适中。从每个杆状病毒属中选择Sevp39同源蛋白的氨基酸序列进行比对,结果显示出6个高度保守的Cys,分别是Cys17、Cys28、Cys35、Cys48、Cys131、Cys168。利用Psipred在线软件分析Sevp39蛋白的二级结构,结果显示Sevp39可能形成7个α螺旋和14个β折叠,6个高度保守的Cys中只有Cys17参与了β折叠的形成。Cys在蛋白质中常形成二硫键,其中存在于蛋白质内部的二硫键通过影响蛋白空间结构而决定其功能;存在于蛋白之间的二硫键,形成蛋白之间一种共价键相互作用的方式。在对猴空泡病毒(Simian vaculating virus)、乳头状瘤病毒(Papillomavirus)的研究中均发现,核衣壳主要蛋白通过保守Cys之间的二硫键构成寡聚体来参与衣壳的装配[29-30]。因此,推测Sevp39蛋白6个保守的Cys参与了蛋白空间结构的形成,在Sevp39的功能中具有重要作用。后续将致力于VP39蛋白中保守Cys的功能研究。
Eleven representative sequences were selected and aligned using Clustal X 1.83 and were edited with GeneDoc software.The VP39 protein sequences used are as follows:SeMNPV (NP_037835.1),AcMNPV (NP_054119.1),HyphantriacuneaNPV (HcNPV,YP_473253.1),LymantriadisparMNPV (LdMNPV,NP_047729.1),SpodopteralituraMNPV (SpltMNPV,NP_258349.1),AgrotissegetumGV (AgseGV,YP_006258.1),Xestiac-nigrumGV (XecnGV,NP_059259.1),NeabNPV (YP_667940.1),NeleNPV (YP_025289.1),NeseNPV (YP_025196.1) and CuniNPV (NP_203328.1).Amino acids with black shading denote 100% conservation in all sequences.Those with dark gray and light gray shading represent 80% and 60% conservation in all sequences,respectively.The asterisks indicate the location of the conservative cysteine amino acids.
图4 Sevp39蛋白与其他昆虫杆状病毒VP39氨基酸序列的同源性比较

图5 Sevp39二级结构预测结果
