小伞山羊草染色体的FISH核型分析
2019-09-13张素勤2
2 张素勤2
(1.贵州大学农学院, 贵阳 550025; 2.国家小麦改良中心贵州分中心, 贵州 贵阳550025)
山羊草属(Aegilops)植物主要分布于小亚细亚、巴基斯坦、地中海和中亚,现在欧洲、非洲大陆、地中海沿岸、帕米尔高原和中亚西亚广为分布[1]。普通小麦(TriticumaestivumL.)由3个染色体组组成,其中B和D 2个染色体组来源于山羊草属,山羊草属与小麦属(Triticum)的亲缘关系最近,山羊草属几乎所有的种均可与小麦杂交[2]。山羊草属含有许多对改造栽培小麦品质及农艺性状十分有价值的基因,例如抗叶锈病、条锈病、赤霉病、白粉病及眼斑病、早熟、抗倒伏、抗虫害、抗寒、抗旱、高蛋白质等优异基因,是小麦改良的1个丰富基因库,在小麦进化中起着重要作用[2-3]。小伞山羊草(Aegilopsumbellulata,UU,2 n=2 x=14)是山羊草属二倍体物种,该物种的U基因组是小麦和相关物种的几个基本基因组之一[4-6]。充分发掘、利用小伞山羊草的优良基因,通过远缘杂交技术导入小麦对提高栽培小麦品质与抗性等具有重要意义。
荧光原位杂交(Fluorescence in Situ hybridization,FISH)技术是远缘杂种鉴定、异源多倍体起源及进化等相关研究的有力工具[7]。通过DNA重复序列可以用于染色体辨别、基因组组成及进化分析,外源遗传物质检测等多项研究[8]。Adonina 等[9]对拟斯卑尔脱山羊草(Aegilopsspeltoides)杂交后代的染色体组进行了FISH核型分析。Schneider[10]等采用pAs 1和pSc 119.2探针对小麦-山羊草附加系的染色体进行了FISH鉴定。Tang等[11-12]开发出的寡核苷酸Oligo-pSc 119.2-1与Oligo-pTa-535-1探针可以作为有效区分小麦及其远缘物种的染色体。宫文萍等[13]用Oligo-pSc119.2-1、Oligo-pTa-535-1和(GAA)8为探针的FISH分别对小麦-单芒山羊草双二倍体和中国春-单芒山羊草附加系进行了分析,并建立了其FISH核型。董磊等[14]利用Oligo-pTa 535和Oligo-pSc 119. 2探针对多份拟斯卑尔脱山羊草进行FISH分析,准确区分拟斯卑尔脱山羊草的不同染色体,建立了拟斯卑尔脱山羊草的FISH核型。本试验以寡核苷酸Oligo-pSc 119.2-1与(GAA)7重复序列为探针的双色FISH技术对小伞山羊草根尖细胞染色体进行核型分析,以期得到小伞山羊草的FISH核型,为小麦遗传研究及新品种培育提供参考。
1 材料与方法
1.1 材 料
试验材料为小伞山羊草(Aegilopsumbellulata,UU,2 n=2 x=14),具有早熟、耐逆,抗锈病、白粉病,抗蚜虫等优点,保存于国家小麦改良中心贵州分中心实验室。
1.2 染色体制片
将种子置于铺有湿润滤纸的培养皿中于常温下发芽,待根长至2 cm左右时,剪取根尖用N2O处理2 h,在90%冰醋酸中固定10 min,用蒸馏水冲洗3遍后切取根尖分生区置于37 ℃水浴酶解50 min,用70%酒精冲洗3遍,捣碎根尖后加入冰醋酸滴片,在显微镜(OLMPUS BX 60)下镜检,选取分裂相好的片子保存于-20 ℃冰箱备用。
1.3 荧光原位杂交分析
FISH技术参照Han等[15]的方法,小伞山羊草的染色体命名参照Friebe等[6]和Mirzaghaderi等[16]的方法。Oligo-pSc 119.2-1(绿色)与(GAA)7(红色)探针由Thermo Fisher Scientific(上海)公司合成。选择FISH效果好的细胞用Cellsens Standard摄像系统照相。选取5个分裂相好且染色清晰的细胞用于FISH核型分析,以得到准确可靠的数据。
2 结果与分析
本试验分别以Oligo-pSc 119.2-1(绿色)与(GAA)7(红色)重复序列为探针对小伞山羊草根尖细胞染色体进行了FISH分析(图1)。结果发现,小伞山羊草共包含7对染色体,其中1 U和5 U染色体各含有1对随体。Oligo-pSc-119.2-1信号主要分布在染色体的端部与近端部,不同染色体之间的信号强度有所差别。(GAA)7信号较Oligo-pSc 119.2-1的亮度高,而且分布更丰富,许多染色体着丝点附近(GAA)7信号尤其强烈。

注:Oligo-pSc 119.2-1探针为绿色,(GAA)7探针为红色;背景染色体由DAPI复染为蓝色;比例尺为10 μm。图1 小伞山羊草根尖细胞染色体的FISH核型
小伞山羊草1 U染色体两端部具有清晰的Oligo-pSc 119.2-1绿色信号(图2);着丝点处有(GAA)7红色信号。2 U染色体上Oligo-pSc 119.2-1信号位于染色体两端部;(GAA)7信号则位于着丝点处、长臂中间与端部。3 U染色体上信号丰富,明亮的Oligo-pSc 119.2-1信号位于染色体两端部;(GAA)7信号处于着丝点附近、长臂中间与近端部。4 U染色体上Oligo-pSc 119.2-1信号位于染色体两端部;(GAA)7信号较丰富,主要分布在着丝点处,短臂中间及端部。5 U染色体在着丝点与长臂近端部有(GAA)7信号。6 U染色体上(GAA)7信号位于染色体着丝点、长臂端部、短臂近端部,其中长臂端部信号最为明亮;Oligo-pSc 119.2-1信号只有1对,因与长臂端部(GAA)7强信号重叠而不易辨认。7 U染色体上存在较多的Oligo-pSc 119.2-1与(GAA)7信号,其中Oligo-pSc 119.2-1信号位于长臂中间与短臂端部;(GAA)7信号分别分布在着丝点处、长臂中间及短臂端部。
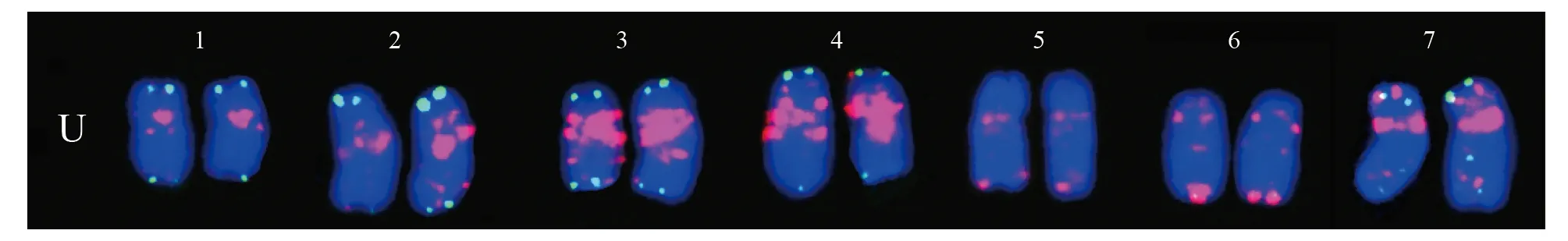
图2 小伞山羊草U基因组的FISH核型
Oligo-pSc 119.2-1信号主要分布在小伞山羊草染色体两端部,表明该U基因组的端部常含有pSc119.2重复序列。因为小伞山羊草的一些(如1 U、2 U、3 U和4 U)染色体的Oligo-pSc 119.2-1信号具有相似性,所以只采用Oligo-pSc 119.2-1探针的单色FISH不能鉴别所有染色体。将Oligo-pSc-119.2-1与(GAA)72种探针配合使用可以有效地鉴别每一染色体。
3 讨 论
小麦远缘杂交可将野生植物的基因导入小麦中,丰富小麦的基因库,以创造出新的小麦种质资源及优良小麦新品种[17]。小麦远缘杂交是染色体工程的基础,而山羊草属是小麦远缘杂交的重要材料,小麦与山羊草属远缘杂交可获得小麦新资源。Mirzaghaderi等采用pSc 119.2探针对小伞山羊草进行了FISH核型分析,发现pSc 119.2信号主要分布于染色体端部[16]。本试验利用Oligo-pSc-119.2-1探针对小伞山羊草进行了FISH分析,也发现Oligo-pSc-119.2-1信号主要分布在染色体的端部,获得了与Mirzaghaderi等[16]相似的结果;但是5 U和6 U染色体上Oligo-pSc-119.2-1信号存在差别,可能是试验材料不同的缘故。
小麦中GAA-卫星序列的杂交模式与N带模式相同[18],N带模式与C带基本相同[19]。Friebe等[6]对小伞山羊草进行C-带带型分析,发现在染色体着丝粒处都存在C-带,其余分布在端部,臂中间部位的C-带较少。本试验小伞山羊草(GAA)7信号与Friebe等[6]建立的C-带核型图有相似性,但也有一些不同,如2 U、3 U、4 U和 7 U染色体的(GAA)7信号与C-带都分布较广,在染色体臂与着丝点处都存在;而1 U和5 U染色体的(GAA)7信号则较C-带少,其原因是C带技术主要显示染色体上的异染色质,其含有大量丰富的高度重复的DNA顺序,而(GAA)7只是1种DNA重复序列。小伞山羊草FISH核型与C-带核型图相比,探针信号与染色体背景反差大、条带丰富、视觉效果明显好于C-带核型,有效提高了染色体核型分析的效果[14]。
本试验Oligo-pSc-119.2-1信号主要分布在小伞山羊草染色体的端部与近端部。(GAA)7信号主要集中于染色体着丝点与近着丝点处,不但比Oligo-pSc-119.2-1信号的亮度强,分布丰富,而且在染色体上的分布可与Oligo-pSc-119.2-1信号互补。因此将Oligo-pSc-119.2-1与(GAA)72种探针配合使用以准确地辨别小伞山羊草的所有染色体,建立了小伞山羊草的FISH核型,可为小麦遗传育种提供参考。
