茶树SBPase基因的电子克隆与生物信息学分析
2019-09-06徐煲铧刘瑞彬余绍东张应中
徐煲铧 刘瑞彬 余绍东 张应中


摘要通过EST序列电子克隆技术,获得参与茶树光合作用的SBPase基因的eDNA拼接序列,对序列进行了生物信息学分析和功能预测,构建了系统进化树。本研究结果可为今后开展山茶属植物光合基因的功能研究奠定重要基础。
关键词 茶树;SBPase基因;生物信息学
中图分类号 S571.1
文献标识码 A
文章编号 1007-5739(2019)07-0017-03
茶树(Camellia sinensis)属山茶科山茶属,为多年生常绿木本植物。叶子呈椭圆形,边缘有锯齿,叶间开五瓣白花;果实扁圆,呈三角形,果实开裂后露出种子。春、秋2季可采摘茶树嫩叶制茶。
目前对主要经济作物和粮食作物的光合作用研究较多,对茶树光合作用的研究相对较少,且研究主要集中在影响茶树光合作用的生理生态因素及茶树品种、树龄.叶位及冠层间光合特性比较等方面",与光合作用相关的分子生物学研究,例如酶的基因序列和蛋白质结构等分子水平上的研究就更少。
光合生物固定CO2的限速步主要集中在暗反应,而其中的Calvin循环(也称C3循环)是所有光合生物碳素光合流动过程的中心环节。SBPase基因的表达产物为景天庚酮糖-1,7-二磷酸酶(sedoheptulose-bisphosphatase,SBPase)。该酶是卡尔文循环过程中的关键酶,不可逆地催化景天庚酮糖-1,7-二磷酸去磷酸化,生成景天庚酮糖-7-磷酸。景天庚酮糖-7-磷酸经过一系列酶促反应转变为CO2的受体核酮糖-1,5-二磷酸。因此,SBPase是植物光合作用途径的主要限速酶之一(231。
电子克隆(in silico cloning)是近几年伴随着基因组计划和表达序列标签EST(expressed sequence tag)计划发展起来的基因克隆新方法。但是数据库中的EST数据最高精确度为97%1。许多植物物种的ESTs数据库还没有建立起来,实现全长cDNA的电子克隆还有一定的难度。
1材料与方法
1.1生物信息学数据库
本研究使用的数据库为美国生物技术信息中心(NCBI:http://www.ncbi.nlm.nih.gov)。
1.2分析工具
基本BLAST检索:ttp://www.ncbi.nlm.nih.gov/blast;开放阅读框分析:http:/www.ncbinm.ih.gvgorflorfig.gi;氨基酸序列翻译:http://web.expasyorg/translate/;
酶切图谱分析:http://ools.neb.com/NEBeutter2/;亚细胞定位分析:http://www.genscript.com/wolf-psorthtml;蛋白质序列理化性质分析:http://web.expasy.org/protparam/;
跨膜区域分析:http://www.cbs.dtu.dk/services/TMHMM/;蛋白质卷曲螺旋分析:http://embnet.vital-it.ch/software/COILS_form.html;
信号肽分析工具:http://www.cbs.dtu.dk/service/SignalP-3.0/;
蛋白质三级结构预测:Swiss-Model:http:/wissmodel.expasy.org/;
蛋白质二级结构预测:http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html。
1.3生物信息学分析软件
本研究使用的生物信息学分析软件主要包括BIOEDIT、MEGACLUSTALX等。
1.4电子克隆
在NCBI数据库中分别以拟南芥光合作用酶SBPase的氨基酸序列作为探针序列,采用tBLASTn程序在茶树EST数据库中进行比对,获得与探针序列相似性较高的EST序列,然后进行序列装配,得到重叠群contig。再将装配结果在NCBI的茶树EST数据库中进行反复检索与装配,直到没有发现更多的EST序列出现后,停止装配,通过结构分析,最终得到完整的茶树SBPase基因电子克隆eDNA序列。
1.5生物信息学分析
运用BIOEDIT软件分析基因序列的组成和特征。运用NCBI在线ORF分析工具ORFFinder分析序列的开放阅读框。运用EXPASY网站translatetools工具将待查询的基因序列的编码区翻译成氨基酸序列,然后对其进行结构和功能预测。运用在线的NEBecutterV2.0工具对基因序列进行酶切图谱分析。
将待测的氨基酸序列提交至Expasy数据库中的ProtParam工具中,得到序列的氨基酸分子式、半衰期、不稳定性指数等值。利用BIOEDIT软件,对编码蛋白质的氨基酸组成特点和分子量进行分析和疏水性预测。运用Expasy中的TMHMMServerV.2.0程序对蛋白进行跨膜结构预测。数值≤0.5表示该区域为跨膜区域的可能性很小,数值>0.5表示该区域为跨膜区域的可能性较大。
本研究采用SOMPA算法对编码蛋白质的二级结构进行了预测。将待测的氨基酸序列提交至在线的COILS Server服务器中,分析蛋白质卷曲螺旋。运用在线的服务器SignalP 3.0 Server对蛋白质序列进行信号肽分析。将序列提交到在線服务器SWISS-MODEL进行蛋白质的三级结构预测。本研究使用NetPhos3.1软件进行serine、threonine和tyrosine磷酸化位点预测。运用在线服务器WoLFPSORT,进行蛋白质的亚细胞定位分析。
运用NCBI上的BLAST检索工具搜索序列的同源性序列,将具有较高同源性的序列用ClustalX程序进行序列比对。再将比对结果,使用Mega程序构建系统进化树。
2结果与分析
2.1茶树SBPase基因cDNA序列及编码氨基酸序列
通过数据库检索与装配,得到茶树SBPase基因的eDNA电子克隆序列(图1)。序列分析结果显示,SBPase基因全长为1621bp,132~1301为该基因的编码区,编码框长度为1170bp,共编码389个氨基酸。ATG为起始密码子,TAA为终止密码子(图2)。
2.2翻译后修饰位点预测及信号肽与跨膜结构分析
翻译,后修饰位点预测结果显示,SBPase蛋白磷酸化位点共有48个,其中Ser磷酸化位点有32个、Thr磷酸化位点有12个,Tyr磷酸化位点有4个。Ser磷酸化位点分别位于19、24、272930、32、3436、37、3942、44、49、57、61、66、67、77、106、110、120、148、166、176、255、308、310、311.336、341、360、376.Thr磷酸化位点分别位于23、63、7086、186、197、216、217、281.307、354371。Tyr磷酸化位点分别位于268、280、334、372。
蛋白信号肽分析结果显示,由神经网络模型分析得出的C(0.370)、Y(0.200)、S(0.636)值可知SBPase编码蛋白的Y、C值不太高,信号肽曲线不典型,表明该蛋白质没有信号肽。同时,隐马尔夫模型分析结果也进一步证明了该结论。跨膜结构分析结果显示,SBPase跨膜区域存在的可能性较小,预测SBPase蛋白不存在跨膜区域,属于非跨膜蛋白。2.3蛋白质二级与三级结构预测
本研究运用了SOMPA算法对蛋白二级结构进行了预测,结果如表1所示。
将蛋白序列提交到SWISS-MODEL,得到蛋白质的三维预测结构模型(图3)。其中,深红色表示为a螺旋,黄色表示为β折叠,淡蓝色表示为转角,其他残基为白色。得到的结果与二级结构预测基本吻合。蛋白质卷曲螺旋分析显示,没有较明显的卷曲螺旋结构,45~70、100~110等处有不太明显的卷曲螺旋结构。
2.4编码蛋白质的理化性质分析与亚细胞定位
SBPase基因编码的蛋白质分子式为Cr121299N4g9OsS17,原子总数为5959,分子量为42227.40,不稳定性指数为34.83,为稳定性蛋白,主要氨基酸残基数为Ser40、Leu38、Gly35,碱性氨基酸残基数为43,酸性氨基酸残基数为44;等电点为6.55,平均疏水性为-0.085。
利用BIOEDIT软件对基因编码的蛋白质理化性质进行分析,根据负值越大表示蛋白亲水性越强、正值越大表示蛋白疏水性越强、介于-0.5~0.5的主要为两性氨基酸的规律问,预测结果(图4)为SBPase酶的最高值为1.7,位于第322位,表示疏水性最强;最低值-1.8出现在第358位,亲水性最强。结合ProtPram预测结果,可推测茶树SBPase酶为亲水性蛋白。
根据亚细胞定位分析结果,预测SBPase蛋白分布在叶绿体内。
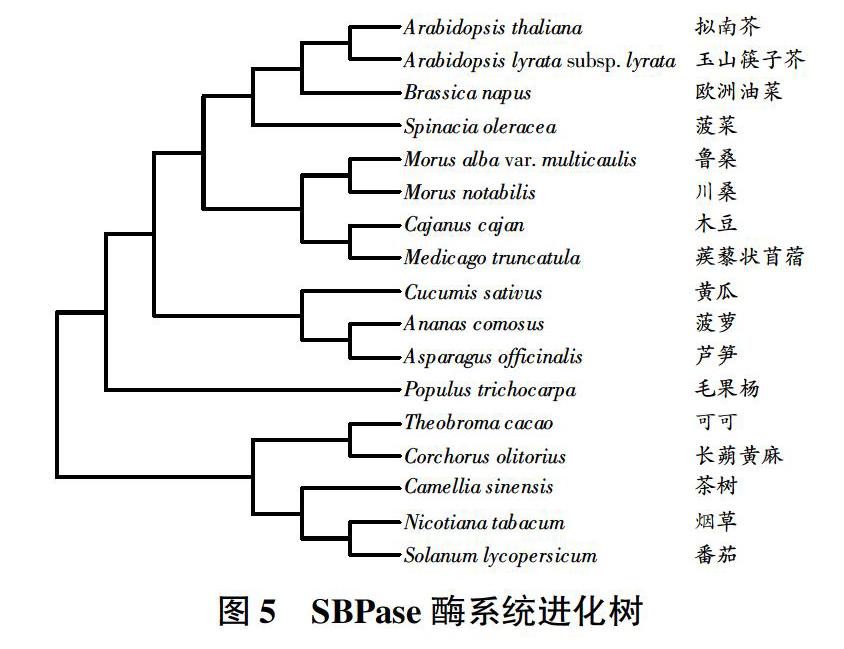
2.5系统进化分析
将SBPase氨基酸序列提交到数据库中,构建系统进化树(图5),可以看到有多条氨基酸序列与SBPase氨基酸序列有较高的相似性,其中茶树SBPase氨基酸序列与番茄(Solanum lycopersicum)、毛果杨(Populus trichocarpa)、可可(Theobroma cacao)、长蒴黄麻(Corchorus olitorius,')、烟草(Nicotianatabacum)相似性较高,分别为82%、80%、82%、82%、81%。经比对发现,SBPase蛋白属于FIG家族,具有1个典型的PLN02462保守结构域。
3结论与讨论
通过EST序列电子拼接技术,获得茶树SBPase基因cDNA序列,序列全长为1681bp,开放阅读框长度为1170bp,可编码389个氨基酸,理论分子量为42227.40,平均疏水性为-0.085,预测等电点为6.55,不稳定性指数为34.83。SBPase为稳定性蛋白,在细胞内有较好的稳定性,能参与植物的光合作用。从蛋白质跨膜结构分析上看,SBPase蛋白不含有明显的跨膜区,属于非跨膜蛋白。SBPase蛋白的二级结构主要由螺旋、折叠和线圈构成,卷曲占少量比例,属于mixed型。SWISS-MODEL预测的三级结构主要由螺旋、折叠和线圈构成,卷曲占少量比例。由蛋白质信号肽分析可知,SBPase蛋白信号肽曲线不典型,所以推测该蛋白没有明显的信号肽。该蛋白不是分泌蛋白,只在细胞内参与反应。结果显示,茶树SBPase酶在系统进化上与烟草SBPase酶关系较近。
刘逊亮发现,SBPase蛋白的结构变化可以抑制植物的光合碳同化效率,进而影响植物的生长发育叶绿体生成和结实率。转基因结果显示,超表达杨树SBPase基因,可以促进拟南芥碳水化合物的积累,利于RuBP的形成和淀粉等多糖的合成,增强植物的光合作用8。开展茶树SBPase基因的电子克隆与生物信息学分析,可以为山茶属植物相关光合作用基因的克隆及结构与功能研究提供支撑。
4参考文獻
[1]孙君,朱留刚,林志坤,等.茶树光合作用研究进展[J].福建农业学报,2015,30(12):1231-1237.
[2]冀宪领,盖英萍,马建平,等.桑树景天庚酮糖-1,7-二磷酸酶基因的
克隆原核表达与植物表达载体的构建[J].林业科学,2008,44(3):62-69.
[3]袁传忠,韩雪娟,李轶群,等.桑树景天庚酮糖-1,7-二磷酸酶基因的遗传转化及生物学功能分析[J].蚕业科学,2013(3):413-419.
[4]何志颖,姚玉成.EST技术及其在基因全长eDNA克隆上的应用策略[J].国际遗传学杂志,2002,25(2):67-69.
[5]孙淼,赵茂林.利用表达序列标签电子克隆cDNA全序列的策略[J].生物技术通报,2010(1):49-52.
[6]张开雪,任伟超,闫嵩,等软枣猕猴桃GDP-L半乳糖磷酸酶电子克隆与生物信息学分析[J].中国药师,2016,19(11):2066-2069.
[7]刘逊亮.氧化胁迫下拟南芥卡尔文循环关键酶SBPase的羰基化修饰及其功能研究[D].北京:中国科学院大学,2012.
[8]王俊秀,周扬颜,韩潇,等.超表达杨树SBPase基因促进拟南芥光合作用及营养生长[J].北京林业大学学报,2018(3):26-33.
