谷子磷酸烯醇式丙酮酸羧化酶基因(PEPC)对逆境胁迫的响应
2019-09-02赵晋锋王高鸿杜艳伟李颜方王振华赵根有余爱丽
赵晋锋,王高鸿,杜艳伟,李颜方,王振华,赵根有,余爱丽
(山西省农业科学院 谷子研究所,特色杂粮种质资源发掘与育种山西省重点实验室,山西 长治 046011)
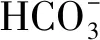
谷子(Setariaitalica(L.)P. Beauv.)是起源于我国的C4禾本科作物,不仅具有蒸腾系数低、净光合强度和水分利用效率高、抗逆性强、适应性广等特点,而且谷子基因组小,高度保守,DNA重复度低,非常便于研究[13]。同时谷子在其生长周期中会经常遇到众多非生物逆境胁迫,比如干旱、盐渍、低温、高温、洪涝以及病虫害侵染等,这些逆境严重影响了作物的生长发育和产量品质[14-16]。目前为止,尽管PEPC基因相关研究开展颇多,但是谷子中PEPC基因在非生物逆境下的表达研究报道很少。
本研究发掘了谷子中的SiPEPC(Seita.1G020700)基因,并对其氨基酸序列、蛋白特征、功能、信号途径、顺势应答元件等参数特征进行分析和预测,随后分析了该基因在幼苗期逆境胁迫下的动态表达模式以及在拔节、抽穗、灌浆3个生育时期不同光照处理和干旱胁迫下的表达,旨在为进一步分析SiPEPC基因在谷子逆境应答信号途径中的功能和机制以及为利用基因工程方法改善作物光合速率和提高产量提供数据支持。
1 材料和方法
1.1 试验材料
供试谷子品种为豫谷1号。
试验所用 LATaqDNA聚合酶,逆转录酶和RNA酶抑制剂均购自宝生物工程有限公司;引物及其他常规生化试剂购自生工生物工程(上海)有限公司。
1.2 试验方法
1.2.1 试验设计 在幼苗生长至三叶期时,分别对其进行 20% PEG,盐(250 mmol/L NaCl),ABA(100 μmol/L)和低温(4 ℃) 胁迫处理,于0,1,3,6,12,24 h整株取样[17]。另外,在旱棚种植豫谷 1 号,光照处理为当植株出苗后用黑色遮阳网分别遮挡1层(光照Ⅰ处理),2层(光照Ⅱ处理)至成熟收获;对照生育期内正常浇水;干旱处理只浇3次关键水,分别为拔节、抽穗、灌浆期取样后浇水,其他时间采用自然控水方式控水;其他农田管理措施相同。所有样品叶片取样后立即在-80 ℃冰箱中速冻备用。试验设2次生物学重复。
1.2.2 引物设计和植物总RNA提取 植物总RNA提取参照生工TRIzol试剂盒说明书,试验中涉及的其他试剂配制参照《分子克隆》第3版[18]。引物设计用 Primer Primer 5.0软件根据SiPEPC、SiGAPB转录序列设计SiPEPC及对照SiGAPB荧光实时定量PCR引物。
1.2.3SiPEPC基因发掘和生物信息学分析 利用拟南芥中已鉴定PEPC基因序列在JGI 谷子数据库(Ver.2.1)进行比对发现siPEPC基因(Seita.1G020700)。SiPEPC蛋白理化特性、氨基酸组成、一二级结构等用ExPASy网站ProtParam pI/Mw在线工具预测;SiPEPC潜在功能用Profun 2.2 Server预测,信号肽用SignalP 4.1 Server 进行分析;SiPEPC编码蛋白亚细胞定位用Psort在线工具预测;SiPEPC氨基酸同源性序列用Blast工具在NCBI上查找。同源基因序列用ClustalX 1.83软件比对分析,不同物种PEPC基因(玉米ZmPEPC、高粱SbPEPC、谷子SiPEPC、水稻OsPEPC、拟南芥AtPEPC、大豆GmPEPC、北美云杉PsPEPC、小立碗藓PpPEPC,其GenBank序列号分别为NP_001105503.1、XP_002451855.1、RCV04685.1、AAG00180.1、AAC24594.1、NP_001241357.1、ABR17346.1和XP_024395991.1)进化树用Mega 4.1软件构建并比较分析[19]。
1.2.4 光照强度测定 在不同生育期用光强测定仪(GM1040)于晴天上午 9:00-10:00测量光照强度,测量10次数值,取平均值。具体生育期测量光照强度值如表1所示。
1.2.5 Real-time PCR 分析 总RNA样品反转录合成cDNA,均一化后作为实时定量PCR 模板,以SiGAPB作为内参基因。经预试验优化后,PCR反应程序为:95 ℃ 预变性2 min;95 ℃变性7 s,61 ℃退火10 s,72 ℃延伸 15 s,共 45 个循环。SiPEPC在逆境胁迫下转录水平变化采用相对定量 2-ΔΔCt方法计算,设3次重复[20]。
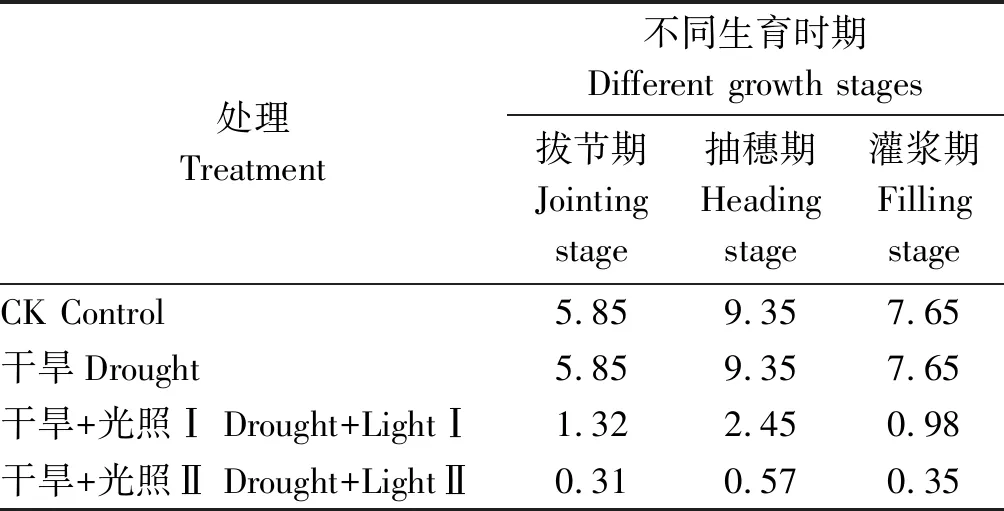
表1 不同处理各生育期光照强度值Tab.1 Light intensity values of treatments at different growth stages ×104 lx
2 结果与分析
2.1 SiPEPC基因序列的获得与参数分析
通过 NCBI 数据库比对从谷子数据库中得到谷子 PEPC(Seita.1G020700)基因,蛋白结构域在线分析显示,该基因存在 PEPcase(PF00311)结构域(164-965 Aa),因此,确认该基因为PEPC家族成员(图1)。该基因组序列长6 652 bp,转录序列3 366 bp,CDS 序列2 898 bp,编码965个氨基酸,无内含子和可变剪切。同源序列分析显示,不同物种PEPC基因非常保守,相似性较高,所有PEPC蛋白一致性(Identity)为89.09%。

图中黑色背景氨基酸表示相同氨基酸残基,灰色背景氨基酸表示相似氨基酸残基(≥60% similarity);序列中磷酸烯醇式丙酮酸羧化酶结构域用黑色连字符方框标出。The black background amino acids represent the same amino acid residues, and the gray background amino acids represent similar amino acid residues(≥60% similarity);The phosphoenolpyruvate carboxylase domain in the sequence is indicated by a black hyphen box.
2.2 SiPEPC 基因的生物信息学分析
2.2.1 SiPEPC蛋白参数分析SiPEPC(Seita.1G020700)基因蛋白分子式为 C4906H7796N1360O1449S37,分子质量为 110.20 ku,等电点(pI)为5.77,平均疏水性(GRAVY)-0.351,脂肪系数(AI)92.41,不稳定指数44.74。二级结构预测显示,螺旋(包括 α-,pi-and 3_10-helix)58.65%,β-折叠链 3.11%,loop 环为 38.24%。溶剂可及性分析显示,膜外 40.41%,膜内 48.91%,镶嵌 10.67%。亚细胞位置预测结果推测,SiPEPC(Seita. 1G020700)基因所编码的蛋白被定位在细胞质中(预测置信度97%)。另外, Psort 软件预测该基因可能存在线粒体基质、微体(过氧物酶体)、细胞核、叶绿体类囊体膜,其预测概率大小依次为 51.6%,30.0%,30.0%,28.0%。根据SignalP 4.1 Server软件预测可知,SiPEPC第36位丙氨酸残基具有最高的原始剪切位点,分值为 0.107;第11位谷氨酰胺信号肽分值最高,为 0.130;最高的综合剪切位点位于第1位甲硫氨酸残基,其值为 0.197。综合来看,SiPEPC氨基酸残基的加权平均值较小,为0.138(小于 0.500),推测SiPEPC基因编码的蛋白为非分泌蛋白。
2.2.2 谷子SiPEPC基因启动子区域顺式元件分析和功能预测 顺式元件分析发现,在SiPEPC(Seita. 1G020700)基因启动子区域主要包括光应答元件(3-AF1 binding site、ATCT-motif、Box 4、G-Box、GATA-motif、LAMP-element、TCT-motif),激素类应答元件 ABRE(Abscisic acid responsiveness),P-box(Gibberellin-responsive element),TCA-element (Salicylic acid responsiveness),逆境类应答元件 TC-rich repeats(Defense and stress responsiveness)以及其他类元件,包括厌氧诱导 ARE(Anaerobic induction),缺氧特异诱导的增强子 GC-motif(Enhancer-like element involved in anoxic specific inducibility),生长素 TGA-element(Auxin-responsive element)。Profun 2.2 Server 软件预测显示,SiPEPC 蛋白具有 C-C裂解酶活性、磷酸烯醇式丙酮酸羧化酶活性、镁离子结合等活性,可能参与单体代谢、有机物代谢、固碳、三羧酸循环、需氧呼吸、前体代谢物和能量的产生、细胞呼吸、氧化还原以及初级代谢等生物过程。
2.2.3 PEPC氨基酸序列进化树分析 用Neighbor-joining方法构建谷子、拟南芥、玉米、高粱、水稻、北美云杉、小立碗藓等物种中的PEPC氨基酸序列进化树。序列比对结果显示SiPEPC与高粱、玉米和水稻中PEPC基因同源性非常高,序列一致性分别为96.21%,96.11%和93.65%;而SiPEPC与拟南芥、大豆、北美云杉、小立碗藓的序列一致性则相对略低,分别为82.79%,81.86%,82.99% 和74.59%。进化树结果也显示,SiPEPC与高粱、玉米和水稻中PEPC基因亲缘关系较近,而与拟南芥、大豆、北美云杉、小立碗藓的亲缘关系较远(图2)。
每个位点氨基酸替代值在底部用0.01刻度值标出。Amino acid substitution values of each site were labeled with a 0.01 scale value at the bottom.
2.3 不同非生物逆境胁迫下SiPEPC 基因表达分析
如图3所示,苗期4种胁迫处理下SiPEPC基因表达量变化模式不完全相同,但在受到胁迫后均被诱导,其表达量均有所上调。在ABA处理时,表达量呈现下调上调交替出现,在 12 h 被诱导达到最高,为对照的8.20倍;在低温诱导时,表达量持续上升,在24 h达到最高,为对照的9.70倍;在PEG和NaCl处理时,表达量整体呈上升趋势,均在12 h达到最高,分别为相应对照表达量的4.85,9.96倍,在24 h 其表达量均急剧下调。
2.4 不同生育期 SiPEPC 基因在干旱和光照处理下的表达分析
如图 4 所示,拔节期干旱条件下(光照5.85万lx)SiPEPC基因表达被诱导;干旱+光照 Ⅰ(1.32万lx)处理下表达量下调;干旱+光照Ⅱ(0.31万lx)处理条件下该基因表达量上调至对照的 2.74 倍。抽穗期干旱条件下(光照9.35万lx)SiPEPC基因表达被诱导上调;干旱+光照Ⅰ(2.45万lx)以及干旱+光照Ⅱ(0.57万lx)处理条件下该基因表达量均下调。灌浆期干旱条件(光照7.65万lx),干旱+光照Ⅰ(0.98万lx)以及干旱+光照Ⅱ(0.35万lx)处理条件下该基因表达量均下调。
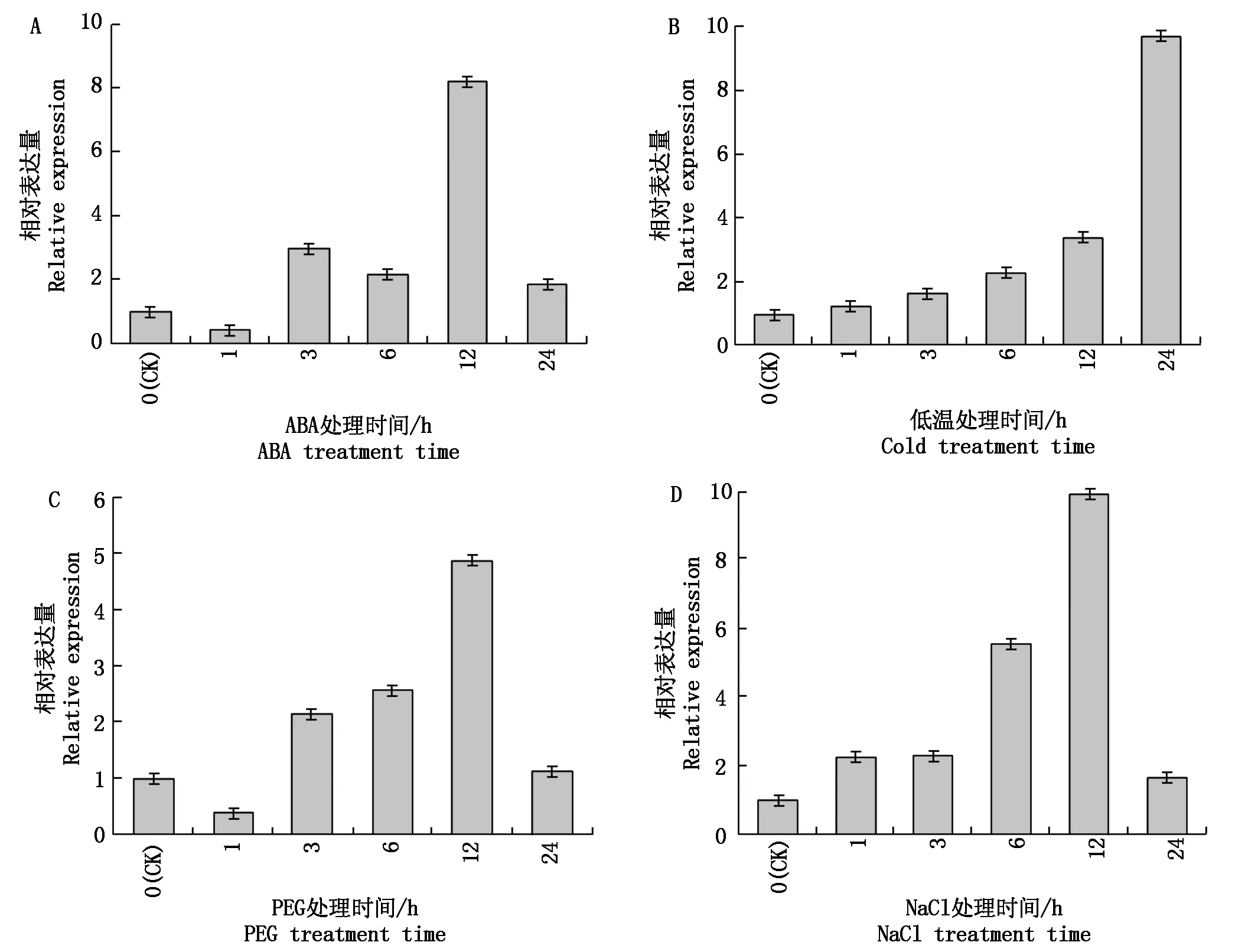
图3 SiPEPC(Seita. 1G020700)在不同逆境处理下的 Real-time PCR 表达分析Fig.3 Real-time PCR expression analysis of SiPEPC(Seita. 1G020700)under different stress treatments
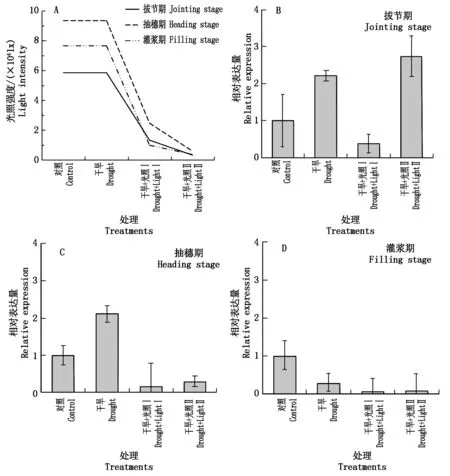
A.不同生育期光照强度值;B.拔节期不同处理SiPEPC基因表达;C.抽穗期不同处理 SiPEPC 基因表达;D.灌浆期不同处理 SiPEPC 基因表达。A.The light intensity of different growth stage;B.The expression of SiPEPC under different treatments at jointing stage;C. The expression of SiPEPC under different treatments at heading stage;D.The expression of SiPEPC under different treatments at filling stage.
3 结论与讨论
3.1 SiPEPC 基因编码磷酸烯醇式丙酮酸羧化酶蛋白
本研究通过生物信息学方法得到抗旱耐瘠作物谷子的SiPEPC基因 (Seita.1G020700), 该基因序列长6 652 bp,转录序列 3 366 bp,CDS 序列 2 898 bp,编码965个氨基酸,无内含子和可变剪切,功能域分析揭示,164-965位氨基酸为磷酸烯醇式丙酮酸羧化酶结构域 (PEPcase Motif),因此,推测该基因为谷子 PEPC 家族成员。不同物种中PEPC蛋白序列比对发现,PEPC 蛋白非常相似,所有比对PEPC序列整体一致性为89.09%,具有非常保守的序列结构。另外,SiPEPC与高粱、玉米和水稻中PEPC基因同源性非常高,其序列一致性分别为96.21%,96.11%,93.65%;而与拟南芥、大豆、北美云杉、小立碗藓的序列一致性则相对较低,分别为82.79%,81.86%,82.99%,74.59%。此结果和这些物种在植物分类上的亲缘关系是一致的(高粱、玉米、谷子是禾本科 C4 作物,水稻是禾本科单子叶作物,拟南芥属十字花科双子叶作物,大豆属于豆科双子叶作物,而北美云杉和小立碗藓分别属于裸子植物和苔藓植物的典型代表)。另外,SiPEPC和玉米、高粱水稻同源基因在系统发育进化树中也聚在一起,也证明了这一推论。此外,在北美云杉 (裸子植物) 和小立碗藓(苔癣类植物)中存在 PEPC 同源基因,说明了PEPC基因的祖先在裸子植物和被子植物分离之前就已存在。
3.2 SiPEPC基因参与多种逆境应答
研究表明,PEPC转基因株系的抗旱、耐盐等抗逆性能得到了明显提高,而且在逆境胁迫下维持了较高的光合速率[21-25]。顺式元件分析也发现,在SiPEPC(Seita. 1G020700)基因启动子区域逆境类应答元件和激素类元件。所以,比较关注该基因在逆境下对逆境胁迫的响应。因此,本研究分别用20% PEG、ABA(100 μmol/L)、低温(4 ℃)、盐(250 mmol/L NaCl)等逆境处理豫谷1号幼苗,然后用 Real-time PCR 分析其在不同处理下的表达情况,结果表明,SiPEPC基因在 4 种胁迫处理下其表达量均有显著上调,在 ABA、低温、PEG和NaCl胁迫时,其诱导表达量最高值分别为对照的8.20,9.70,4.85,9.96倍,说明SiPEPC基因在谷子苗期对逆境响应明显,可能在逆境应答信号途径中调整植物的光合作用来响应不同逆境胁迫。为了解该基因在不同生育期逆境胁迫和不同光照强度下的表达情况,本研究在旱棚种植材料设计试验用Real-time PCR进行了分析,结果表明,在拔节期和抽穗期干旱正常光照下,SiPEPC基因表达量均被诱导上调(分别为对照2.20倍和2.10倍),但是在灌浆期其表达量是下调的(对照的0.28倍),说明SiPEPC基因在干旱正常光照下的拔节期和抽穗期对干旱的响应比灌浆期强烈;而在干旱光照Ⅰ(中等光照)和光照Ⅱ(弱光照)的抽穗期和灌浆期SiPEPC基因表达被下调;但是在干旱和光照Ⅱ条件下的拔节期中SiPEPC表达量被上调,达对照的2.74倍。说明该基因在干旱条件下中等和弱光照条件下的抽穗期和灌浆期可能起作用较小。这些试验结论进一步验证了顺势元件分析结果,证明了SiPEPC基因在植物逆境应答中起一定作用,尤其是在苗期受到不同逆境的强烈诱导。
进一步研究表明,SiPEPC基因在拔节期和抽穗期正常光照下参与了对干旱胁迫的响应,而干旱条件下的中等光照和弱光照处理下SiPEPC基因表达在不同生育期几乎都受到严重抑制,有可能是弱光处理严重影响了干旱条件下的基因表达。功能预测分析显示,SiPEPC可能参与单体代谢、有机物代谢、固碳、三羧酸循环、需氧呼吸、前体代谢物和能量的产生、细胞呼吸、氧化还原以及初级代谢等生物过程,其中大部分功能与光合作用有关的。逆境胁迫下SiPEPC基因是如何参与调控这些生物过程仍有待进一步详细验证。
植物生长发育过程中经常面临各种非生物逆境胁迫的挑战,如干旱、高盐、低温等,这些逆境胁迫对植物的生长发育影响最为明显,是制约农作物产量和品质的主要限制因素[26]。本研究对预测谷子SiPEPC基因的氨基酸序列、蛋白特征、功能、信号途径、顺势应答元件等参数特征进行系统的预测和分析,并对该基因在苗期不同逆境处理及不同生育期干旱胁迫下的动态表达情况进行了初步分析,结果表明,SiPEPC因表达量在谷子幼苗期被ABA、低温、PEG、高盐胁迫诱导均上调;SiPEPC基因在谷子拔节期和抽穗期正常光照强度下参与了对干旱胁迫的响应。因此,推测SiPEPC(Seita.1G020700)基因参与了谷子对非生物逆境的应答,可能在干旱和其他逆境胁迫信号途径中起关键作用。研究结果将为今后开展SiPEPC基因功能和利用研究提供试验数据支持。
