青藏高原青稞育成品种的亲缘系数分析
2019-08-27李健丰先红蔡一林
李健,丰先红,蔡一林
青藏高原青稞育成品种的亲缘系数分析
李健1,2,丰先红2,蔡一林1
(1西南大学农学与生物科技学院,重庆 400715;2甘孜藏族自治州农业科学研究所,四川康定 626000)
【】分析青藏高原青稞育成品种的遗传多样性,评估其遗传基础,探索拓宽其遗传基础的对策,推动青稞新品种选育工作。【】以1950s后,影响力高、推广面积大的105个藏区青稞育成品种和3个引进品种为研究对象,采用亲缘系数分析法分析参试品种的遗传基础,进而以亲缘系数为基础,采用兰氏距离、类平均法对参试品种进行聚类分析;采用累积直系亲缘系数,筛选藏区主要骨干亲本。【】18.01%的品种组合间存在亲缘关系,各组合亲缘系数的变异范围为0.000—0.750,组合平均亲缘系数为0.053,显著高于中国大麦主栽品种以及北美大麦品种。根据参试品种的育成时间,以十年为期,将参试品种划分为7个时期,1970s和1980s 2个时期,参试育成品种的组合平均亲缘系数较高,分别达到0.115和0.107;1990s和最近十年,参试育成品种的组合平均亲缘系数较低,分别为0.040和0.032。西藏自治区、青海省、四川省、甘肃省的参试育成品种数分别为48、27、17和12个;甘肃省的参试育成品种组合平均亲缘系数最高,达到0.220,西藏自治区、青海省、四川省的参试育成品种组合平均亲缘系数较为接近,分别为0.094、0.122和0.138。聚类分析的结果显示,参试品种聚为7类,Ⅱ、Ⅲ类群共包含25个参试品种,各类群的组合平均亲缘系数较低;其他5个类群共包含83个参试品种,各类群的组合平均亲缘系数较高。40个参试品种的累积直系亲缘系数大于0,其中9个育成品种是藏区青稞的重要骨干亲本,其衍生了67个参试品种。9个骨干亲本及其67个衍生种占参试育成品种总量的72.38%。1960s,骨干亲本及其衍生种的数量占该时期参试品种总量的41.76%,之后的4个时期,骨干亲本及其衍生种的数量占各时期参试育成品种总量的比例分别达到80.00%、80.77%、76.47%和72.22%;最近十年,该指标虽有所下降,但仍然高达68.75%。对于西藏自治区、青海省、四川省和甘肃省,骨干亲本及其衍生品种数占各地区参试育成品种总量的比例分别达到79.17%、62.96%、70.59%和75.00%。参试育成品种所使用的亲本合计118个,其中本地亲本97个,占比82.20%,外引亲本15个,占比12.71%;剩余6个亲本的来源信息缺失。【】藏区青稞的9个骨干亲本及其67个衍生品种构成了育成品种的主体,外引大麦种质利用率低,从而导致青藏高原青稞育成品种的遗传多样性较低,遗传基础相对狭窄、脆弱。
青藏高原;青稞;育成品种;亲缘系数;遗传多样性
0 引言
【研究意义】青稞既是青藏高原主要粮食作物,又是酿酒原料和牲畜饲料。在国际环境复杂多变的条件下,藏区“粮食安全”的实质就是以“青稞安全”为核心的食物安全[1]。青稞新品种选育工作,在藏区已经开展了近70年,以系统选择、杂交育种为技术支撑,培育了大批优良品种。由于长期对产量、品质、抗性等性状的连续定向选择,很可能造成育成品种的遗传基础日益狭窄,遗传多样性下降,不利于当前青稞新品种的选育[2-3]。【前人研究进展】近年来,利用分子标记(SSR、RAPD、SRAP)、同工酶、醇溶蛋白等技术,对青稞种质资源的遗传多样性进行了评估[4-10]。吴昆仑[4]研究认为,青藏高原青稞种质资源具有丰富的遗传多样性。曾兴权等[5]研究认为,青稞育成品种的平均遗传多样性高于西藏青稞地方品种的平均遗传多样性。孟凡磊等[9]、杨平等[10]研究认为西藏、四川的青稞育成品种遗传多样性较低,遗传基础相对狭窄。【本研究切入点】由于参试材料和研究方法的差异,研究结果不尽相同;虽然认为西藏、四川的青稞育成品种遗传基础相对狭窄,但对其成因还缺乏了解。其次,利用分子标记进行遗传多样性评价,如果存在标记数量少、密度低,与表型性状关联度低等缺陷,其结果的参考价值、实用价值有限,而大幅提升标记密度,同时强化与表型性状的关联度,又存在技术要求高、费用高的问题。亲缘系数分析是对遗传多样性进行评价的简便方法。近年来,对于自花授粉、谱系信息较为完整的作物,利用亲缘系数结合分子标记技术分析小麦、大麦、大豆、水稻、亚麻等作物遗传多样性,发现亲缘系数法与分子标记技术的分析结果具有较高的一致性[2,11-15]。另外,亲缘系数分析可以直观地展示育成品种遗传多样性的演变进程,揭示其成因,为品种选育工作提供参考。【拟解决的关键问题】青稞是高度自花授粉作物,且育成品种有较为完整的谱系信息。本研究选择1950s后藏区选育的105个推广面积较大、影响力较高的青稞育成品种以及3个引进品种,收集、整理其谱系信息,利用亲缘系数法分析青藏高原青稞育成品种的遗传多样性,了解其演变进程,探索其成因和改良对策,从而推动当前青稞新品种选育工作。
1 材料与方法
1.1 材料
根据文献报道,选择1950年后,推广面积较大、影响力较高且谱系信息相对完整的藏区青稞育成品种105个,引进品种3个,作为参试品种(电子附表1)。
1.2 方法
根据参试品种的谱系信息,参考王春江等[16]和刘章雄等[17]的方法,计算108个品种成对组合的亲缘系数(coefficient of parentage,COP)。计算原则如下:①一个品种分别从其双亲得到一半的基因;②所有祖先种亲本及其后代品种都是纯合的;③最早的祖先品种(系)间的亲缘系数为0;④混合授粉材料各花粉供体享有相同的等同于雌配子的亲缘系数概率,例如,A与5个父本混合授粉材料杂交得到B,则B与A的亲缘系数为1/2;B与各父本的亲缘系数均为1/2×5;⑤一个品种与其系选所得品种间的亲缘系数为0.75;⑥自然突变和诱导突变材料与其祖先的亲缘系数为0.75;⑦含有部分相同亲本的旁系品种间的亲缘系数计算公式为RSD=∑ (1/2)n1+n2;R代表品种S和品种D之间的亲缘系数,n1和n2分别代表S品种和D品种的共同亲本与品种S和品种D间的世代数;⑧一个品种与其自身的亲缘系数为1.00;⑨亲本与后裔品种间的亲缘系数可以表示其遗传物质的传递比率即遗传贡献率。
根据参试品种的谱系信息,计算各品种的累积直系亲缘系数(cumulative direct coefficient of parentage,CD-COP)。首先分别计算该品种与其所有衍生种的COP系数,然后对该品种与其所有衍生种的COP系数求和。
利用DPS7.05软件,采用兰氏距离、类平均法,对COP矩阵(108×108)进行聚类分析。
2 结果
2.1 参试品种亲缘系数分析
108个参试品种形成5886对组合,其中,有1060对组合的COP值不为0,占比18.01%;各组合亲缘系数的变异范围为0.000—0.750,5 886对组合累积亲缘系数为314.146,平均值为0.053;品种间亲缘系数高于均值的组合数为771对,占比13.10%。
2.1.1 各时期参试品种亲缘系数分析 以十年为期,将藏区青稞选育工作划分为7个时期。由于1950年前,藏区青稞育种工作进展极为缓慢,1950s的4个参试品种均为历史遗留的农家种,参试品种各组合间的COP值为0。1960s,参试品种数达到12个,形成78对品种间组合,组合累积亲缘系数为1.500,组合平均亲缘系数为0.019。1970s,参试品种数小幅增加至15个,形成组合120对,组合累积亲缘系数上升至13.750,组合平均亲缘系数骤然提升至各时期的最高水平,达到0.115。1980s,参试品种数达到26个,为各时期的最高值,形成351对组合,组合累积亲缘系数为37.516,组合平均亲缘系数小幅下降至0.107。1990s,参试品种数下降至17个,形成组合153对,组合累积亲缘系数陡然下降至6.078,组合平均亲缘系数也大幅下降至0.040。21世纪初,参试品种数为18个,形成组合171对,组合累积亲缘系数回升至13.469,组合平均亲缘系数大幅回升至0.079。最近十年,参试品种数为16个,形成组合136对,组合累积亲缘系数为4.414,组合平均亲缘系数为0.032,这两项指标均处于最近40年的历史最低值(图1)。
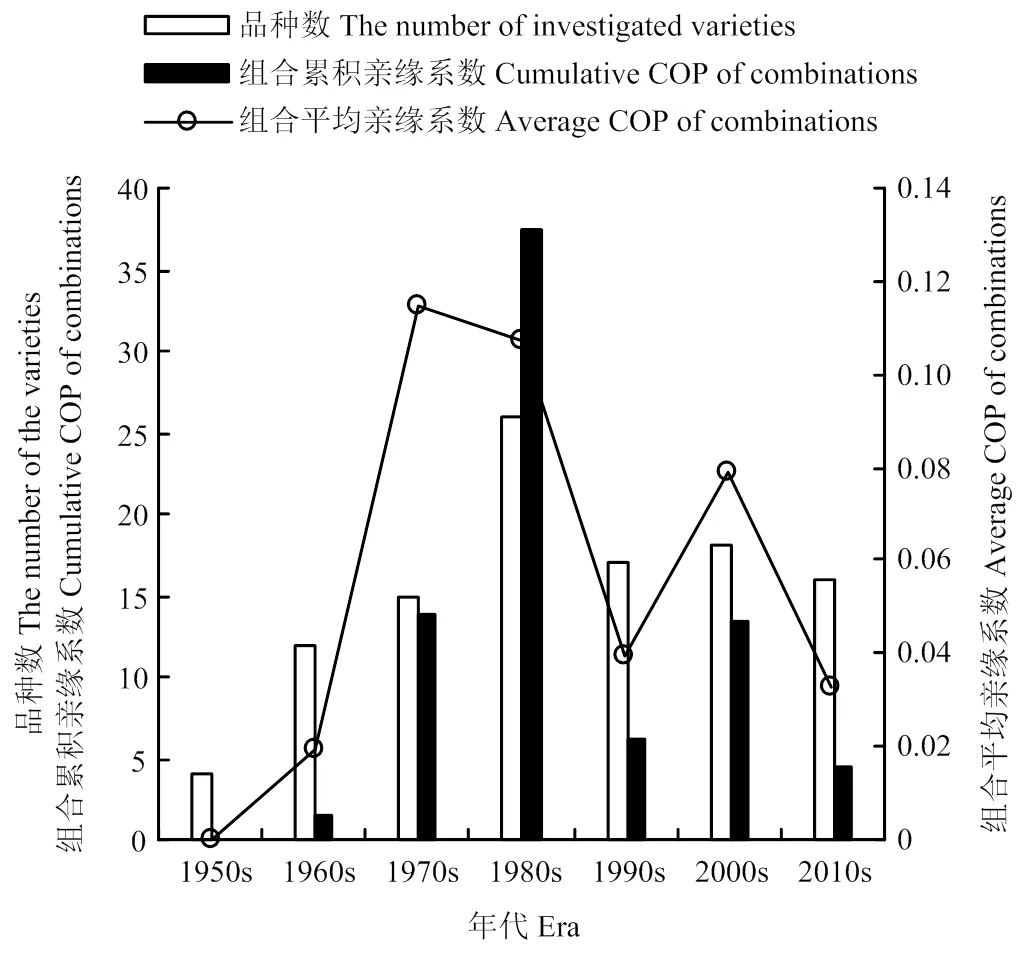
图1 各时期参试品种亲缘系数分析
2.1.2 各地区参试品种亲缘系数分析 西藏自治区参试品种数达到48个,组合累积亲缘系数为110.969,是藏区参试品种最多,组合累积亲缘系数最高的地区。青海省参试品种数、组合累积亲缘系数位列藏区第二,分别为27个和46.160。计算各地区参试品种的组合平均亲缘系数,结果表明,甘肃省最高,达到0.220,西藏自治区、青海省、四川省的参试品种组合平均亲缘系数较为接近,分别为0.094、0.122和0.138(图2)。
2.2 参试品种亲缘系数聚类分析
根据参试品种的亲缘系数,采用兰氏距离、类平均法进行聚类分析。108个参试品种聚为7个类群,不同类群间存在显著差异(电子附图1)。
2.2.1 不同类群所包含品种呈现明显的地域差异 通过分析各类群所包含品种的地理来源,发现Ⅰ类群仅包含四川省、甘肃省的育成品种,占比分别为80.00%和20.00%;Ⅱ、Ⅲ类群所包含品种的地理来源呈多样化分布,西藏自治区、青海省的育成品种数占比较高,四川省、甘肃省、云南省的育成品种数占比较低;Ⅳ、Ⅴ类群以西藏自治区育成品种为主体,其中,Ⅴ类群所包含品种全部来源于西藏自治区;Ⅵ类群所包含品种以甘肃省育成品种为主体;Ⅶ类群50.00%的品种来自西藏自治区,40.00%的品种来自青海省,10.00%的品种来自四川省(图3)。

图3 各类群参试品种地理来源分布
2.2.2 群内组合累积亲缘系数、平均亲缘系数差异显著 Ⅰ类群包含10个品种,群内组合累积亲缘系数为17.500,群内组合平均亲缘系数为0.318。Ⅱ类群包含12个品种,组合累积亲缘系数和组合平均亲缘系数均为0。Ⅲ类群包含13个品种,群内组合累积亲缘系数和平均亲缘系数都非常低,遗传多样性相对较高。Ⅳ、Ⅴ类群分别包含18个和11个品种,Ⅳ、Ⅴ类群的组合累积亲缘系数接近,但Ⅴ类群的组合平均亲缘系数为0.358,大幅高于Ⅳ类群。Ⅵ类群包含14个品种,组合累积亲缘系数大幅升高,组合平均亲缘系数为0.391,是各类群的最高值;Ⅶ类群包含30个品种,群内组合累积亲缘系数高达116.930,组合平均亲缘系数为0.252(图4)。
2.2.3 不同类群品种构成分析 Ⅱ、Ⅲ类群的群内组合平均亲缘系数较低,所包含品种以农家种或农家系选种、引进品种为主,其他少数品种为影响力较大、推广面积大但谱系信息部分缺失的品种。
Ⅰ、Ⅳ、Ⅴ、Ⅵ、Ⅶ类群的群内组合平均亲缘系数较高;群内所包含品种中,存在亲缘关系的品种数占比也较高。Ⅰ类群90.00%的品种与813和康青3号存在亲缘关系。Ⅳ类群66.67%的品种与南繁2号、果洛存在亲缘关系。Ⅴ类群所有品种与白玉紫芒、藏青336存在亲缘关系。Ⅵ类群所有品种与肚里黄存在亲缘关系。Ⅶ类群所有品种与昆仑1号、藏青稞存在亲缘关系。因此,Ⅰ、Ⅳ、Ⅴ、Ⅵ、Ⅶ类群均存在骨干亲本,各类群以各自的骨干亲本及其衍生种为主体,将83个参试品种划分为5类(图5)。
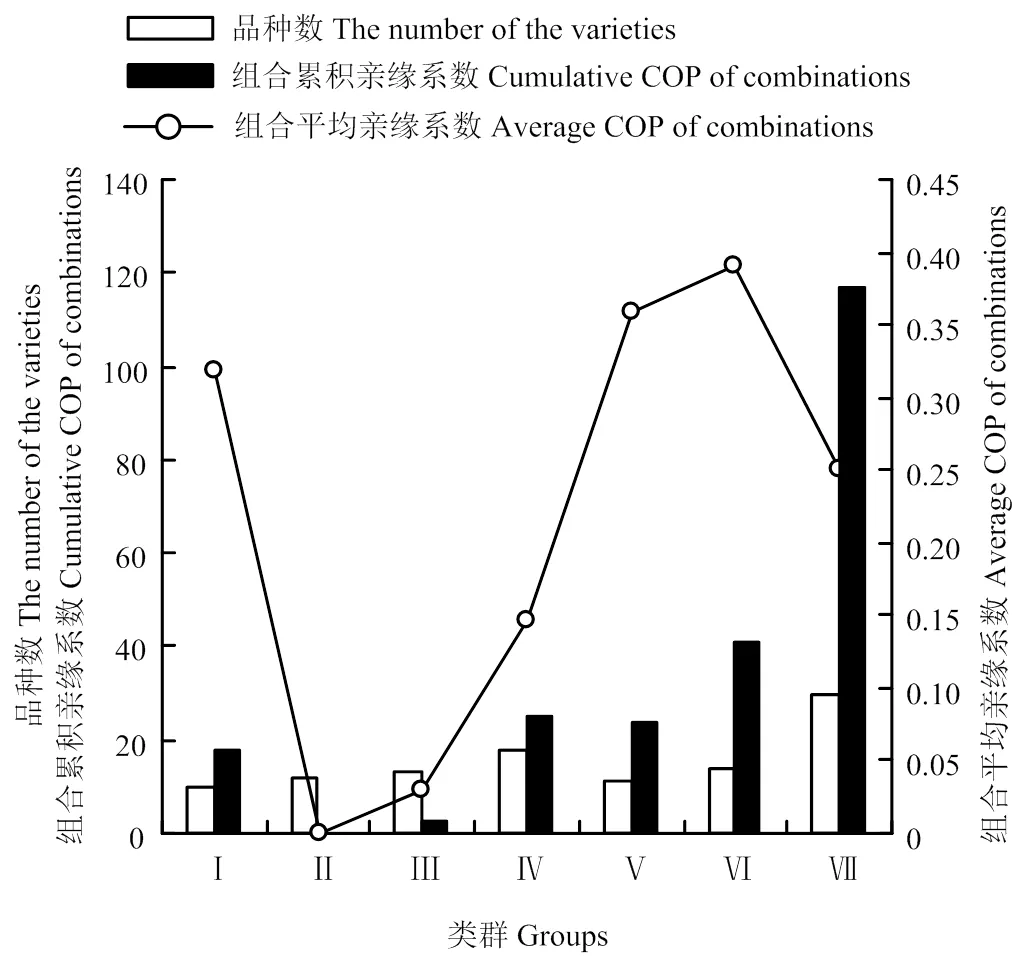
图4 各类群参试品种亲缘系数分析
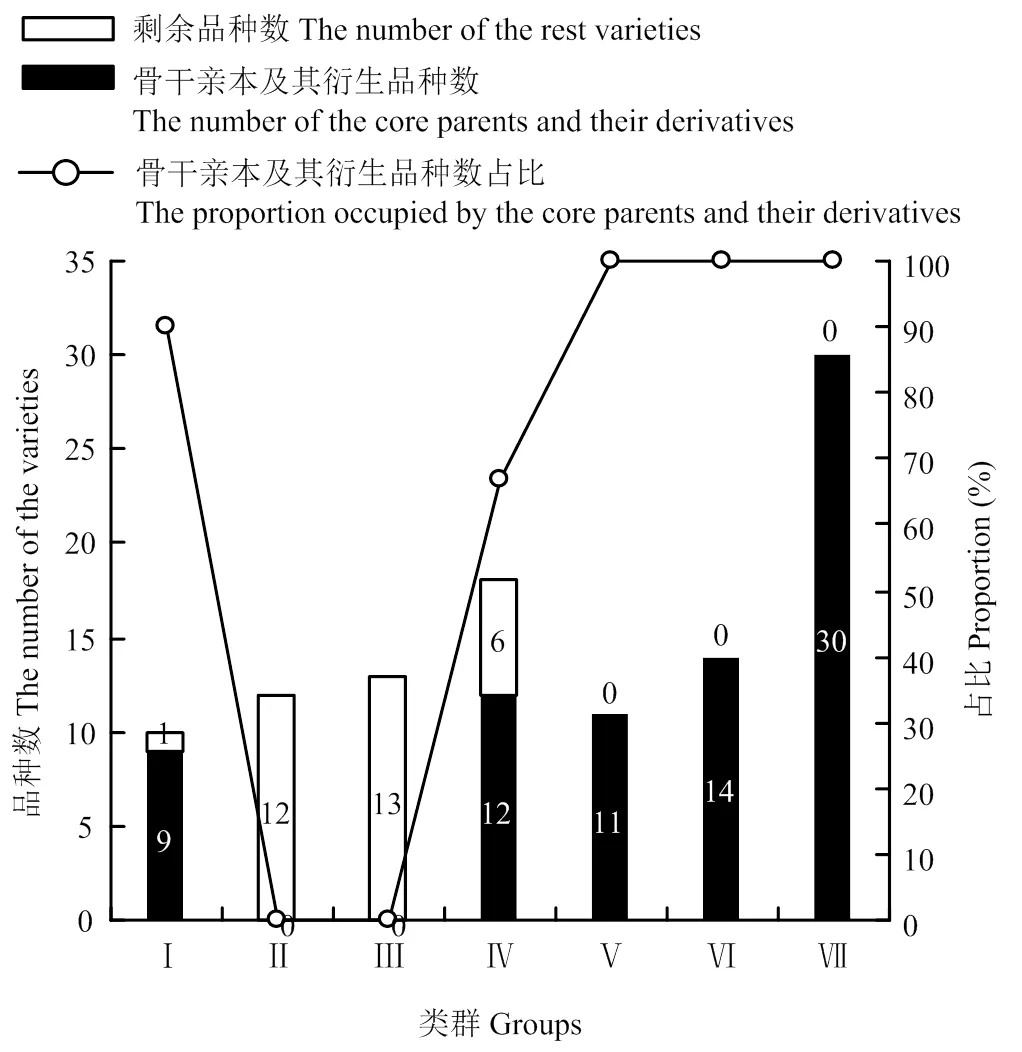
图5 各类群骨干亲本及其衍生品种数占比
2.3 藏区青稞骨干亲本分析
根据各参试品种的累积直系亲缘系数,筛选出骨干亲本。结果表明,各参试品种的累积直系亲缘系数变异范围为0.000—7.563;其中,40个品种的累积直系亲缘系数不为0;昆仑1号、肚里黄、藏青336、藏青稞、白玉紫芒、果洛、康青3号、813、南繁2号的累积直系亲缘系数位居前列,是藏区主要的骨干亲本(电子附表2)。上述骨干亲本及其衍生种的数量合计达76个,占参试育成品种总量的72.38%。
2.3.1 骨干亲本及其衍生种的时间分布 1960s,是藏区青稞品种选育的起步阶段,10年间,5个参试品种(藏青稞、藏青336、白玉紫芒、813和肚里黄)成为第一批骨干亲本,占该时期参试品种数的41.67%。1970s,育成昆仑1号和果洛2个骨干亲本,第一批骨干亲本迅速在藏区青稞品种选育活动中发挥显著作用。1970s,骨干亲本及其衍生种占该时期参试育成品种总量的80.00%。1980s是参试育成品种集中出现的时期,该时期育成青稞品种26个,其中21个是由9个骨干亲本及其衍生种组成,占比80.77%。随着时期的推移,9个骨干亲本及其衍生种占同时期参试育成品种总量的比例呈逐期下降趋势,但是降幅非常小。其后的3个时期,9个骨干亲本及其衍生种数量占同时期参试品种总量的比例分别为76.47%、72.22%和68.75%(图6)。1970s—1990s,以9个骨干亲本为亲本选育而成的品种数占参试育成品种总量的比例较高,分别达到66.67%、61.54%和70.59%;21世纪初,44.44%的参试育成品种仍以9个骨干亲本为亲本。最近十年,虽然以9个骨干亲本为亲本选育而成的参试品种数占比下降至12.50%,但是仍然有56.25%的参试育成品种以9个骨干亲本为祖先种(图6和图7)。
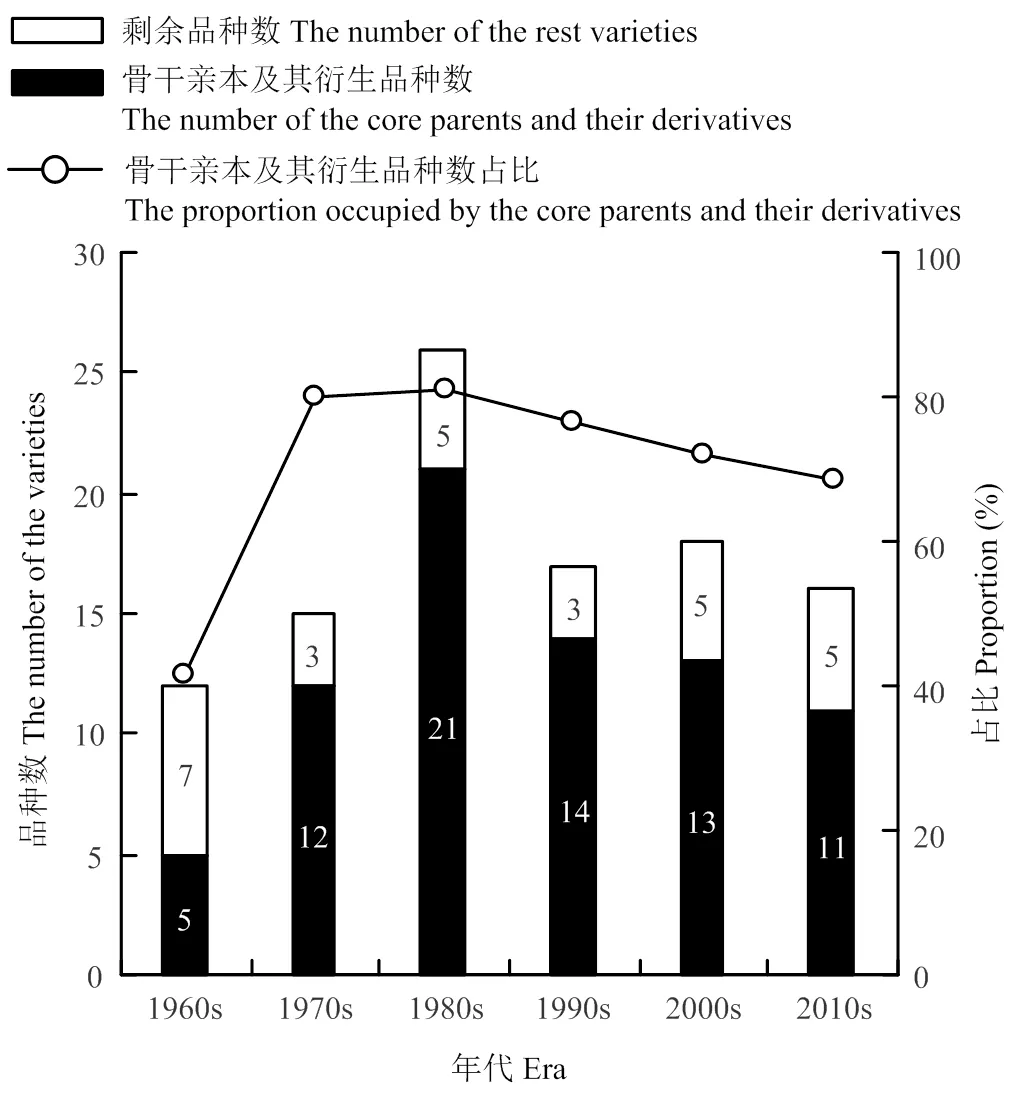
图6 骨干亲本及其衍生品种数占各时期参试品种总量的比例
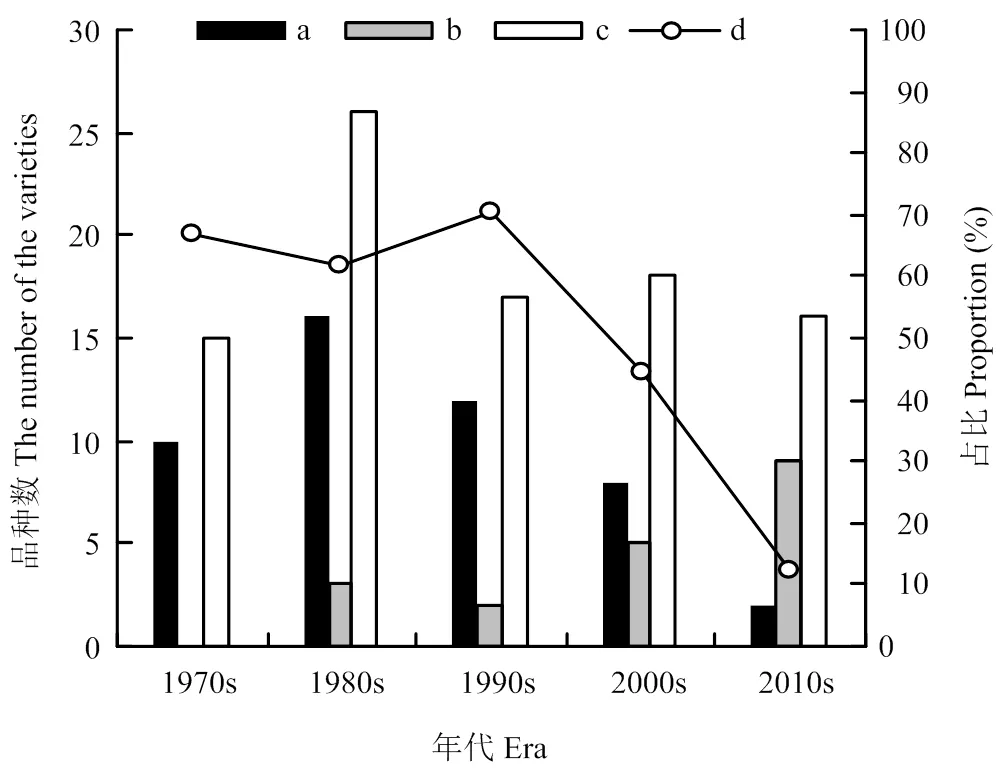
a:以9个骨干亲本为亲本选育而成的参试育成品种数;b:以9个骨干亲本为祖先种选育而成的参试育成品种数;c:各时期参试育成品种数;d:各时期以9个骨干亲本为亲本选育而成的品种数占参试育成品种总量的比例
2.3.2 骨干亲本及其衍生种的地域分布 通过分析骨干亲本及其衍生种的地理来源,发现76个品种存在明显的地域划分,不同地域高频使用的骨干亲本也各不相同。西藏自治区育成的骨干亲本为藏青336、白玉紫芒、果洛和南繁2号,主要引进骨干亲本为昆仑1号和藏青稞;6个骨干亲本及其衍生品种数占西藏自治区参试育成品种数的79.17%。青海省育成的骨干亲本为昆仑1号和藏青稞,引进的骨干亲本为肚里黄,3个骨干亲本及其衍生品种数占青海省参试育成品种数的62.96%。四川省育成的骨干亲本为813和康青3号,引进的骨干亲本为昆仑1号和肚里黄,4个骨干亲本及其衍生品种数占四川省参试育成品种数的70.59%。甘肃省育成的骨干亲本为肚里黄,引进的骨干亲本为康青3号,2个骨干亲本及其衍生品种数占甘肃省参试育成品种数的75.00%(图8)。
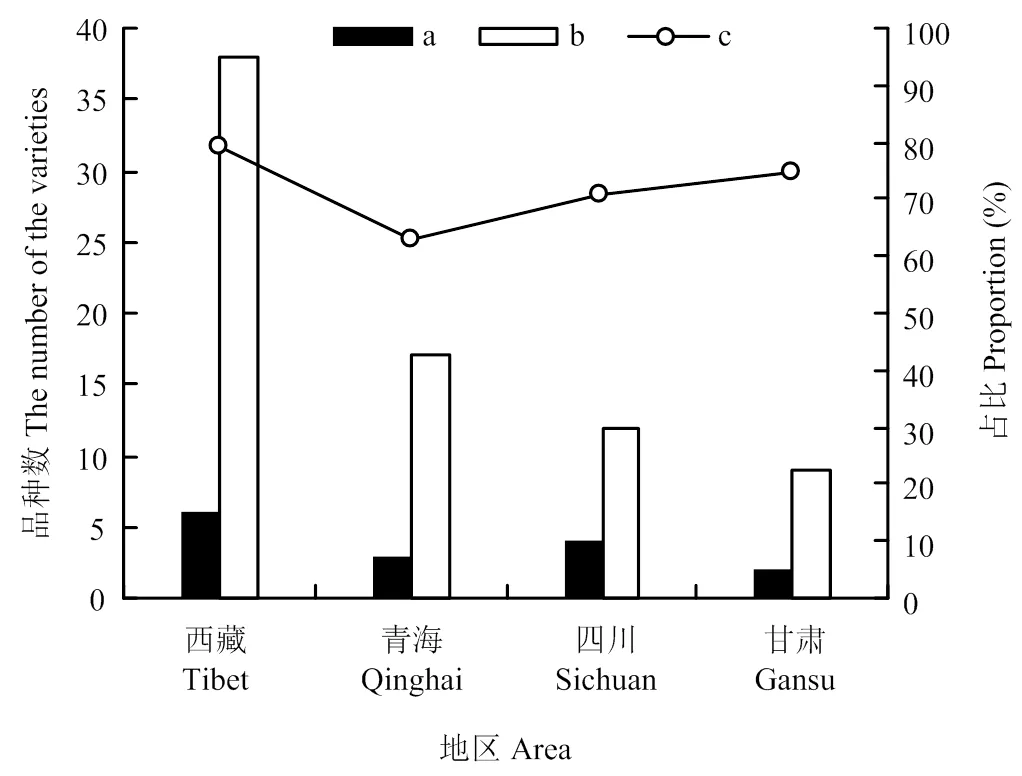
a:常用骨干亲本数;b:骨干亲本及其衍生品种数;c:骨干亲本及其衍生品种数占各地区参试育成品种总量的比例
2.4 参试育成品种选育方法及选育周期分析
通过对参试育成品种的选育方法进行分析,结果表明,79.05%的参试育成品种是通过杂交育种技术选育而成,19.05%的参试育成品种是通过系统选育而成,仅有1.90%的参试育成品种是结合杂交育种技术和辐射诱变技术选育而成。利用系统选育技术选育而成的品种主要分布在1950s—1970s,占比达到80.00%。利用杂交育种技术选育而成的品种分布在1970s以后;结合辐射诱变技术和杂交育种技术选育而成的品种分布在1980s和1990s(图9)。
52个参试品种包含选育周期信息。选育周期在5—10年的品种占比为25.00%,共13个品种,其中,76.92%的品种审定时间集中在1980s及1980s以前。选育周期在10—15年的品种占比为51.92%,共27个品种,其中,96.30%的品种审定时间集中在1980s及1980s以后。选育周期在15—20年的品种占比为23.07%,共12个品种,其审定时间全部集中在最近20年。
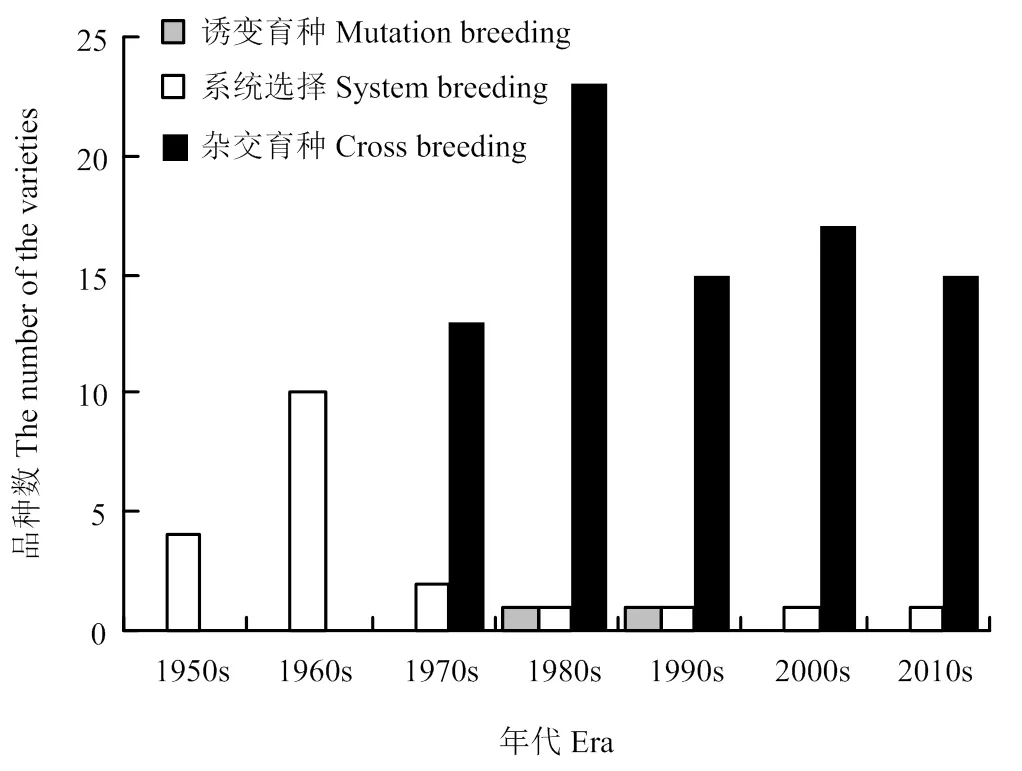
图9 参试育成品种选育方法分析
3 讨论
3.1 藏区青稞育成品种的遗传多样性分析
2018年,曾兴权等[18]对177份大麦种质资源进行重测序分析,其中包含69个藏区青稞地方品种和35个藏区青稞育成品种;结果表明,大麦野生种的遗传多样性最高,东、西方大麦种质资源的遗传多样性较高,而青稞地方品种和育成品种的遗传多样性最低,遗传基础相对狭窄。
2016年,郭焕强等[19]对中国155个大麦主栽品种进行亲缘系数分析,结果表明,155个大麦品种的组合平均亲缘系数为0.013。2008年Mikel等[20]对1970—2006年间育成的北美大麦品种进行亲缘系数分析,结果显示,363个大麦品种的组合平均亲缘系数为0.023,其中69个啤大麦品种的组合平均亲缘系数为0.111,274个饲用大麦品种的组合平均亲缘系数为0.020。与本研究结果相比,108个藏区青稞品种的组合平均亲缘系数为0.053,显著高于中国大麦主栽品种以及北美大麦品种的组合平均亲缘系数。因此,青藏高原青稞育成品种的遗传多样性低,遗传基础相对狭窄、脆弱。
3.2 藏区青稞育成品种遗传基础狭窄的成因及对策
3.2.1 骨干亲本在藏区青稞育种中的重要作用 在麦类作物育种工作中,骨干亲本都起到了不可替代的重要作用。阿夫、阿勃、欧柔、南大2419、矮孟牛、小偃6号等16个小麦骨干亲本,对中国小麦品种的选育做出了突出贡献,阿勃衍生了217个品种,阿夫衍生了165个品种[21]。1950年后,藏区选育的9个骨干亲本,在各个时期、各个地区的青稞选育工作中,都起到了不可替代的重要作用。
1970s,以矮化育种为理论指导,以萧山立夏黄、沧州裸大麦、喜马拉1号为矮源与藏青稞、白玉紫芒等当地骨干亲本组配,培育了以昆仑1号为代表的突破性青稞品种,大幅提高了青稞单产[22]。同时,又以昆仑1号为亲本与当地骨干亲本组配,培育衍生品种4个,骨干亲本及其衍生种数量占1970s参试品种数的80%。1980s,仅以昆仑1号为骨干亲本选育而成的衍生种数量就达到8个,占同时期参试品种总量的30.77%;9个骨干亲本及其67个衍生种占同时期参试品种总量的80.77%。21世纪初,72.22%的参试育成品种仍然由9个骨干亲本及其衍生种构成,44.44%的参试育成品种仍然以9个骨干亲本为亲本。
藏区各地参试育成品种的谱系信息表明,西藏自治区79.17%的参试育成品种由6个骨干亲本(藏青336、藏青稞、白玉紫芒、昆仑1号、果洛和南繁2号)及其衍生种构成;青海省62.96%的参试育成品种由3个骨干亲本(昆仑1号、肚里黄和藏青稞)及其衍生种构成;四川省70.59%的参试育成品种由4个骨干亲本(813、肚里黄、昆仑1号和康青3号)及其衍生种构成;甘肃省75%的参试育成品种由2个骨干亲本(肚里黄和康青3号)及其衍生种构成。
由于9个本地骨干亲本在藏区青稞育种工作中的重要作用,使得藏区在青稞播种面积有所下降的情况下,青稞总产量到达解放初期的3倍以上,青稞平均单产到达解放初期的4倍左右。
但是,由于骨干亲本的高频、长期使用,客观上造成了藏区青稞育成品种的遗传多样性大幅下降,遗传基础趋于狭窄。1970s、1980s和21世纪初的3个时期,参试品种的组合平均亲缘系数到达0.115、0.107和0.079,是藏区青稞育成品种遗传多样性最低的3个时期。但是,1990s和最近十年,参试育成品种的组合平均亲缘系数分别为0.040和0.032,育成品种的遗传基础得以拓宽(图1)。通过对2个时期参试品种的构成进行分析,结果表明,育种目标的调整是2个时期参试品种遗传多样性提高的主要原因。1974年,藏区由单一的春青稞育种思路发展为冬青稞、春青稞并重的育种思路。1990s,优良冬青稞品种大量涌现,该时期38.88%的参试品种为冬青稞品种,使得参试品种的组合平均亲缘系数显著下降,藏区青稞育成品种的遗传基础得以拓宽。基于青稞育种的经验总结,矮秆、早熟很难实现高产;其次,21世纪初,藏区的农业基础设施、耕作水平大幅提升,防灾抗灾能力明显增强。因此,通过提高生物产量,延长灌浆时间以提高青稞单产的育种理论得以普及。最近十年,以藏青2000为代表的“中高秆”、“中晚熟”品种开始出现。由于育种理论的进一步发展,导致最近十年参试育成品种的组合平均亲缘系数大幅下降,藏区青稞育成品种的遗传基础得以再次拓宽。因此,加强青稞基础研究,适时调整育种目标,有利于青稞育成品种遗传基础的拓宽和突破性青稞品种的选育。
3.2.2 由于生态条件和最终用途的显著差异,外引大麦种质的利用率低 DAI等[23]利用DarT标记技术,对不同来源的大麦种质进行遗传多样性分析,结果表明,西藏大麦栽培种与西藏野生大麦的遗传相似性高于地中海地区的野生大麦和栽培大麦。ZENG等[24]通过对青稞地方品种“拉萨勾芒”的基因组进行测序并与Morex的基因组数据进行比对分析,结果显示,“拉萨勾芒”所包含的104个基因家系存在显著扩张,这些基因家系与大麦的抗逆性、抗寒性相关;信号通路分析显示这些基因存在于环境应答或环境适应信号通路之中。由于青藏高原特殊的自然环境,推动了本地青稞种质的适应性进化,而外引大麦品种则表现出较差的环境适应性,很难直接推广使用。1960s以后,没有一个外引大麦品种,在藏区进行大面积推广使用[25]。
由于籽粒最终用途的显著差异,导致外引大麦种质作为供体亲本而加以利用的频率很低。在北美、欧洲、澳洲以及国内其他地区,大麦主要作为酿酒原料和饲料[26],由于麸皮对大麦籽粒、麦芽具有一定的保护作用且裸粒性状为隐性性状,因而皮大麦种质的数量远高于裸大麦种质[27-29]。根据中国作物种质信息网的查询结果,中国国家种质库中保存的6 452份大麦种质中,皮大麦种质的数量占比高达91.00%。在青藏高原,青稞籽粒的最终用途是食品原料,因而,裸粒性状是刚性育种目标。裸大麦与皮大麦组配,将有大量不符合育种目标的有麸后代产生,降低了选育效率;增加了皮大麦种质在藏区青稞育种工作中的利用难度。即便引入裸大麦种质,在藏区特殊的生态条件下,多棱型品种的产量表现普遍优于二棱型品种。因此,育种人员更倾向于使用多棱型大麦种质与本地青稞种质组配;否则,育种效率会显著下降。105个藏区育成品种所使用的亲本种质为118个,分析各亲本种质的地理来源信息,结果表明,82.20%的亲本属于藏区本地青稞种质,5.09%的种质原产地信息不详,从国内外其他地区引进的种质仅占12.71%。引进的大麦种质中,多棱裸大麦种质、二棱皮大麦种质、二棱裸大麦种质的数量占比分别为6.67%、13.33%和33.34%。
因此,由于适应性和最终用途的显著差异,外引大麦种质在藏区青稞育种工作中的使用频率很低,是导致藏区青稞育成品种遗传多样性较低的重要原因。
Chen等[30]通过筛选120个代表性小麦品种并对其进行全基因组重测序分析,结果表明,现代中国小麦育成品种与欧洲小麦地方种质的遗传相似性甚至高于与中国小麦地方种质的遗传相似性;由此可见,高效利用外引种质,对于现代麦类作物育种的重要作用。随着育种技术的不断发展,利用分子标记辅助育种技术,“speed breeding”快速加代技术等先进育种手段[31],提高外引优异突变与藏区本地种质的融合速度,充分发挥外引种质在遗传改良过程中的作用,是拓宽藏区青稞育成品种遗传基础,推动藏区青稞新品种选育的重要途径。
4 结论
藏区青稞的9个骨干亲本及其67个衍生品种构成了育成品种的主体,外引大麦种质利用率低,从而导致青藏高原青稞育成品种的遗传多样性较低,遗传基础相对狭窄、脆弱。
[1] 赵贯锋, 余成群, 钟志明, 武俊喜, 李少伟, 孙维, 苗彦军. 西藏食物安全战略初探. 西藏科技, 2016(5): 17-21.
ZHAO G F, YU C Q, ZHONG Z M, WU J X, LI S W, SUN W, MIAO Y J. The exploratory research of Tibetan food security strategy., 2016(5): 17-21. (in Chinese)
[2] Martin J M, Blake T K, Hockett E A. Diversity among north American spring barley cultivars based on coefficients of parentage., 1991, 31(5): 1131-1137.
[3] Ren x f, Nevo e, Sun d f, Sun g l. Tibet as a potential domestication center of cultivated barley of China., 2013, 8(5): e62700.
[4] 吴昆仑. 青稞种质资源的SSR标记遗传多样性分析. 麦类作物学报, 2011, 31(6): 1030-1034.
WU K L. Genetic diversity analysis of hulless barley germplasm by SSR markers., 2011, 31(6): 1030-1034. (in Chinese)
[5] 曾兴权, 王玉林, 徐齐君, 原红军, 扎西罗布, 尼玛扎西. 利用SSR引物分析西藏青稞种质资源的遗传多样性. 麦类作物学报, 2013, 33(2): 260-267.
ZENG X Q, WANG Y L, XU Q J, YUAN H J, ZAXi L B, NIMa z X. Assessment of Genetic diversity in tibetan hulless barley germplasm (L. var. nudum HK. F.) by SSR Primers., 2013, 33(2): 260-267. (in Chinese)
[6] 巴桑玉珍, 刘新春, 付国勇, 李东梅, 王丹丹, 强小林, 冯宗云. 青藏高原青稞耐寒种质资源基于SSR标记的遗传多样性及群体结构分析. 麦类作物学报, 2017, 37(1): 40-47.
BASANG Y z, LIU X C, FU G Y, LI D M, WANG D D, QIANG X L, FENG Z Y. Genetic Diversity and population structure analysis of hulless barley with cold tolerance from the Qinghai-Tibetan plateau revealed by SSR markers., 2017, 37(1): 40-47. (in Chinese)
[7] 杨菁, 迟德钊, 吴昆仑, 何桂芳. 青海省栽培青稞SSR标记遗传多样性研究. 安徽农业科学, 2010, 12(8): 4307-4309.
YANG J, CHI D Z, WU K L, HE G F. Genetic diversity of SSR markers in cultivatedL. in Qinghai province., 2010, 38(8): 4307-4309. (in Chinese)
[8] 孟亚雄, 孟祎林, 汪军成, 司二静, 张海娟, 任盼荣, 马小乐, 李葆春, 杨轲, 王化俊. 青稞遗传多样性及其农艺性状与SSR标记的关联分析. 作物学报, 2016, 42(2): 180-189.
MENG Y X, MENG Y L, WANG J C, SI E J, ZHANG H J, REN P R, MA X L, LI B C, YANG K, WANG H J. Genetic diversity and association analysis of agronomic characteristics with SSR markers in hulless barley., 2016, 42(2): 180-189. (in Chinese)
[9] 孟凡磊, 强小林, 佘奎军, 唐亚伟, 胡银岗. 西藏主要农区青稞品种的遗传多样性分析. 作物学报, 2007, 33(11):1910-1914.
MENG F L, QIANG X L, SHE K J, TANG Y W, HU Y G. Genetic diversity analysis among hulless barley varieties from the major agricultural areas of Tibet., 2007, 33(11): 1910-1914. (in Chinese)
[10] 杨平, 刘仙俊, 刘新春, 李俊, 王希文, 何守朴, 李刚, 杨武云, 冯宗云. 利用SRAP标记研究四川高原青稞育成品种的遗传多样性. 遗传, 2008, 30(1): 115-122.
YANG P, LIU X J, LIU X C, LI J, WANG X W, HE S P,LI G, YANG W Y, FENG Z Y. Genetic diversity analysis of the developed qingke (hulless barley) varieties from the plateau regions of Sichuan Province in China revealed by SRAP markers., 2008, 30(1): 115-122. (in Chinese)
[11] Almanza-Pinzón M I, Khairallah M, Fox P N, WarburtonM L. Comparison of molecular markers and coefficients of parentage for the analysis of genetic diversity among spring bread wheat accessions., 2003, 130(1): 77-86.
[12] Casas A M, Igartua E, Valles M P, Molina-Cano J L. Genetic diversity of barley cultivars grown in Spain, estimated by RFLP, similarity and coancestry coefficients., 2010, 117(5): 429-435.
[13] Cui Z L, Carter T E, Burton J W. Genetic base of 651 Chinese soybean cultivars released during 1923 to 1995., 2000, 40(5): 1470-1481.
[14] Khera P, Singh A K, Priyadarshi R, Khandekar D, ALLU R K, HIREMATH C, KUMAR R, MOHAN R, ULAGANATHAN K, Shenoy V. Genetic variability in trait-specific rice germplasm groups based on coefficient of parentage, SSR markers and fertility restoration., 2015, 13(1): 56-67.
[15] You F M, Duguid S D, Lam I, Cloutier S, Rashid K Y, Booker H M. Pedigrees and genetic base of flax cultivars registered in Canada., 2016, 96(5): 837-852.
[16] 王江春, 胡延吉, 余松烈, 王振林, 刘爱峰, 王洪刚. 建国以来山东省小麦品种及其亲本的亲缘系数分析. 中国农业科学, 2006, 39(4):664-672.
WANG J C, HU Y J, YU S L, WANG Z L, LIU A F, WANG H G. Relationship coefficient analysis among winter wheat varieties and their parents in shandong province after liberation., 2006, 39(4): 664-672. (in Chinese)
[17] 刘章雄, 石孙, 李卫东, 陈立军, 常汝镇, 邱丽娟. 1983—2010年北京市大豆育成品种的亲缘关系分析. 作物学报, 2013, 39(9): 1693-1700.
LIU Z X, Shi S, LI W D, CHEN L J, CHANG R Z, QIU L J. Analysis of parental relationship for soybean cultivars released from 1983 to 2010 in Beijing., 2013, 39(9): 1693-1700. (in Chinese)
[18] Zeng X, Guo Y, Xu Q, Mascher M, Guo G, Li S, Mao L, Liu Q, Xia Z, Zhou J, Yuan H, Tai S, Wang Y, Wei Z, Song L, Zha S, Li S, Tang Y, Bai L, Zhuang Z, He W, Zhao S, Fang X, Gao Q, Yin Y, Wang J, Yang H, Zhang J, Henry R J, Stein N, Tashi N. Origin and evolution of qingke barley in Tibet., 2018, 9(1): 5433.
[19] 郭焕强, 王文峰, 姚全杰, 孙丹, 姚小波, 王翠玲, 田世富, 包海柱, 王凤涛, 冯晶,蔺瑞明,徐世昌. 大麦主栽品种亲缘系数和对叶斑病的抗性分析. 植物遗传资源学报, 2016, 17(4): 586-598.
GUO H Q, WANG W F, YAO Q J, SUN D, YAO X B, WANG C L, TIAN S F, BAO Hai Z, WANG F T, FENG J, LIN R M, XU S C. Analysis on coefficient of parentage of major barley varieties and their spot blotch resistance., 2016, 17(4): 586-598. (in Chinese)
[20] MIKEL M A, KOLB F L. Genetic diversity of contemporary North American barley., 2008, 48(4): 1399-1407.
[21] 盖红梅, 王兰芬, 游光霞, 郝晨阳, 董玉琛, 张学勇. 基于SSR标记的小麦骨干亲本育种重要性研究. 中国农业科学, 2009, 42(5): 1503-1511.
GE H M, WANG L F, YOU G X, HAO C Y, DONG Y C, ZHANG X Y. Fundamental roles of cornerstone breeding lines in wheat reflected by SSR random scanning., 2009, 42(5): 1503-1511. (in Chinese)
[22] 张京. 中国大麦育种主要矮源的遗传等位测验. 作物学报, 1998, 24(1): 42-46.
ZHANG J. AIlelism tests for the dwarf genes in the three main dwarf sources of barley., 1998, 24(1): 42-46. (in Chinese)
[23] DAI F, NEVO E, WU D, COMADRAN J, ZHOU M, QIU L, CHEN Z, BEILES A, CHEN G, ZHANG G. Tibet is one of the centers of domestication of cultivated barley., 2012, 109(42): 16969-16973.
[24] ZENG X, LONG H, WANG Z, ZHAO S, TANG Y, HUANG Z, WANG Y, XU Q, MAO L, DENG G, YAO X, LI X, BAI L, YUAN H, PAN Z, LIU R, CHEN X, WANGMU Q, CHEN M, YU L, LIANG J, DUNZHU D, ZHENG Y, YU S, LUOBU Z, GUANG X, LI J, DENG C, HU W, CHEN C, TABA X, GAO L, LV X, ABU Y B, FANG X, NEVO E, YU M, WANG J, TASHI N. The draft genome of Tibetan hulless barley reveals adaptive patterns to the high stressful Tibetan Plateau., 2015, 112(4): 1095-1100.
[25] 巴桑玉珍, 强小林. 西藏青稞育种的成就与经验分析. 西藏农业科技, 2004, 26(1): 26-36.
BASHANG Y Z, Qiang x l. Tibet the highland barley teachs the achievement and the experience analysis of the kind., 2004, 26(1): 26-36. (in Chinese)
[26] Newman W C, Newman R K. A brief history of barley foods., 2006, 51(1): 4-7.
[27] MILNER S G, JOST M, TAKETA S, MAZON E R, HIMMELBACH A, OPPERMANN M, WEISE S, KNUPFFER H, BASTERRECHEA M, KONIG P, SCHULER D, SHARMA R, PASAM R K, RUTTEN T, GUO G, XU D, ZHANG J, HERREN G, MULLER T, KRATTINGER S G, KELLER B, JIANG Y, GONZALEZ M Y, ZHAO Y, HABEKUß A, FARBER S, ORDON F, LANGE M, BORNER A, GRANER A, REIF J C, SCHOLZ U, MASCHER M, STEIN N. Genebank genomics highlights the diversity of a global barley collection., 2019, 51: 319-326.
[28] Schmidt M, Kollers S, Maasberg-Prelle A, Großer J, Schinkel B, Tomerius A, Graner A, Korzun V. Prediction of malting quality traits in barley based on genome-wide marker data to assess the potential of genomic selection., 2016, 129(2): 203-213.
[29] TAKETA S, AMANO S, TSUJINO Y, SATO T, SAISHO D, KAKEDA K, NOMURA M, SUZUKI T, MATSUMOTO T, SATO K, KANAMORI H, KAWASAKI S, TAKEDA K. Barley grain with adhering hulls is controlled by an ERF family transcription factor gene regulating a lipid biosynthesis pathway., 2008, 105(10): 4062-4067.
[30] Chen H, Jiao C, Wang Y, Wang Y, Tian C, Yu H, Wang J, Wang X, Lu F, Fu X, Xue Y, Jiang W, Ling H, Lu H, Jiao Y. Comparative population genomics of bread wheat () reveals its cultivation and breeding history in China., 2019: 519587.
[31] WATSON A, GHOSH S, WILLIAMS M J, CUDDY W S, SIMMONDS J, REY M D, HATTA M A M, HINCHLIFFE A, STEED A, REYNOLDS D, ADAMSKI N M, BREAKSPEAR A, KOROLEV A, RAYNER T, DIXON L E, RIAZ A, MARTIN W, RYAN M, EDWARDS D, BATLEY J, RAMAN H, CARTER J, ROGERS C, DOMONEY C, MOORE G, HARWOOD W, NICHOLSON P, DIETERS M J, DELACY I H, ZHOU J, UAUY C, BODEN S A, PARK R F, WULFF B B H, HICKEY L T. Speed breeding is a powerful tool to accelerate crop research and breeding., 2017, 4(1): 23-29.
Coefficient of Parentage Analysis among Naked Barley Varieties in Qinghai-Tibet Plateau
LI Jian1,2, FENG Xianhong2, CAI Yilin1
(1College of Agronomy and Biotechnology, Southwest University, Chongqing 400715;2Ganzi Tibetan Autonomous Prefecture Institute of Agricultural Science, Kangding 626000, Sichuan)
【】The present analysis of genetic diversity of bred naked barley varieties of the Qinghai–Tibet Plateau was conducted to evaluate their genetic diversity, to explore measures to broaden the genetic basis, and to promote the breeding of naked barley varieties. 【】In total, 105 bred naked barley varieties grown commonly and broadly across Qinghai-Tibet plateau, which has been surveyed since 1950, and 3 introduced varieties were selected as study materials. The genetic diversity of the investigated varieties was estimated by coefficient of parentage (COP), and then based on these COP values, a clustering analysis was conducted using Lance and Williams distance and UPGMA. Ultimately, the core parents in Qinghai-Tibet plateau were determined by examining the cumulative direct coefficient of parentage (CD-COP). 【】Overall, 18.01% of variety combinations were genetically related. The COP of all combinations ranged from 0.000 to 0.750, with an average of 0.053, which was significantly higher than that of the main barley varieties of China and North American. Based on the released time, the investigated varieties were divided into seven decade-long periods since 1950. The average COP value of the combinations that was made by the investigated varieties bred in the 1970s and 1980s were higher, reaching 0.115 and 0.107, respectively, while this index was lower at the 1990s and the recent decade, reaching 0.040 and 0.032, respectively. The numbers of investigated varieties from the Tibet Autonomous Region, Qinghai Province, Sichuan Province, and Gansu Province were 48, 27, 17, and 12, respectively. The average COP of the combinations made by the varieties from Gansu Province was highest, reaching 0.220, while the average COP of the combinations made by the bred varieties from Tibet Autonomous Region, Qinghai Province, and Sichuan Province were similar, reaching 0.094, 0.122, and 0.138, respectively. Cluster analysis placed the investigated varieties into seven groups, of which 25 were in groups II and III, with a relatively low average COP of combinations within each group, while 83 were in the other five groups, which had higher average COP of combinations. The cumulative direct COP of 40 investigated varieties were more than 0; of these, 9 bred varieties were identified as important core parents in Qinghai-Tibet plateau, from which 67 varieties had been derived. 9 core parents and their 67 derivatives account for 72.38% of the number of investigated bred varieties. In the 1960s, the number of core parents and their derivatives accounted for 41.76% of the number of investigated varieties. For the next four decades, the proportions of core parents and their derivatives out of all investigated bred varieties in each period reached 80.00%, 80.77%, 76.47%, and 72.22%, respectively. In the last decade, the index has declined further, but still reaches 68.75%. In the Tibet Autonomous Region, Qinghai Province, Sichuan Province, and Gansu Province, the proportion of core parents and their derivatives out of the number of the investigated bred varieties reached 79.17%, 62.96%, 70.59%, and 75.00%, respectively. The total number of parents used in breeding was 118, including 97 local parents (82.20%), 15 introduced parents (12.71%), and 6 parents lacking source information. 【】9 core parents and their 67 derivatives constitute the majority of naked barley varieties. The utilization of introduced barley germplasm has been inefficient. Consequently, the genetic diversity of naked barley varieties cultivated across the Qinghai–Tibet Plateau is relatively lower.
Qinghai-Tibet Plateau; naked barley; bred varieties; coefficient of parentage; genetic diversity
2019-04-03;
2019-06-08
国家现代农业产业技术体系(CARS-05-B)、甘孜州应用技术研究与开发(2013)111
李健,E-mail:lijian5028@163.com。
蔡一林,E-mail:caiyilin1789@163.com
(责任编辑 李莉)
