植物质膜蛋白质组学研究进展
2019-08-20陈亚琳谢成建杨星勇
陈亚琳 谢成建 杨星勇

摘要:植物质膜(plasma membrane,简称PM)是连接细胞内外的活性屏障,而PM蛋白作为PM的重要组成成分之一,对植物细胞膜功能有着重要的作用。植物PM蛋白质组学的研究有利于对PM的功能进行更加深入的探索。本文主要对近几年来在植物PM蛋白质组学的相关研究进行综合性论述,包括其研究方法、功能及其面临的问题等,以期为植物PM蛋白质组学的后续研究以及PM功能的深入探讨提供参考。
关键词:PM蛋白;蛋白质组;PM蛋白功能;微结构域
中图分类号: Q942.6文献标志码: A
文章编号:1002-1302(2019)08-0007-05
蛋白质组学以直接参与生命活动的所有蛋白质作为研究目标,其主要研究技术包括双向电泳技术(two-dimensional electrophoresis,简称2-DE)、质谱鉴定技术、蛋白质组生物信息学、核磁共振、噬菌体展示技术、蛋白芯片、酵母双杂交系统等。植物质膜(plasma membrane,简称PM)蛋白作为植物蛋白质的一个重要部分,蛋白组学技术为植物PM蛋白研究提供了重要的技术支撑。目前,通过PM蛋白的微结构域和蛋白质组学分析来研究PM蛋白的功能已成为常用的研究手段之一,也包括用拉曼射线对膜结构域进行分析[1]以及研究大豆(Glycine max)中的[WTBX][STBX]GmFWL1基因编码的膜微结构域的相关蛋白质等[2]。
为了系统性地了解植物PM蛋白的研究进展,本研究针对植物PM蛋白组学相关的问题与挑战开展讨论,归纳整理近几年在植物PM蛋白质组学方面取得的研究进展。
1植物质膜功能
植物PM作为植物细胞的活性屏障,是植物细胞与外部环境之间沟通的桥梁,主要负责调节细胞内外的物质运输、信号转导等等[3]。植物中多种代谢活动的协调依赖于通过膜接触点(membrane contact sites,简称MCS)建立的组织间通讯网络[4],植物特有的MCS在植物组织发育、胞间运输和应激反应中发挥的重要作用尚有待深入研究。如内质网(endoplasmic reticulum,简称ER)与PM的接触位点(endoplasmic reticulum-plasma membrane contact sites,简称EPCs)连接的蛋白质复合物及其结构有待阐释[5]。此外,有一种蛋白质亚复合物ESCRT-Ⅲ能够介导PM的破碎和修复过程[6];溶酶体与PM融合也能够帮助机械损伤的PM重新修复[7],这些有赖于植物膜蛋白研究的新发现对于植物PM功能研究具有重要作用。
作为植物PM重要组成成分的植物PM蛋白通过嵌入脂双层与脂质一起构成植物PM的分子结构,并在植物物质运输、离子交换、代谢等过程中起着重要作用。如在拟南芥(Arabidopsis thaliana)中有一种[WTBX][STBX]VPS36介导的细胞PM蛋白质能够对其向地性产生作用[8],另有一种PM局部转运蛋白NIP1介导拟南芥中铝吸收、转运,以及铝耐受性[9];此外,复合物AP2M参与定位PM的病害抗性蛋白介导的效应物触发的免疫(effector-triggered immunity,简称ETI),是通过PM上的免疫受体复合物介导的内吞作用完成的[10]。近年,研究者利用不同的方法力求将PM蛋白的成分、结构和功能了解清楚,包括通过对PM蛋白质谱的分析探究其表达特征和功能,以及植物某种PM蛋白对整个PM蛋白组成和植物生理生化功能产生的影响[11],其中,如何使质谱检测结果达到最优化,也是PM蛋白研究的一个重点。
关于PM的结构,在流动镶嵌模型[12]的基础上有了更深入的阐释,即蛋白质能够瞬时被捕获在脂筏和肌动蛋白-细胞骨架相关结构中的区域化分层中[13]。而PM蛋白可根据其在PM中的分布位置分为外周PM蛋白、内在PM蛋白和膜锚定蛋白。PM蛋白不能在脂双层翻转,但它可以在脂双层中侧向扩散并持续扩散。其中,外在PM蛋白是指蛋白质通过非共价离子与磷脂分子的极性头部结合或者通过其与内在蛋白的相互作用而形成,主要存在于膜的外表面或内表面[14]。内在PM蛋白因其部分蛋白穿过或嵌入脂双层,因此也被叫做跨PM蛋白,它是利用非极性氨基酸与磷脂分子的疏水区相互作用从而固定于膜上,由于其疏水结构域之间的疏水作用,使得它与脂双层的结合非常紧密。单个跨膜螺旋的蛋白质代表了PM蛋白中功能最丰富和多样的一种PM蛋白,如参与细胞通信、信号转导、转运和代谢调节、免疫反应等多种生物功能[10,15-16]。脂锚定蛋白是指共价结合的脂质锚定一些外周PM蛋白,使之既能插入脂双层又能在膜表面固定蛋白质,可分为2类:糖磷脂酰肌醇(glycosyl phosphatidy linositol,简称GPI)连接的蛋白,以及与插入PM内小叶的长碳氢链结合的脂锚定蛋白;这种脂质和蛋白结合形成的特定PM蛋白复合物在PM蛋白的运动中可能会起到限定性作用。此外,可以通过分析在轴突初始段(axon initial segment,简称AIS)的PM蛋白的运动的纳米分区而了解PM蛋白的运动规律[17]。
PM蛋白功能是多种多样的。(1)作为受体,如参与许多信号转导途径的跨膜螺旋蛋白-G蛋白偶联受体,它在视力、脑功能和运动中都发挥着重要作用,且有几种与此受体相关的G蛋白的功能具有胆固醇依赖性[18-21]。(2)介导各种信号转导途径,在植物PM内侧有一种异基三聚体G蛋白,可以在各种内膜位置发出信号,调节植物代谢活动[22]。此外,植物PM蛋白冷应答蛋白激1(cold-responsive protein kinase 1,簡称CRPK1)通过磷酸化一种14-3-3蛋白,将冷信号从PM转移到细胞核以调节植物应对冷胁迫的应激反应[23]。(3)作为物质进出细胞的载体,成为细胞与外界交换的桥梁。如ERM蛋白家族是连接PM与细胞骨架之间的重要调节因子,在细胞形态调节、膜流动性和细胞迁移等许多细胞过程中起作用[24-28]。此外,表面抗原与特异抗体结合的免疫沉淀技术的发展促进了与PM蛋白相互作用的蛋白质的深入研究和相关蛋白的鉴定[29],这有利于推动PM蛋白在生物代谢,特别是植物代谢过程中作用研究。
2植物细胞PM蛋白的研究方法
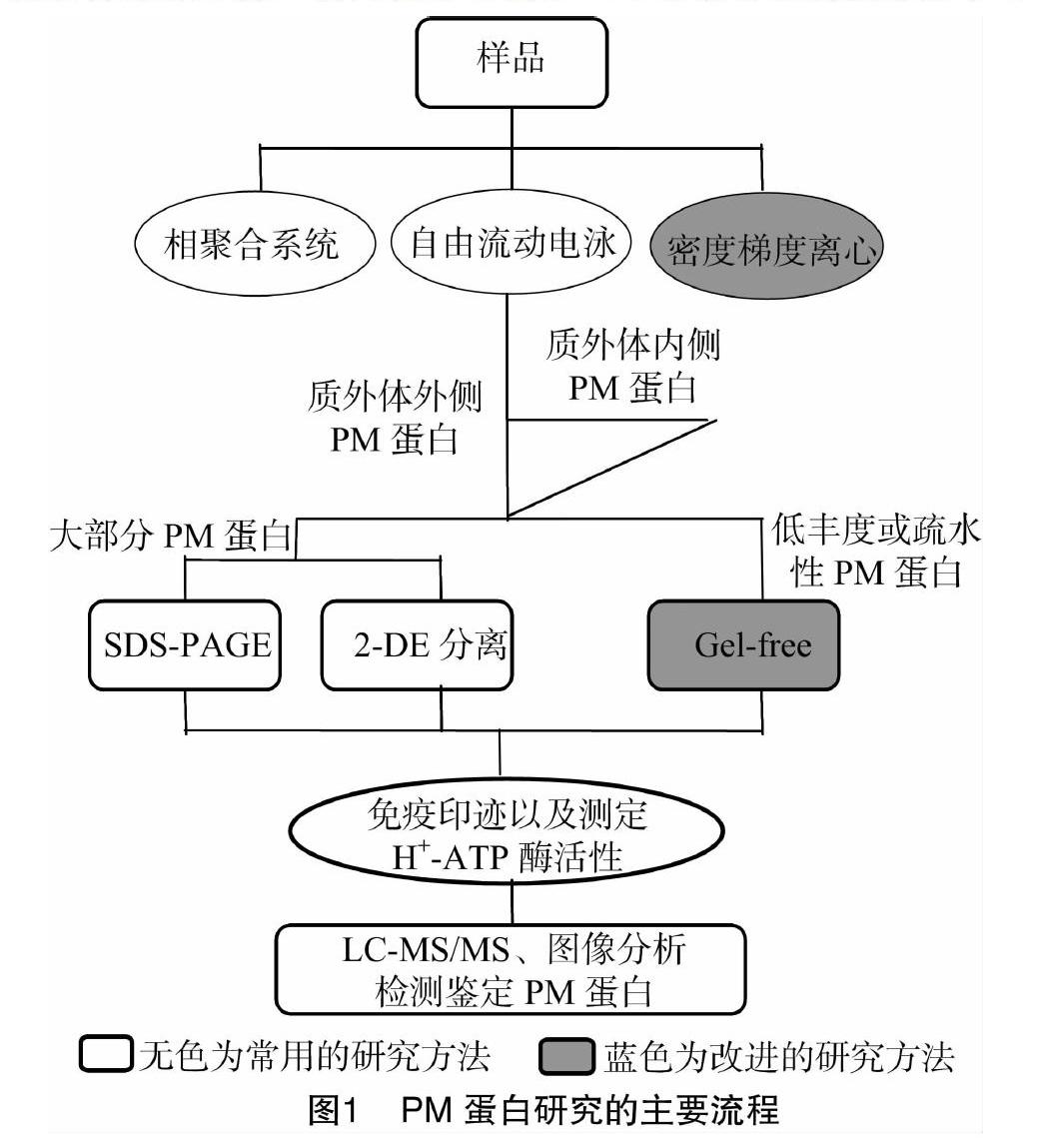
PM蛋白分离和提取的技术方法主要包括:密度梯度离心、自由流动电泳和相聚合系统[30]。在这些方法中,两相分配是用于提取高纯度的PM囊泡蛋白最为常用的技术,PM囊泡是一种细胞裂解过程中形成的膜衍生的囊泡[3]。两相分配是相聚合系统中的一种方法,其分离PM蛋白的大致流程见图1。
2.1PM蛋白提取
在兩相分配系统中,生物物质与成相组分通过疏水键、离子键、氢键等相互作用而不同程度的分配在两相中。因此,必须根据不同的材料使用相应的相,常用的相包括21 g相、27 g相等[31-33]。而自由流动电泳通常则是和水两相分配结合使用,以保证提取的PM蛋白质量[34]。利用密度梯度离心富集PM蛋白时,虽避免了前2种方法复杂繁琐的过程,但可能因离心导致膜中的其他杂质影响PM蛋白质量[35]。利用此方法提取PM蛋白时,要注意去除杂质或者与PM蛋白相关的一些非特异性蛋白质。
上述3种方法提取的植物PM蛋白存在差异(图2)。PM囊泡蛋白主要是存在于质外体外侧部分,当需要利用内侧PM蛋白时,使用的方法需要利用一种非离子型去垢剂 Brij-58,它可使PM囊泡发生内向翻转来达到此目的[30]。由于Brij-58提取质体内侧PM蛋白的效果比较明显,且其提取的PM蛋白纯度较高,用Brij-58处理总微粒体以减少细胞器蛋白的污染,可以提高PM蛋白的丰度[36]。Brij-58还能够与丙烯酸或其他类似物质形成微凝胶,从而对蛋白质进行吸附或解吸[37]。
2.2PM蛋白分离
当提取出PM蛋白后,通过双向电泳或是无凝胶技术分离目的蛋白或肽。2-DE是将等电聚焦电泳(isoelectric focusing,简称IEF)以及十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gel electrophoresis,简称SDS-PAGE)二者相结合的高分辨分离蛋白方法[35,38]。但2-DE存在对低丰度蛋白和疏水性蛋白不敏感的问题。鉴于2-DE的局限性,无凝胶分离蛋白技术得到了应用,它是在蛋白质鉴定之前将蛋白质裂解以产生肽,而肽可以通过高效液相色谱法(high performance liquid chromatography,简称HPLC)分离,该方法操作快,覆盖面更广。具体试验研究过程中,在如何对无凝胶技术和2-DE这2种蛋白质分离技术进行选择过程中,仍然存在争议,因为无凝胶技术虽然能弥补 2-DE 在低丰度蛋白上的缺陷,扩大其分离范围,提高分离效率,但2-DE能够提供完整的蛋白质的映射,同步反映了由于蛋白质同种类型和翻译后修饰的变化的移位。因此这2种方法各有优劣,在试验过程中需根据实际情况进行[39]。
当PM蛋白分离后,通过免疫印迹分析以及H+-ATP酶水解活性测定,以确定所分离PM蛋白的纯度,通过不同的标记酶的抑制剂作用于酶,观察释放的磷的含量从而测定ATP酶的活性[40]。
2.3LC-MS/MS
用胰蛋白酶将获取的蛋白质消化后通过液相色谱串联质谱(liquid chromatograph-mass spectrometer/mass spectrometer,LC-MS/MS)分析,然后在相关的蛋白质数据库中检索相关蛋白,这种鉴定方法基本在大部分PM蛋白提取试验中都适用,LC-MS/MS分析是现阶段蛋白质研究的常用方法,其过程方便快捷,操作也比较简单,且利用此方法分析出来的结果准确率高。在提取PM蛋白的整个过程中必须尽可能地使所提取的PM蛋白纯度更高,为了达到这个目的,现在可用的方法是使用相关的去污剂来达到这个效果,它对于溶解和纯化PM蛋白具有很好的效果[41]。
3植物PM蛋白组的功能研究
细胞膜被认为是液相平面,其中脂质和蛋白质理论上可以自由扩散。而细胞壁有可能限制蛋白质的扩散,特别是对于[CM(25]具有较大的细胞外结构域的蛋白质,即使蛋白质和细胞壁组分之间没有结合或者是相互作用[42]。此外,PM蛋白在植物物质运输方面也起到相当重要的作用,通过分析比较相关PM蛋白前后积累量的变化发现,部分PM蛋白在膜运输的过程中起调节作用[38]。PM内在蛋白(plasma membrane intrinsic protein,简称PIP)主要位于PM中,促进水和小的不带电溶质的被动扩散,可以通过研究PIP调节水分运动来了解植物细胞如何调节其膜渗透性[43]。有研究对PIP的合成进行RNA干扰,结果发现PIP不仅在植物水分跨膜运输过程中具有重要作用,而且能够通过改变PM蛋白的组成响应其在膜运输中的作用,即影响植物的蒸腾作用和气孔运动[44]。因此,PM蛋白在帮助植物物质运输方面起着重要作用。
植物PM蛋白质在植物细胞功能诠释中具有非常重要的作用。如通过蛋白组学手段,以模式植物拟南芥为研究对象,并结合PM蛋白质微结构域方面开展相关研究,并取得一系列研究结果[30]。在研究植物PM蛋白功能方面,不同试验情况采用不同的研究手段。
利用经典的SDS-PAGE技术对拟南芥PM蛋白成分研究,发现生长素结合蛋白1(auxin binding protein 1,简称ABP1)并不是拟南芥生长发育所必需的,也不是植物生长发育必需的[45]。植物是一种固着生物,但却生长在一个快速变化的环境中,在其生长和发育过程中经常会遇到各种环境胁迫,包括生物和非生物胁迫[46]。在植物生物胁迫方面,为揭示拟南芥感染灰葡萄孢菌(Botrytis cinerea)后的响应机制,Costa等利用两相分配的方式研究发现,部分PM蛋白能够响应某种病原体感染,并部分具有应激机制[47]。PM蛋白在植物响应和耐受盐胁迫过程中发挥着重要的作用[48],Li等通过密度梯度离心,对响应盐胁迫的甜菜相关PM蛋白进行分析,发现了植物响应盐胁迫以及盐胁迫耐受性的部分相关机制[35]。此外,有研究人员通过分析玉米(Zea mays L.)PM蛋白在正常水环境以及水胁迫环境下的分布,并依据生理、转录组和细胞壁蛋白质组学等的分析阐释了玉米根适应水胁迫的压力进行生长调节的机制[49]。盐胁迫和水胁迫都只是非生物胁迫的一部分,而植物如何适应各种不良环境,PM蛋白又在其中扮演着怎样的角色,在以后的植物学研究中尚有很多亟待解决的问题。
除此之外,隨着PM蛋白技术的不断改进,越来越多的植物PM蛋白功能被不断发现。如通过两相分配结合无凝胶技术提取质膜蛋白,并通过鸟枪蛋白质组学分析[50]揭示了铁元素的缺乏对于甜菜质膜蛋白的重要影响;利用无标记和基于MaxQuant的蛋白质组学方法[40]发现了甘蓝型油菜(Brassica napus)对磷缺乏适应性机制的基因型差异;Preger等通过密度梯度离心与无凝胶技术结合,发现了大豆质膜上的一种b型细胞色素受生长素响应基因[WTBX][STBX]AIR2调控,并控制其开花特异性[51]。随着PM蛋白研究技术的不断发展,植物PM蛋白的功能将会越来越清晰。
4植物PM蛋白质组研究的问题与挑战
作为细胞与外界的连接纽带,PM具有物质运输,信号传导,细胞壁生物合成和分泌的一系列重要功能。而作为构成PM重要成分的蛋白质,它在PM的众多功能中又具体扮演着什么样的角色,这是我们目前要去深入探讨的一个问题。随着研究的深入,研究人员一步一步揭开了关于植物PM蛋白质的神秘面纱,其结构和功能也越来越多地被了解。目前针对植物PM蛋白质组的相关研究,主要集中在对模式植物或经济作物的PM蛋白质结构及其在植物生理功能方面的阐释。
虽然有关PM的研究越来越多,但有很大比例都是关于PM的结构和其微结构域的研究。关于PM蛋白的研究相对偏少,植物PM蛋白的研究更少。PM蛋白研究的一个难点是PM蛋白的表面相对疏水,甚至需要洗涤剂从膜中提取,有时又非常不稳定。这导致包括表达、溶解、纯化以及相关蛋白的鉴定和翻译后修饰的确定就更加困难[29]。关于植物PM蛋白研究存在的困难还比较多,包括在植物中ER-PM接触位点的结构和功能,目前也仅仅只鉴定出一些蛋白复合物[5],PM蛋白参与其中作用的具体种类和作用机制仍未得到解决。近年来,已经发现肌动蛋白、血影蛋白和锚蛋白的轴突周期性模式形成了190 nm的间隔的环状结构;然而,这种结构是否与扩散屏障功能相关还不清楚[17],这对研究PM蛋白在PM中的运动轨迹也造成了阻碍。
虽然在植物PM蛋白质组研究的很多方面仍旧存在很多困难,但在研究人员的不懈努力下,PM蛋白质组的研究仍然稳步前进中,并且取得了一定进展。比如最近引入基于代谢标记(metabolic labeling)和点击化学(click chemistry)的生物化学结合的方法能够通过超分辨率显微镜对不同膜组分进行直观观察[52-54]。这个方法一定程度上克服了PM蛋白研究的一部分困难。除此之外,植物PM蛋白质组在PM功能、在各种代谢中的不同作用以及植物各种环境胁迫应激过程中,不同的作用种类及其结构以及作用机制仍然有很多问题需要解决。尽管植物PM蛋白质组学的研究还比较有限,但有理由相信,未来植物PM蛋白质组学研究结果会越来越多、方法会越来越成熟,越来越多的PM蛋白功能会被阐释。
参考文献:
[1]Matthews J R,Shirazinejad C R,Isakson G A,et al. Structural analysis by enhanced raman scattering[J]. Nano Letters,2017,17(4):2172-2177.
[2]Qiao Z Z,Brechenmacher L,Smith B A,et al. The [WTBX][STBX]GmFWL1 (FW2-2-like) nodulation gene encodes a plasma membrane microdomain-associated protein[J]. Plant,Cell & Environment,2017,40(8):1442-1455.
[3]Lamontagne E D,Collins C A,Peck S C,et al. Isolation of microsomal membrane proteins fromarabidopsis thaliana[J]. Current Protocols in Plant Biology,2016,1(1):217-234.
[4]Perez-Sancho J,Tilsner J,Samuels A,et al. Stitching organelles:organization and function of specialized membrane contact sites in plants[J]. Trends in Cell Biology,2016,26(9):705-717.
[5]Wang P H C,Hussey P J. Plant endoplasmic reticulum-plasma membrane contact sites[J]. Trends in Plant Science,2017,22(4):289-297.
[6]Christ L,Raiborg C,Wenzel E M,et al. Cellular functions and molecular mechanisms of the ESCRT Membrane-Scission machinery[J]. Trends in Biochemical Sciences,2017,42(1):42-56.
[7]Raiborg C,Stenmark H. Plasma membrane repairs by small GTPase Rab3a[J]. Journal of Cell Biology,2016,213(6):613-615.
[8]Hsu Y W,Jauh G Y. VPS36-Mediated plasma membrane protein turnover is critical for Arabidopsis root gravitropism[J]. Plant Signaling & Behavior,2017,12(4):e1307495.
[9]Wang Y,Li R,Li D,et al. Nip1; 2 is a plasma Membrane-Localized transporter mediating aluminum uptake,translocation,and tolerance in Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America,2017,114(19):5047-5052.
[10]Hatsugai N,Hillmer R,Yamaoka S A,et al. The mu subunit of Arabidopsis adaptor protein-2 is involved in effector-triggered immunity mediated by membrane-localized resistance proteins[J]. Molecular Plant-Microbe Interactions,2016,29(5):345-351.
[11]Li A,Wang R Y,Li X L,et al. Proteomic profiling of cellulase-aid-extracted membrane proteins for functional identification of cellulose synthase complexes and their potential associated-components in cotton fibers[J]. Scientific Reports,2016,6:26356.
[12]Singer S J,Nicolson G L. The fluid mosaic model of the structure of cell membranes[J]. Science,1972,175(4023):720-731.
[13]Kusumi A,Fujiwara T K,Chadda R,et al. Dynamic organizing principles of the plasma membrane that regulate signal transduction:[JP3]commemorating the fortieth anniversary of singer and nicolsons Fluid-Mosaic model[J]. Annual Review of Cell & Developmental Biology,2012,28(1):215-250.
[14]王慶达,林其谁. 膜蛋白的拓扑学[J]. 生物化学与生物物理进展,1996,23(3):213-217.
[15]Hubert P,Sawma P,Duneau J P,et al. Single-spanning transmembrane domains in cell growth and Cell-Cell interactions[J]. Cell Adhesion & Migration,2014,4(2):313-324.
[16]Almén M,Nordstrm K V,Fredriksson R,et al. Mapping the human membrane proteome:a majority of the human membrane proteins can be classified according to function and evolutionary origin[J]. BMC Biology,2009,7(1):50.
[17]Albrecht D,Winterflood C M,Sadeghi M,et al. Nanoscopic compartmentalization of membrane protein motion at the axon initial segment[J]. Journal of Cell Biology,2016,27(1):37-46.
[18]Smith A W. Lipid-protein interactions in biological membranes:a dynamic perspective[J]. Biochimica et Biophysica Acta-Biomembranes,2012,1818:172-177.
[19]Bennett M P,Mitchell D C. Regulation of membrane proteins by dietary lipids:effects of cholesterol and docosahexaenoic acid acyl chain-containing phospholipids on rhodopsin stability and function[J]. Biophysical Journal,2008,95(3):1206-1216.
[20]Cherezov V,Rosenbaum D M,Hanson M A,et al. High-Resolution crystal structure of an engineered human 2-Adrenergic G protein-coupled receptor[J]. Science,2007,318(5854):1258-1265.
[21]Yao Z,Kobilka B. Using synthetic lipids to stabilize purified Β2 adrenoceptor in detergent micelles[J]. Analytical Biochemistry,2005,343(2):344-346.
[22]Klayman L M,Wedegaertner P B. Inducible inhibition of G reveals localization-dependent functions at the plasma membrane and golgi[J]. Journal of Biological Chemistry,2017,292(5):1773-1784.
[23]Liu Z Y,Jia Y X,Ding Y L,et al. Plasma membrane CRPK1-Mediated phosphorylation of 14-3-3 proteins induces their nuclear import to fine-tune CBF signaling during cold response[J]. Molecular Cell,2017,66(1):117.
[24]Shabardina V,Kramer C,Gerdes B A,et al. Mode of Ezrin-Membrane interaction as a function of PIP2 binding and pseudophosphorylation[J]. Biophysical Journal,2016,110(12):2710-2719.
[25]Bretscher A E K,Fehon R G. Erm proteins and Merlin:integrators at the cell cortex[J]. Nature Reviews Molecular Cell Biology,2002,3(8):586-599.
[26]Mangeat P R C,Martin M. Erm proteins in cell adhesion and membrane dynamics:authorscorrection[J]. Trends in Cell Biology,1999,9(5):187-192.
[27]Tsukita S,Yonemura S. Cortical actin organization:Lessons from ERM (ezrin/radixin/moesin) proteins[J]. Journal of Biological Chemistry,1999,274(49):34507-34510.
[28]Sauvanet C,Wayt J,Pelaseyed T,et al. Structure,regulation,and functional diversity of microvilli on the apical domain of epithelial cells[J]. Annual Review of Cell & Developmental Biology,2015,31(1):593-621.
[29]Kadota Y M A,Zipfel C. Immunoprecipitation of plasma membrane Receptor-Like kinases for identification of phosphorylation sites and associated proteins,1363[M]. New Jersey:Humana Press,2016:133-144.
[30]Yadeta K A E J,Coaker G. Advancements in the analysis of the Arabidopsis plasma membrane proteome[J]. Frontiers in Plant Science,2013,4(7):86.
[31]Dunkley T J,Hester S,Shadforth I P,et al. Mapping the Arabidopsis organelle proteome[J]. Proceedings of the National Academy of Sciences of the United States of America,2006,103(17):6518.
[32]Larsson C W S,Kjellbom P. Preparation of high-purity plasma membranes[J]. Methods in Enzymology,1987,148:558-568.
[33]Santoni V. Plant plasma membrane protein extraction and solubilization for proteomic analysis[J]. Plant Proteomics,2007,355:93-109.
[34]de Michele R,Mcfarlane H E,Parsons H T,et al. Free-flow electrophoresis of plasma membrane vesicles enriched by two-phase partitioning enhances the quality of the proteome from Arabidopsis [JP3]seedlings[J]. Journal of Proteome Research,2016,15(3):900-913.
[35]Li H Y,Pan Y,Zhang Y X,et al. Salt stress response of membrane proteome of sugar beet monosomic addition line M14[J]. Journal of Proteomics,2015,127:18-33.
[36]Collins C A,Leslie M E,Peck S C,et al. Simplified enrichment of plasma membrane proteins from arabidopsis thaliana seedlings using differential centrifugation and brij-58 treatment[J]. Methods in Molecular Biology,2017,1564:155.
[37]Liu Y L,Xing L J,Zhang Q S,et al. Thermo- and salt-responsive poly(NIPAm-co-AAc-Brij-58) microgels:adjustable size,stability under salt stimulus,and rapid protein adsorption/desorption[J]. Colloid and Polymer Science,2016,294(3):617-628.
[38]Nie L L,Feng J J,Fan P X,et al. Comparative proteomics of root plasma membrane proteins reveals the involvement of calcium signalling in NaCl-facilitated nitrate uptake in Salicornia europaea[J]. Journal of Experimental Botany,2015,66(15):4497-4510.
[39]Barua P,Subba P,Lande N V,et al. Gel-based and gel-free search for plasma membrane proteins in chickpea (Cicer arietinum L.) augments the comprehensive data sets of membrane protein repertoire[J]. Journal of Proteomics,2016,143(SI):199-208.
[40]Chen S S,Luo Y,Ding G D,et al. Comparative analysis of Brassica napus plasma membrane proteins under phosphorus deficiency using label-free and MaxQuant-based proteomics approaches[J]. Journal of Proteomics,2016,133(17):144-152.
[41]Orwick-Rydmark M A T,Linke D. The use of detergents to purify membrane proteins[J]. Current Protocols in Protein Science,2016,84(8):1-4.
[42]Martiniere A,Lavagi I,Nageswaran G A,et al. Cell wall constrains lateral diffusion of plant plasma-membrane proteins[J]. Proceedings of the National Academy of Sciences of the United States of America,2012,109(31):12805-12810.
[43]Hachez C,Besserer A,Chevalier A S. Insights into plant plasma membrane aquaporin trafficking[J]. Trends in Plant Science,2013,18(6):344-352.
[44]Bi Z,Merl-Pham J,Uehlein N,et al. RNAi-mediated downregulation of poplar plasma membrane intrinsic proteins (PIPs) changes plasma membrane proteome composition and affects leaf physiology[J]. Journal of Proteomics,2015,128(42):321-332.
[45]Gao Y,Zhang Y,Zhang D,et al. Auxin binding protein 1 (ABP1) is not required for either auxin signaling or Arabidopsis development[J]. Proceedings of the National Academy of Sciences of the United States of America,2015,112(7):2275-2280.
[46]Zhu J K. Abiotic stress signaling and responses in plants[J]. Cell,2016,167(2):313-324.
[47]Costa A,Barbaro M R,Sicilia F A,et al. AIR12,a b-type cytochrome of the plasma membrane of Arabidopsis thaliana is a negative regulator of resistance against Botrytis cinerea[J]. Plant Science,2015,233(20):32-43.
[48]Mansour M M F S K,Allam H. Role of the plasma membrane in saline conditions:lipids and proteins[J]. The Botanical Review,2015,81(4):416-451.
[49]Voothuluru P,Anderson J C,Sharp R E,et al. Plasma membrane proteomics in the maize primary root growth zone:novel insights into root growth adaptation to water stress[J]. Plant,Cell & Environment,2016,39(9):2043-2054.
[50]Gutierrez-Carbonell E,Takahashi D,Luethje S,et al. A shotgun proteomic approach reveals that Fe deficiency causes marked changes in the protein profiles of plasma membrane and detergent-resistant microdomain preparations from beta vulgaris roots[J]. Journal of Proteome Research,2016,15(8):2510-2524.
[51]Preger V,Tango N,Marchand C,et al. Auxin-responsive genes AIR12 code for a new family of plasma membrane b-type cytochromes specific to flowering plants[J]. Plant Physiology,2009,150(2):606-620.
[52]Mateos-Gil P,Letschert S,Doose S,et al. Super-Resolution imaging of plasma membrane proteins with click chemistry[J]. Frontiers in Cell and Developmental Biology,2016,4:98.
[53]Letschert S,Goehler A,Franke C A,et al. Super-resolution imaging of plasma membrane glycans[J]. Angewandte Chemie-International Edition,2014,53(41):10921-10924.
[54]Saka S K,Honigmann A,Eggeling C,et al. Multi-protein assemblies underlie the mesoscale organization of the plasma membrane[J]. Nature Communications,2014,5:4509.
