利用CRISPR/Cas9系统定向改良水稻稻瘟病抗性
2019-07-29徐鹏王宏涂燃冉刘群恩吴玮勋傅秀民曹立勇沈希宏
徐鹏 王宏 涂燃冉 刘群恩 吴玮勋 傅秀民 曹立勇 沈希宏
利用CRISPR/Cas9系统定向改良水稻稻瘟病抗性
徐鹏 王宏 涂燃冉 刘群恩 吴玮勋 傅秀民 曹立勇 沈希宏*
(中国水稻研究所/浙江省超级稻研究重点实验室/水稻生物学国家重点实验室, 杭州 311401;*通讯联系人, E-mail: xihongshen@126.com)
【】CRISPR/Cas9基因编辑技术是作物遗传改良的有效工具。本研究通过对水稻、和稻瘟病相关基因进行定点编辑,以期获得能够稳定遗传的抗稻瘟病水稻材料。【】利用CRISPR/Cas9基因编辑技术,以、和为靶基因,构建共编辑载体pC1300-2×35S::Cas9-g-g-g,用农杆菌转化长粒粳稻恢复系L1014,筛选获得稳定遗传的纯合突变体用于稻瘟病抗性鉴定。在T0代转基因株系中,、和突变频率分别为75%、85%和65%,突变基因型多为双等位突变。筛选到的不含T-DNA成分的T1代能够稳定遗传给T2代,并从中获得单突变纯合株系及、和的三突变纯合株系。稻瘟病抗性鉴定结果表明,与野生型相比,突变株系的抗性显著提高。同时,接种后纯合突变体株系内水杨酸、茉莉酸和乙烯等信号转导途径相关基因的表达量均上调。据此,我们推测纯合突变株系对稻瘟病的抗性增强可能与其对稻瘟病菌的响应被激活有关。利用CRISPR/Cas9技术获得了能够稳定遗传和具有较高稻瘟病抗性的纯合突变株系,为水稻稻瘟病抗性改良提供了良好的材料。
水稻;CRISPR/Cas9;;;;稻瘟病抗性
水稻是世界上食用人口最多的农作物。水稻产量每年都会受制于稻瘟病的暴发,稻瘟病严重时甚至导致颗粒无收,制约了水稻生产的发展。因此,提高稻瘟病抗性以减少水稻产量损失对于缓解日益增加的世界人口压力显得尤为重要[1],在生产实践中常使用化学农药和抗性品种来控制稻瘟病的发生[2],但两者的弊端日益显现。一方面,农药的使用,既污染了环境又增加了生产成本;另一方面,水稻稻瘟病是由子囊菌[(Hebert) Barr,无性世代为(Cooke) Sacc.]引起的真菌性病害,且稻瘟菌生理小种具有高度变异的潜力,其新基因型的产生势必降低了对水稻品种中抗性基因的敏感度[2],田间优势生理小种出现,优良抗性品种往往种植3~5年后其抗性会逐渐丧失[3]。在水稻育种上,结合分子标记辅助技术,通过杂交、回交方式能够将多个抗性基因聚合到同一品种中来弥补单一抗性基因抗性的弱化,但该育种过程周期较长,在将目标基因导入时难以避免连锁累赘,造成育种效率较低。
CRISPR/Cas9系统[4-7]是近年来发展较为迅速、操作简便及成本低而被广泛应用的基因编辑技术。该技术原理是借助gRNA与目标序列的特异性识别,引导Cas9核酸酶在靶位点切割DNA使之双链断裂[8],进而激发胞内DNA修复系统进行同源重组和非同源重组修复时引起的碱基特异突变(碱基插入、缺失和替换)[7]。近年来,利用该系统已经对水稻[9-12]、拟南芥[12-14]、小麦[10,12,15-16]等不同物种进行了相关性状的遗传改良。Xie等[4]通过定向编辑水稻基因来改良水稻对病原菌的抗性;Shen等[17]利用CRISPR/Cas9系统同时敲除8个水稻性状相关基因,获得了众多不同基因组合的突变体;Fei等[18]通过定向基因降低直链淀粉含量来改良稻米品质。CRISPR/Cas9技术能够在育种过程中迅速得到安全、稳定遗传的纯和突变,从而在植物基因组编辑中得到广泛的应用。
前人研究表明,是一个隐性抗稻瘟基因位点[19],产物富含脯氨酸,包含重金属结合结构域和蛋白互作结构域,其抗性的产生是抗性品种的基因在富脯氨酸区分别有21 bp和48 bp的缺失[20]。是一个AP2/ERF类转录因子,受稻瘟病菌诱导而负调控稻瘟病菌的抗性[21]。基因编码928个氨基酸,其抗感差异的产生是由于第918位的丙氨酸到丝氨酸的变化[22],且基因介导的抗性必须有(t)的特异性信号识别发挥作用[23]。本研究利用CRISPR/Cas9系统对长粒粳稻恢复系L1014中、和为靶基因进行定点编辑,进而获得有利用价值的纯合突变体,为水稻稻瘟病抗性育种提供材料。
1 材料与方法
1.1 试验材料与载体
转基因受体材料长粒粳稻恢复系L1014及其T0转基因植株于2017年夏季种植在中国水稻研究所转基因试验基地,常规水肥管理,T1植株于冬季种植在温室。表达载体pC1300-Cas9和SK-gRNA中间载体由中国水稻研究所王克剑实验室提供。
1.2 gRNA靶位点设计
利用CRISPR-P网站(http://cbi.hzau.edu.cn/cgi- bin/CRISPR)进行gRNA靶位点的设计,选择得分较高且靠近ATG起始位点的引物序列,分在和的第1外显子上设计19 bp的靶位点序列且其PAM序列为NGG(图1)。其中,3个靶位点的设计都靠近ATG起始密码子,以便更容易造成目标基因的功能丧失。最后利用NCBI对水稻基因组进行BLAST来分析确认靶位点的特异性。
1.3 CRISPR/Cas9表达载体的构建
根据靶位点序列,分别合成引物-g-F/- g-R、-g-F/-g-R和-g-F-g-R (表1),然后等量混合变性退火形成具有黏性末端的片段。利用Ⅰ(Ferment公司)酶切SK-gRNA中间载体,用T4连接酶将、和接头和被Ⅰ酶切回收的SK-gRNA线性载体进行连接,转化大肠杆菌,通过测序(杭州擎科梓熙生物技术有限公司)挑选出正确的连接载体。分别用Ⅰ/Ⅰ、Ⅰ/Ⅰ和Ⅰ/Ⅱ酶切SK-gRNA-、SK-gRNA-和SK-gRNA-三个中间载体获得目的片段,同时用Ⅰ/HⅠ酶切pC1300- Cas9载体,再利用T4连接酶连接酶切产物构建成pC1300-2×35S::Cas9-g-g-g表达载体,通过测序确定正确的表达载体(图2),最终利用农杆菌转化受体材料获得转基因T0植株。
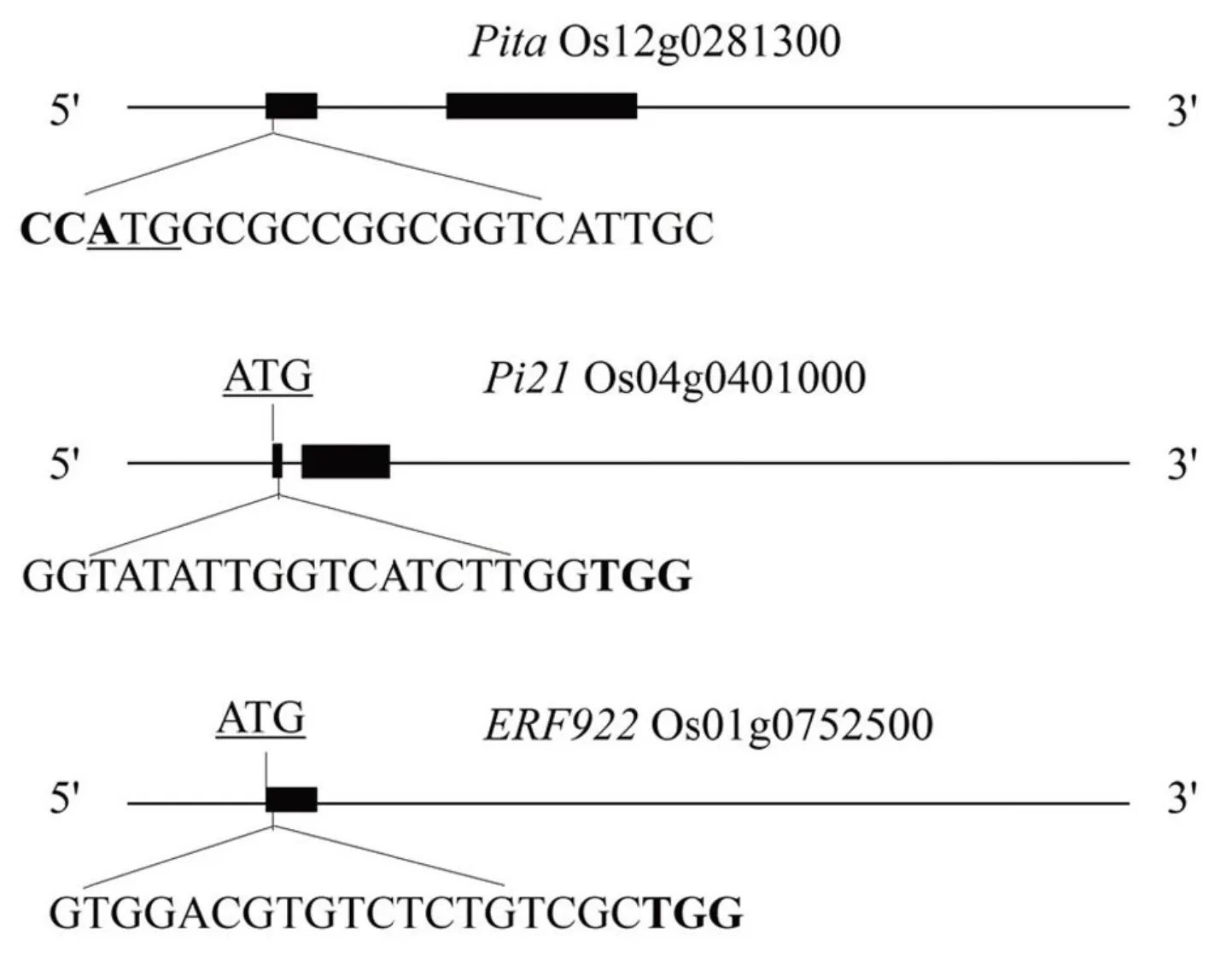
带下划线的字母为起始密码子,加粗字母为PAM序列。
Fig. 1. Schematic diagram of the targeted sites inand.
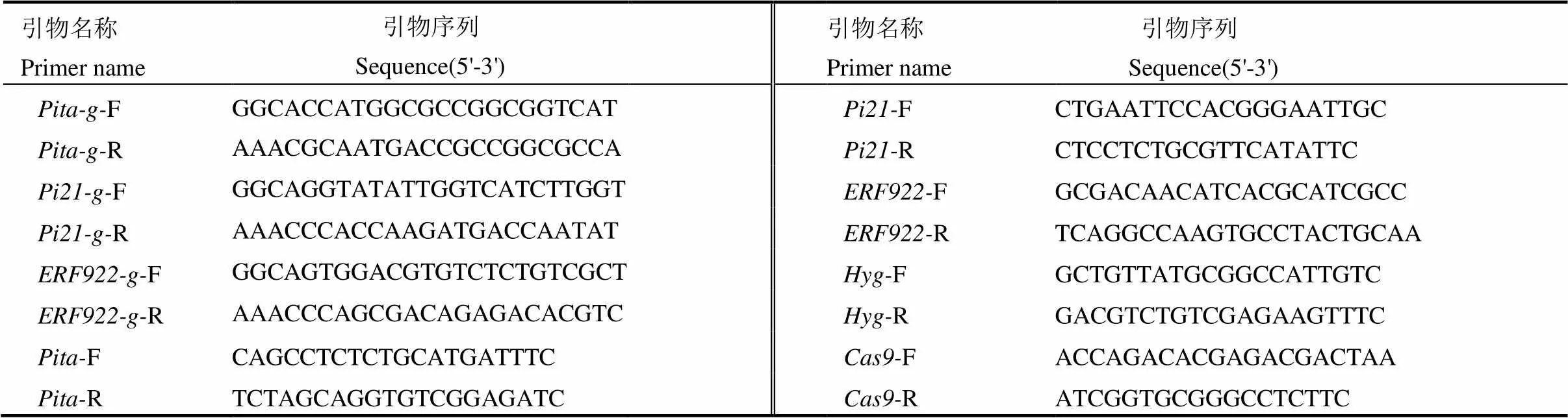
表1 本研究所用引物
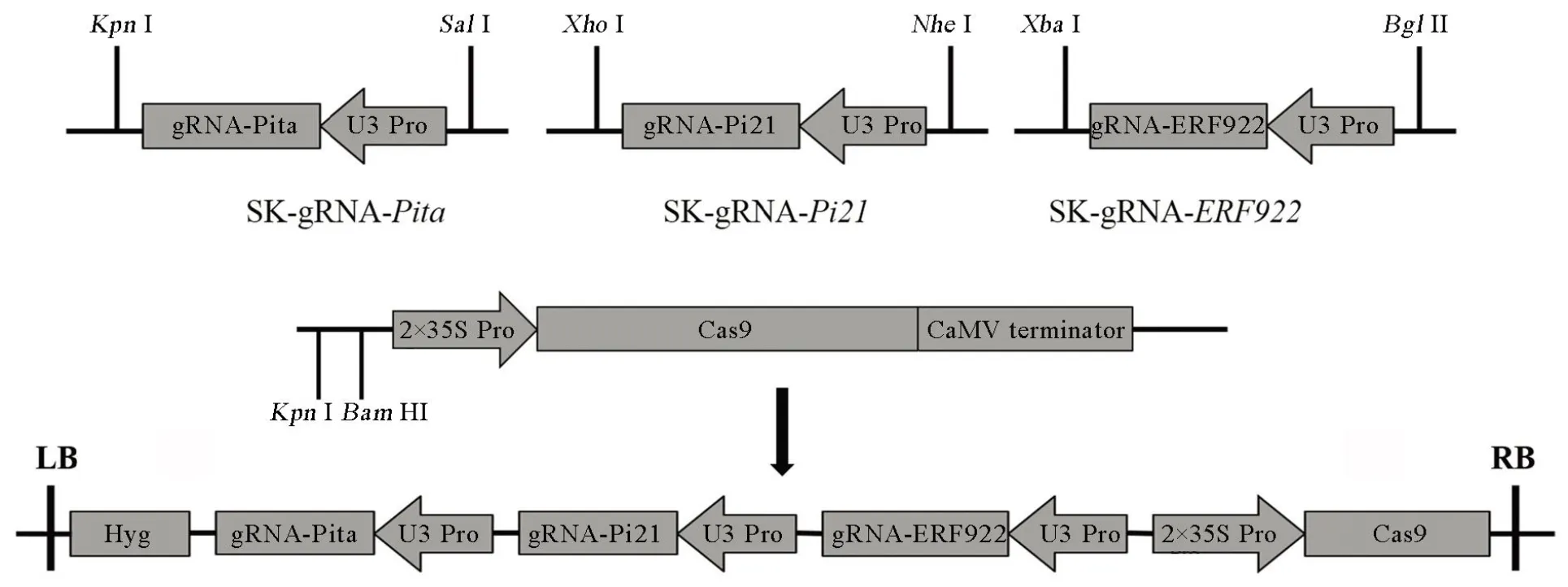
图2 表达载体构建流程
Fig. 2. Flow diagram of the expression vector construction.
1.4 T0代阳性株的获得及靶位点检测
用农杆菌株转化受体材料长粒粳稻L1014获得T0植株。剪取T0植株叶片利用CTAB方法提取基因组DNA。分别在、和靶序列两侧设计扩增引物(表1)来扩增T0阳性植株的目的条带,将PCR产物送杭州擎科梓熙生物技术有限公司测序,以L1014基因组序列作为参照。测序出现单峰则为野生型或纯合突变,再通过与野生型序列比对确定;测序出现双峰,利用在线软件DSDecode进行序列解码。
1.5 T1代无转基因成分筛选
利用载体特异引物对F/R和Cas9F/Cas9-R扩增突变植株基因组序列,两对引物均扩增不出条带的为无转基因成分的突变植株。
1.6 稻瘟病菌的培养及孢子液的配制
在超净工作台内将从−20℃冰箱取出的带有稻瘟菌生理小种18-6-5(本实验室保存)的纸片放置在燕麦培养基上,把培养基倒置放进霉菌培养箱,25℃~27℃下黑暗培养至菌丝长满培养皿,用灭菌的棉签刮掉菌丝,灭菌水清洗净菌丝后室温晾干,然后在培养皿上盖两层已灭菌的纱布,盖紧培养皿,25℃~27℃下光照培养至大量分生孢子产生。
用0.02%的吐温20洗脱培养好的稻瘟菌分生孢子(可将洗脱的孢子液倒入同一菌种的平板继续洗,如此洗脱2~3次),用灭菌的棉签轻轻刮下稻瘟菌,然后用灭菌的两层纱布过滤得到孢子收集液。将孢子液混匀后,在血球计数板上计数,把孢子悬浮液调至2×105个/mL(在10×10倍视野下孢子数为20~30个)用于稻瘟菌接种备用[20]。
1.7 稻瘟菌喷雾接种
接种前用0.02%的吐温洗脱液润洗喷雾瓶,将培养好的水稻幼苗(播种后两周)放进透明的收纳箱内,取配制好的孢子溶液斜向上喷向空中使之均匀落在水稻叶片上,尽量使每片叶片上形成雾化的小水滴,喷完后立即用保鲜膜封口并盖上盖子。放置于培养箱,保持28℃恒温,95%恒湿,黑暗培养24 h后恢复正常光照周期,每天对水稻苗进行一次少量喷水以促进发病,接种7 h后调查发病情况。以丽江新团黑谷作为感病对照。

表2 对水稻防卫相关基因进行qRT-PCR分析所用的引物
1.8 接种后取样及防卫相关基因的表达量分析
在稻瘟菌接种后的0、12、24 h分别对野生型和突变株系进行混合取样。在各个取样时间点对3次重复接种的幼苗随机取样5片左右(每个株系水稻幼苗的倒1叶),迅速置于液氮中,−80℃下保存。
利用植物总RNA提取试剂盒(RNAprep pure Plant Kit)提取样品总RNA,用反转试剂盒(ReverTra Aceq PCR RT Master Mix with gDNA Remover,ToYoBo,日本)进行第1链cDNA合成,反应体系和反应条件参照说明书。荧光定量PCR(Quantitative Real-Time PCR, qRT-PCR)扩增程序如下:95℃下预变性30 s;95℃下变性5 s,58℃下30 s,72℃下15 s,共35个循环,定量PCR引物见表2。以(登录号为LOC_Os03g08020)作为内参基因,用2−∆∆法进行基因相对表达量的计算。
2 结果与分析
2.1 T0转基因阳性株的获得
将携带3个靶点的表达载体pC1300-2×35S:: Cas9-g-g-g通过农杆菌转化受体材料L1014以获取T0转基因株系。用载体特异引物F/R和Cas9F/Cas9-R检测转基因苗中T-DNA区以筛选转基因阳性株系。结果表明,21株T0转基因苗中有20株阳性苗,阳性率达95.2%。
2.2 靶位点突变情况分析
为了分析L1014中、和的突变情况,用靶点检测引物分别扩增三个靶序列的上下游,将PCR产物进行测序分析。测序结果表明,有15株阳性苗在靶位点位置发生了突变,突变频率为75%;有17株阳性苗在靶点发生突变,突变频率为85%;有13株阳性苗在靶点发生突变,突变频率为65%。其中,突变基因型包括纯和突变、双等位突变,突变类型包括碱基插入和缺失,以碱基插入为主(73.5%);突变基因型包括纯合突变、杂合突变和双等位突变,突变类型包括碱基的插入、缺失和替换,其中以碱基缺失为主,突变频率高达85.3%;突变基因型包括纯合突变、杂合突变和双等位突变,突变类型包括碱基的插入、缺失、替换及同时替换缺失(表3)。
2.3 无转基因成分的纯合突变株的获得
为获得纯合突变植株且不含T-DNA转基因成分,将T0代阳性株自交繁殖下一代构成T1代突变群体。提取T1代植株DNA样品,用载体特异引物F/R和Cas9F/Cas9-R扩增T1代突变体基因组序列,两对引物不能同时扩增出615 bp和600 bp的DNA片段的植株即不含转基因成分的突变株(图3为部分T1植株的鉴定)。结果筛选出2种单基因突变和1种和三基因突变不含有T-DNA成分,纯合突变类型见图4。利用靶点检测引物分别扩增无突变植株的靶位点上下游DNA序列。PCR产物测序表明,T1代突变体靶位点突变情况与T0代是一致的,说明这2种单基因突类型和1种和三基因突类型在剔除Cas9载体骨架后是能够稳定遗传的。把以上三种纯合突变体通过自交构建T2代纯合突变株系,分别命名为D2101、D2102和D0301。随后对这三种纯合突变系进行接种鉴定。
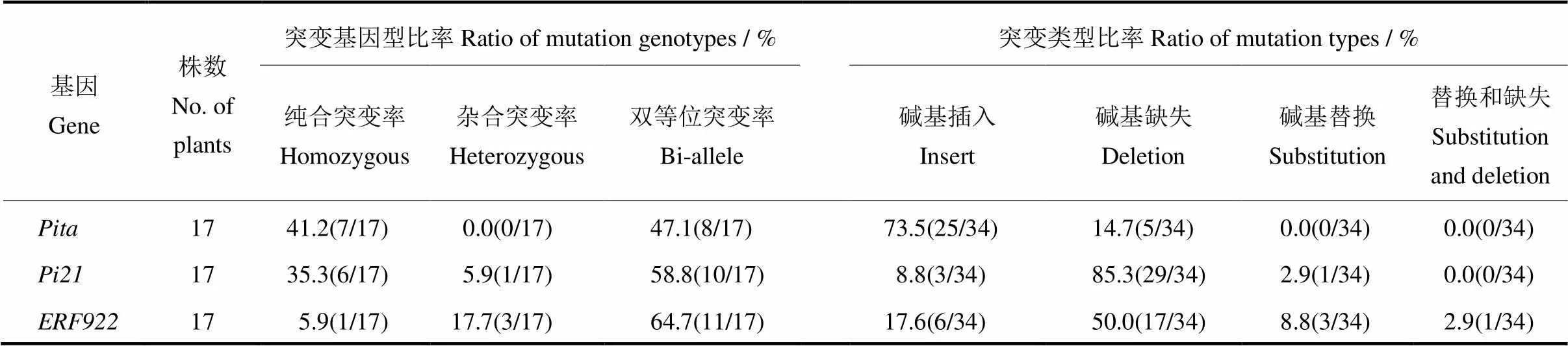
表3 T0突变体突变基因型和突变类型频率
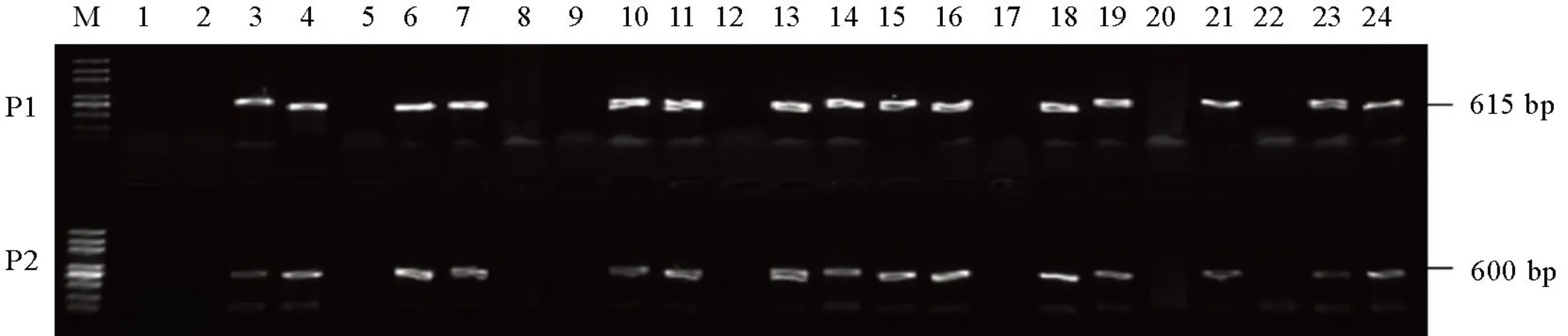
M−DNA标记; 1−双蒸水; 2−野生型; 3−阳性对照; 4~24−T1突变单株; P1−引物Hyg-F和Hyg-R; P2−引物Cas9-F和Cas9-R。
Fig. 3. Screening of transgenic-free mutant plantsby PCR in T1.
Fig. 4. Three mutation types of homozygotes in T2.
2.4 纯合突变系编辑基因的qRT-PCR检测及氨基酸序列分析
对上述纯合D2101、D2102和D0301突变系的、和进行qRT-PCR检测,并分析其相对表达量(图5)。在D2101和D2102单基因纯合突变株系中的表达量相对于野生型无显著变化,在D0301三基因纯合突变株系的表达量却显著低于野生型。在D2101和D2102单基因纯合突变株系中的表达量极显著低于在野生型的表达,与野生型相比,在D0301三基因纯合突变株系的表达量显著降低。在D2101和D2102单基因纯合突变株系中的表达量趋势类似于,即在单基因突变株系中的表达量与野生型相比没有显著的改变,而在D0301三基因纯合突变株系中,其相对表达量极显著低于野生型。说明、和三个基因在被Cas9系统靶向编辑后,其转录水平可能受到影响,RNA可能在体内发生部分降解。

*和**分别表示野生型与纯合突变体的差异达0.05和0.01显著水平(t检验)。
Fig. 5. Expression analyses of,andin homozygous mutant plants.
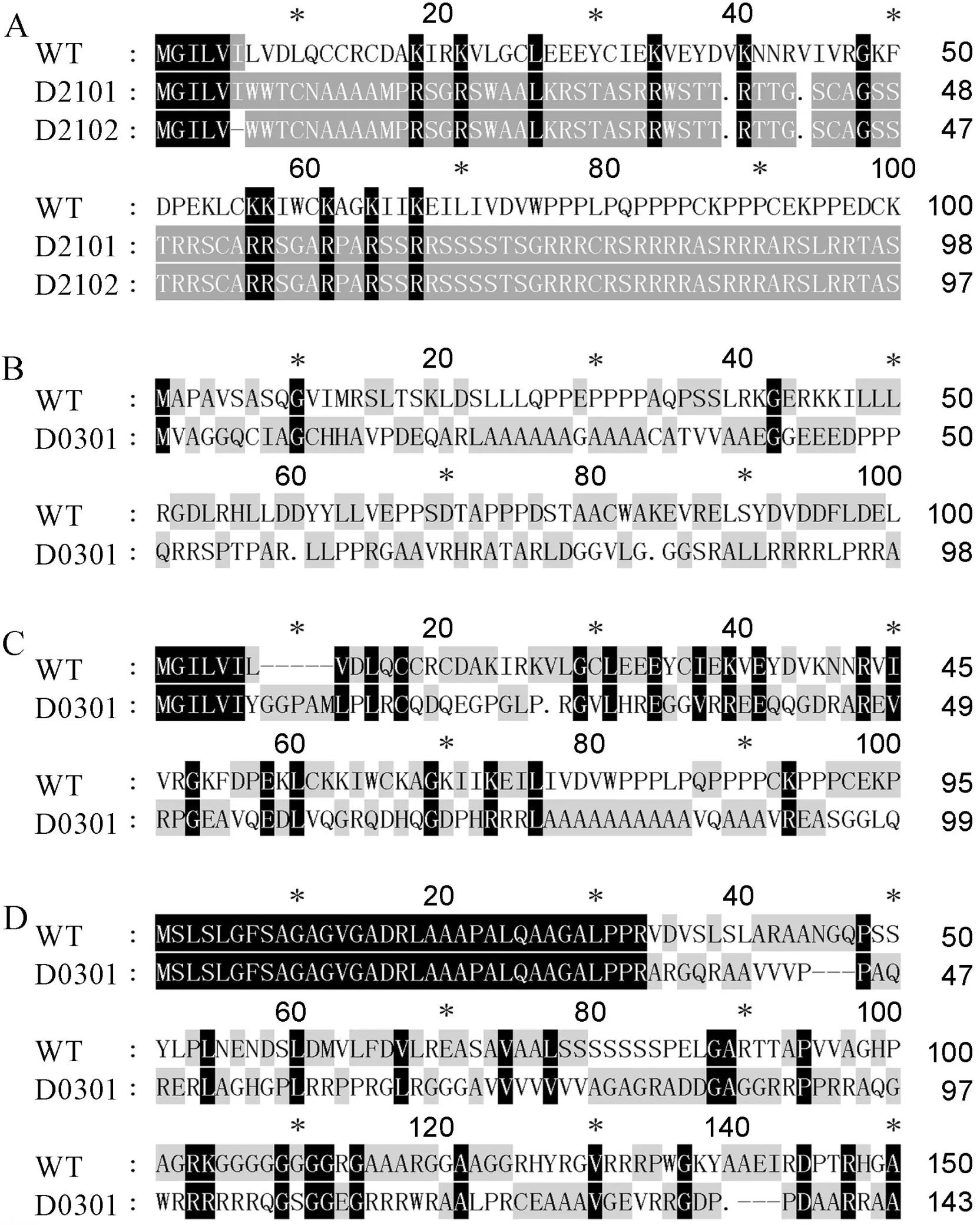
A为D2101和D2102纯合突变株系中Pi21氨基酸序列的比对;B, C和D分别为D0301纯合突变株系中Pita、Pi21和ERF922的氨基酸序列比对。
Fig. 6.,andgeneamino acid sequences in homozygous mutant lines and the wild type.
同时,对D2101和D2102纯合突变株系的基因及D0301纯合突变株系的、和基因编码蛋白的氨基酸序列进行比对分析(图6)。由于靶位点设计在目的基因ATG起始密码子附近,经过基因编辑后,靶位点序列出现碱基的插入和缺失,导致蛋白的翻译提前终止,从而影响蛋白的完整性。表明基因编辑不但能最终影响蛋白的水平,对基因的转录水平可能也存在一定的影响。
2.5 纯合突变株系的稻瘟病抗性鉴定
为了鉴定CRISPR/Cas9基因编辑技术创制的纯合突变株系对稻瘟病菌的抗性,我们选择单基因突变的两种不同突变类型(D2101在第1外显子有1 bp缺失,D2102在第1外显子缺失4 bp)以及、和三基因突变的一种突变类型用来接种鉴定。对3种不同突变类型的T2代纯和突变株系于苗期用喷雾法接种稻瘟菌生理小种18-6-5,接种7 d后对病斑叶片进行拍照并统计病斑面积(图7)。从图中可以看出,野生型叶片侵染较为严重,有明显的病斑并有所扩散,而纯和突变株的病斑明显小于野生型,只是侵入叶片形成病斑而未进一步扩散。对病斑面积进一步量化时,纯合突变株系的相对病斑面积跟野生型存在显著差异。以上结果表明,纯合突变株系在一定程度上提高了对稻瘟菌生理小种18-6-5的抗性,但是否对其他稻瘟菌生理小种产生抗性,仍需进一步的接种鉴定。
2.5 基因编辑株系的防卫相关基因表达量分析
植物响应病原菌侵入时,体内会激发一系列病程相关基因的表达来抵御病原菌的扩散。野生型和纯合突变株系的防卫相关基因的差异表达结果如图8和图9。本研究对SA合成及信号传导途径中的、与,JA合成相关基因,乙烯应答转录因子等基因进行突变株系的表达差异分析。D2101突变株系的、、基因表达量在接种24 h后较野生型极显著上调;D2102突变株系的、、及基因在接种12 h后的表达水平高于野生型,其中的表达量在接种12 h后是野生型的220倍;而的表达水平在D2102突变株系中接种12 h后较野生型显著下降,接种24 h后却显著高于野生型。和在未接种时表达水平已显著高于野生型,接种12 h和24 h后其表达水平持续上调;D0301突变株系的和基因表达模式类似于D2101和D2102单突突变株系,即在未接种时基因产物有所积累,在接种12 h和24 h后其表达产物持续积累。突变体本身的某些防卫相关基因在基因编辑后被激发表达,另外,与野生型相比,在病原菌侵入水稻时,突变株系中的以上防卫相关基因在接种初期(12 h和24 h)表达量更高。
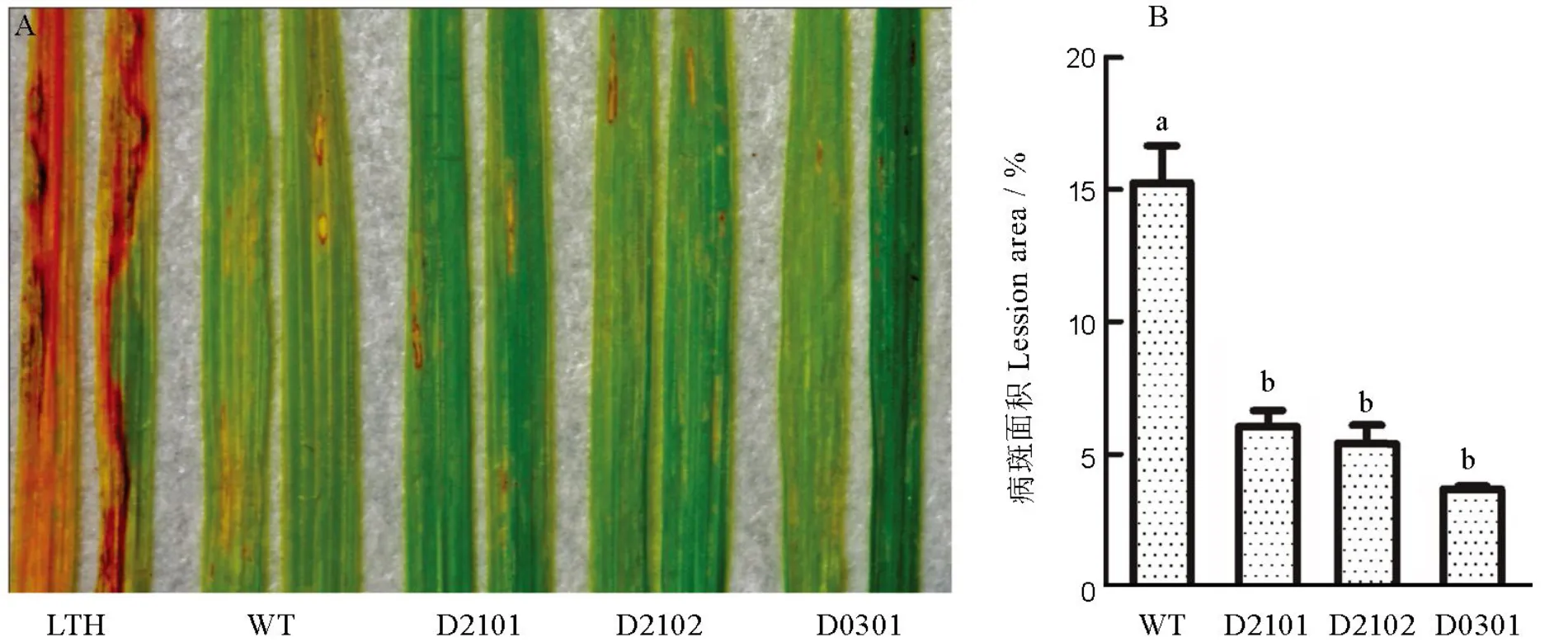
A−纯和突变株系和野生型在苗期的接种表型;B−野生型和纯合突变株系的相对病斑面积(均值±标准误,n=3)。柱上不同小写字母表示野生型与纯合株的差异达0.01显著水平(t检验)。LTH−丽江新团黑谷;WT−L1014野生型;D2101和D2102−Pi21纯合株系;D0301−Pita、Pi21和ERF922纯合株系。
Fig. 7. Identification of homozygous mutant lines and the control varieties after inoculation.
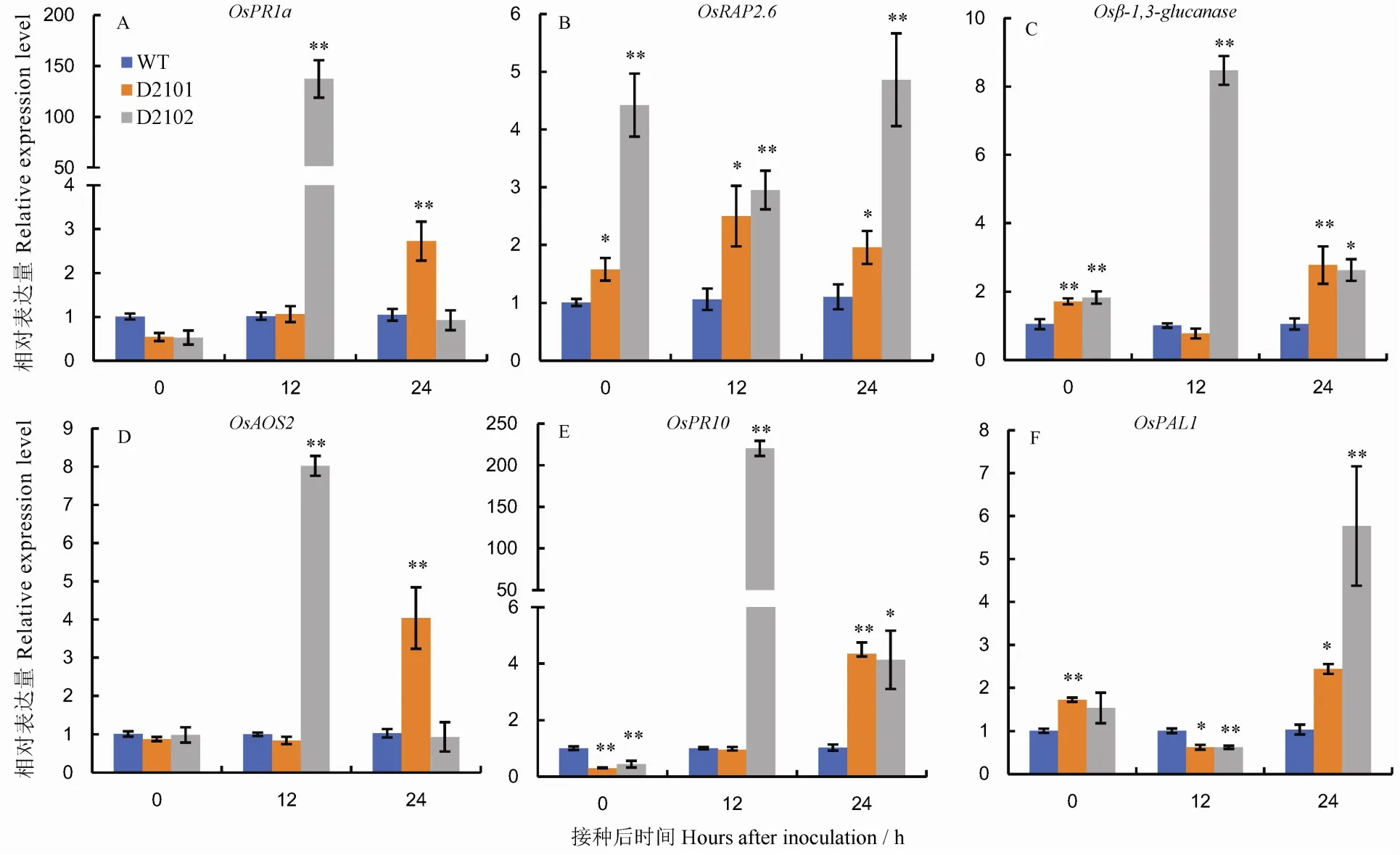
*和**表示野生型和纯合株系在接种后同一时期0.05和0.01水平上的差异。
Fig. 8. Relative expression levels of defense-related genes in the wild type (L1014, WT) and the homozygous mutant lines inoculated with(Mean±SE,=3).
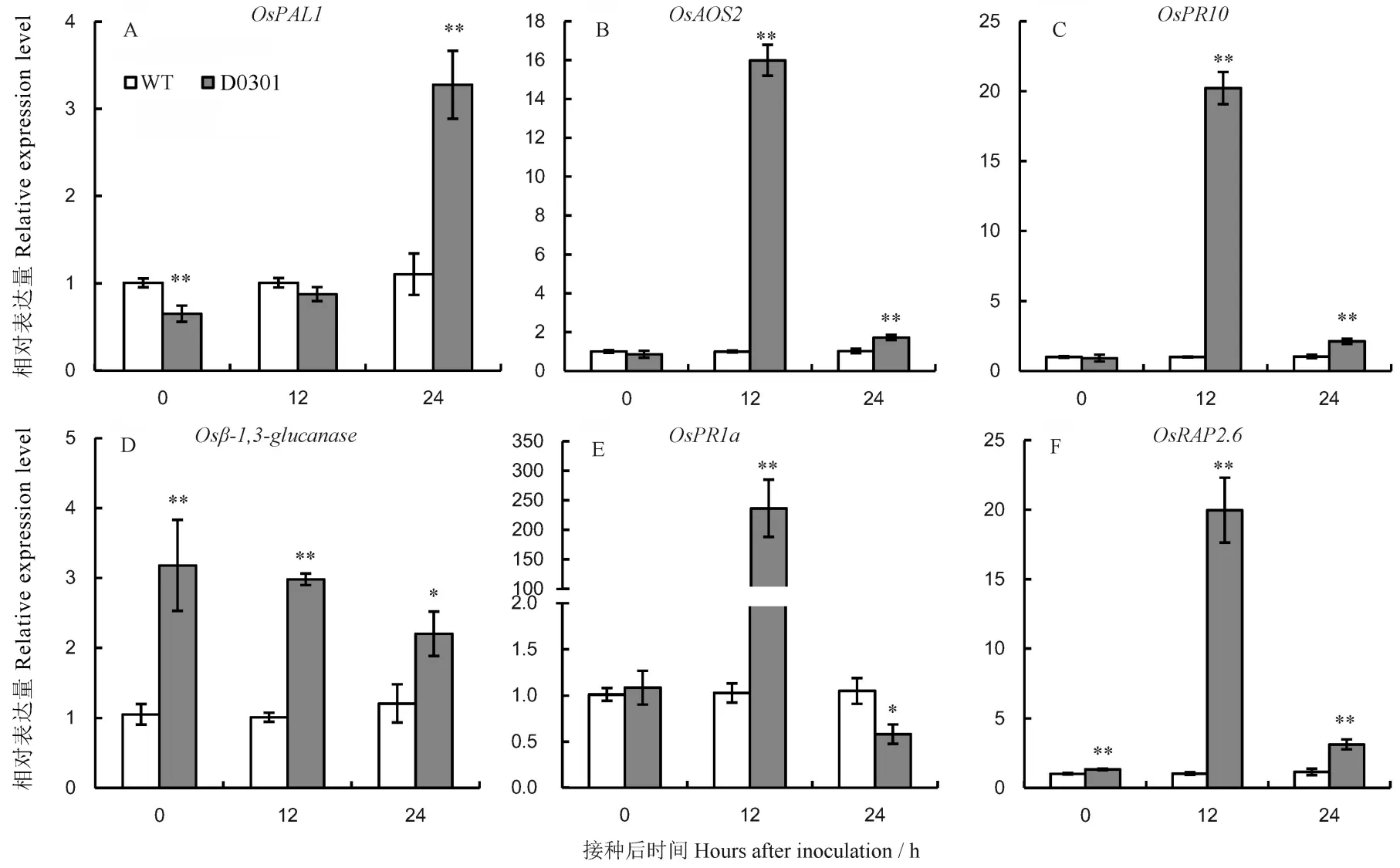
*和**表示在接种后同一时期野生型与纯合株系的差异达0.05和0.01显著水平。
Fig. 9. Relative expression level of defense-related genes in the wild type(L1014) and the homozygous mutant line D0301, inoculated with(Mean±SE,=3).
3 讨论
近年来,CRISPR/Cas9基因编辑技术的发展和应用正方兴未艾,研究者不断对该系统进行优化来提高编辑效率继而扩大其应用范围。由于该技术具有操作简便、成本较低特点,目前被广泛应用于动物[24, 25]、植物[9, 12, 26, 27]和微生物[28]中。Shen[29]利用CRISPR/Cas9系统成功构建了水稻的,和三基因表达载体,分析了基因编辑效率及三个基因的突变情况;随后利用该系统同时编辑8个水稻相关性状基因[17],得到了丰富的突变株系。本研究也表明,该技术在水稻抗性品种改良上是较为有效的育种工具。
本研究利用CRISPR/Cas9基因编辑技术对长粒粳稻恢复系L1014进行、和三个稻瘟病抗性相关基因的共编辑,以期得到具有利用价值的纯合突变系。在T0代中和三基因的突变频率分别为75%、85%和65%。突变类型以碱基插入为主(频率为73.5%),和突变类型以碱基缺失为主(频率分别为85.3%和58.8%),突变基因型以双等位突变频率最高(频率分别为47.1%,58.8%和64.7%)。王芳权等[30]利用CRISPR/Cas9基因编辑技术编辑水稻基因的两个靶位点,突变频率分别为78.57%和92.86%,杨海河等[31]构建的单突突变频率为86.7%; Wang等[32]对进行基因编辑,其突变类型多为碱基缺失且突变基因型也多为双等位突变为主。本研究中的突变频率和的突变情况跟前人研究结果基本一致。我们发现,的突变往往伴随着和的突变,三个基因的突变与期望有所偏离,从而趋向于同时发生突变。因此,该研究中只得到单突和、和三突变这两种基因型组合。
对获得的两种基因组合的纯合突变株系进行稻瘟病抗性鉴定,利用CRISPR/Cas9基因编辑技术产生的单突株系和、和三突变株系在接种稻瘟菌后能够表现出相应的抗性。在对基因编辑纯合株系的防卫相关基因表达进行分析时,发现与野生型相比,突变株系的SA、JA和乙烯信号途径被激活从而引发防卫相关基因的高表达。SA合成相关基因在三突变纯合系接种24 h后其表达量显著上调,在单突变纯合系中,表达量在接种12 h后有所下降,但在接种24 h后表达量显著增高;SA信号转导途径相关基因在D2102单突变纯合系和三突变纯合系在接种12 h后,表达量迅速达到极显著水平,而在D2101单突变纯合系接种24 h后表达量才达到显著水平;JA合成相关途径基因的表达与在单突变纯合系的表达模式类似,在三突变纯合系接种12 h后表达水平显著提高,而在接种24 h后表达水平又迅速降低。纯合突变株系在未接种时和基因表达水平已显著高于野生型,在接种后的12 h和24 h后表达更是持续积累。前人研究报道,基因编码葡聚糖酶,能够降解真菌细胞壁[33],基因为乙烯应答转录因子,与RACK1互作参与水稻先天免疫反应[34]。这些基因在接种前后表达量的变化能够促使植物快速的响应和抵御病原菌的侵染和扩散。是个显性抗性基因,具有稻瘟病菌的生理小种特异性,其抗性由所介导[23],而和介导的抗性属于基础抗性,具有抗谱广和持续时间长的特点。本研究中、和三突变纯合系也能产生相应的抗性,可能是和介导的基础抗性效应在起主要作用。根据Zhao等[35]发现,不仅是发挥抗性功能所需,其本身也能对更多的生理小种产生抗性,包括识别的生理小种,植物长期进化过程中产生多种抗性机制来适应病原菌的侵染,、和三突纯合系产生相应的抗性也可能是功能丧失后,由来弥补病原菌入侵带来的损害。
谢辞:感谢中国水稻研究所王克剑老师提供SK-gRNA中间载体和pC1300-Cas9表达载体。
[1] Liu J, Wang X, Mitchell T, Hu Y, Liu X, Dai L, Wang G L. Recent progress and understanding of the molecular mechanisms of the rice-interaction., 2010, 11(3): 419-427.
[2] Borrelli V, Brambilla V, Rogowsky P, Marocco A, Lanubile A. The enhancement of plant disease resistance using CRISPR/Cas9 technology., 2018, 9: 1245.
[3] Wilson R A, Talbot N J. Under pressure: investigating the biology of plant infection by., 2009, 7(3): 185-195.
[4] Xie K, Yang Y. RNA-guided genome editing in plants using a CRISPR-Cas system., 2013, 6(6): 1975-1983.
[5] Shan Q, Wang Y, Li J, Zhang Y, Chen K, Liang Z, Zhang K, Liu J, Xi J J, Qiu J L, Gao C. Targeted genome modification of crop plants using a CRISPR-Cas system., 2013, 31(8): 686-688.
[6] Cong L, Ran F A, Cox D, Lin S, Barretto R, Habib N, Hsu P D, Wu X, Jiang W, Marraffini L A, Zhang F. Multiplex genome engineering using CRISPR/Cas systems., 2013, 339(6121): 819-823
[7] Islam W. CRISPR-Cas9: An efficient tool for precise plant genome editing., 2018, 39: 47-52.
[8] Vriend L E M, Prakash R, Chen C, Vanoli F, Cavallo F, Zhang Y, Jasin M, Krawczyk P M. Distinct genetic control of homologous recombination repair of Cas9-induced double-strand breaks, nicks and paired nicks., 2016, 44(11): 5204-5217.
[9] Mao Y, Zhang H, Xu N, Zhang B, Gou F, Zhu J K. Application of the CRISPR-Cas system for efficient genome engineering in plants., 2013, 6(6): 2008-2011.
[10] Shan Q, Wang Y, Li J, Gao C. Genome editing in rice and wheat using the CRISPR/Cas system., 2014, 9(10): 2395-2410.
[11] Zhang J, Zhang H, Botella J R, Zhu J K. Generation of new glutinous rice by CRISPR/Cas9-targeted mutagenesis of the Waxy gene in elite rice varieties. J, 2018, 60(5): 369-375.
[12] Jiang W, Zhou H, Bi H, Fromm M, Yang B Weeks D P. Demonstration of CRISPR/Cas9/sgRNA-mediated targeted gene modification in, tobacco, sorghum and rice., 2013, 41(20): e188.
[13] Fauser F, Schiml S, Puchta H. Both CRISPR/Cas-based nucleases and nickases can be used efficiently for genome engineering in., 2014, 79(2): 348-359.
[14] Jiang W, Yang B Weeks D P. Efficient CRISPR/Cas9- mediated gene editing in Arabidopsis thaliana and inheritance of modified genes in the T2and T3generations., 2014, 9(6): e99225.
[15] Wang Y, Cheng X, Shan Q, Zhang Y, Liu J, Gao C, Qiu J L. Simultaneous editing of three homoeoalleles in hexaploid bread wheat confers heritable resistance to powdery mildew., 2014, 32(9): 947-951.
[16] Zhang Y, Bai Y, Wu G, Zou S, Chen Y, Gao C, Tang D. Simultaneous modification of three homologs of TaEDR1 by genome editing enhances powdery mildew resistance in wheat., 2017, 91(4): 714-724.
[17] Shen L, Hua Y, Fu Y, Li J, Liu Q, Jiao X, Xin G, Wang J, Wang X, Yan C, Wang K. Rapid generation of genetic diversity by multiplex CRISPR/Cas9 genome editing in rice., 2017, 60(5): 506-515.
[18] Fei Y Y, Yang J, Wang F Q, Fan F J, Li W Q, Wang J, Xu Y, Zhu J Y, Zhong W G. Production of two elite glutinous rice varieties by editinggene., 2019, 26(2): 118-124.
[19] Fukuoka S, Okuno K. QTL analysis and mapping of, a recessive gene for field resistance to rice blast in Japanese upland rice., 2001, 103(2-3): 185-190.
[20] Fukuoka S, Saka N, Koga H, Ono K, Shimizu T, Ebana K, Hayashi N, Takahashi A, Hirochika H, Okuno K, Yano M. Loss of function of a proline-containing protein confers durable disease resistance in rice., 2009, 325(5943): 998-1001.
[21] Liu D, Chen X, Liu J, Ye J, Guo Z. The rice ERF transcription factor OsERF922 negatively regulates resistance toand salt tolerance., 2012, 63(10): 3899-3911.
[22] Bryan G T, Wu K S, Farrall L, Jia Y, Hershey H P, McAdams S A, Faulk K N, Donaldson G K, Tarchini R, Valent B. A single amino acid difference distinguishes resistant and susceptible alleles of the rice blast resistance gene., 2000, 12(11): 2033-2046.
[23] Jia Y, Martin R. Identification of a new locus,, required for rice blast resistance gene-mediated resistance., 2008, 21(4): 396-403.
[24] Tu Z, Yang W, Yan S, Guo X, Li X. CRISPR/Cas9: A powerful genetic engineering tool for establishing large animal models of neurodegenerative diseases., 2015, 10: 35.
[25] Hruscha A, Krawitz P, Rechenberg A, Heinrich V, Hecht J, Haass C, Schmid B. Efficient CRISPR/Cas9 genome editing with low off-target effects in zebrafish., 2013, 140(24): 4982-4987.
[26] Cai Y, Chen L, Liu X, Guo C, Sun S, Wu C, Jiang B, Han T, Hou W. CRISPR/Cas9-mediated targeted mutagenesis of GmFT2a delays flowering time in soya bean., 2018, 16(1): 176-185.
[27] Yu Z, Chen Q, Chen W, Zhang X, Mei F, Zhang P, Zhao M, Wang X, Shi N, Jackson S, Hong Y. Multigene editing via CRISPR/Cas9 guided by a single-sgRNA seed in., 2018, 60(5): 376-381.
[28] Jakočiu̅nas T, Rajkumar AS, Zhang J, Arsovska D, Rodriguez A, Jendresen C B, Skjødt M L, Nielsen A T, Borodina I, Jensen M K, Keasling J D. CasEMBLR: Cas9-facilitated multiloci genomic integration of in vivo assembled DNA parts in., 2015, 4(11): 1226-1234.
[29] 沈兰. 基于CRISPR/Cas9的水稻多基因编辑及其在育种中的应用. 扬州: 扬州大学, 2017.
Shen L. CRISPR/Cas9-mediated multiples genome editing in rice () and the application in rice breeding. Yangzhou: Yangzhou University, 2017. (in Chinese with English abstract)
[30] 王芳权, 范方军, 李文奇, 朱金燕, 王军. 利用CRISPR/Cas9技术敲除水稻基因的效率分析. 中国水稻科学, 2016, 30(5): 469-478.
Wang F Q, Fan F J, Li W Q, Zhu J Y, Wang J. Knock-out efficiency analysis ofgene using CRISPR/Cas9 in rice., 2016, 30(5): 469-478. (in Chinese with English abstract)
[31] 杨海河, 毕冬玲, 张玉, 邹小维, 高晓庆, 袁正杰, 曲海艳, 何海燕, 瞿绍洪. 基于CRISPR/Cas9技术的水稻基因编辑材料的创制及稻瘟病抗性鉴定. 分子植物育种, 2017, 15(11): 4451-4465.
Yang H H, Bi D L, Zhang Y, Zou X W, Gao X Q, Yuan Z J, Qu H Y, He H Y, Qu S H. Generation of ricegene editing lines based on CRISPR/Cas9 technology and evaluation of their blast resistance., 2017, 15(11): 4451-4465.(in Chinese with English abstract)
[32] Wang F, Wang C, Liu P, Lei C, Hao W, Gao Y, Liu Y, Zhao K. Enhanced rice blast resistance by CRISPR/Cas9-targeted mutagenesis of the ERF transcription factor gene., 2016, 11(4): e0154027.
[33] Zhang Y, Zhao J, Li Y, Yuan Z, He H, Yang H, Qu H, Ma C, Qu S. Transcriptome analysis highlights defense and signaling pathways mediated by ricegene with partial resistance to., 2016, 7: 1834.
[34] Wamaitha M J, Yamamoto R, Wong H L, Kawasaki T, Kawano Y, Shimamoto K.transcription factor contributes to rice innate immunity through its interaction with Receptor for Activated Kinase-C1(RACK1)., 2012, 5: 35.
[35] Zhao H, Wang X, Jia Y, Minkenberg B, Wheatley M, Fan J, Jia M H, Famoso A, Edwards J D, Wamishe Y, Valent B, Wang G, Yang Y. The rice blast resistance geneencodes an atypical protein required for broad-spectrum disease resistance., 2018, 9(1): 2039.
Orientation Improvement of Blast Resistance in Rice via CRISPR/Cas9 System
XU Peng, WANG Hong, TU Ranran, LIU Qunen, WU Weixun, FU Xiumin, CAO Liyong, SHEN Xihong*
(,;Corresponding author,:)
【】CRISPR/Cas9 gene editing is an effective biotechnology for crop genetic improvement. This study aims toeditandgenes forobtaining valuable and stable genetic materials with resistance to rice blast. 【】The target genesandwere selected for constructing the pC1300-2×35S::Cas9-g-g-gexpression vector by CRISPR/Cas9 gene editing technology, and transformed by-mediated method into long-grainricerestore lines L1014. Stable genetic mutant lines were selected and their resistance to rice blast was identified.【】 In the T0transgenic lines, the mutation rates ofandwere 75%, 85% and 65%, respectively, and most of the mutant lines are biallelic mutations.The homozygous mutation lines without T-DNA components within the screened T1were selected and transferred to T2stably. Evaluation analysis of resistance to rice blast suggested that the mutant lines showed significantly higher resistance tocompared with the wild type. The expression level of defense-related genes involved in signaling pathways of salicylic acid, jasmonic acid and ethylene metabolisms was up-regulated in the mutant lines compared to the wild type after inoculation of the physiological races of. Therefore, we hypothesized that the increased resistance of homozygous mutant lines to rice blast might be related to the activation of the response to. 【】The homozygous mutant lines with stable inheritance and high blast resistance were obtained by CRISPR/Cas9 system, which would provide ideal materials for improving blast resistance in rice.
rice; CRISPR/Cas9;blast resistance
Q755; S435.111.4+1; S511.034
A
1001-7216(2019)04-0313-10
10.16819/j.1001-7216.2019.9043
2019-04-10;
2019-05-24。
中国农业科学院创新工程超级稻育种创新团队和水稻杂种优势研究机理研究创新团队资助项目(CAAS-ASTIP-2013-CNRRI);转基因重大专项(2016ZX08001-002)。
