甘蓝BoPINs家族基因的特征和表达分析
2019-07-23王玉奎张贺翠白晓璟廉小平施松梅刘倩莹左同鸿朱利泉
王玉奎 张贺翠 白晓璟 廉小平 施松梅 刘倩莹 左同鸿 朱利泉,*
甘蓝家族基因的特征和表达分析
王玉奎1,**张贺翠1,**白晓璟1廉小平2施松梅2刘倩莹1左同鸿1朱利泉1,*
1西南大学农学与生物科技学院, 重庆 400715;2西南大学园艺园林学院, 重庆 400100
为了探索植物生长素极性运输载体蛋白编码基因家族参与甘蓝自交不亲和性的成员数目与参与方式, 本文通过转录组分析获得家族在甘蓝自花和异花授粉后的表达情况, 利用分子生物学技术和生物信息学方法对该家族的基因结构、蛋白进化亲缘关系和表达模式等特征进行分析。结果表明, 甘蓝基因家族包含8个成员, 含有5~9个外显子和4~8个内含子; 其编码的蛋白质的氨基酸残基数在350~650之间, 相对分子质量为38~70 kD; 除了BoPIN5和BoPIN8不含中间亲水区以外, 其余6个BoPINs家族成员都含有位于两端的疏水区和中间亲水环, 它们可能以膜锚定蛋白的形式发挥作用; 甘蓝与芜菁、拟南芥基因家族亲缘关系较近; 染色体定位分析表明,、和与S位点之间发生不同程度的连锁; 启动子活性分析表明, BoPINs家族蛋白参与甘蓝SI反应, 可能受IAA、ABA等激素相互交叉影响;、、、、、、、和在柱头中表达量均较高; 数据表达谱和荧光定量分析表明, 8个家族成员中的6个基因在自花授粉后下调表达; 自花授粉后柱头生长素含量降低, 与SI反应呈负相关。因此, 在甘蓝家族的8个成员中有6个基因家族成员可能在膜上以负调节方式调控自交不亲和反应。
甘蓝; 生长素; 自花授粉;家族; 自交不亲和
生长素是植物生长发育的重要调节剂, 在植物的胚胎发育、顶端分生组织和微管组织分化等过程中扮演重要角色, 尤其是在调控雌蕊发育、花粉萌发和花粉管生长方面发挥重要作用[1-2]。生长素在细胞之间呈不对称分布-即存在生长素浓度梯度, 这种分布模式是生长素合成代谢及极性运输的共同结果。主要以特定质膜结合极性运输载体介导生长素在细胞间移动实现在组织中的不对称分布[3]。外源生长素常常抑制花器官的发育, Okada等[4]研究发现生长素极性运输抑制剂N-1-氨甲酰苯甲酸萘酯(N-1- naphthylphthalamic acid, NPA)处理会导致拟南芥原基分化和发育异常, 输出载体PIN1(pin-formed 1)的弱突变体花发育异常、花柱缺失、雌蕊内没有胚珠, 而强突变体将不能起始花发育, 形成针状花序。花原基形成后, 生长素在花的发育和性别方面担负着重要角色, 直接或间接地参与花粉与雌蕊之间的相互作用[5-7]。有研究表明, 通过改变可可树中生长素浓度梯度可能会影响或控制自交不亲和性(self-incompatibility)[8]。生长素反应因子(auxin response factor, ARF3)以非细胞自主调节方式抑制生长素应答报告基因表达, 从而促进十字花科植物自花花粉的排斥反应[9]。这是自交不亲和途径与生长素信号传导途径之间存在交叉的重大突破。迄今为止, 对于生长素相关基因是否参与甘蓝自交不亲和途径鲜有报道。PINs家族作为生长素极性运输载体调节生长素分布的重要家族蛋白, 通过调节生长素的时空分布影响器官发生、胚胎发育、根系发育和花系统形成[10]。拟南芥PINs家族蛋白的功能、定位以及基因缺失导致的生理表型除了PIN6外均已报道[11]。但该家族是否可能通过改变植物体内的生长素浓度梯度和生长素极性分布来影响自交不亲和反应目前还不是十分清楚。本研究利用转录组测序技术分析甘蓝亲和授粉和不亲和授粉后得到的家族基因的表达情况, 以确定该家族中某些生长素极性运输载体基因是否可能参与自交不亲和反应; 利用模式植物中功能已知的拟南芥AtPINs蛋白家族为参考序列, 对BoPIN蛋白家族的基因进行结构分析、进化分析、组织特异性表达分析和功能分析, 以期进一步明确家族基因的特征及其与自交不亲和性之间的可能关系。
1 材料与方法
1.1 试验材料及处理
甘蓝高度自交不亲和系A4和F1植株由西南大学十字花科研究所提供, 于2017年3月下旬在开花前1~2 d对长势一致的A4植株人工去雄, 然后分别取A4和F1植株的花粉对A4植株的柱头人工授粉, 称为自花授粉(self-pollnation, SI)和异花授粉(cross-pollnation, CP)。授粉时间为0、15、30和60 min。授粉结束后快速扫去柱头上的花粉, 立即置液氮中保存。分别提取上述处理的甘蓝柱头RNA, 并送北京百迈克生物科技有限公司进行转录组测序。
1.2 甘蓝BoPIN家族基因序列的收集
通过拟南芥数据库中获得拟南芥AtPIN家族蛋白的氨基酸序列, 进行BLASTp检索; 同时与转录组数据所获得CDS序列比对, 获取甘蓝家族转录组号。
1.3 数字表达谱测序分析
基于转录组数据中家族的FPKM (Fragments Per Kilobase of transcript per Million fragments mapped)值结果, 采用Multiple Array Viewer软件绘制热图, 进行表达聚类分析。
1.4 甘蓝BoPIN家族成员的序列分析、在染色体上的分布分析和系统进化树构建
用Editseq软件进行ORF查询, 利用ExPASy (http://Hwww.expasy.org/tools)中的proPram (http://web. expasy.org/protparam/)分析BoPINs蛋白的理化性质。使用WOLF PSORT (http://www.genscript.com/wolf-psort.html)分析亚细胞定位。采用Mapchart分析BoPINs家族成员在染色体上的分布。利用GSDS在线软件(http://gsds1.cbi. pku.edu.cn/)分析基因结构。利用Smart (http://smart.embl- heidelberg.de/)和NCBI Conserved Domain Search对BoPINs家族成员进行蛋白结构域预测和保守序列分析。利用MEGA6.0软件选用邻接法(Neighbor-Joining, NJ)进行系统进化分析, 校验参数为Bootstrap=1000。
1.5 差异表达基因的荧光定量PCR检测
按照THUNDERBIRD SYBR Green Realtime PCR Master Mix试剂盒说明书, 利用Primer Premier 6.0软件设计家族荧光定量PCR引物(附表)。反应体系包含1 μL浓度为100 ng μL-1cDNA模板、SYBR Green Realtime PCR Master Mix 10 μL、上下游引物各0.5 μL和无RNA酶水8 μL。qPCR程序为95℃预变性5 min; 95℃变性30 s, 58℃退火30 s, 72 ℃延伸30 s, 共40个循环, 以上反应每个样品3次平行重复。选取作为内参基因, 按照2–ΔΔCT法计算家族的相对表达量[12]。
1.6 BoPINs家族启动子活性分析
从NCBI甘蓝基因组中下载基因转录起始位置上游1500 bp的序列, 并利用PlantCARE数据库(http:// bioinformatics.psb.ugent.be/webtools/plantcare/html/)进行顺式作用元件分析。
1.7 BoPINs家族的组织特异性表达分析
对自花授粉后的柱头、花瓣、萼片、叶片、花蕾和花药各个组织进行荧光定量PCR检测, 分析在家族不同组织中的表达模式。利用热图软件进行表达聚类分析。
2 结果与分析
2.1 BoPINs家族的基本特征分析
通过拟南芥数据库家族的氨基酸序列进行BLASTp比对(取E-value≤1×10–20), 结合甘蓝基因组数据库序列, 从甘蓝基因组数据库中共鉴定出8个家族基因[13-15]。根据它们的染色体位置, 将、和的同源拷贝依次命名为、、、、和。其编码多肽链的氨基酸残基数在350~650之间, 相对分子质量为38~70 kD。编码氨基酸链最长的是BoPIN2, 其相对分子质量为70.46 kD; 编码氨基酸链最短的是BoPIN8, 其相对分子质量为38.22 kD。BoPINs蛋白的理论等电点(pI)在6~9之间。不稳定系数分析发现, 大部分BoPINs蛋白为稳定蛋白(不稳定系数小于40), 只有BoPIN1-1和BoPIN5属于不稳定蛋白。根据亲水性指数(GRAVY为负值表示亲水性, 正值表示疏水性, 介于-0.5 ~ +0.5的为两性蛋白)的原则, 发现BoPIN5和BoPIN8蛋白为疏水性蛋白, 其余BoPIN家族蛋白均为两性蛋白[16]。亚细胞定位预测分析表明,BoPIN1-1、BoPIN1-2、BoPIN2、BoPIN3、BoPIN4、BoPIN7-1和BoPIN7-2定位于细胞膜; BoPIN5、BoPIN6和BoPIN8定位于内质网[17-18](表1)。SignalP在线工具分析显示, BoPINs家族蛋白都不存在信号肽, 均为非分泌型蛋白。
2.2 甘蓝BoPINs的基因结构和蛋白系统进化分析
根据转录组数据中的CDS序列, 结合NCBI数据库中的甘蓝基因组信息, 序列比对分析甘蓝家族成员的基因结构特征。由图1可知,、、和均具有6个外显子和5个内含子;具有9个外显子和8个内含子;具有7个外显子和6个内含子;具有5个外显子和4个内含子; 因此甘蓝家族成员的基因均含有数目不等的外显子和内含子, 且数目差别明显, 甘蓝BoPINs蛋白结构特征存在于其外显子编码的氨基酸序列变异中。以系数0.54为标准, BoPIN1-1、BoPIN3-1、BoPIN3-2、BoPIN4、BoPIN5、BoPIN7-1和BoPIN7-2的进化关系相近, BoPIN5和BoPIN8进化关系相近。
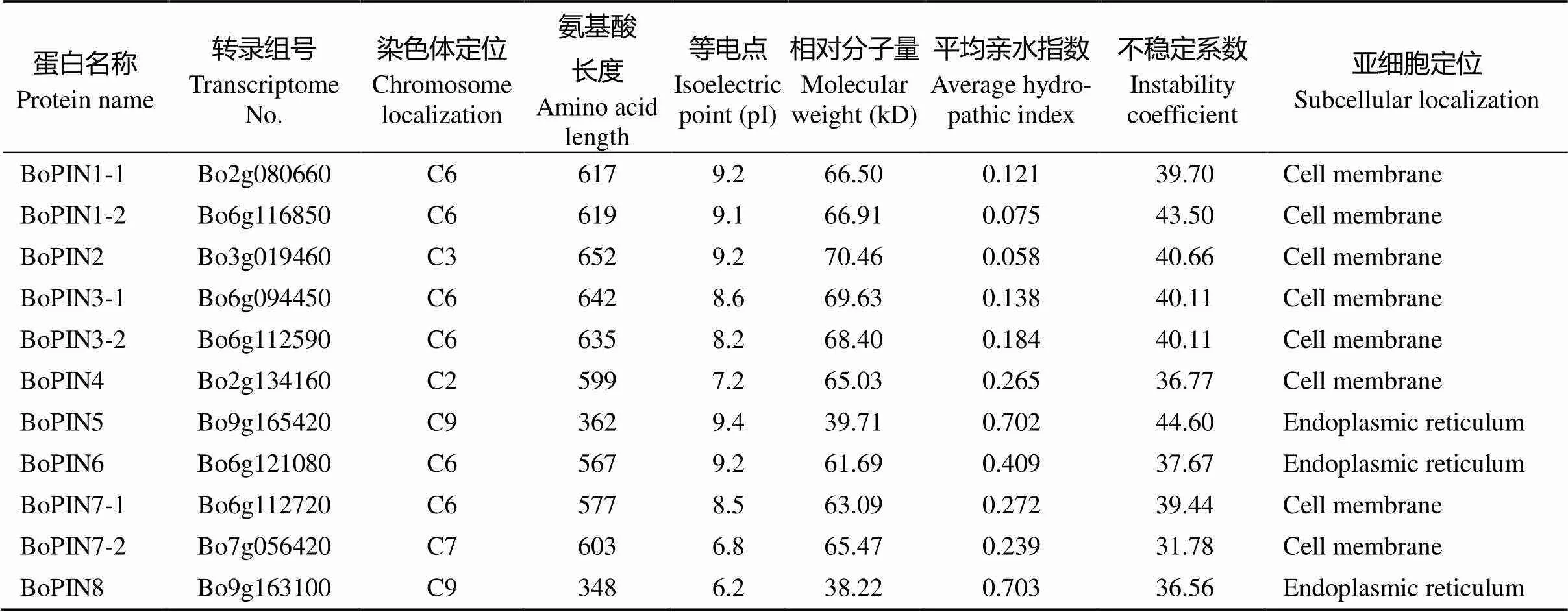
表1 甘蓝BoPINs家族蛋白信息
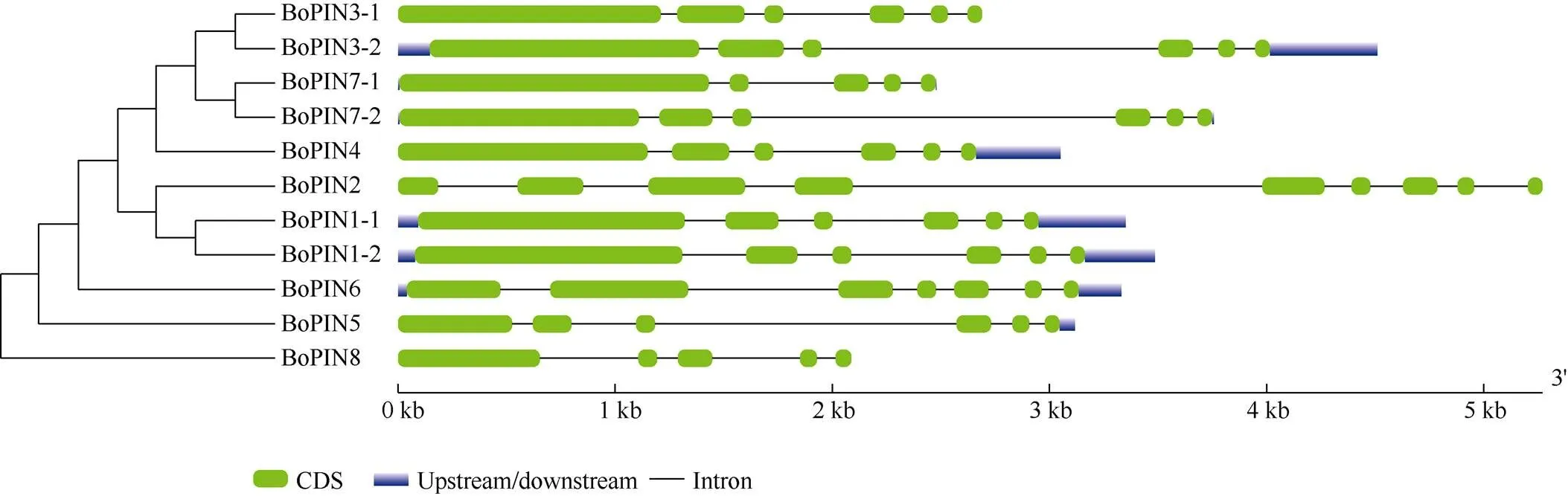
图1 甘蓝BoPINs的进化树和基因结构图
左侧为进化树, 用MEGA6.0软件构建; 右侧为甘蓝家族的基因结构图。
The phylogenetic tree is constructed with MEGA6.0 software on the left and the gene structure map of thefamily on the right.
为了更好理解不同植物PIN蛋白之间的进化关系, 利用MEGA6.0软件对十字花科植物(甘蓝、拟南芥、芜菁、萝卜)、单子叶植物(玉米、水稻) 6种植物共55个PIN蛋白构建系统进化树。表明甘蓝BoPINs与芜菁BrPINs、拟南芥AtPINs和萝卜亲缘关系最近, 与玉米ZmPINs、水稻OsPINs亲缘关系较远。以水稻OsPIN9为外群, 按照亲缘关系远近的原则分为6组, 第I组是14个十字花科特异性组, BoPIN3和BoPIN7在进化过程中发生基因复制事件; 第II、III组均有单子叶植物和十字花科植物聚类在一起, 表明PIN1、PIN2在物种进化过程中未发生基因丢失事件; 第IV组包括4个十字花科物种, 表明PIN6只存在于十字花科植物中, 而不存在于以玉米、水稻为代表的单子叶植物中; 第V、VI组中AtPIN5和AtPIN8的直系同源物分别对应甘蓝BoPIN8和BoPIN5 (图2)。

图2 甘蓝家族BoPINs的系统进化树
2.3 甘蓝BoPINs家族基因成员的染色体定位
、、、、有1个拷贝;、和有2个同源拷贝,、、、、和分布在不同染色体上。从图3可以看出, 甘蓝自交不亲和关键元件S位点糖蛋白(S-locus glycoprotein, SLG)、S半胱氨酸富集蛋白(S-locus cystine-rich protein, SCR)以及S位点受体激酶(S-locus receptor kinase, SRK)和家族成员主要集中在第6染色体上, 芸薹属自交不亲和反应受多态性S位点复等位基因控制。在第6染色体上存在一个多态性S位点, 而该基因家族成员、、和都位于该多态性位点附近。按照1,000,000 nt为1 cM计算, 遗传图距分别是13.8、4.21、11.37和31.1 cM。这不同程度地连锁反映了该家族基因与S位点的遗传距离, 遗传距离近的可能存在顺式作用, 遗传距离远的可能存在反式作用。
2.4 BoPINs家族自花授粉和异花授粉后的授粉依赖性表达
从转录组数据得到基因家族在自花和异花授粉0、15、30和60 min的表达模式。将家族基因的FPKM值均一化, 采用Multiple Array Viewer 软件绘制热图(图4)。在自花授粉后下调, 在异花授粉后表达量变化不明显。在自花授粉15~60 min剧烈下调, 在60 min后表达量达到最低。在自花授粉0~60 min过程中下调, 而异花授粉后15~60 min表达量上调, 在异花授粉60 min时,达到了在未授粉时的表达量。、与表达模式一致。和在自花授粉0~60 min过程表达量下调, 而异花授粉后15~60 min表达量上调, 而后急剧下调。综上所述, 在自花授粉0~60 min过程中,、、、、和处于下调状态, 这与自交不亲和关键元件SRK (Bo6g101190)在自花授粉0~60 min的表达模式是一致的。

图3 BoPINs家族在甘蓝染色体的分布
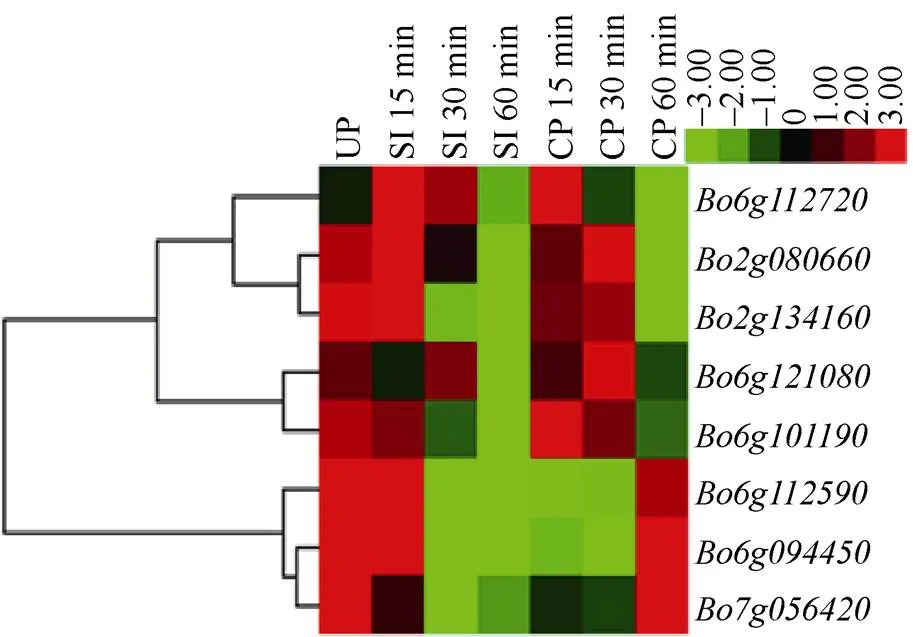
图4 BoPINs基因家族在自花授粉和异花授粉后的表达模式
为了进一步确定甘蓝家族在自花授粉和异花授粉处理后的表达模式, 利用荧光定量PCR对该基因家族进行表达分析。、、、、和在自花授粉后显著下调表达, 在60 min后达到最低, 而在异花授粉后表达差异不大。在自花授粉后下调表达, 在异花授粉后显著上调表达。在自花授粉后的表达模式与异花授粉后的表达模式呈现出相反的趋势。在自花授粉后显著上调表达, 在30 min达到最高而后急剧下调表达, 异花授粉后表达量差异不显著(图5), 说明、、、、、和的表达受到SI的抑制, 而基因的表达既受到SI抑制还受到CP促进,基因的表达受到与相反的调控,基因受SI抑制具有先期时限性。
2.5 BoPINs家族的启动子顺式作用元件分析
利用NCBI数据库选取转录起始位点上游1500 bp的序列, 结合PlantCARE在线软件分析该家族启动子的顺式作用元件研究是否受自花授粉显著诱导。由表2可知,包含光、ABA、IAA、水杨酸、脱落酸、赤霉素、胁迫响应相关的顺式作用元件。其中、、和含有生长素响应相关元件, 除了其余基因家族成员都含有胁迫响应相关元件。表明BoPINs家族蛋白可能受IAA、ABA等激素相互交叉影响参与甘蓝SI反应[19-20]。
2.6 BoPINs家族的组织特异性表达分析
为了探究在自花授粉后甘蓝基因家族在不同组织中的表达量, 利用qRT-PCR技术对甘蓝基因家族在花蕾、花药、萼片、花瓣、叶片和柱头6种组织进行特异性表达分析, 将荧光定量所得数据均一化进行数据表达谱分析。在萼片中表达量最高, 在花瓣中表达量最低;在花药中表达量最高, 在花瓣中表达量最低;在柱头的表达量最高, 在花粉和花瓣的表达量较低;和在柱头的表达量最高, 花粉次之, 在花瓣的表达量最低;在花蕾和萼片的表达量最高, 在柱头和花瓣的表达量最低。、、、、、、和在柱头的表达量高于其他组织(图6)。结合该家族在授粉后各个时期的表达模式表明该家族部分成员可能参与自交不亲和反应。
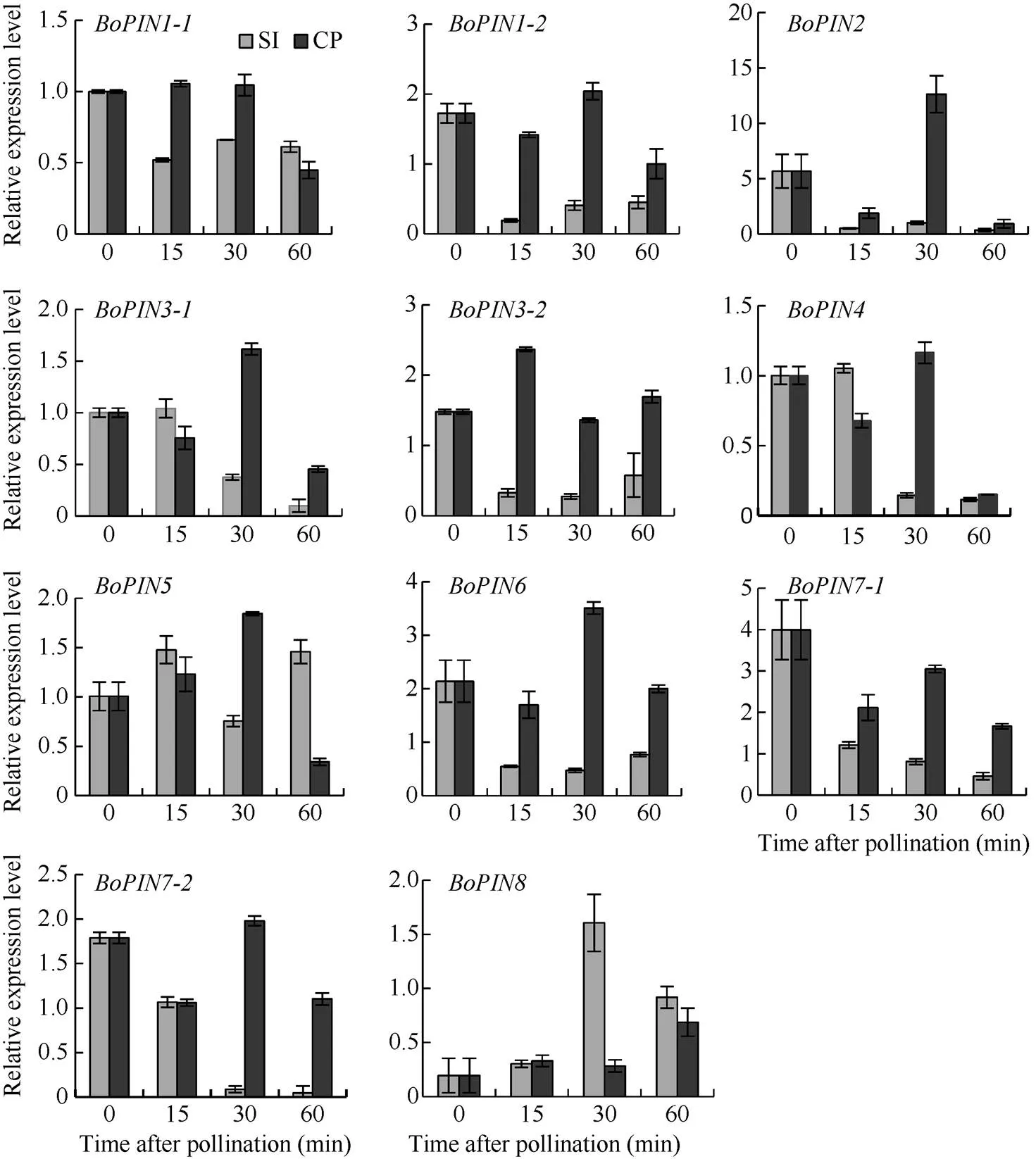
图5 甘蓝BoPINs家族在自花和异花授粉后的表达分析
SI: self-pollination; CP: cross-pollination.
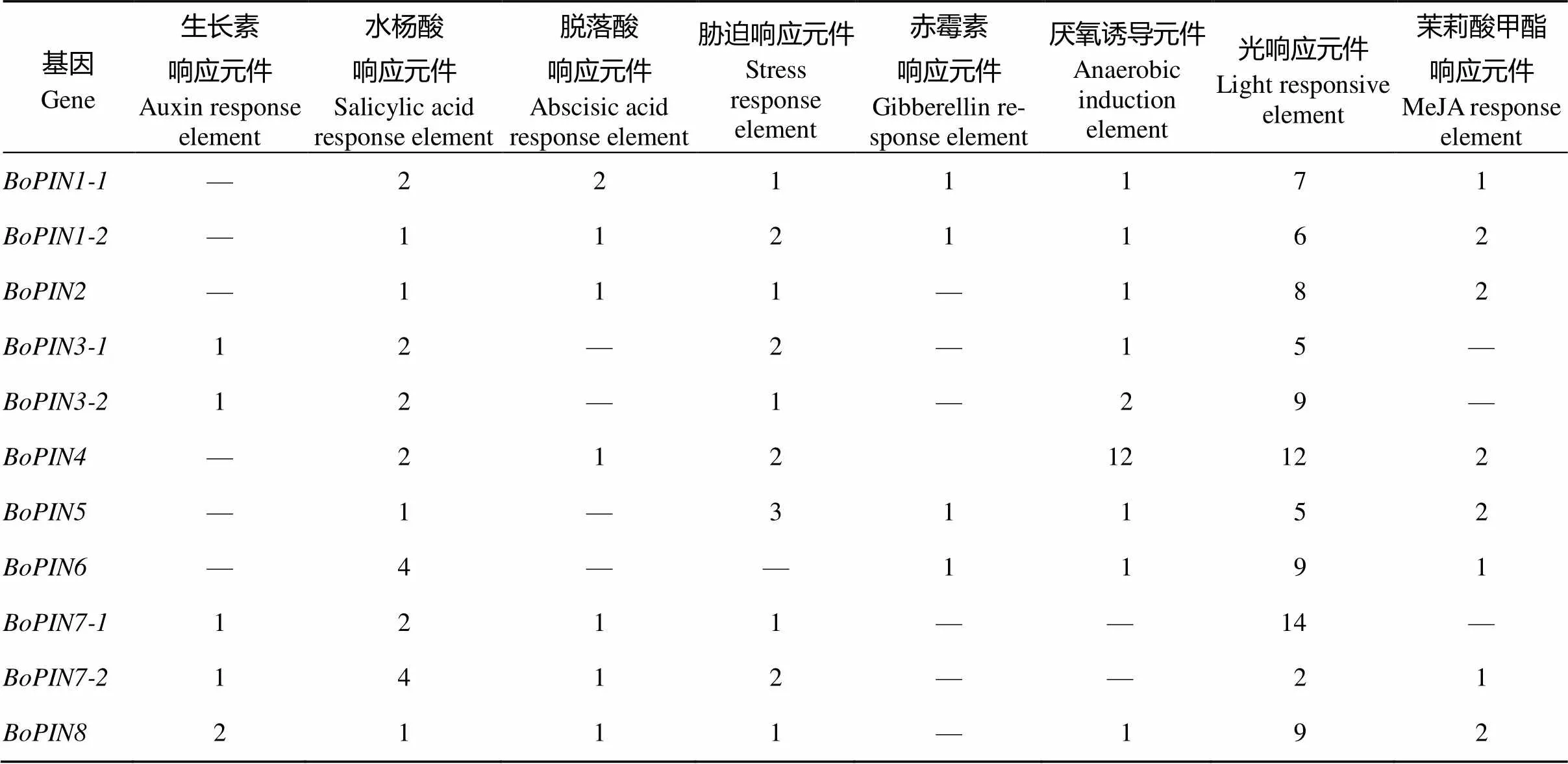
表2 BoPINs基因启动子的顺式作用元件

图6 BoPINs基因家族成员在自花授粉后不同组织中的表达量分析
2.7 甘蓝柱头授粉后生长素含量的变化
色氨酸氨基转移酶(Tryptophan aminotransferase, TAA1)通过吲哚丙酮酸途径(indole-3-pyruvate, IPA)参与生长素的合成, 它将Trp转化成IPA, 从而参与生长素生物合成。TAA1的mRNA的含量与生长素含量成正比关系[21-22]。本研究利用转录组数据库中的TAA1的mRNA来表征柱头授粉后的生长素含量变化。结果表明(图7), 在自花授粉后TAA1的mRNA含量一直降低, 异花授粉后mRNA含量变化不明显, 说明甘蓝柱头在自花授粉后生长素含量降低, 与SI反应呈负相关。
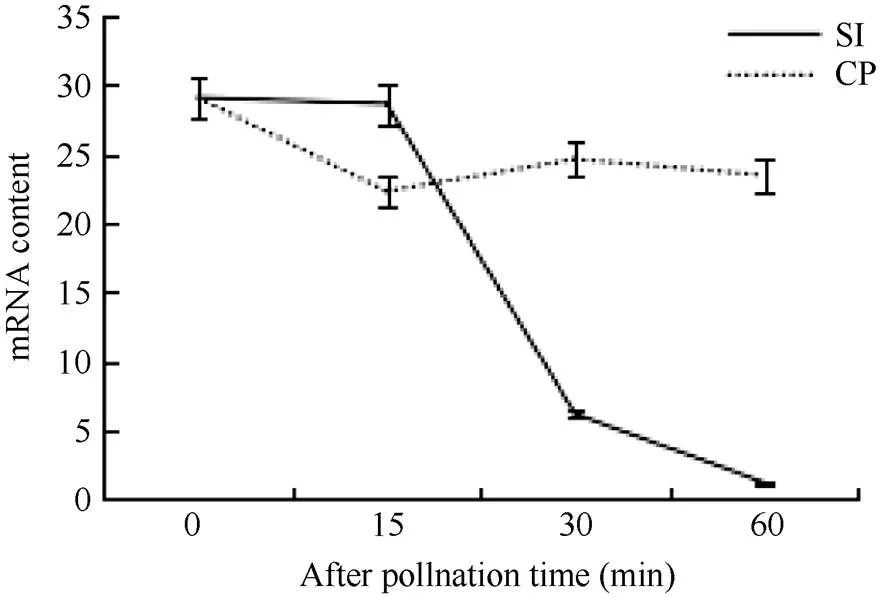
图7 柱头授粉后生长素生物合成关键酶TAA1的mRNA含量变化
3 讨论
本研究在自花授粉的转录组数据中首次获得甘蓝生长素输出载体基因家族8个成员。在其他植物中, 研究发现生长素在花粉发育、花粉管生长和花序维管组织发育等生物过程起着关键作用, 已有研究表明, 梨自花授粉后柱头内的IAA含量变化较显著, 在可可树自花授粉后生长素含量减少, 乙烯含量增加, 可可树自交不亲和性受生长素调节控制[23-24]。IAA在烟草自交不亲和的花粉与柱头的识别过程中出现了相似的显著变化[25], 在本文中首次发现,、、、、等成员的表达量与自交不亲和性之间存在负相关, 从表达量和功能研究两方面印证了柱头表面细胞内未知的生长素响应元件的抑制促进了柱头对自身花粉的抑制作用[26]。
生长素输出载体基因编码一类生长素极性运输跨膜蛋白, 它是细胞内IAA运输到胞外的关键载体因子。PINs家族作为生长素信号传导通路中主要的输出载体蛋白, 通过对生长素浓度梯度极性分布调节, 参与包括SI在内的诸多植物生理过程。本研究通过转录组分析, 获得了8个甘蓝的家族成员。除了BoPIN5和BoPIN8蛋白缺失了中间亲水区, 其他蛋白均含有两端疏水结合区和中间亲水环。进化分析表明, 除BoPIN5与AtPIN8亲缘关系最近、BoPIN8和AtPIN5亲缘关系最为密切之外, 其他BoPINs家族都与AtPINs家族同属同一进化分支, 也存在较近的亲缘关系。启动子活性分析表明基因家族受多种植物激素相互交叉影响参与自交不亲和反应。染色体定位分析表明,主要定位于第2、第4、第6和第9染色体上, 其中、和定位于第6染色体上, 而自交不亲和关键元件、、均位于第6染色体上, 与S位点基因存在紧密连锁性。可能参与自花授粉响应, 调节柱头乳突细胞的生长素浓度梯度和花器官内生长素的时空分布。PINs家族蛋白在自花授粉后下调表达可能是胞内生长素的浓度反馈调节基因表达引起[27]。生长素对基因表达调节可能与(plethora)基因有关, PLT蛋白是一类含有AP2结构域的转录因子, 是根尖干细胞发生、发育主要决定因子。在双突变体中, 根尖干细胞缺失, 根系发育异常, PIN1、PIN4和PIN7的蛋白表达量明显降低。这种反馈模式对于维持植物细胞生长素梯度平衡至关重要[28]。PIN蛋白表达量受诸多因素影响, 如生长素的反馈调节、生长素自身分布和生长素信号[29-30]。PIN蛋白作为ARF和Aux/IAA蛋白下游信号元件, ARF直接或间接地促进PIN蛋白的表达, Aux/IAA蛋白通过泛素蛋白降解途径抑制PINs蛋白表达[31]。有研究表明ARF3 (ETTIN)和ARF5 (MONOPTEOS) 可以直接靶向激活、和的表达, 特别是ARF3作为增强SI反应的雌蕊发育调节因子,、和可能作为靶向基因下调生长素反应促进自花花粉的抑制[32-33]。PIN蛋白的极性定位与囊泡运输密切相关, 当用囊泡运输抑制剂BFA (brefeldin)处理时, 膜定位的PIN1、PIN3等可逆地在胞质内聚集, 用生长素抑制剂(NPA)处理会导致PIN1的持续内在化[34]。拟南芥GNOM/EMB30缺失突变体中定位异常, 表现出极性运输缺失相似的表型, 例如胚胎发育异常、器官融合等。GNOM介导下游的SNX1 (sorting nexin 1)调节VPS29 (vaculolar protein sorting 29)蛋白, 从而调节PIN1在胞内的极性定位, 影响生长素的极性运输[35-36]。通过酵母双杂技术筛选出SNX1能与SRK发生相互作用, 进一步研究证明了PIN1可能作为自交不亲和关键元件SRK的下游信号元件抑制生长素反应增强SI[37-38]。PID (PINOID)过表达植株中PIN蛋白极性定位发生变化, 植物体内生长素浓度梯度紊乱, 胚胎和幼苗根发育严重缺陷, 与磷酸酶PP2A (protein phosphatase 2A)功能缺失突变体表型类似。PID蛋白激酶与磷酸酶PP2A以相反的方式调节PIN蛋白的磷酸化与去磷酸化, 改变PIN亲水环磷酸化位点的氨基酸序列, 可以破坏PIN蛋白的极性定位, 从而影响生长素的极性运输[39]。总而言之, 植物体内生长素极性运输的调节可通过调控PIN蛋白的量或定位实现对生长素浓度分布的精细调控。
、、、、、、、和在自花授粉后柱头中表达量较高, 且在自花授粉后显著下调表达, 表明在自花授粉后未知生长素信号反应蛋白反馈抑制家族极性运输生长素, 导致柱头乳突细胞内生长素含量降低, 生长素含量与自交不亲和性呈负相关, 最终激活SRK下游未知信号通路导致自交不亲和反应。
[1] Vieten A, Sauer M, Brewer P B, Friml J. Molecular and cellular aspects of auxin-transport-mediated development., 2007, 12: 160–168.
[2] Petrášek J, Friml J. Auxin transport routes in plant development., 2009, 136: 2675–2688.
[3] Reinhardt D, Pesce E R, Stieger P, Mandel T, Baltensperger K, Bennett M, Traas J, Friml J. Regulation of phyllotaxis by polar auxin transport., 2003, 426: 255–260.
[4] Okada K, Ueda J, Komaki M K, Bell C J, Shimura Y. Requirement of the auxin polar transport system in early stages of Arabidopsis floral bud formation., 1991, 3: 677–684.
[5] Hall I V, Forsyth F R. Production of ethylene by flowers following pollination and treatments with water and auxin., 1967, 45: 1163–1166.
[6] Safavian D, Zayed Y, Indriolo E, Chapman L, Ahmed A, Goring D R. RNA silencing of exocyst genes in the stigma impairs the acceptance of compatible pollen in Arabidopsis., 2015, 169: 2526–2538.
[7] Zhang C, Li G, Chen T, Feng B, Fu W, Yan J, Islam M R. Heat stress induces spikelet sterility in rice at anthesis through inhibition of pollen tube elongation interfering with auxin homeostasis in pollinated pistils., 2018, 11: 14, doi: 10.1186/s12284- 018-0206-5.
[8] Hasenstein K H, Zavada M S. Auxin modification of the incompatibility response in., 2001, 112: 113–118.
[9] Tantikanjana T, Nasrallah J B. Non-cell-autonomous regulation of crucifer self-incompatibility by Auxin Response Factor ARF3., 2012, 109: 19468–19473.
[10] Vanneste S, Friml J. Auxin: a trigger for change in plant development., 2009, 136: 1005–1016.
[11] Benkova E, Michniewicz M, Sauer M, Teichmann T, Seifertova D, Jurgens G, Friml J. Local, efflux-dependent auxin gradients as a common module for plant organ formation., 2003, 115: 591–602.
[12] Livak K J, Schmittgen T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2−ΔΔCTmethod., 2001, 25: 402–408.
[13] 王占军, 杨立伟, 徐忠东, 欧祖蓝, 袁华玲, 任意飞. 麻疯树基因家族的鉴定与生物信息学分析. 分子植物育种, 2015, 13: 1111–1122. Wang Z J, Yang L W, Xu Z D, Ou Z L, Yuan H L, Ren Y F. Identification and bioinformatics analysis of thegene family of., 2015, 13: 1111–1122 (in Chinese with English abstract).
[14] Liu Y, Wei H. Genome-wide identification and evolution of the PIN-FORMED (PIN) gene family in., 2017, 60: 564–571.
[15] Křeček P, Skůpa P, Libus J, Naramoto S, Tejos R, Friml J. The PIN-FORMED (PIN) protein family of auxin transporters., 2009, 10: 249, doi: 10.1186/gb-2009-10-12-249.
[16] Bendtsen J D, Jensen L J, Blom N, Heijne G, Brunak S. Feature-based prediction of non-classical and leaderless protein secretion., 2004, 17: 349–356.
[17] Mravec J, Skůpa P, Bailly A, Hoyerova K, Krecek P, Bielach A, Petrasek J, Zhang J, Gaykova V, Stierhof Y D, Rolcik J, Stierhof D, Luschnig C,Benkova E, Zazimalova E, Geisler M, Friml J. Subcellular homeostasis of phytohormone auxin is mediated by the ER-localized PIN5 transporter., 2009, 459: 1136–1140.
[18] Friml J. Subcellular trafficking of PIN auxin efflux carriers in auxin transport., 2010, 89: 231–235.
[19] Baker R P, Hasenstein K H, Zavada M S. Hormonal changes after compatible and incompatible pollination inL., 1997, 32: 1231–1234.
[20] Ono K, Morimoto T, Akagi T, Wunsch A, Tao R. Genome re-sequencing of diverse sweet cherry () individuals reveals a modifier gene mutation conferring pollen-part self- compatibility., 2018, 59: 1265–1275.
[21] Zhou Z Y, Zhang C G, Wu L, Zhang C G, Chai J, Wang M, Jha A, Jia P F, Cui S J, Yang M, Chen R. Functional characterization of thegene and dissection of hormonal actions in the Arabidopsis root., 2011, 66: 516–527.
[22] Kakei Y, Nakamura A, Yamamoto M, Ishida Y, Yamazaki C, Sato A, Nara M N, Soeno K, Shimada Y. Biochemical and chemical biology study of rice OsTAR1 revealed that tryptophan aminotransferase is involved in auxin biosynthesis: identification of a potent OsTAR1 inhibitor, pyruvamine 2031., 2017, 58: 598–606.
[23] Cazzonelli C I, Vanstraelen M, Simon S, Yin K, Arthur A, Nisar N, Tarle G, Cuttriss A J, Searle L R, Mathesius U, Masle J, Friml J, Pogson B J. Role of the Arabidopsis PIN6 auxin transporter in auxin homeostasis and auxin-mediated development., 2013, 8: e70069.
[24] 齐国辉, 徐继忠, 张玉星. 鸭梨自交不亲和性与花柱内源激素关系的研究. 河北农业大学学报, 2007, 30(1): 31–34. Qi G H, Xu J Z, Zhang Y X. Study on the relationship between self-incompatibility and the endogenous hormones in style of Yali., 2007, 30(1): 31–34 (in Chinese with English abstract).
[25] Bavrina T V, Milyaeva E L, Machácčková I, Pustovoitova T N, Gurko N A, Kasumova I V, Zhdanova N E. Effect of phytohormone biosynthesis genes (ipt and iaaM+ iaaH) on the sexual reproduction of transgenic tobacco plants., 2002, 49: 484–491.
[26] Chen D, Zhao J. Free IAA in stigmas and styles during pollen germination and pollen tube growth of., 2008, 134: 202–215.
[27] Vieten A, Vanneste S, Wiśniewska J, Benkova E, Benjamins R, Beeckman T, Luschnig C, Friml J. Functional redundancy of PIN proteins is accompanied by auxin-dependent cross-regulation of PIN expression., 2005, 132: 4521–4531.
[28] Blilou I, Xu J, Wildwater M, Willemsen V, Paponov I, Friml J, Heidstra R, Aida M, Palme K, Scheres B. The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots., 2005, 433: 39–44.
[29] Aida M, Beis D, Heidstra R, Willemsen V, Blilou I, Galinha C, Nussaume L, Noh Y S, Amasino R, Scheres B. Thegenes mediate patterning of the Arabidopsis root stem cell niche., 2004, 119: 109–120.
[30] Geisler M, Blakeslee J J, Bouchard R, Lee O R, Vincenzetti V, Bandyopadhyay A, Titapiwatanakun B, Peer W A, Bailly A, Richard E L, Ejendal K F K, Smith A P, Baroux C, Grossniklaus U, Muller A, Hrycyna C A, Dudler R, Murphy A S, Murphy A S. Cellular efflux of auxin catalyzed by the Arabidopsis MDR/PGP transporter AtPGP1., 2005, 44: 179–194.
[31] Petrasek J, Mravec J, Bouchard R, Blakeslee J J, Abas M, Seifertova D, Wisniewska J, Tadele Z, Kubes M, Covanova M, Dhonukshe P, Skupa P, Benkova E, Perry L, Krecek P, Lee O R, Fink G R, Geisler M, Murphy A S, Luschnig C, Zazimalova E, Friml J. PIN proteins perform a rate-limiting function in cellular auxin efflux., 2006, 312: 914–918.
[32] Lavenus J, Guyomarc’h S, Laplaze L. PIN transcriptional regulation shapes root system architecture., 2016, 21: 175–177.
[33] Simonini S, Bencivenga S, Trick M. Auxin-induced modulation of ETTIN activity orchestrates gene expression in Arabidopsis., 2017, 29: 1864–1882.
[34] Geldner N, Friml J, Stierhof Y D, Jurgens G, Palme K. Auxin transport inhibitors block PIN1 cycling and vesicle trafficking., 2001, 413: 425–428.
[35] Geldner N, Anders N, Wolters H, Keicher J, Kornberger W, Muller P, Delbarre A, Ueda T, Nakano A, Jurgens G. The Arabidopsis GNOM ARF-GEF mediates endosomal recycling, auxin transport, and auxin-dependent plant growth., 2003, 112: 219–230.
[36] Jaillais Y, Fobis-Loisy I, Miege C, Rollin C, Gaude T. AtSNX1 defines an endosome for auxin-carrier trafficking in Arabidopsis., 2006, 443: 106–109.
[37] Vanoosthuyse V, Tichtinsky G, Dumas C, Gaude T, Cock J M. Interaction of calmodulin, a sorting nexin and kinase-associated protein phosphatase with thelocus receptor kinase., 2003, 133: 919–929.
[38] Roux M, Zipfel C. Receptor kinase interactions: complexity of signaling. In: Receptor-like Kinases in Plants.Springer, 2012. pp 145–172.
[39] Michniewicz M, Zago M K, Abas L, Weijers D, Schweighofer A, Meskiene M G, Ohno C, Zhang J, Huang F, Schwab R, Weigel D, Meyerowitz E M, Luschnig C, Offringa R, Friml J. Antagonistic regulation of PIN phosphorylation by PP2A and PINOID directs auxin flux., 2007, 130: 1044–1056.
Characteristics and expression analysis offamily genes in
WANG Yu-Kui1,**, ZHANG He-Cui1,**, BAI Xiao-Jing1, LIAN Xiao-Ping2, SHI Song-Mei2, LIU Qian-Ying1, ZUO Tong-Hong1, and ZHU Li-Quan1,*
1College of Agriculture and Biotechnology, Southwest University, Chongqing 400715, China;2College of Horticulture and Gardening, Southwest University, Chongqing 400100, China
In order to explore the number and expression of thegene family participating in self-incompatibilty of, their expression after self-pollination and cross-pollination were detected by transcriptome analysis, and the corresponding gene structure, phylogenetic tree and expression patterns of the family were further analyzed by bioinformatics. This gene family contained 5-9 exons and 4-8 introns. The amino acid of the encoding protein residues were between 350 and 650 and had molecular weights ranging from 38 kD to 70 kD. Except that BoPIN5 and BoPIN8 did not contain internal hydrophilic cytoplasmic regions, the remaining six BoPINs proteins contained a hydrophobic region at both ends and an internal hydrophilic ring, showing they located on membrane. The evolutionary analysis indicated thatwere closely related to theand thegene family. Chromosome localization analysis indicated that,,, andmembers of the family were linked to S-loucs to different degrees. Tissue-specific expression analysis indicated that,,,,,,,, andhad higher expression levels in the stigma. Data expression profiling and fluorescence quantitative analysis indicated that six of the eightgenes were down-regulated after self-pollination. All these results indicate that six members of the eightgene family members on the membrane may participate in the self-incompatibility response ofin a negative regulatory manner.
; auxin; self-pollination; BoPINs family; self-incompatibility
2018-10-15;
2019-04-15;
2019-04-26.
10.3724/SP.J.1006.2019.84129
朱利泉, E-mail: zhuliquan@swu.edu.cn, Tel: 023-68250794
**同等贡献(Contributed equally to this work)
E-mail: wangyuk0808@163.com
本研究由国家自然科学基金项目(31572127)和中央高校基本科研业务费(XDJK2017C023)项目资助。
This study was supported by the National Natural Science Foundation of China (31572127) and the Basic Research Fund of the Central University (XDJK2017C023).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20190426.0914.002.html
