花生籽仁大小相关性状QTL定位
2019-07-23曾新颖郭建斌赵姣姣陈伟刚邱西克罗怀勇周晓静姜慧芳黄家权
曾新颖 郭建斌 赵姣姣 陈伟刚 邱西克 黄 莉 罗怀勇 周晓静 姜慧芳,* 黄家权
花生籽仁大小相关性状QTL定位
曾新颖1,2,**郭建斌2,**赵姣姣2陈伟刚2邱西克2黄 莉2罗怀勇2周晓静2姜慧芳2,*黄家权1,*
1海南大学热带农林学院 / 海南省热带资源可持续利用重点实验室, 海南海口 571003;2中国农业科学院油料作物研究所/ 农业农村部油料作物生物学与遗传育种重点实验室, 湖北武汉 430062
花生籽仁大小相关性状是决定花生产量的直接因素。为发掘与花生籽仁大小相关的QTL, 本研究以中花16×J11构建的RIL群体为材料, 得到了一张包含289个SSR标记、21个连锁群、覆盖长度为947.3 cM的遗传连锁图谱。连续2年对籽仁大小相关性状鉴定表明, 各性状在群体中变异广泛, 呈典型正态分布, 且大部分性状间显著相关。结合本研究构建的遗传图谱, 利用WinCart2.5进行QTL定位分析, 2年共检测到66个QTL, 贡献率为3.23%~ 33.01%。与籽仁长(SL)、籽仁宽(SW)、籽仁长宽比(LWR)和百仁重(HSW)相关的QTL分别有18、16、18和14个。在这些QTL中, A05染色体上的区间A05A1500−A05A1530同时存在控制籽仁长(和)和百仁重的相关的QTL (); B06染色体上的区间A06B135−A06B113同时存在控制籽仁宽(和)和百仁重相关的QTL (), 这些稳定存在的主效QTL将为花生产量相关性状的精细定位和分子育种奠定基础。
花生; 遗传图谱; 籽仁大小; QTL
花生作为重要的油料作物之一, 高产一直是其主要育种目标之一, 其中, 花生籽仁大小是决定花生产量的直接因素[1-2]。研究花生籽仁大小相关的QTL, 选择具有不同QTL的亲本杂交, 对于提高花生的籽仁产量有重要意义[3]。
近年来, 有关花生重要农艺性状QTL定位的研究已取得了进展。Wilson等[4]利用野生花生和栽培种花生的回交群体在2个以上环境下检测到17个与花生脂肪酸含量相关的QTL。Wang等[5]在2个RIL群体中共检测到控制脂肪酸组分的164个主效QTL以及27个上位性QTL, 贡献率为0.16%~40.56%。Khedikar等[6]在含有268个株系的群体中鉴定到11个与花生抗晚叶斑病(LLS)相关QTL和12个与抗锈病相关QTL。Pandey等[7]检测到9个抗早叶斑病(ELS)、22个抗晚叶斑病和11个抗番茄斑委病毒(TSWV)QTL, 贡献率为6.36%~15.60%。在产量相关性状方面, Luo等[8]利用195份材料的RIL群体在4年环境下共检测到与花生出仁率相关的25个QTL; Chen等[9]鉴定了92个与产量性状相关的QTL; 此外, Shirasawa等[10]鉴定了7个与荚果相关性状相关的QTL; Ravi等[11]报道了4个控制荚果重的主效QTL和2个控制籽仁重的主效QTL。目前关于花生QTL的研究主要集中在抗病和品质性状方面, 而关于花生产量相关性状QTL定位方面的研究相对较少。本研究选取籽仁大小差异显著的中花16和J11为父母本杂交构建的RIL群体, 鉴定籽仁大小相关性状的QTL, 为花生产量性状的进一步提高奠定基础。
1 材料与方法
1.1 试验材料
以“中花16×J11”构建的重组自交系群体(RIL)F5、F6共188个家系为材料, 其中中花16籽粒较大, J11籽粒较小。
1.2 试验方法
2017年和2018年在中国农业科学院油料作物研究所试验农场种植RIL群体F5、F6代材料及其亲本常规田间管理。收获晒干后剥壳, 随机选取30粒成熟饱满的种子, 采用万深SC-G考种仪测量种子长、宽及百粒重, 重复3次, 计算平均值。
1.3 SSR标记鉴定
取亲本和RIL群体幼嫩叶片, 采用改良CTAB法提取其DNA样本[12]。利用亲本间有差异的SSR引物, 扩增RIL群体DNA, 对扩增产物进行聚丙烯酰胺凝胶电泳, 其中PCR体系为10 μL, 反应程序为Touchdown; 电泳产物经银染显色。与母本、父本相同的基因型条带分别以“A”、“B”表示, 杂合条带以“H”表示, 缺失记为“-”。
1.4 图谱构建及QTL鉴定
根据RIL群体基因型数据, 运用JoinMap 4.0软件构建遗传连锁图谱。以2017和2018两年所得的群体表型数据, 包括花生的籽仁长、籽仁宽、长宽比以及百仁重, 采用WinCart2.5进行相关性状的QTL扫描, MapChart软件汇总整理所得结果。
2 结果与分析
2.1 花生籽仁表型性状的变异
亲本中花16的籽仁长、籽仁宽和百仁重均显著大于J11, 且2年结果表现一致(表1)。RIL群体的籽仁长、籽仁宽、籽仁长/宽比和百仁重均表现出很大的变异, 其中籽仁长的变异系数最大, 2年分别为10.73%和10.66%; 籽仁宽的变异系数最小, 2年分别为6.34%和6.03%。2年环境下的花生籽仁长、籽仁宽、籽仁长宽比和百仁重均呈正态分布, 且其峰度和偏度绝对值均小于0.45 (表1和图1)。

表1 亲本及RIL群体籽仁相关表型性状统计分析
P1: 中花16; P2: J11。P1: Zhonghua 16; P2: J11. LWR: ratio of seed length to width.
籽仁长、籽仁宽和百仁重两两之间呈极显著正相关。其中, 2018年百仁重与籽仁长相关系数最大, 为0.856; 2017年籽仁宽与籽仁长相关系数最小, 为0.45 (图2)。2年环境下籽仁长宽比与籽仁宽均呈负相关, 其中2017年呈显著负相关, 2018年相关性不显著, 但籽仁长宽比与其他2个性状均呈显著正相关(图2)。上述结果表明, 在不同环境下, 籽仁长、籽仁宽和百仁重这3个性状之间均存在相互影响。方差分析表明, 除籽仁宽在环境间差异不显著外(=0.0564,=0.813), 其余性状在环境间均表现出显著差异; 4个性状在家系间均表现出极显著差异。与籽仁长、百仁重和籽仁长宽比3个性状相比, 籽仁宽受环境影响相对较小(表2)。

图1 籽仁长、籽仁宽、百仁重以及籽仁长宽比4个性状在2017和2018两年环境下的频率分布
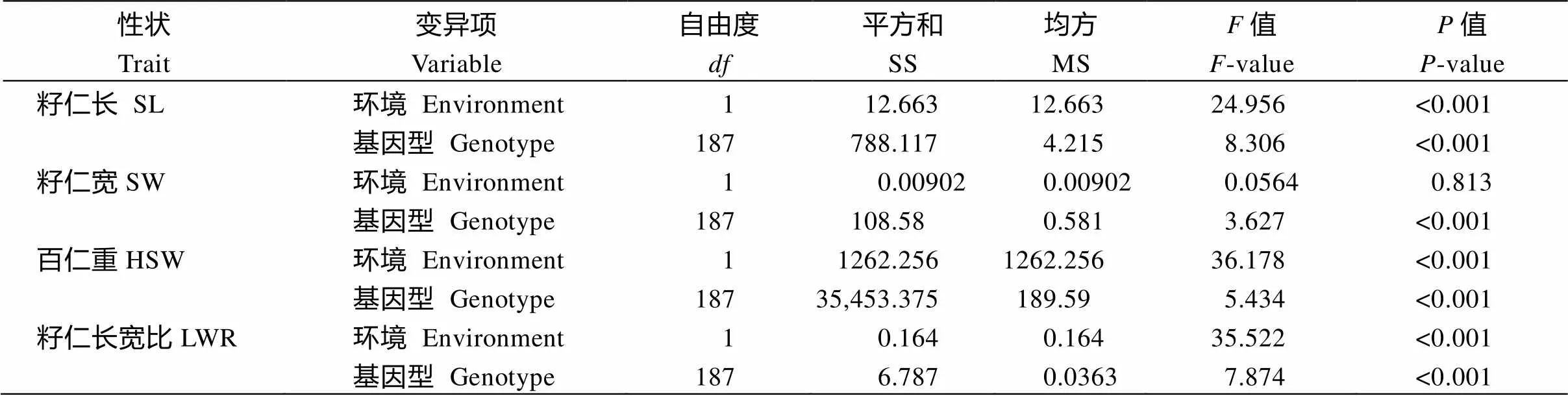
表2 花生籽仁大小相关性状的方差分析
SL: seed length; SW: seed width; HSW: hundred seed weight; LWR: ratio of seed length to width.
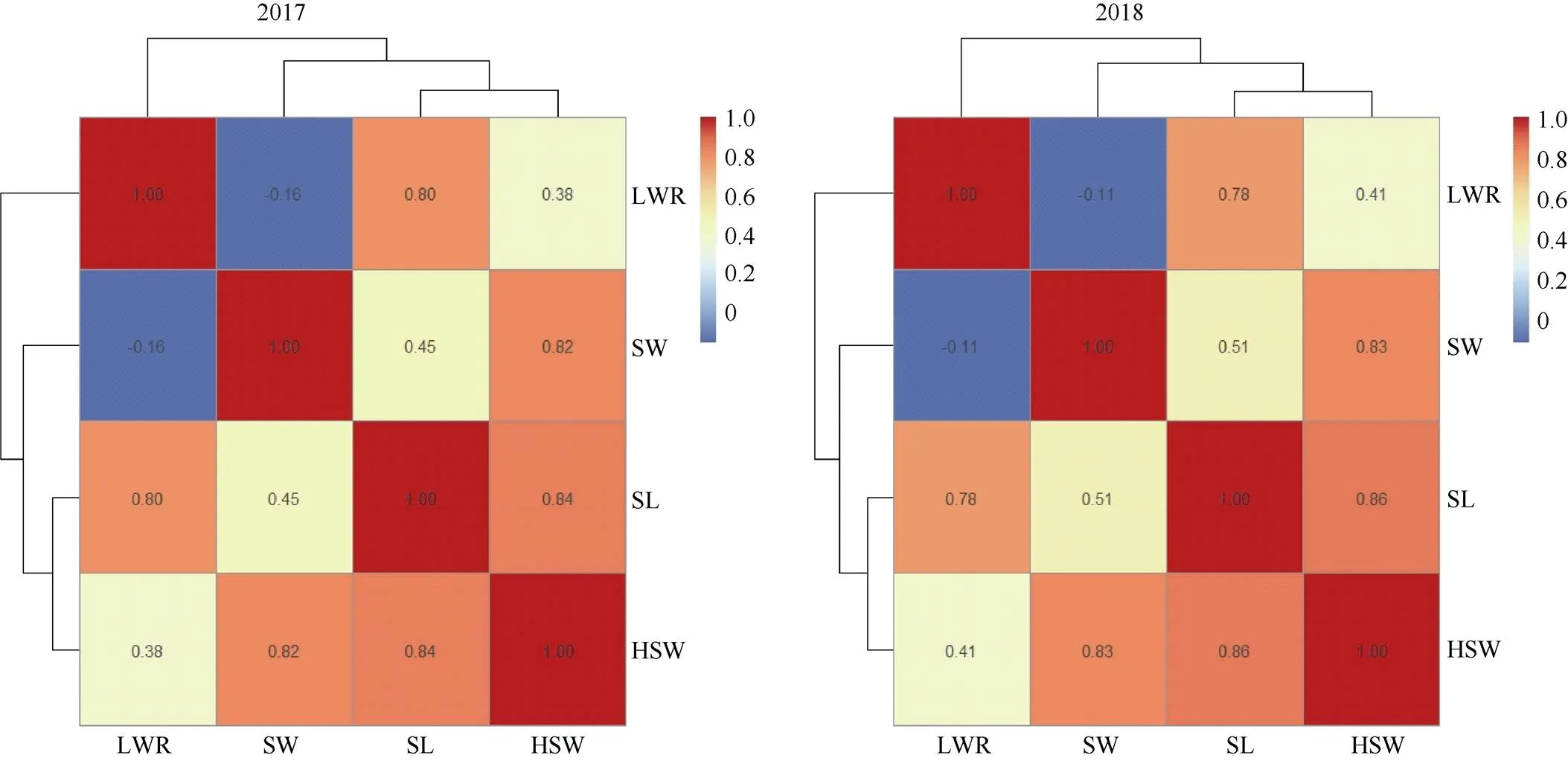
图2 籽仁大小性状的相关性分析热图
LWR: 籽仁长宽比; SW: 籽仁宽; SL: 籽仁长; HSW: 百仁重。
LWR: ratio of seed length to width; SW: seed width; SL: seed length; HSW: hundred seed weight.
2.2 遗传连锁图谱构建
利用4500对本实验室开发的基因组SSR引物对亲本中花16和J11进行多态性筛选, 共得到434对具有多态性的引物。继续用434对多态性引物扩增RIL群体, 329对引物在RIL群体中也表现出良好的多态性。通过JoinMap4.0软件对329个基因型数据进行作图, 得到了一张包含289个SSR分子标记、21个连锁群的花生遗传连锁图谱(图3), 整个连锁群覆盖长度为947.3 cM, 标记间平均距离为3.28 cM。其中A04被分成了A04a和A04b两个连锁群。在所有连锁群中, 长度最短的染色体为A04a (4.91 cM), 最长染色体为B06 (84.17 cM), 标记个数最少的染色体为A04b (3个), 标记最多的染色体为B06 (38个)(表3)。
2.3 花生籽仁大小QTL定位
结合2年表型数据, 采用WinCart2.5软件, 在2个环境下共检测到66个QTL, 分布在12个连锁群上, 表型贡献率为3.23%~33.01%。
2.3.1 籽仁长QTL 共检测到18个籽仁长相关的QTL, 其中2017年检测到7个, LOD值为2.66~ 18.2, 贡献率为3.6%~33.0%, 其中有2个QTL (和)贡献率大于10%, 为主效QTL; 2018年检测到11个, LOD值为3.19~23.00, 贡献率为3.30%~32.66%, 有3个主效QTL (、和)。在这些QTL中, 2017年检测到的和2018年检测到的均在区间A05A1500~A05A1530, LOD值分别为18.20和22.99, 贡献率分别为33.01%和31.06%; 2017年检测到的和2018年检测到的均在区间A06B467~A06B552, LOD值分别为2.72和3.87, 贡献率分别为3.60%和3.95% (表4)。
2.3.2 籽仁宽QTL 共检测到16个与籽仁宽相关的QTL, 2017年检测到12个, LOD值为2.57~8.53, 贡献率为4.08%~14.27%, 其中4个为主效QTL (、、和); 2018年检测到4个, LOD值为2.96~5.08, 贡献率为4.50%~12.23%, 有1个主效QTL ()。在这些QTL中, 2017年检测到的和2018年检测到的均在区间A06B135~A06B113, LOD值为8.53和7.64, 贡献率分别为14.27%和12.23% (表4)。
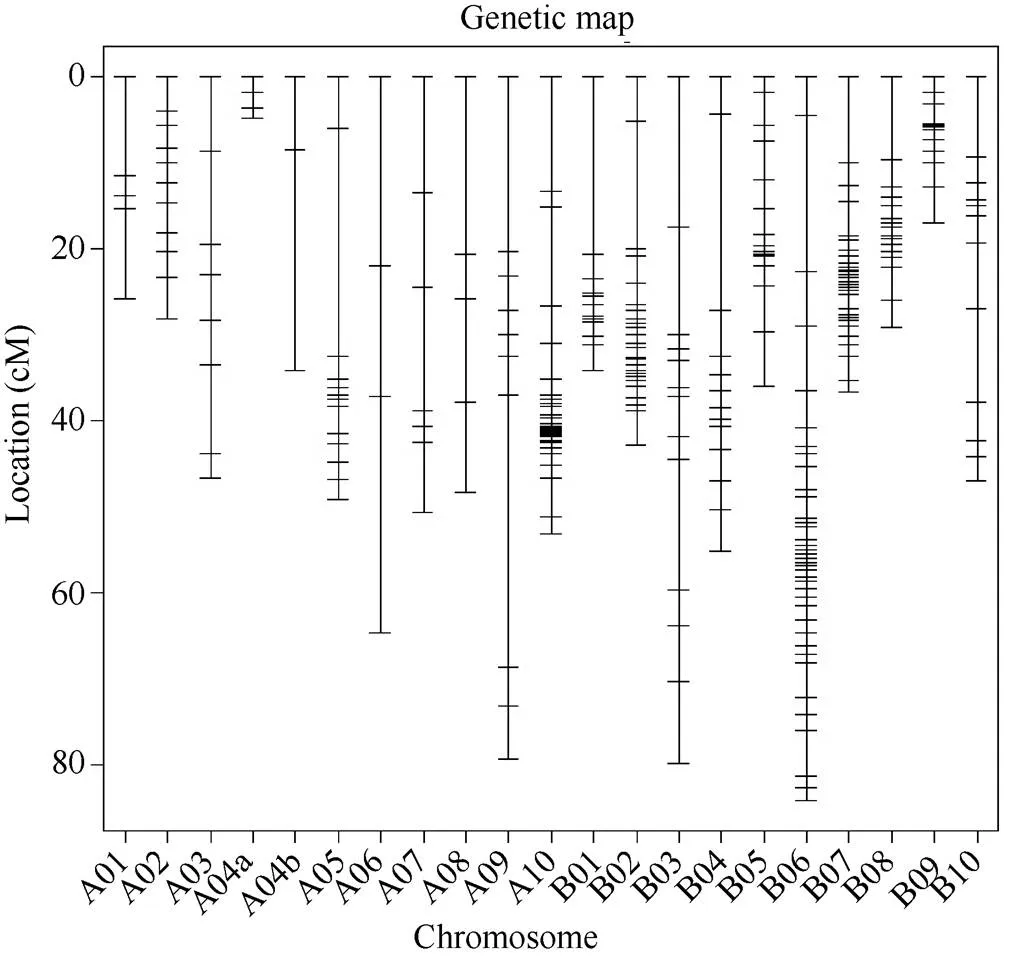
图3 花生遗传连锁图谱
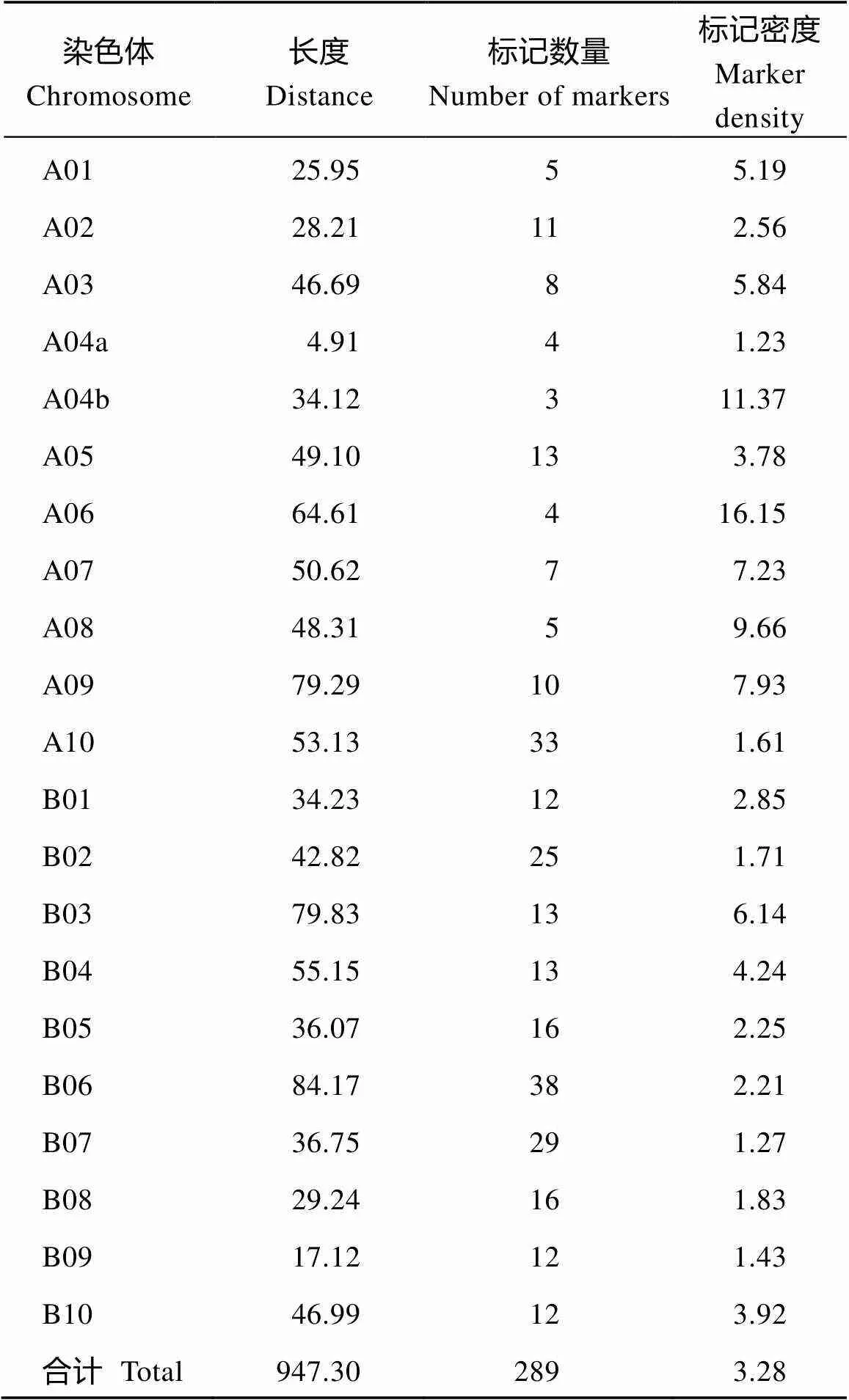
表3 SSR标记在遗传图谱上的分布情况
2.3.3 籽仁长/宽QTL 共18个与籽仁长宽比相关的QTL, 2017年检测到9个, LOD值为2.71~14.87, 贡献率为3.23%~20.30%, 其中4个为主效QTL (、、和); 2018年检测到9个, LOD值为2.75~13.97, 贡献率为3.56%~19.11%, 其中5个为主效QTL (、、、和)。在这些QTL中, 2017年检测到的和2018年检测到的均在区间A02B113− A02A138, LOD值分别为7.23和11.59, 贡献率为10.33%和17.08%; 2017年检测到的和2018年检测到的均在区间A05A1053~ A05A1150, LOD值分别为7.07和8.42, 贡献率分别为11.03%和12.28% (表4)。
2.3.4 百仁重QTL 共检测到14个与百仁重相关的QTL, 2017年检测到6个, LOD值为3.12~11.61, 贡献率为5.26%~19.67%, 1个主效QTL (); 2018年检测到8个, LOD值为2.74~15.31, 贡献率为3.43%~23.54%, 其中3个主效QTL (、和)的贡献率均在10%以上。
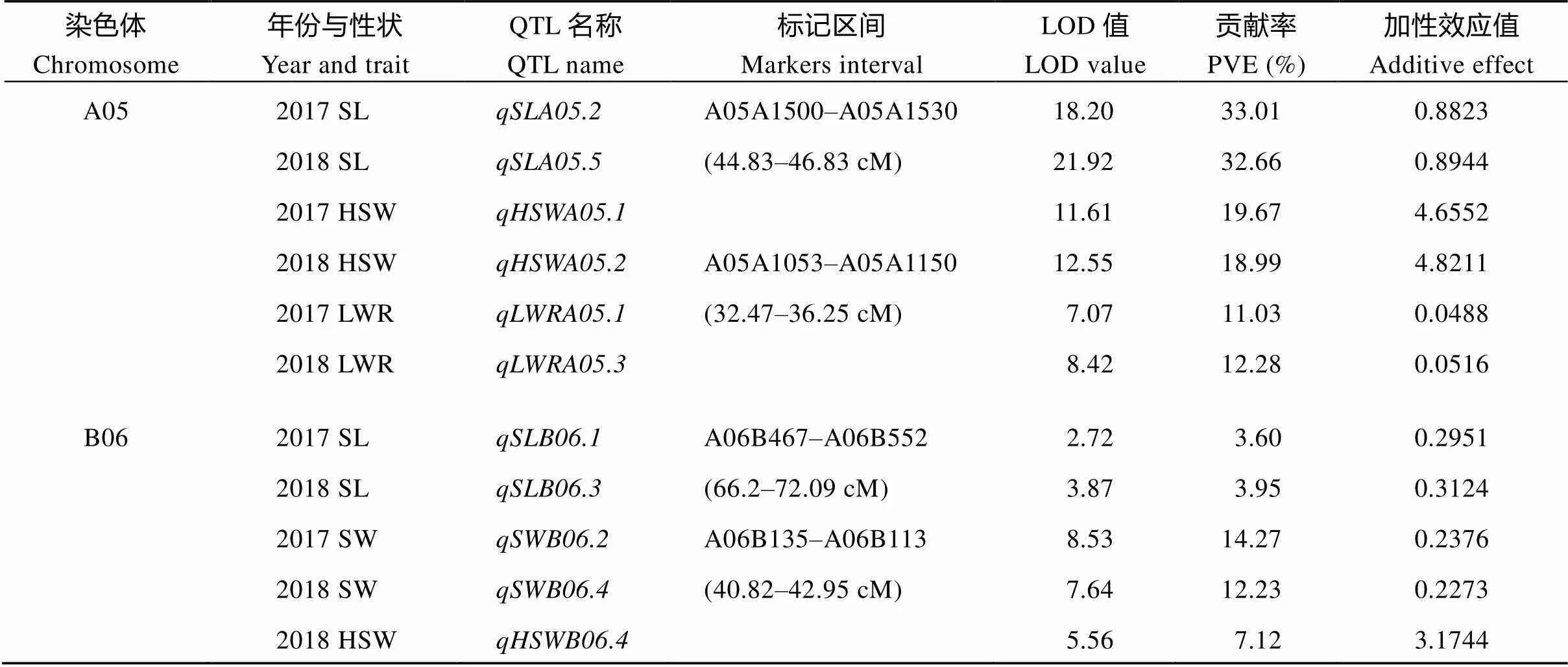
表4 不同环境下重复检测到的QTL
SL: 籽仁长; SW: 籽仁宽; HSW: 百仁重; LWR: 籽仁长宽比。
SL: seed length; SW: seed width; HSW: hundred seed weight; LWR: ratio of seed length to width; PVE: explained phenotypic variance.
2.4 不同性状QTL共定位
由图4可知, 在A05染色体上的区间A05A1500~ A05A1530同时检测到控制籽仁长和百仁重相关的QTL (、和), 贡献率均在10%以上, 最高达33.01%; LOD值均在10以上, 最高达21.92。在B06染色体上的区间A06B135~ A06B113同时检测到控制籽仁宽和百仁重相关的QTL (、和), LOD值均在5以上, 贡献率最高达14.27%。
3 讨论
花生籽仁大小相关性状与产量密切相关。本研究表明, 花生籽仁相关性状存在广泛变异, 籽仁长、籽仁宽和百仁重3个性状之间相关性显著(<0.001), 且2年表现一致, 说明籽仁长和籽仁宽是决定百仁重的重要因素, 其中籽仁长对百仁重的影响相对较大。这与Chen等[13]和Luo等[14-15]的研究结果类似, Chen等[13]报道花生籽仁长和籽仁宽2个性状在3个环境下均呈显著正相关, 且3年结果基本一致; Luo等[14-15]报道在3个环境下荚果长、荚果宽和百果重之间呈显著正相关且3年表现一致。
本研究通过遗传连锁图谱的构建, 结合2年群体表型数据, 对4个籽仁大小相关性状进行了QTL分析, 发现重复QTL区间主要分布在A05和B06两个染色体上, 且区间内QTL的贡献率较高。前人对花生产量性状QTL的定位主要集中在A05染色体上[10-11,13-14], 将本研究在A05染色体上检测到的与籽仁大小相关的QTL与Luo等[14]和李振动等[16]鉴定的QTL对比, 发现Luo等[14]的研究中在区间A05A1368~A05A1500内定位到控制荚果长和荚果宽相关的QTL, 而本研究在标记A05A1500附近检测到与籽仁长和籽仁宽相关的QTL。通过分析花生资源品种信息, 发现荚果大小和籽仁大小存在极显著正相关, 推测该区间存在控制籽仁大小相关的QTL, 而这些QTL在不同群体中被检测到, 证明了本研究结果的可靠性。此外, 由于所用标记的不同, 很多相关性状的QTL很难与本研究结果比较。对B06上的QTL分布情况未见有报道, 因此本研究在B06染色体上检测到的QTL为新发现的QTL。在这些QTL中, 有4个被重复定位到, 说明其结果的可靠性。这些稳定且贡献率较高的QTL, 将为改良籽仁大小相关性状提供重要参考。
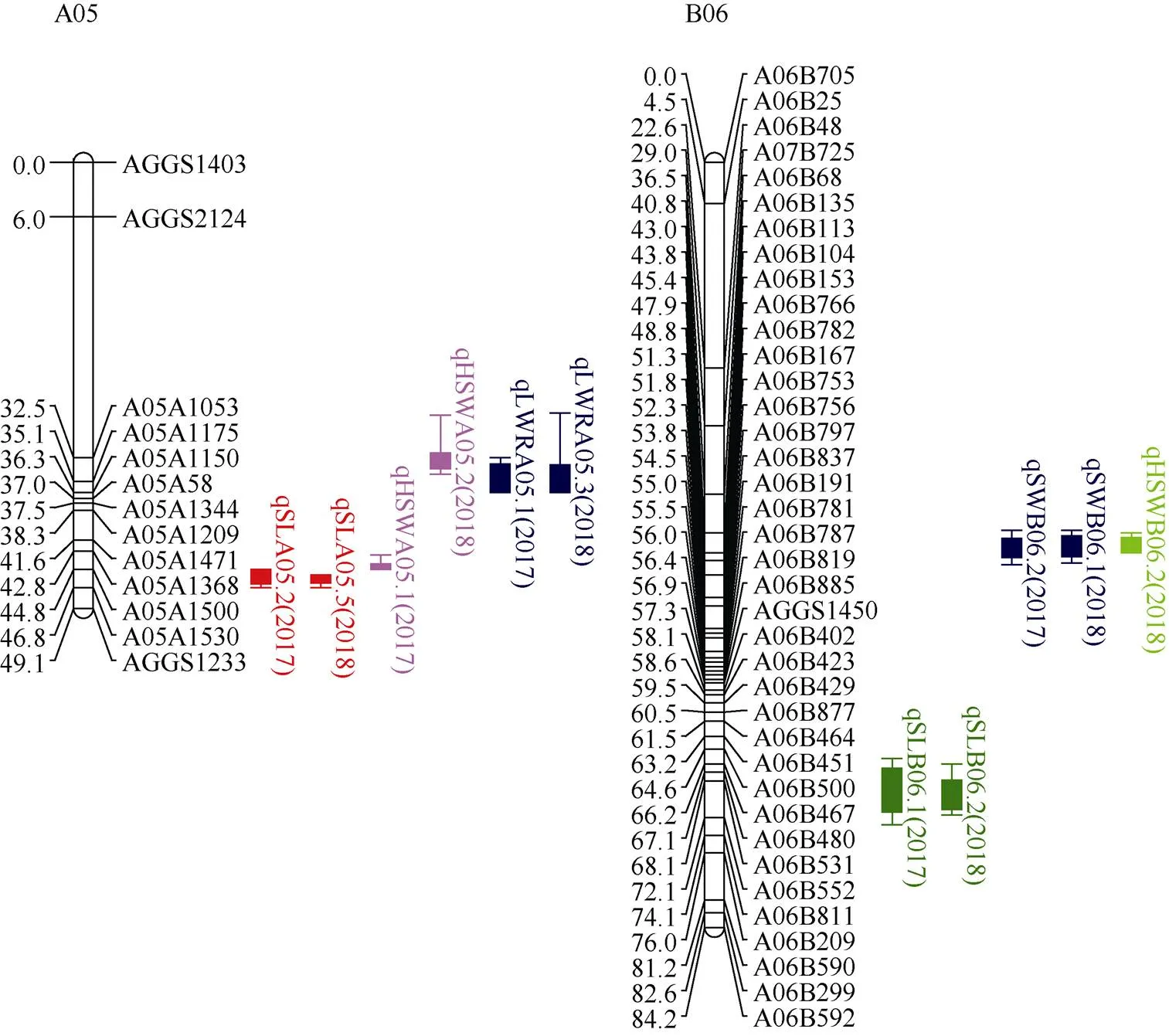
图4 A05和B06上QTL共定位区间
根据QTL定位结果, 2年环境下在A05和B06上重复检测到控制籽仁长、籽仁宽和长宽比性状的QTL, 表明这些QTL是稳定存在的。此外, 在A05上的区间A05A1209~A05A1500内同时定位到控制籽仁长和百仁重相关的QTL; 在B06上的区间A06B135~A06B113内同时定位到控制籽仁宽和百仁重的QTL; 在区间A05A1053~A05A1150内同时定位到控制籽仁长宽比和百仁重相关的QTL, 说明控制这些性状的QTL紧密连锁或基因存在多效性, 这一结果与相关性分析结果一致。相关性状在同一位点上的共定位在一定程度上证明了该位点的稳定性, 因此, 本研究中籽仁相关的多个性状共定位在A05上的区间A05A1209~A05A1500和B06上的区间A06B135~A06B113, 是与籽仁大小相关的稳定区间。本研究检测到的稳定且贡献率较高的QTL将为籽仁大小相关性状的改良和精细定位奠定基础。
4 结论
检测到与花生籽仁大小相关性状的QTL共67个, 其中A05染色体上的区间A05A1500~ A05A1530同时定位到控制籽仁长和百仁重的相关的QTL, B06染色体上的区间A06B135~A06B113同时定位到控制籽仁宽和百仁重相关的QTL。
[1] Gomes R L F, Lopes  C A. Correlations and path analysis in peanut., 2005, 5: 105–112.
[2] Selvaraj M G, Narayana M, Schubert A M, Ayers J L, Baring M R, Burow M D. Identification of QTLs for pod and kernel traits in cultivated peanut by bulked segregant analysis., 2009, 12: 13.
[3] Getahun A, Yang X L, He M J, Cui S L, Mu G J, Liu L F. Advances of genetic map construction and QTL mapping in peanut., 2017, 46: 1–10.
[4] Wilson J N, Chopra R, Baring M R, Selvaraj M G, Simpson C E, Chagoya J, Burow M D. Advanced backcross quantitative trait loci (QTL) analysis of oil concentration and oil quality traits in peanut (L.)., 2017, 10: 1–17.
[5] Wang M L, Khera P, Pandey M K, Wang H, Qiao L X, Feng S P, Tonnis B, Barkley N A, Pinnow D, Holbrook C C, Culbreath A K, Varshney R K, Guo B Z. Genetic mapping of QTLs controlling fatty acids provided insights into the genetic control of fatty acid synthesis pathway in peanut (L.)., 2015, 10: e0119454.
[6] Khedikar Y P, Gowda M V C, Sarvamangala C, Patgar K V, Upadhyaya H D, Varshney R K. A QTL study on late leaf spot and rust revealed one major QTL for molecular breeding for rust resistance in groundnut (L.)., 2010, 121: 971–984.
[7] Pandey M K, Wang H, Khera P, Vishwakarma M K, Kale S M, Culbreath A K, Holbrook C C, Wang X J, Varshney R K, Guo B Z. Genetic dissection of novel QTLs for resistance to leaf spots and tomato spotted wilt virus in peanut (L.)., 2017, 8: 25.
[8] Luo H Y, Xu Z J, Li Z D, Li X P, Lv J W, Ren X P, Huang L, Zhou X J, Chen Y N, Yu J Y, Chen W G, Lei Y, Liao B S, Jiang H F. Development of SSR markers and identification of major quantitative trait loci controlling shelling percentage in cultivated peanut (L.)., 2017, 130: 1635–1648.
[9] Chen Y N, Ren X P, Zheng Y L, Zhou X J, Huang L, Yan L Y, Jiao Y Q, Chen W G, Huang S M, Wan L Y, Lei Y, Liao B S, Huai D X, Wei W H, Jiang H F. Genetic mapping of yield traits using RIL population derived from Fuchuan Dahuasheng and ICG6375 of peanut (L.)., 2017, 37: 17.
[10] Shirasawa K, Koilkonda P, Aoki K, Hirakawa H, Tabata S, Watanabe M, Hasegawa M, Kiyoshima H, Suzuki S, Kuwata C, Naito Y, Kuboyama T, Nakaya A, Sasamoto S, Watanabe A, Kato M, Kawashima K, Kishida Y, Kohara M, Kurabayashi A, Takahashi C, Tsuruoka H, Wada T, Isobe S.polymorphism analysis for the development of simple sequence repeat and transposon markers and construction of linkage map in cultivated peanut., 2012, 12: 80.
[11] Ravi K, Vadez V, Isobe S, Mir R R, Guo Y, Nigam S N, Gowda M V C, Radhakrishnan T, Bertioli D J, Knapp S J, Varshney R K. Identification of several small main-effect QTLs and a large number of epistatic QTLs for drought tolerance related traits in groundnut (L.)., 2011, 122: 1119–1132.
[12] 王传堂, 黄粤, 杨新道, 姜勇, 张建成, 陈殿绪, 闵平, 禹山林. 改良CTAB法和高盐低pH值法提取花生DNA的效果. 花生学报, 2002, 31: 20–23. Wang C T, Huang Y, Yang X D, Jiang Y, Zhang J C, Chen D X, Min P, Yu S L. Isolation of DNA from peanut: comparison between modified CTAB and high salt, low pH methods., 2002, 31: 20–23 (in Chinese with English abstract).
[13] Chen W G, Jiao Y Q, Cheng L Q, Huang L, Liao B S, Tang M, Ren X P, Zhou X J, Chen Y N, Jiang H F. Quantitative trait locus analysis for pod- and kernel-related traits in the cultivated peanut (L.)., 2016, 17: 25.
[14] Luo H Y, Ren X P, Li Z D, Xu Z J, Li X P, Huang L, Zhou X J, Chen Y N, Chen W G, Lei Y, Liao B S, Pandey M K, Varshney R K, Guo B Z, Jiang X G, Liu F, Jiang H F. Co-localization of major quantitative trait loci for pod size and weight to a 3.7 cM interval on chromosome A05 in cultivated peanut (L.)., 2017, 18: 58.
[15] Luo H Y, Guo J B, Ren X P, Chen W G, Huang L, Zhou X J, Chen Y N, Liu N, Xiong F, Lei Y, Liao B S, Jiang H F. Chromosomes A07 and A05 associated with stable and major QTLs for pod weight and size in cultivated peanut (L.)., 2017, 131: 267–282.
[16] 李振动, 李新平, 黄莉, 任小平, 陈玉宁, 周小静, 廖伯寿, 姜慧芳. 栽培种花生荚果大小相关性状QTL定位. 作物学报, 2015, 41: 1313–1323. Li Z D, Li X P, Huang L, Ren X P, Chen Y L, Zhou X J, Liao B S, Jiang H F. Mapping of QTLs for pod size related traits in cultivated peanut (L.)., 2015, 41: 1313–1323 (in Chinese with English abstract).
Identification of QTL related to seed size in peanut (L.)
ZENG Xin-Ying1,2,**, GUO Jian-Bin2,**, ZHAO Jiao-Jiao2, CHEN Wei-Gang2, QIU Xi-Ke2, HUANG Li2, LUO Huai-Yong2, ZHOU Xiao-Jing2, JIANG Hui-Fang2,*, and HUANG Jia-Quan1,*
1Institute of Tropical Agriculture and Forestry, Hainan University / Hainan Key Laboratory for Sustainable Utilization of Tropical Bioresources, Haikou 571003, Hainan, China;2Oil Crops Research Institute of Chinese Academy of Agricultural Sciences / Key Laboratory of Biology and Genetic Improvement of Oil Crops, Ministry of Agriculture and Rural Affairs, Wuhan 430062, Hubei, China
Seed size-related traits are the direct factors determining the yield of peanut. To identify the QTL related to seed size-related traits, a recombinant inbred lines (RIL) population (188 progenies) derived from Zhonghua 16 × J11 was used in this study. A genetic linkage map of 947.3 cM in length was constructed, containing 21 linkage groups and 289 SSR markers. Seed size-related traits showed extensive variations in two years’ phenotyping. Between most of the traits with significant correlation. Based on the genetic map, we detected 66 QTL with the explained phenotypic variance (PVE) of 3.23%–33.01% were detected using the WinCart 2.5 software. The number of QTL for seed length (SL), seed width (SW), ratio of seed length to width (LWR) and hundred seed weight (HSW) were 18, 16, 18, and 14, respectively. Notably, the A05A1500–A05A1530 interval on Chromosome A05 harbored three QTL, i.e.andfor SL andfor HSW, and the A06B135–A06B113 interval on B06 harbored three QTL as well, i.e.andfor SW andfor HSW. These stable and major QTL pave a way for fine mapping of peanut yield-related traits and molecular breeding.
peanut; genetic mapping; seed size; QTL
2018-12-18;
2019-04-15;
2019-05-08.
10.3724/SP.J.1006.2019.84173
黄家权, E-mail: jqhuang@163.com; 姜慧芳, E-mail: peanutlab@oilcrops.cn, Tel: 027-86711550
**同等贡献(Contributed equally to this work)
曾新颖, E-mail: 15799032956@163.com; 郭建斌, E-mail: guojianbin1990@163.com
本研究由国家自然科学基金项目(31461143022, 31761143005, 31571713, 31801403, 31871666), 农业部农作物种质资源保护项目(2017NWB033), 国家农作物种质资源共享服务平台(NICGR2017-36)和国家现代农业产业技术体系建设专项(CARS-13-种质资源评价)资助。
This study was supported by the National Natural Science Foundation of China (31461143022, 31761143005, 31571713, 31801403, 31871666), the Crop Germplasm Resources Protection Project (2017NWB033), the Plant Germplasm Resources Sharing Platform (NICGR2017-36), and the China Agriculture Research System (CARS-13-Germplasm Resource Evaluation).
URL:http://kns.cnki.net/kcms/detail/11.1809.S.20190505.1358.004.html
