采用生物信息学方法筛选与肺腺癌患者预后相关的LncRNAs*
2019-07-15李云慧黄俊杰
陶 冶,李云慧,黄俊杰,梁 彬△
(1.中国医科大学生命科学学院生物信息学教研室,辽宁沈阳 110122;2.北部战区总医院和平院区检验科,辽宁沈阳 110003)
据2018年185个国家的统计结果显示,肺癌占所有癌症病例的11.6%,占癌症总死亡人数的18.4%,位列我国癌症发病率之首[1]。非小细胞肺癌是肺癌常见形式,包括3种组织病理学亚型:腺癌、鳞状细胞癌和大细胞癌。肺腺癌是最常见的变异型,约占肺癌病例的40.0%[2-3]。目前,虽然在肺癌手术、化疗和分子靶向治疗技术方面取得了进展,但肺癌仍然是全球癌症死亡的主要原因[3-4]。长链非编码RNA(lncRNAs)是大于200 bp的非编码RNA,主要通过表观遗传学、转录及转录后水平调控基因表达[5]。研究结果显示,lncRNAs是重要的调控因子,调控细胞增殖、分化、侵袭以及转移[6]。本研究利用TCGA中肺腺癌患者的RNA测序数据,采用生物信息学技术手段,筛选肺腺癌患者差异表达的lncRNAs,探讨差异表达的lncRNAs与肺腺癌临床参数的相关性及与肺腺癌患者预后的关系。
1 资料与方法
1.1数据下载 从TCGA数据库(https://cancergenome.nih.gov/)下载肺腺癌患者癌组织和癌旁组织的lncRNAs测序数据,cBioPortal数据库(http://www.cbioportal.org/)下载相应的临床资料。其中男219例,年龄38~88 岁,平均(65.66±9.77)岁;女259例,年龄40~87岁,平均(65.75±9.72)岁。样本的纳入标准:(1)经病理证实为原发性肺腺癌,术前未经过任何放化疗;(2)具有完整的临床分期资料及预后信息。共计478例肺腺癌患者纳入分析,临床特点见表1。本研究中肺腺癌的分期采用TNM分期方法,其中T分期与肿瘤浸润深度相关,分期越高,浸润深度越大;N分期与淋巴结转移情况相关,分期越高,转移淋巴结越多;M分期与肿瘤远处转移相关。肺腺癌患者在年龄和性别上差异无统计学意义,具有可比性。
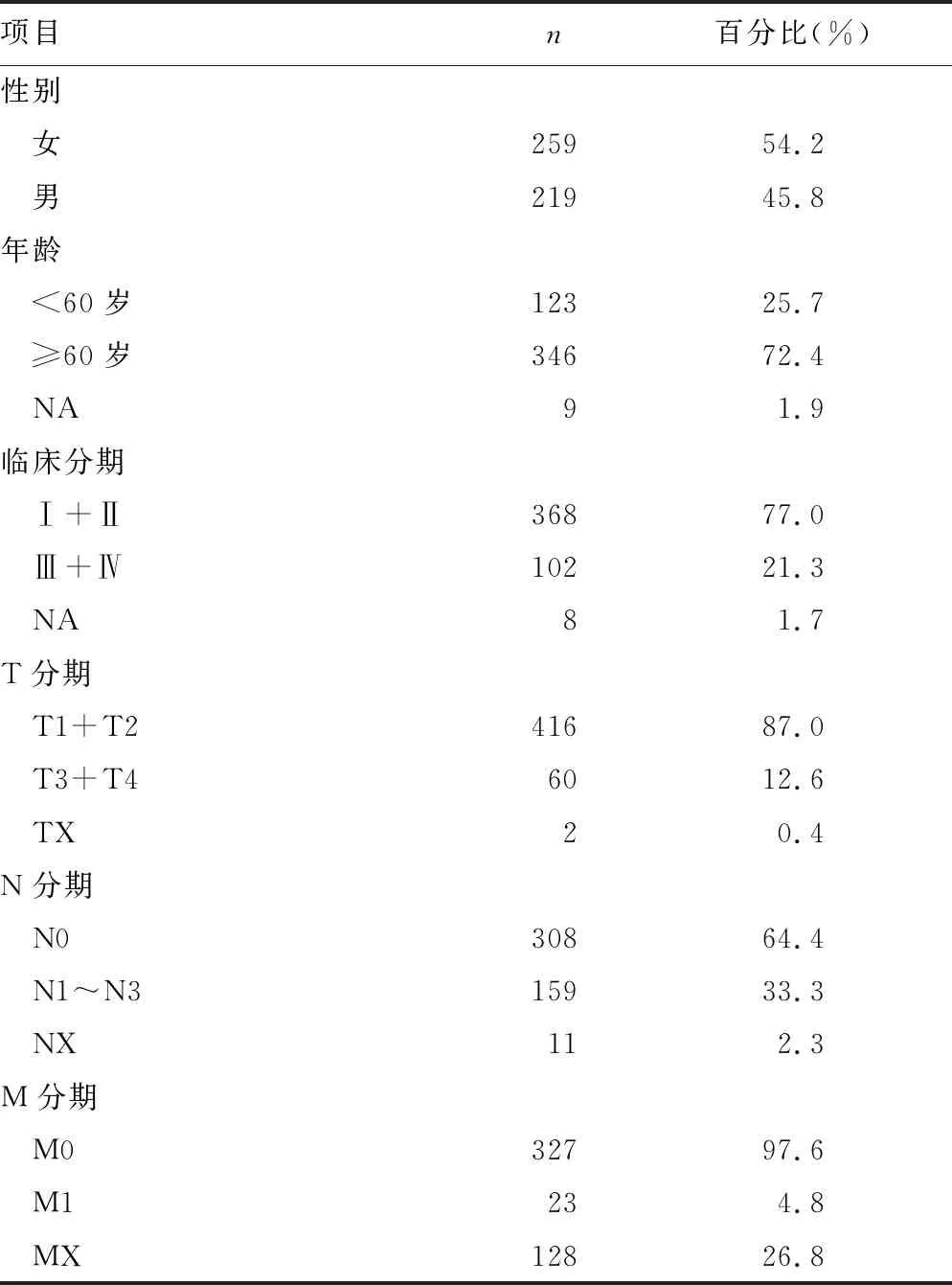
表1 肺腺癌患者的临床特点
注:NA表示无法确定;TX表示原发肿瘤的情况无法评估;NX表示区域淋巴结情况无法评估;MX表示肿瘤有无远处转移无法评估
1.2数据处理 应用R语言程序包对TCGA数据库下载的lncRNAs数据信息进行差异分析,筛选肺腺癌组织和癌旁组织中差异表达的lncRNAs。筛选标准:log2|差异倍数(FC)≥1,P<0.05。
1.3筛选与肺腺癌患者预后相关的lncRNAs 对肺腺癌患者癌组织中多个差异表达的lncRNAs进行分组,根据每个lncRNA的测序数据中值分成高表达组和低表达组。应用Kaplan-Meier生存曲线和Log-Rank检验方法分别对高表达和低表达的lncRNA与总体生存期(OS)的相关性进行分析,筛选出与总体生存期显著相关的lncRNAs。应用单因素和多因素Cox回归模型对与OS相关的lncRNAs行进一步分析。
1.4差异表达lncRNAs与临床参数的关系 根据患者年龄、TNM分期、总体生存状态进行分组,分析与OS相关的lncRNAs与各临床参数的关系。
1.5统计学处理 采用SPSS20.0统计学软件包进行统计学分析。计数资料采用率(%)表示。应用Kaplan-Meier曲线法进行生存分析,Log-Rank方法检验各lncRNA高表达和低表达组生存率差异是否具有统计学意义。多因素预后分析采用Cox比例风险回归模型。lncRNA表达和临床参数之间的关系采用χ2检验。P<0.05表示差异有统计学意义。
2 结 果
2.1差异表达lncRNAs的筛选 应用R语言程序包筛选出癌组织和癌旁组织差异表达的lncRNAs有168个,其中上调的lncRNA有128个,下调的lncRNAs有40个。癌组织和癌旁组织中差异表达lncRNAs的分布见图1。
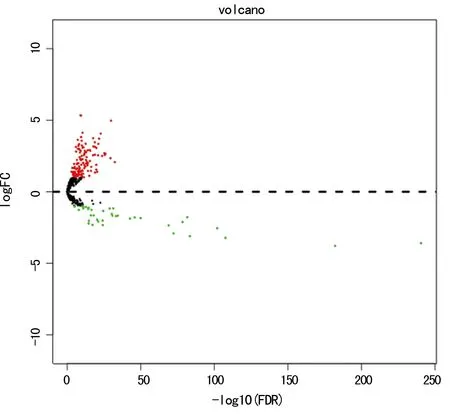
注:FDR表示错误发现率;FC表示差异倍数;红色表示上调lncRNAs,绿色表示下调lncRNAs
图1癌组织和癌旁组织差异表达的lncRNAs分布火山图
2.2与OS相关的lncRNAs筛选 根据每个lncRNA的表达中值,分成高表达组和低表达组。采用Kaplan-Meier生存曲线和Log-Rank检验分析168个差异表达的lncRNAs与肺腺癌患者预后的关系。结果显示,RP11-490M8.1(P=0.024)、RP11-132A1.4(P=0.001)、DRAIC(P=0.007)、LINC00942(P=0.001)、TMPO-AS1(P=0.005)、Z83851.4(P<0.001)和LINC01133(P=0.022)与肺腺癌患者的预后呈显著相关。高表达RP11-490M8.1、RP11-132A1.4、LINC00942、TMPO-AS1、Z83851.4和LINC01133与预后差显著相关,而低表达DRAIC与预后差显著相关。见图2。
采用单因素和多因素Cox回归分析结果进一步证实,差异表达的lncRNAs与肺腺癌患者的预后相关,高表达RP11-490M8.1(P=0.047)、RP11-132A1.4(P=0.049)、LINC00942(P=0.033)、TMPO-AS1(P=0.006)、Z83851.4(P=0.032)、LINC01133(P=0.035)和低表达DRAIC(P=0.023)是肺腺癌患者预后不良的独立预后因素。见表2。
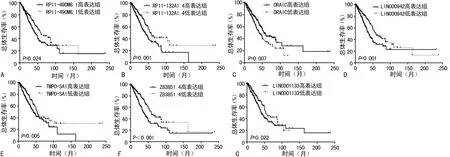
注:A为RP11-490M8.1;B为RP11-132A1.4;C为DRAIC;D为LINC00942;E为TMPO-AS1;F为Z83851.4;G为LINC01133
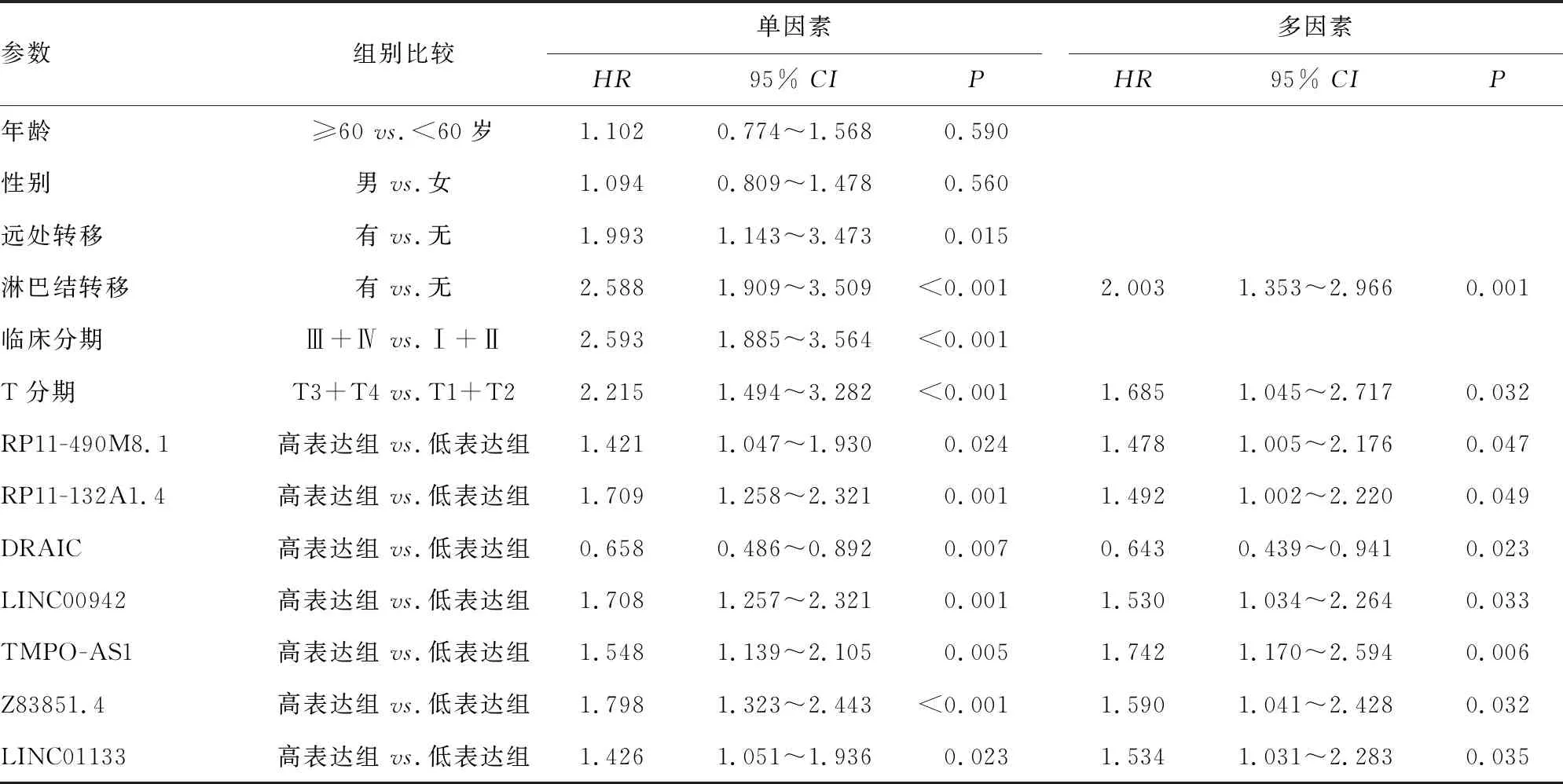
表2 肺腺癌患者差异表达lncRNAs的单因素和多因素Cox回归分析
注:HR表示风险比值
2.3与预后显著相关的lncRNAs和临床参数的关系 对与预后显著相关的7个lncRNAs和各临床参数之间的关系进行分析,结果显示,RP11-132A1.4 表达水平与年龄(P=0.016)、淋巴结转移(P=0.016)、临床分期(P<0.001)和T分期(P=0.001)显著相关;DRAIC表达水平与年龄(P=0.027)显著相关;LINC00942表达水平与淋巴结转移(P<0.001)和临床分期(P=0.001)显著相关;TMPO-AS1表达水平与临床分期(P=0.008)显著相关;Z83851.4表达水平与淋巴结转移(P=0.008)显著相关;LINC01133与T分期(P=0.049)显著相关。见表2。
3 讨 论
lncRNAs是一种长度大于200个核苷酸,缺乏完整的特异性开放阅读框,无编码蛋白质功能的核酸分子。大量研究结果显示lncRNAs通过染色质重塑,组蛋白修饰和RNA代谢等多种生物学过程在表观遗传、转录和转录后水平调控基因表达[7-8]。lncRNA发挥内源性“miRNA 海绵”功能与miRNA相互作用,参与靶基因的表达调控;反之,miRNA通过RNA诱导沉默复合物调控lncRNAs发挥生物学功能,两者又可竞争结合mRNAs,共同参与疾病的发生发展过程[9]。
lncRNAs与多种癌症相关,其异常表达和突变与肿瘤的增殖、凋亡、侵袭、迁移及药物敏感性密切相关。研究结果显示,在多种肿瘤患者中,某些lncRNAs表达发生改变,可作为癌症诊断及预后的标志物[10]。TCGA癌症数据库是由美国国家癌症研究所及国家人类基因组研究所联合建立,其中包含丰富的数据类型和肿瘤类型。本研究采用生物信息学技术手段,筛选肺腺癌患者差异表达的lncRNAs 168个进行预后分析,总体生存分析显示:RP11-490M8.1、RP11-132A1.4、DRAIC、LINC00942、TMPO-AS1、Z83851.4和LINC01133是肺腺癌患者独立预后的不良因素。YANG等[11]报道LINC01133通过充当miR-106a-3p的ceRNA来调控APC的表达和Wnt/β-catenin通路,抑制胃癌的进展和转移。也有研究证明,LINC01133低表达影响结直肠癌的转移和预后[12]。ZANG等[13]报道敲除LINC01133减少非小细胞肺癌肿瘤细胞的增殖、迁移和侵袭以及诱导细胞凋亡发挥抑瘤作用。也有研究发现LINC01133是食管鳞状细胞癌预后的生物标志物和治疗靶点[14]。HUANG等[15]报道,在前列腺癌细胞中TMPO-AS1过度表达促进细胞周期的进展、肿瘤细胞迁移和增殖,抑制细胞凋亡。也有研究证明,DRAIC/PCAT29的表达水平与前列腺癌的发生和进展有关[16]。其他的lncRNAs目前还未有报道。
此外,本研究发现RP11-132A1.4、LINC00942和Z83851.4与淋巴结转移显著相关,RP11-132A1.4、LINC00942和TMPO-AS1与临床分期显著相关,RP11-132A1.4和LINC01133与T分期显著相关,提示上述lncRNAs可能参与肺腺癌的发生发展过程,在肺腺癌恶性进程中发挥重要作用。
4 结 论
本研究对TCGA数据库中肺腺癌患者大数据进行分析,筛选出与肺腺癌预后显著相关的lncRNAs:RP11-490M8.1、RP11-132A1.4、DRAIC、LINC00942、TMPO-AS1、Z83851.4和LINC01133。本研究结果提示上述lncRNAs分别与各临床参数存在显著相关性,可能为肺腺癌机制的研究提供理论依据,为寻找肺腺癌诊断和预后新的分子标志物以及个性化治疗方案提供新的思路。
